El diagnóstico molecular es una área dinámica en constante desarrollo que ha revolucionado el diagnóstico clínico. La detección y cuantificación específica de material genético en una muestra biológica ha mostrado un significativo impacto en todas las áreas de la salud, sobre todo en las áreas de las enfermedades infecciosas y el cáncer. El desarrollo de nuevas tecnologías, más rápidas y precisas, ha transformado al diagnóstico molecular en una herramienta clave para el equipo clínico en directo beneficio del paciente. Esta revisión se enfoca en el impacto de la biología molecular en el diagnóstico, describiendo algunos aspectos históricos, las principales técnicas utilizadas y sus proyecciones.
Molecular diagnostics is a dynamic area in constant development that has revolutionized the diagnosis in health care centers. Detection and quantification of genetic material in a sample has shown an enormous impact on all areas of health, with emphasis on infectious diseases and cancer. The development of faster and more accurate new technologies has become molecular diagnosis a key tool for the clinical team in direct benefit to the patient. This review focuses on the impact of molecular biology in the diagnosis, describing some historical aspects, the main techniques used and their projections.
El diagnóstico es una etapa crítica en el manejo de un paciente, que implica decisiones médicas que definirán el progreso del cuidado de un individuo. De esta forma, el apoyo diagnóstico es un servicio fundamental para el equipo médico, y por ende, para la institución que lo realiza. De forma general, los laboratorios clínicos que ofrecen estos test diagnósticos se agrupan en una sección de la institución, en espacios con una infraestructura adecuada para dar un servicio de calidad. Con el desarrollo de técnicas de diagnóstico molecular, la sección de laboratorio clínico ha debido incorporar nuevos laboratorios con el fin de implementar técnicas de biología molecular al servicio de los pacientes. Hoy en día, el laboratorio de biología molecular es el área diagnóstica de mayor dinamismo y crecimiento dentro los laboratorios clínicos, revolucionando el sistema de salud, liderando la investigación biomédica y optimizando los tratamientos médicos.
En esta revisión se enfocará en el impacto de la biología molecular en el diagnóstico, describiendo algunos aspectos históricos, las principales técnicas utilizadas, sus limitaciones, nuevas técnicas y sus proyecciones.
DIAGNÓSTICO MOLECULAREl concepto de “diagnóstico molecular” es un término amplio que incluye técnicas de biología molecular en beneficio de la salud humana, detectando y/o cuantificando secuencias genéticas específicas de ácido desoxirribonucleico (ADN), ácido ribonucleico (ARN) o proteínas. Inicialmente, el concepto de “Biología Molecular” se aplicó a los trabajos realizados sobre el ADN. Esta molécula, descubierta por el biólogo y médico suizo Johan Friedrich Miescher en el año 1869, capturó la atención de la comunidad científica, luego que los investigadores James D. Watson, Francis Crick y Rosalind Franklin descubrieran la estructura del ADN en el año 1953 (Watson y Crick, 1953). Este descubrimiento abrió un nuevo horizonte para futuras generaciones en diferentes áreas de la ciencia, incluida la investigación biomédica.
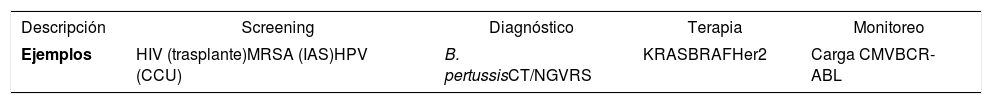
El campo del diagnóstico molecular ha tenido un sostenido crecimiento en los últimos años, sobre el 12% anual, esperando que para el 2017 alcance un mercado por sobre los US$60 billones. En la actualidad, el diagnóstico molecular se ha enfocado principalmente en el diagnóstico de enfermedades infecciosas (50-60%), sin embargo, existe un aumento progresivo de técnicas moleculares en el área de cáncer y enfermedades genéticas, convirtiendo al diagnóstico molecular en una de las áreas de diagnóstico de mayor dinamismo y crecimiento, revolucionando las estrategias para el tratamiento de diversas patologías y condiciones de salud, ofreciendo técnicas con altos estándares de calidad que entregan al equipo clínico información crítica para el cuidado de los pacientes (Tabla 1).
Ejemplo de técnicas de diagnóstico molecular para las diferentes etapas del cuidado del paciente
| Descripción | Screening | Diagnóstico | Terapia | Monitoreo |
|---|---|---|---|---|
| Ejemplos | HIV (trasplante)MRSA (IAS)HPV (CCU) | B. pertussisCT/NGVRS | KRASBRAFHer2 | Carga CMVBCR-ABL |
HIV, virus de la inmunodeficiencia humana; MRSA, Staphylococcus aureus resistente a meticilina; IAS, infecciones asociadas a atención de salud; virus del papiloma humano; CCU, cáncer del cuello uterino; CT, Chlamydia trachomatis; NG, Neisseria gonorrhoeae; CMV, citomegalovirus.
El desarrollo de nuevas técnicas de diagnóstico en medicina tiene su origen en los problemas clínicos que requieren con urgencia un método de diagnóstico, idealmente con una alta sensibilidad y especificidad, que pueda ser accesible a la población. La investigación básica en conjunto con el desarrollo tecnológico, ha permitido el diseño de instrumentos y métodos para responder a las necesidades clínicas. En el caso del diagnóstico molecular, la detección de segmentos de ADN en una muestra problema fue la primera estrategia utilizada en clínica. Mediante técnicas de hibridación de Southern blot, utilizando fragmentos de ADN denominadas sondas, fue posible detectar la presencia de una región de ADN que contenía una secuencia complementaria a la sonda en una muestra problema. Posteriormente, la utilización de enzimas de restricción, tijeras moleculares que cortan el ADN en regiones específicas, junto con el desarrollo de métodos de secuenciación simples, amplió el número de técnicas disponibles para su uso en clínica. Por ejemplo, las técnicas de Southern blot se convirtieron en el gold standard para la detección de reordenamiento de los genes que codifican para las cadenas ligeras y pesadas de las inmunoglobulinas, así como también la familia de receptores de células T, los que eran asociados a linfomas de células T y B. En enfermedades infecciosas, utilizando esta estrategia experimental se diseñó un test aprobado por la FDA para la detección de Chlamydia trachomatis y Neisseria gonorrheae (Tsongalis y Silverman, 2006).
Una de las principales limitaciones de estas técnicas eran los días de arduo trabajo con personal de alto desarrollo técnico, lo que impedía su aplicación masiva en la práctica clínica de rutina. No fue hasta la década de los 80́donde para muchos el mayor invento científico de la biología molecular originó un punto de inflexión en el diagnóstico molecular.
LA REACCIÓN DE LA POLIMERASA EN CADENA (RPC)“….One friday night I was driving, as was my custom, from Berkeley up to Mendocino where I had a cabin far away from everything off in the woods. My girlfriend, Jennifer Barnett, was asleep. I was thinking. Since oligonucleotides were not that hard to make anymore, wouldn’t it be simple enough to put two of them into the reaction instead of only one such that one of them would bind to the upper strand and the other to the lower strand with their three prime ends adjacent to the opposing bases of the base pair in question….”. El párrafo citado corresponde a un fragmento del discurso del bioquímico Kary B. Mullis al recibir el premio Nobel de química en el año 1993 por diseñar la reacción de la polimerasa en cadena (RPC), quizás el mayor aporte en la era del diagnóstico molecular (Saiki y cols., 1988). De forma general, esta técnica permite la amplificación de una región específica de ADN utilizando partidores o secuencias de ADN que delimitan la zona de amplificación. A partir de una copia de la región a amplificar se obtienen millones de copias, lo que permite su detección y de esta forma se evidencia la presencia de la región de ADN en una muestra determinada. Dada la naturaleza de la técnica, la alta especificidad de la RPC viene dada por la hibridación de los partidores complementarios a la secuencia blanco y su elevada sensibilidad a la baja cantidad de ADN que requiere para iniciar la amplificación.
Desde su invención, se han descrito varias variantes de la RPC que han optimizado el diagnóstico clínico. En la tabla 2 se detallan las variantes de RPC más utilizadas en el diagnóstico clínico, destacando sus principales características y aplicaciones.
Tipos de RPC, características y aplicaciones
| Tipo de RPC | Características | Aplicaciones |
|---|---|---|
| RPC Estándar | Amplificación de un segmento de ADN utilizando dos partidores.La detección de la amplificación es mediante geles de agarosa. | Detección cualitativa de un segmento de ADN. |
| RPC Múltiple | Amplificación de 2 o más segmentos de ADN utilizando varios partidores en un sola reacción de amplificación. La detección de la amplificación es mediante geles de agarosa. | Detección cualitativa de varios segmentos de ADN en una sola reacción de RPC |
| RPC-RFLP(Restriction fragment length polymorphisms) | RPC estándar con paso posterior de digestión con enzimas de restricción. | Detección de polimorfismos genéticos (SNPs). |
| RT (Reverse transcriptase)-RPC | Síntesis de cADN a partir de ARN mediante transcripción reversa, seguido de una RPC. | Expresión de genes.Detección de virus ARN. |
| RPC-TR (Real time) o qRPC | RPC estándar donde se utilizan tinciones o sondas con fluoróforos para la detección de los fragmentos amplificados.Puede ser del tipo multiplex. | Detección cualitativa de uno o varios segmentos de ADN.Cuantificación de ADN en la muestra (cargas) o expresión de genes (asociada a una reacción de transcripción reversa). |
Tal como fue mencionado al inicio, el desarrollo de test de diagnóstico molecular ha sido liderado por el área de enfermedades infecciosas y cáncer. A continuación, se describe el impacto del diagnóstico molecular en estas áreas.
DIAGNÓSTICO MOLECULAR EN ENFERMEDADES INFECCIOSASLas enfermedades infecciosas se han transformado en la “punta de lanza” para el desarrollo de test de diagnóstico molecular, siendo más del 50% de las técnicas disponibles hoy en día. La principal explicación a este desarrollo se debe a la dificultad de detectar un patógeno mediante la microbiología clásica. Los largos periodos de crecimiento, las condiciones de cultivo y la obtención de una muestra adecuada son los mayores factores que afectan el diagnóstico microbiológico tradicional. Considerando que las técnicas de biología molecular se basan en la detección de segmentos de ADN, no es necesaria la presencia de un microorganismo viable en la muestra, sino que solo su material genético. Los altos valores de sensibilidad de especificidad de la técnicas de diagnóstico molecular, junto con lo rapidez con la que se pueden obtener los resultados las han transformado en las técnicas de elección para el diagnóstico de enfermedades infecciosas y en muchos casos se consideran los estándares de oro (gold standard) para el diagnóstico de patologías infecciosas, sobre todo en infecciones virales, desplazando al cultivo como método de referencia (Espy y cols., 2006). Esta situación ha llevado a desarrollar técnicas que presenten certificaciones que entregan al equipo clínico seguridad en la calidad de los resultados, ya que en muchos casos estas técnicas definirán el manejo del paciente. Muchas de las técnicas disponibles han evolucionado desde test de uso exclusivo para investigación (RUO: research use only) a productos para el diagnóstico in vivo (IVD: in vitro diagnosis) con certificación de la comunidad europea (marcados como CE) y la Food and Drug Administration (FDA) en EEUU (marcados como FDA-cleared).
Bordetella pertussis es el agente etiológico de la coqueluche (tos ferina). El diagnóstico de este patógeno se realiza mediante técnicas de cultivo microbiológico, ensayos de inmunofluorescencia y diagnóstico molecular, principalmente PCR-TR. El cultivo es una técnica con valores de sensibilidad entre un 12-60%, pero con una alta especificidad, requiriendo medios de transporte adecuados para obtener un resultados días después de obtener la muestra. La inmunofluoresencia presenta niveles de sensibilidad y especificidad similares al cultivo, con la ventaja de obtener un resultado en el transcurso del día. Las técnicas moleculares, por su parte, presenta niveles de sensibilidad entre 70-99% y una especificidad de 86-100%, con un tiempo de detección dentro del rango de 1-4 horas. Esta situación ha llevado a utilizar la RPC para Bordetella pertussis como la técnica de rutina para la detección de este patógeno (Leber, 2014).
Las infecciones por citomegalovirus (CMV) corresponden a una importante causa de morbilidad y mortalidad en pacientes trasplantados. En trasplante de precursores hematopoyéticos, se ha descrito que hasta un 35% de los receptores pueden presentar infección activa por CMV, mientras que en trasplante de órganos sólidos, se ha descrito una incidencia de infección activa y enfermedad por CMV de 36% y 19%, respectivamente. De estos antecedentes, se desprende la necesidad de contar y con métodos de diagnóstico que permitan la detección del CMV, como también su cuantificación para evaluar el tratamiento antiviral. Diversas técnicas del tipo RPC-TR han mostrado una alta sensibilidad y especificidad cuando se compara con técnicas de cultivo viral o antigenemia, transformándola en una herramienta ideal para el diagnóstico, tratamiento y seguimiento de estos pacientes (Farfan y cols., 2011).
DIAGNÓSTICO MOLECULAR EN CÁNCEREl diagnóstico de cáncer en órganos sólidos y hematológicos se ha transformado en un gran desafío, principalmente por el número de alteraciones genéticas descritas, la búsqueda de herramientas que permitan predecir la evolución de la neoplasia y la elección del tratamiento farmacológico adecuado.
El cromosoma Filadelfia, originado por una translocación del cromosoma 9 y 22 que origina una fusión entre los genes BCR y ABL (fusión BCR-ABL) está presente en aproximadamente el 95% de los casos leucemia mieloide crónica y el 25-30% de los casos leucemia linfoblástica aguda en el adulto. Mediante técnicas de diagnóstico molecular es posible detectar la fusión BCR-ABL que indica la presencia del cromosoma Filadelfia. Además, mediante técnicas de RPC-TR es posible monitorear la enfermedad cuantificando los transcritos de la fusión BCR-ABL normalizados con la expresión del gen ABL (Cross y cols., 2015).
Para el caso del cáncer colorectal, la detección de mutaciones en el gen que codifica para la proteína K-Ras (KRAS) permite determinar la respuesta a la terapia utilizando anticuerpos contra el receptor del factor de crecimiento epidérmico (EGFR). Los pacientes que presentan ciertas mutaciones en el gen KRAS se asocian a la activación de la vía se señalización intracelular que depende del EGFR, lo que clínicamente se traducen en una mala respuesta al tratamiento con terapias anti-EGFR. Por lo tanto, el diagnóstico molecular de mutaciones en KRAS es un test realizado en pacientes que podrían verse beneficiados por esta terapia (Dietel y cols, 2015). Actualmente, existen varios test diagnósticos disponibles en el mercado, la mayoría con certificación IVD que aseguran la calidad de los resultados.
DIAGNÓSTICO MOLECULAR DEL TIPO POINT OF CARELa necesidad de contar con un diagnóstico rápido y preciso ha llevado al desarrollo de plataformas de diagnóstico molecular del tipo point of care (POC). Este desarrollo ha estado fuertemente influenciado por la necesidad de implementar test de diagnóstico molecular en lugares donde no existe la infraestructura necesaria para realizarlo o no se cuenta con un laboratorio de referencia de fácil acceso. Para el caso del diagnóstico molecular, los productos POC deben integrar los procesos de extracción y purificación de ácidos nucleicos directamente desde la muestra, con los procesos de amplificación e identificación de los blancos. La primera versión comercial de un test POC fue el sistema Xpert de la empresa Cepheid en el año 2006 y correspondía a un test aprobado por la FDA para la detección de Streptococcus grupo B en mujeres embarazadas en periodo de tiempo de 55 minutos, con valores de sensibilidad y especificidad por sobre el 90%.
Por otra parte, la amenaza latente de un ataque de bioterrorismo en la década anterior, llevó al gobierno de EE.UU. a invertir millones de dólares en proyectos para desarrollar nuevos métodos diagnóstico para la detección de patógenos con el potencial de ser utilizados como armas biológicas, tales como el ántrax. La empresa BioFire Diagnostics, Inc., tomó el desafío de diseñar una plataforma POC para la detección de varios patógenos en una sola reacción para ser utilizada por personal sin mayor entrenamiento en técnicas de diagnóstico. En el año 2011, la FDA aprobó para su uso en clínica el panel Filmarray respiratorio, un sistema con la capacidad de detectar 20 patógenos respiratorios virales y bacterianos en una plataforma integrada que requiere una manipulación de dos minutos por parte del operador y la obtención de resultados en un lapso de una hora (Babady, 2013). El desarrollo posterior de paneles para la detección de patógenos gastrointestinales (Filmarray gastrointestinal), microorganismo asociados a sepsis (Filmarray BCID) o meningitis/encefalitis (Filmarray ME), sin lugar a dudas, marcaran un nuevo punto de inflexión en el diagnóstico molecular.
LIMITACIONES DEL DIAGNÓSTICO MOLECULARUna de las grandes limitaciones del diagnóstico molecular es el costo de los test utilizados. De forma general, estos test superan los valores de los ensayos comúnmente utilizados en clínica para el mismo propósito y en la mayoría de ellos no es posible obtener un reembolso por isapres o compañías aseguradoras, siendo el paciente el responsable de costear estos exámenes. Esta situación se ve mayormente reflejada en los hospitales públicos, donde no existe un presupuesto que contemple la implementación de técnicas moleculares. Sin embargo, esta situación ha cambiado en los últimos años, la competencia de mercado y el desarrollo de plataformas más económicas, ha impulsado la implementación de laboratorios de biología molecular tanto en centros privados como público. Dado el impacto de los test de diagnóstico molecular en el cuidado de los pacientes, varios centros de salud han incorporado laboratorios de biología molecular como una inversión y un polo de desarrollo y marketing. Sin embargo, este desarrollo ha evidenciado la falta de sistemas de regulación tanto en los laboratorios que realizan estas actividades como los insumos utilizados para el diagnóstico molecular. De esto se desprende la necesidad de contar con entidades reguladoras que fiscalicen y normalicen la oferta y calidad de los test de diagnóstico molecular, sobre todo en aquellas técnicas con implicancias clínicas, legales y/o sociales.
Dada las diversas aplicaciones del diagnóstico molecular, otra limitación importante es la integración de los laboratorios de biología molecular con el resto de los laboratorios clínicos, de manera de crecer de forma armónica y transversal ante las necesidades del paciente.
NUEVAS HERRAMIENTAS DE DIAGNÓSTICO MOLECULARCon el creciente desarrollo tecnológico en el área de la biomedicina, existen varias plataformas que han demostrado un gran impacto en la investigación biomédica que lentamente se están incorporando a la práctica clínica.
La secuenciación de nueva generación (NGS: Next-Generation sequencing) ha sido recientemente adaptada para su uso como herramienta de diagnóstico molecular y un creciente número de laboratorios y compañías ofrecen servicios, que van desde la búsqueda y caracterización de patógenos, detección de polimorfismos genéticos, diagnóstico de cáncer, estudio de microbiota, entre otras aplicaciones (Tan y cols., 2015).
De forma general, las diferentes plataformas de NGS tiene la capacidad de secuenciar una gran cantidad de material genético en horas a diferencia de los métodos de secuenciación tradicionales (Sanger) en los que se requerían días a semanas. Nuevamente, las enfermedades infecciosas y cáncer son las áreas donde se ha iniciado la implementación de estas herramientas, principalmente en el estudio de brotes y el diagnóstico de neoplasias, respectivamente. Sin embargo, a diferencia de las plataformas ya existentes, la NGS entrega una gran cantidad de información, la que debe ser cuidadosamente analizada por bioinformáticos en conjunto con el equipo médico para enfocar la búsqueda de lo que se quiere diagnosticar.
Por otro lado, el estudio de polimorfismos genéticos (SNP: single nucleotide polymorphisms) ha tenido un fuerte impacto en la biomedicina, debido a que la presencia de estos pequeños cambios en el ADN pueden tener un gran impacto en la respuesta frente a un fármaco, un microorganismo o explicar una condición patológica particular. El estudio de estas variantes genéticas y su impacto en la salud humana ha llevado a plantear el concepto de “medicina personalizada”, que consiste en entregar un tratamiento adecuado al paciente (genoma) adecuado, basado en que cada individuo presenta una genética diferente y única. Actualmente, se han descritos cientos de SNPs, muchos de ellos de importancia clínica. En un principio la detección de los SNPs se basó en técnicas de RFLP-RPC, sin embargo, debido al gran número de SNP's descritos para una condición en particular, fue necesario utilizar otras plataformas diagnósticas como los microarreglos de ADN o microarrays. Esta plataforma consiste en un chip compuesto de una superficie sólida del tamaño de un portaobjetos a la cual se une fragmentos de ADN, los que se unirán o no al encontrarse con una secuencia de ADN complementaria presente en la muestra a analizar. En cada chip es posible analizar cientos de SNPs en un lapso de un día (Chau y Thomas, 2015).
Debido a su costo, las plataformas de NGS y microarrays se han implementado en algunos centros de salud. Sin embargo, existe un gran número de laboratorios y compañías que ofrecen estos servicios y guían al interesado en la elección de test diagnóstico a utilizar y la interpretación de los resultados obtenidos.
COMENTARIO FINALLa biología molecular se instaló en la práctica clínica. Las numerosas ventajas de las técnicas moleculares con respectos a las técnicas clásicas ha impulsado la implementación de laboratorios de biología molecular tanto en el sistema de salud público y privado. A pesar de este crecimiento masivo, existen varios desafíos que deben ser enfrentados de forma de asegurar diagnósticos precisos y de calidad. Por otra parte, es importante informar y educar al equipo médico y a los futuros profesionales de salud sobre este tipo de técnicas, indicando sus ventajas y limitaciones, de forma de fomentar el desarrollo de equipos multidisciplinarios que en el futuro puedan diseñar, implementar, estandarizar, controlar e interpretar estas herramientas diagnósticas.
El autor declara no tener conflictos de interés, en relación a este artículo.
Referencias no citadasSaiki et al., 1988, Espy et al., 2006, Leber, in pressa, Farfán et al., 2012, Cross et al., 2015, Dietel et al., 2015 and Tan et al., 2015.