Despite positive serological outcome, molecular confirmations have encountered little/no success either due to protocol accuracy, primer targets, or choice of sample amongst others. This study aims at providing an optimized protocol for the molecular detection of Zika virus amongst serologically positive respondent using samples from 2 selected hospitals in North Central Nigeria.
Materials and methodsAbout five (5) ml of blood samples was collected from a total of 400 participants for serological analysis, the IgM-positive samples were processed for molecular analysis using target primers from Asian and African lineage while a structured questionnaire was used to evaluate risk factors.
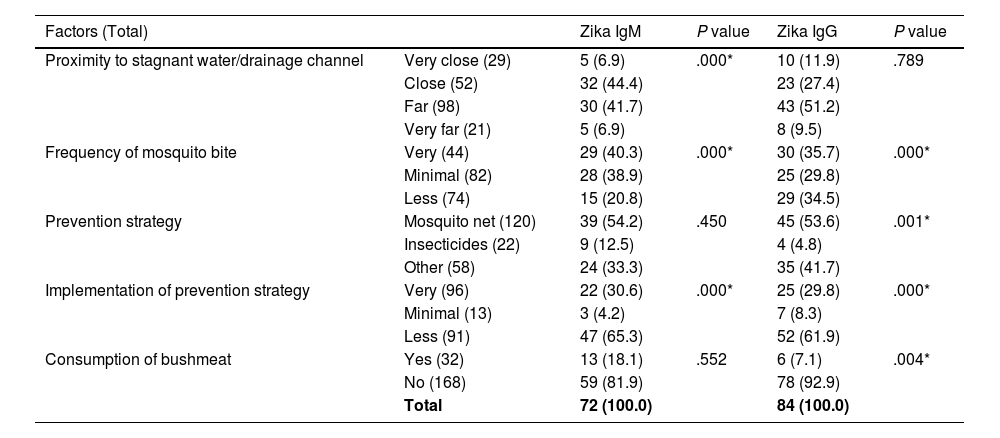
ResultsPrevalence of 19% (38) and 45% (90) IgM and IgG positivity was recorded amongst respondent in Federal Medical Center (FMC), Keffi (R2=1) while 36% (72) and 42% (84) was recorded in General Hospital (GH), Minna (R2=1). The respective risk factors such as proximity of respondent to stagnant water or drainage channel, frequency of mosquito bite, prevention strategy, implementation of the prevention strategies for mosquito, and consumption of bushmeat were significant at set standard of P<0.05. Molecular quantification revealed cut-off values (Ct) from 21.73 to 25.75 for all the 3 targeted protein while sequence analysis showed relatedness to deposited sequences in GenBank.
ConclusionsThe abundance of the viral proteins as well as the genetic relatedness is indicative of presence of multiple strains of the virus or conservation of region across different geolocations. In lieu of the outcome, primers from multiple lineages is thereby recommended to forestall/overcome the challenge of cross-reactivity/false-negativity with Zika virus detection.
A pesar de obtener resultados serológicos positivos, las confirmaciones moleculares han tenido poco o ningún éxito, ya sea debido a la precisión del protocolo, los objetivos de los cebadores y la elección de la muestra, entre otros factores. Este estudio tiene como objetivo proporcionar un protocolo optimizado para la detección molecular del virus Zika entre los encuestados con resultados serológicos positivos utilizando muestras de dos hospitales seleccionados en el centro-norte de Nigeria.
Materiales y MétodosSe recogieron 5 ml de muestras de sangre de un total de 400 participantes para el análisis serológico. Las muestras con resultados positivos para IgM se procesaron para el análisis molecular utilizando cebadores específicos de los linajes asiáticos y africanos, mientras que se utilizó un cuestionario estructurado para evaluar los factores de riesgo.
ResultadosSe registró una prevalencia del 19% (38) y el 45% (90) de positividad para IgM e IgG, respectivamente, entre los encuestados en el Centro Médico Federal (FMC) de Keffi (R2=1), mientras que se registró un 36% (72) y el 42% (84) en el Hospital General (GH) de Minna (R2=1). Los factores de riesgo correspondientes, como la proximidad de los encuestados a agua estancada o canales de drenaje, la frecuencia de las picaduras de mosquitos, las estrategias de prevención y la implementación de las estrategias de prevención para los mosquitos y el consumo de carne de animales silvestres, fueron significativos según el estándar establecido de p<0,05. La cuantificación molecular reveló valores de corte (Ct) que oscilan entre 21.73 y 25.75 para las tres proteínas objetivo, mientras que el análisis de secuencias mostró similitud con secuencias depositadas en GenBank.
ConclusionesLa abundancia de las proteínas virales, así como la similitud genética, indica la presencia de múltiples cepas del virus o la conservación de una región en diferentes ubicaciones geográficas. En base a estos resultados, se recomienda el uso de cebadores de múltiples linajes para evitar/superar el desafío de la reactividad cruzada o los falsos negativos en la detección del virus Zika.