El síndrome de ovario poliquístico es una enfermedad compleja y heterogénea que implica problemas reproductivos y metabólicos. Se ha sugerido una predisposición genética en la etiología de este síndrome. La identificación del gen de la calpaína-10 (CAPN10) como el primer gen asociado a diabetes mellitus tipo 2 suscitó el interés por investigar su posible relación con el síndrome de ovario poliquístico, debido a que este síndrome se asocia a hiperinsulinemia y resistencia a la insulina, 2 anormalidades metabólicas relacionadas con diabetes.
ObjetivoInvestigar si existe asociación entre el SNP-63 y la variante indel-19 del gen CAPN10 y el síndrome de ovario poliquístico en mujeres en edad reproductiva.
Material y métodosEl estudio incluyó a 101 mujeres (55 con síndrome de ovario poliquístico y 46 clínicamente sanas). La variante indel-19 se identificó mediante corrimiento electroforético de los fragmentos amplificados por PCR y el SNP-63 por PCR-RFLP.
ResultadosLas frecuencias alélicas y genotípicas de las 2 variantes no difieren significativamente entre pacientes con síndrome de ovario poliquístico y mujeres del grupo control. El haplotipo 21 (definido por el alelo inserción de la variante indel-19 y el alelo C del SNP-63) se encontró con mayor frecuencia en los 2 grupos de estudio, siendo más frecuente en el grupo de pacientes; sin embargo, esta diferencia no fue estadísticamente significativa (p = 0.8353).
ConclusionesLos resultados sugieren que el SNP-63 y la variante indel-19 del gen CAPN10 no son factores de riesgo para síndrome de ovario poliquístico en nuestro grupo de pacientes.
Polycystic ovary syndrome is a complex and heterogeneous disease leading to reproductive, as well as metabolic problems. It has been suggested that there may be a genetic predisposition in the aetiology of polycystic ovary syndrome. The identification of calpain 10 gene (CAPN10) as the first candidate gene for type 2 diabetes mellitus has focused the interest in investigating their possible connection with the polycystic ovary syndrome. This syndrome is associated with hyperinsulinaemia and insulin resistance, two metabolic abnormalities associated with type 2 diabetes mellitus.
ObjectiveTo investigate if there is association between the SNP-63 and the genetic variant indel-19 of CAPN10 gene and polycystic ovary syndrome in women of reproductive age.
Material and methodsThis study included 101 women (55 with polycystic ovary syndrome and 46 without polycystic ovary syndrome). The genetic variant indel-19 was identified by electrophoresis of the amplified fragments by PCR, and the SNP-63 by PCR-RFLP.
ResultsThe allele and genotype frequencies of the two variants do not differ significantly between women with polycystic ovary syndrome and the control women group. The haplotype 21 (defined by the insertion allele of indel-19 variant and C allele of SNP-63) was found with higher frequency in both study groups, being more frequent in the polycystic ovary syndrome patients group, however, this difference was not statistically significant.
ConclusionsThe results suggest that SNP-63 and indel-19 variant of CAPN10 gene do not represent a risk factor for polycystic ovary syndrome in our patient group.
El síndrome de ovario poliquístico es un desorden endocrino heterogéneo considerado como la causa más común de anovulación, hiperandrogenismo e infertilidad en mujeres en edad reproductiva1. Otras de las principales características clínicas del síndrome de ovario poliquístico son la obesidad (principalmente del segmento superior) y anormalidades metabólicas, tales como hiperinsulinemia y resistencia a la insulina, lo que constituye un factor de riesgo para desarrollar diabetes mellitus tipo 2 y enfermedad cardiovascular2,3.
La prevalencia del síndrome de ovario poliquístico en diferentes poblaciones es del 3-7% en mujeres en edad reproductiva y en mujeres con hiperandrogenismo es del 60-80%4. En México se ha reportado una prevalencia del 6%5.
La fisiopatología del síndrome de ovario poliquístico es compleja; sin embargo, las 2 principales alteraciones hormonales observadas en las pacientes con síndrome de ovario poliquístico incluyen: la elevación de los niveles circulantes de la hormona luteinizante y de la insulina (hiperinsulinemia); de hecho, se ha sugerido que existe sinergismo entre ambas, por lo que la hiperestimulación ovárica por la insulina provocaría hiperandrogenismo2. Se ha considerado que el síndrome de ovario poliquístico es un trastorno con un componente genético en su etiología y esta condición ha sido apoyada por la marcada agregación familiar y porque se ha documentado que el grado de concordancia es mayor en las gemelas monocigotas que en las dicigotas2. Las teorías con respecto al modo de herencia del síndrome de ovario poliquístico incluyen un modelo multifactorial en donde la interacción de factores ambientales (nutrición y actividad física) y un grupo de genes causales (genes candidatos de susceptibilidad) contribuyen a su variabilidad fenotípica6.
La asociación del síndrome de ovario poliquístico con resistencia a la insulina y riesgo para desarrollar diabetes mellitus tipo 2 condujo a suponer que el defecto primario causante de este síndrome podría residir en alguno de los mediadores de la ruta metabólica de la respuesta a la insulina7. Desde que el gen de la calpaína-10 (CAPN10) fue identificado por Horikawa et al.8 en el año 2000 como el primer gen asociado a diabetes mellitus tipo 2, surgió el interés por investigar su posible relación con el síndrome de ovario poliquístico, debido a la participación documentada del producto proteico de este gen en la vía de señalización de la insulina6,7,9–11.
La calpaína-10 es una cisteína-proteasa no lisosomal activada por calcio, perteneciente al grupo de calpaínas atípicas por carecer del dominio IV8,12. Esta calpaína se expresa en diferentes tejidos, tales como islotes pancreáticos, músculo esquelético, hígado y adipocitos13. Aunque su función específica aún no ha sido totalmente esclarecida, se ha propuesto que la calpaína-10 es una molécula importante en la función de las células β del páncreas, ya que se ha demostrado que regula la exocitosis de los gránulos secretores de insulina14,15.
También se ha indicado que la calpaína-10 facilita la translocación de GLUT4 a través de un efecto distal sobre la vía de acción de la insulina. Además, la calpaína-10 también ha sido implicada en la reorganización del citoesqueleto de actina que acompaña tanto a la translocación de vesículas GLUT 4 como a la secreción de insulina14,16.
El CAPN10 se localiza en la región distal del brazo largo del cromosoma 2 (2q37.3), y abarca 65,674 nucleótidos en longitud, distribuidos en 15 exones y 14 intrones14,16. De las variantes que han sido identificadas en el gen CAPN10, destacan un SNP localizado en el intrón 3, el SNP-43 (G>A; rs3792267), y otro localizado en el intrón 13, el SNP-63 (C>T; rs5030952), así como una inserción/deleción de 32 pb, referida en la literatura médica como SNP-19, presente en el intrón 6 (variante indel-19; rs3842570), ya que se ha descrito que, de manera independiente o como haplotipo, contribuyen a la susceptibilidad a diabetes mellitus tipo 2 en diversas poblaciones8,17. Algunos estudios se han centrado en la relación del síndrome de ovario poliquístico con variantes en el gen CAPN101,6,9–11,18–24; sin embargo, la influencia de las variaciones genéticas en la susceptibilidad para el desarrollo de patologías multifactoriales como síndrome de ovario poliquístico parece diferir entre poblaciones; por lo mismo, el objetivo de este estudio fue analizar la distribución de alelos y genotipos de 2 de las variantes más comunes del gen CAPN10 (el SNP-63 y la variante indel-19) en pacientes con síndrome de ovario poliquístico y en mujeres sin síndrome de ovario poliquístico para evaluar si existe asociación entre este síndrome y las 2 variantes en mujeres en edad reproductiva.
Material y métodosSe incluyó en el estudio a un total de 101 mujeres (55 con diagnóstico de síndrome de ovario poliquístico y 46 sin síndrome de ovario poliquístico), todas ellas en edad reproductiva (18-38 años) y no emparentadas entre sí. Los tamaños de muestra para los 2 grupos de estudio (55 y 46) superan el mínimo (36 individuos) para un sistema de polimorfismos bialélicos (α 0.05; p = 0.05) de acuerdo a Chakraborty25.
Todas las mujeres incluidas en el estudio eran mestizas, residentes del estado de Jalisco. Previo a la firma de la carta de consentimiento informado de participación en el estudio, las mujeres que aceptaron participar fueron informadas acerca de la finalidad alidad, los beneficios ios y los procedimientos que se llevaron a cabo en el proyecto. El protocolo de estudio fue conducido de acuerdo con la Declaración de Helsinki y fue aprobado por el Comité Local de Investigación y Ética del Instituto Mexicano del Seguro Social (#2005/1/I/064).
Las pacientes con síndrome de ovario poliquístico fueron consecutivamente captadas en los servicios de Fertilidad y Endocrinología de la Unidad Médica de Alta Especialidad del Hospital de Ginecología y Obstetricia del Centro Médico Nacional de Occidente del Instituto Mexicano del Seguro Social. El diagnóstico de síndrome de ovario poliquístico fue de acuerdo con los criterios del consenso ASRM/ESHRE de Rotterdam revisados en 200326, complementado con el abordaje integral del síndrome de ovarios poliquísticos (Guía de práctica clínica, CIE-10:E28, Instituto Mexicano del Seguro Social)27 (tabla 1). De las 55 pacientes con síndrome de ovario poliquístico, 44 cursaron con sobrepeso (índice de masa corporal > y 11 presentaron obesidad (índice de masa corporal ≥ 30).
Criterios revisados de Rotterdam ESHRE/ASRM 2003 complementados con la GPC CIE-10: E28
| Criterios diagnósticos (2 de 3 criterios) | ||
|---|---|---|
| 1. Oligo y/o anovulación | Historial de 8 o menos ciclos menstruales en un año o ciclos menstruales menores de 26 días o más de 35 días y P4 < 4 ng/ml | |
| 2. Signos de hiperandrogenismo | Clínico | Hirsurtismoa |
| Acné | ||
| Patrón alopecia masculino | ||
| Bioquímico | Testosterona total > 84.7 ng/dl | |
| Testosterona libre > 0.75 ng/dl | ||
| Índice de testosterona libre/andrógeno | ||
| DHEA | ||
| DHEAS > 2459 ng/ml | ||
| Relación LH/FSH > 2 | ||
| Niveles disminuidos de globulina transportadora de esteroides sexuales | ||
| 3. Ovarios poliquísticos | Presencia de 12 o más folículos en cada ovario con una medida de 2 a 9mm de diámetro, y/o volumen ovárico incrementado > 10mm (evaluación por ultrasonido) | |
| Exclusión de otras patologíasb | ||
| Síndromes de resistencia a la insulina severa | Síndrome de Cushing | |
| HAIRAN | ||
| Neoplasma secretor de andrógenos | ||
| Hiperplasia suprarrenal congénita | Deficiencia de 21 hidroxilasa (17 hidroxiprogesterona 2-3 ng/ml) | |
| Tumores secretores de andrógenos | Tumores de Sertoli-Leydig, adenomas suprarrenales | |
| Hiperprolactinemia | Elevación de niveles de prolactina | |
| Hipogonadismo hipogodotrófico | Niveles séricos bajos de FSH y niveles disminuidos de E2 | |
| Falla ovárica prematura | Niveles séricos elevados de FSH y niveles disminuidos de E2 | |
| Estudios complementarios para evaluar comorbilidades | ||
| Perfil de lípidos en ayuno | ||
| Glucosa sérica en ayuno | ||
| Insulina sérica en ayuno | ||
| Razón glucosa/insulina < 4.5 indica resistencia a la insulina | ||
DHEA: dehidroepiandrosterona; DHEAS: sulfato de dehidroepiandrosterona; E2: estradiol; FSH: hormona foliculoestimulante; HAIRAN: síndrome acantosis nigricans resistente a la insulina hiperandrogénica; LH/FSH: relación hormona luteinizante/hormona folículo estimulante; P4: progesterona.
a Evaluado por sistema de puntaje estandarizado (Ferriman-Gallwey).
b La exclusión de otras patologías forma parte de la evaluación inicial. Criterios revisados de Rotterdam ESHRE/ASRM 200326. Abordaje integral del síndrome de ovarios poliquísticos27.
Las mujeres del grupo control fueron seleccionadas al azar y tuvieron ciclos menstruales regulares entre 21-28 días, con un índice de masa corporal < 25 y sin historia personal y familiar de síndrome de ovario poliquístico, las cuales asistieron al hospital para chequeo general de salud.
No se incluyó en el estudio a mujeres embarazadas o que estuvieran bajo terapia anovulatoria, antiandrogénica o con corticoides. Las variables de confusión, como actividad física y régimen alimenticio, no fueron consideradas para este estudio.
GenotipificaciónEl ADN genómico fue extraído a partir de leucocitos de sangre periférica de acuerdo con un protocolo estándar28, con algunas modificaciones ciones, y fue almacenado en buffer TrisEDTA pH 8.0 a –20°C hasta su procesamiento.
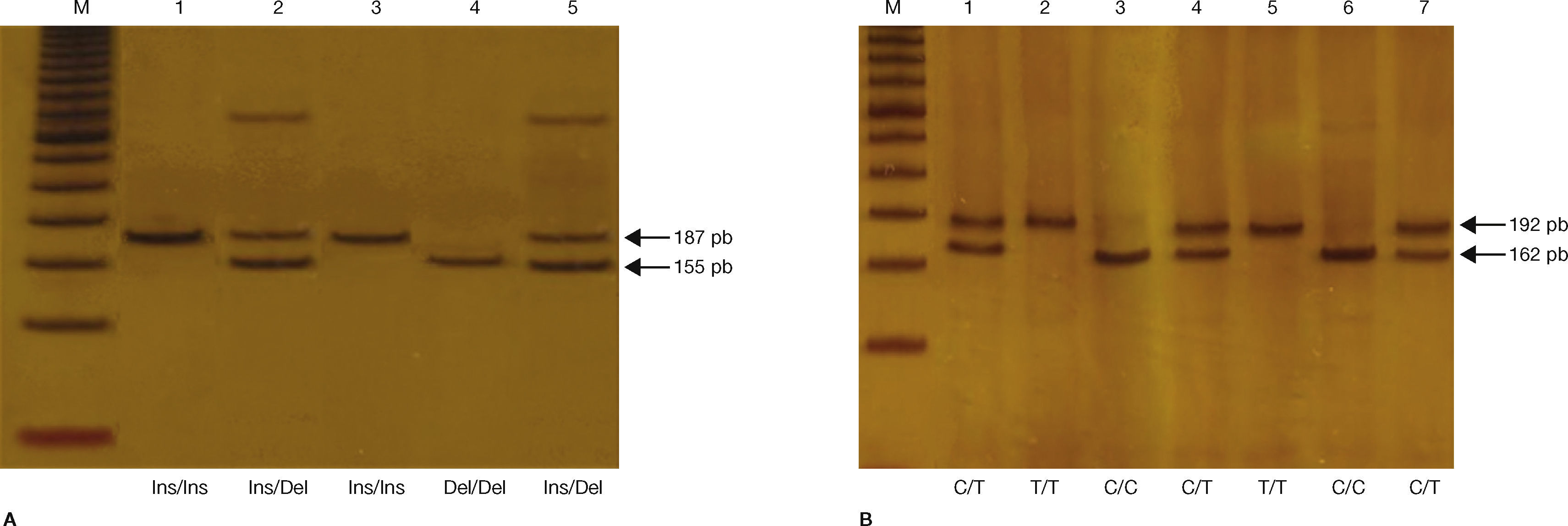
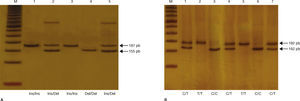
Para la identificación del SNP-63 (C16378T), se amplificó un fragmento de 192 pb, el cual se sometió a digestión con 2 U de la enzima de restricción HhaI a 37°C, de acuerdo con las instrucciones del fabricante (New England Biolabs, Ipswich, EE. UU.), lo que generó un fragmento de 192 pb si el alelo T estaba presente y 2 fragmentos uno de 162, y otro de 30 pb si estaba presente el alelo C29. Para la identificación de la variante indel-19, se amplificó un fragmento de 155 pb en presencia de la deleción (3 repeticiones de 32 pb, alelo 2R), mientras que si estaba presente la inserción el fragmento amplificado era de 187 pb (2 repeticiones de 32 pb, alelo 3R)29 (tabla 2). La diferencia en pares de bases de los productos de PCR y los productos de la digestión enzimática se analizó en geles de poliacrilamida al 6%, teñidos con nitrato de plata (fig. 1). Como control interno de calidad, se seleccionaron muestras con los genotipos de referencia para su corroboración por electroforesis capilar en un secuenciador automático Beckman-Coulter modelo CEQ8800 (California, EE. UU.).
Abordaje metodológico para la identificación de las dos variantes del gen CAPN10
| Variante | Iniciadores | Fragmento amplificado | Enzima de restricción | Fragmentos esperados |
|---|---|---|---|---|
| SNP-63 | Sentido | 192 pb | HhaI | Alelo C 162 +30 pb |
| 5’AAGGGGGGCCAGGGCCTGACGGGGGTGGCG3’Antisentido | GCG^C | Alelo T 192 pb | ||
| Indel-19 | 5’AGCACTCCCAGCTCCTGATC3’Sentido | |||
| inserción/deleciónde | 5’GTTTGGTTCTCTTCAGCGTGGAG3Antisentido | 155 pb | – | Alelo Ins 187 pb |
| 32 pb | 5’CATGAACCCTGGCAGGGTCTAAG3’ | 187 pb | Alelo Del 155 pb |
pb: pares de bases.
A) Genotipificación de la variante indel-19. Productos de PCR separados en gel de poliacrilamida al 6% teñido con nitrato de plata. M: marcador de peso molecular (escalera de ADN de 50 pb). Carriles 1 y 3 homocigoto Ins/Ins (fragmento de 187 pb); carriles 2 y 5 heterocigoto Ins/Del (fragmentos de 187 pb y 155 pb); carril 4 homocigoto Del/Del (fragmento de 155 pb). B) Genotipificación del SNP-63. Productos de PCR digeridos con la enzima Hha1 separados en gel de poliacrilamida al 6% teñido con nitrato de plata. M: marcador de peso molecular (escalera de ADN de 50 pb). Carriles 3 y 6 homocigoto C/C (fragmento de 162 pb); carriles 1, 4 y 7 heterocigoto C/T (fragmentos de 192 pb y 162 pb); carriles 2 y 5 homocigoto T/T (fragmento de 192 pb).
Los haplotipos formados por los 2 polimorfismos del gen CAPN10 fueron inferidos considerando la variante indel-19 en la primera posición y el SNP-63 en la segunda posición. Esto genera 4 posibles haplotipos: haplotipo 11 (alelo deleción de la variante indel-19, alelo C del SNP-63), haplotipo 12 (alelo deleción de la variante indel-19, alelo T del SNP63), haplotipo 21 (alelo inserción de la variante indel-19, alelo C del SNP-63) y haplotipo 22 (alelo inserción de la variante indel-19, alelo T del SNP-63).
Análisis estadísticoLas frecuencias alélicas de las 2 variantes del gen CAPN10 se determinaron por el método de conteo directo de los genotipos observados y se presentaron como frecuencias simples.
La comparación de las frecuencias alélicas, genotípicas y de haplotipos de la variante indel-19 y del SNP-63, entre las pacientes con síndrome de ovario poliquístico y las mujeres del grupo control, así como la determinación del equilibrio de Hardy-Weinberg se realizó mediante la prueba de la chi al cuadrado (χ2). Se consideró como valor estadísticamente significativo si el valor de probabilidad fue menor de 0.05.
El análisis de los datos se realizó utilizando el programa estadístico RxC con 10,000 iteraciones30. Para inferir los haplotipos se utilizó el programa HaplotypeReconstructor_v0.6.
ResultadosLa edad promedio de las mujeres del grupo control fue de 26 años; así mismo el 80% de estas mujeres presentaron ciclos menstruales de 25 a 28 días, mientras que en las pacientes con síndrome de ovario poliquístico el promedio de edad fue de 29 años y el 88% de ellas tuvieron ciclos menstruales de 30 a 60 días.
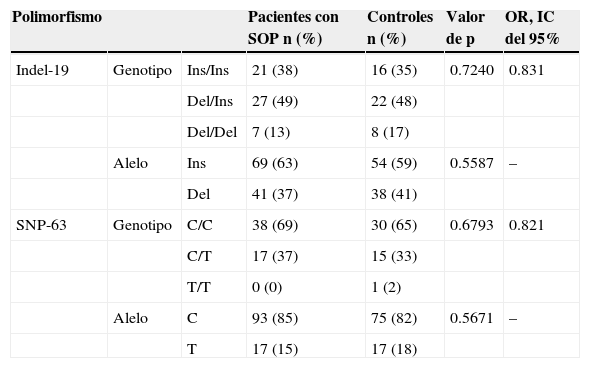
La distribución de alelos y genotipos del SNP-63 y la variante indel-19 en las pacientes con síndrome de ovario poliquístico y las mujeres del grupo control se muestran en la tabla 3. La distribución de genotipos observados de las 2 variantes estuvo de acuerdo con lo esperado con el equilibrio de Hardy-Weinberg (p > 0.05). Las frecuencias genotípicas de la variante indel-19 y del SNP-63 no difieren significativamente entre el grupo de pacientes con síndrome de ovario poliquístico y las mujeres del grupo control (p = 0.7240 y p = 0.6793, respectivamente). El análisis de las frecuencias alélicas demostró que el alelo inserción de la variante indel-19 y el alelo C del SNP-63 se encontraron con mayor frecuencia en el grupo de pacientes con síndrome de ovario poliquístico comparado con el grupo control; sin embargo, esta diferencia no fue estadísticamente significativa (p = 0.5587 y p = 0.5671, respectivamente).
Distribución de alelos y genotipos del SNP-63 y la variante indel-19 en las pacientes con síndrome de ovario poliquístico y las mujeres del grupo control
| Polimorfismo | Pacientes con SOP n (%) | Controles n (%) | Valor de p | OR, IC del 95% | ||
|---|---|---|---|---|---|---|
| Indel-19 | Genotipo | Ins/Ins | 21 (38) | 16 (35) | 0.7240 | 0.831 |
| Del/Ins | 27 (49) | 22 (48) | ||||
| Del/Del | 7 (13) | 8 (17) | ||||
| Alelo | Ins | 69 (63) | 54 (59) | 0.5587 | – | |
| Del | 41 (37) | 38 (41) | ||||
| SNP-63 | Genotipo | C/C | 38 (69) | 30 (65) | 0.6793 | 0.821 |
| C/T | 17 (37) | 15 (33) | ||||
| T/T | 0 (0) | 1 (2) | ||||
| Alelo | C | 93 (85) | 75 (82) | 0.5671 | – | |
| T | 17 (15) | 17 (18) |
Del: deleción; n: número de sujetos de estudio (genotipo) o cromosomas (alelo); Ins: inserción; SOP: síndrome de ovario poliquístico.
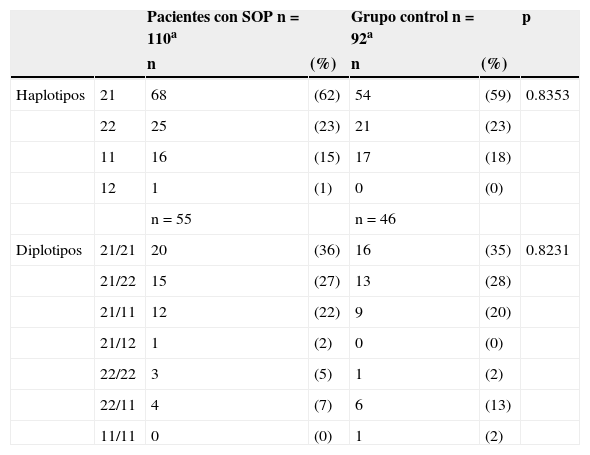
En la población de estudio se observaron los 4 posibles haplotipos. La combinación 21 (alelo inserción de la variante indel-19, alelo C del SNP–63) y el haplodiplotipo 21/21 fueron los que se encontraron con mayor frecuencia en los 2 grupos de estudio (tabla 4), pero al comparar las frecuencias observadas del total de haplotipos y haplodiplotipos entre el grupo de pacientes con síndrome de ovario poliquístico y el grupo control no se encontraron diferencias estadísticamente significativas (p = 0.8353 y p = 0.8231, respectivamente).
Distribución de haplotipos y diplotipos del SNP-63 y la variante indel-19 en pacientes con síndrome de ovario poliquístico y controles
| Pacientes con SOP n = 110a | Grupo control n = 92a | p | ||||
|---|---|---|---|---|---|---|
| n | (%) | n | (%) | |||
| Haplotipos | 21 | 68 | (62) | 54 | (59) | 0.8353 |
| 22 | 25 | (23) | 21 | (23) | ||
| 11 | 16 | (15) | 17 | (18) | ||
| 12 | 1 | (1) | 0 | (0) | ||
| n = 55 | n = 46 | |||||
| Diplotipos | 21/21 | 20 | (36) | 16 | (35) | 0.8231 |
| 21/22 | 15 | (27) | 13 | (28) | ||
| 21/11 | 12 | (22) | 9 | (20) | ||
| 21/12 | 1 | (2) | 0 | (0) | ||
| 22/22 | 3 | (5) | 1 | (2) | ||
| 22/11 | 4 | (7) | 6 | (13) | ||
| 11/11 | 0 | (0) | 1 | (2) |
SOP: síndrome de ovario poliquístico.
n: número de individuos; SOP: síndrome de ovario poliquístico.
a n: número de cromosomas.
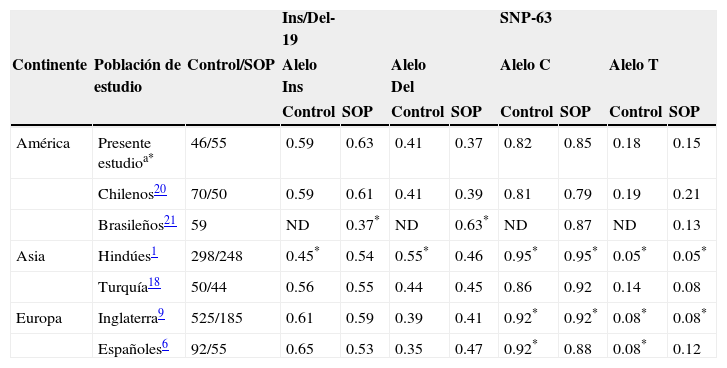
De manera adicional, también se realizó la comparación de las frecuencias alélicas de la variante indel-19 y del SNP–63 observadas en la población aquí estudiada con las frecuencias descritas en otros estudios conducidos en poblaciones de América (Chile y Brasil), Asia (China, Corea, India y Turquía), y Europa (Alemania, España, e Inglaterra) (tabla 5).
Frecuencias alélicas de la variante indel-19 y el SNP-63 del gen CAPN10 en diferentes poblaciones
| Ins/Del-19 | SNP-63 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Continente | Población de estudio | Control/SOP | Alelo Ins | Alelo Del | Alelo C | Alelo T | ||||
| Control | SOP | Control | SOP | Control | SOP | Control | SOP | |||
| América | Presente estudioa* | 46/55 | 0.59 | 0.63 | 0.41 | 0.37 | 0.82 | 0.85 | 0.18 | 0.15 |
| Chilenos20 | 70/50 | 0.59 | 0.61 | 0.41 | 0.39 | 0.81 | 0.79 | 0.19 | 0.21 | |
| Brasileños21 | 59 | ND | 0.37* | ND | 0.63* | ND | 0.87 | ND | 0.13 | |
| Asia | Hindúes1 | 298/248 | 0.45* | 0.54 | 0.55* | 0.46 | 0.95* | 0.95* | 0.05* | 0.05* |
| Turquía18 | 50/44 | 0.56 | 0.55 | 0.44 | 0.45 | 0.86 | 0.92 | 0.14 | 0.08 | |
| Europa | Inglaterra9 | 525/185 | 0.61 | 0.59 | 0.39 | 0.41 | 0.92* | 0.92* | 0.08* | 0.08* |
| Españoles6 | 92/55 | 0.65 | 0.53 | 0.35 | 0.47 | 0.92* | 0.88 | 0.08* | 0.12 |
ND: no disponible; SOP: síndrome de ovario poliquístico.
a Grupo de referencia.
* p < 0.05.
Se ha descrito la asociación del síndrome de ovario poliquístico con resistencia a la insulina y el riesgo para desarrollar diabetes mellitus tipo 2; el presente estudio se llevó a cabo para evaluar la posible asociación de este síndrome con 2 de los polimorfismos del gen CAPN10 (variante indel-19 y SNP-63) implicados en el haplotipo de riesgo (121) para desarrollar diabetes mellitus tipo 2 en mexico-americanos8, y se encontró que la distribución de alelos y genotipos fue similar en pacientes con síndrome de ovario poliquístico y mujeres sin síndrome de ovario poliquístico, lo que permite sugerir que en nuestro grupo de pacientes la variante indel-19 y SNP-63 no representan un factor de riesgo para desarrollar síndrome de ovario poliquístico. Este hallazgo está de acuerdo con lo descrito en 2 estudios previos, uno conducido en Inglaterra9 y el otro en Turquía18, en donde además de analizar el SNP-63 y la variante indel-19, analizaron otras 2 variantes del gen CAPN10 (SNP-44 y SNP-43) y no hubo asociación de ninguna de las 4 variantes con síndrome de ovario poliquístico. Aun cuando existen varios estudios en los que se da evidencia de la implicación de variantes en el gen CAPN10 en el desarrollo de síndrome de ovario poliquístico, la asociación no es siempre con la misma variante, ya que, por ejemplo, en mujeres alemanas se reportó la asociación de este síndrome con el SNP-5611; en mujeres de España6, de Turquia19 y de India1 la asociación fue con el SNP-44, mientras que en 2 poblaciones de América Latina, Chile20 y Brasil21, la asociación fue con el SNP-43.
Existen relativamente pocos estudios en los que se haya investigado la relación entre el síndrome de ovario poliquístico y los haplotipos del gen CAPN10. En un estudio realizado en mujeres alemanas, en las que se analizaron 8 variantes del gen CAPN10 (incluidos indel-19 y SNP-63), se encontró que el haplotipo TGA2AGCA representa un riesgo incrementado para síndrome de ovario poliquístico11. En mujeres de India, donde se analizaron 5 variantes (también incluidos indel-19 y SNP-63), se encontró asociación significativa con el haplotipo 211211. En mujeres españolas, en las que se analizaron 4 variantes (SNP-44, SNP-43, indel-19 y SNP-43), se encontró asociación del haplotipo 1121 pero con hipercolesterolemia en pacientes con síndrome de ovario poliquístico10. En mujeres coreanas donde se analizaron solo 3 variantes (SNP-43, indel-19 y SNP-63), la asociación con síndrome de ovario poliquístico se encontró con el haplotipo 11122. En el presente estudio, al analizar solo 2 variantes, el haplotipo que se encontró con mayor frecuencia en las pacientes con síndrome de ovario poliquístico fue el haplotipo 21 (alelo inserción de la variante indel-19, alelo C del SNP– 63), el cual es parcialmente similar al haplotipo 121 descrito por Horikawa et al., en población mexico-americana8, sin embargo, no se alcanzó valor estadístico significativo cuando se compararon las frecuencias haplotípicas con el grupo control, descartando así su posible asociación con síndrome de ovario poliquístico en nuestro grupo de pacientes estudiadas. Por lo anterior, es necesario analizar otras variantes en el gen CAPN10 para investigar la contribución de un haplotipo extendido en el desarrollo de este síndrome.
Aun cuando nuestros resultados sugieren la no implicación en el desarrollo de síndrome de ovario poliquístico de la variante indel-19 y el SNP-63 del gen CAPN10, ya sea de manera individual o como haplotipo, el presente estudio es de particular importancia ya que, hasta nuestro conocimiento, este es el primer estudio en el que se investiga la posible asociación de variantes del gen CAPN10 y haplotipos con síndrome de ovario poliquístico en población mexicana.
El análisis comparativo de las frecuencias de alelos entre diferentes poblaciones demostró que las frecuencias alélicas de la variante indel-19 en nuestro grupo de pacientes con síndrome de ovario poliquístico fueron diferentes de las encontradas en pacientes con síndrome de ovario poliquístico de la población de Brasil21, así mismo se observaron diferencias entre las frecuencias de nuestro grupo control con el grupo control de la población de la India1. En relación con el SNP-63, además de que también se encontraron diferencias cuando se compararon las frecuencias alélicas observadas, tanto en nuestro grupo de pacientes con síndrome de ovario poliquístico, como en nuestro grupo control con las frecuencias reportadas en la India1, también se encontraron diferencias con las frecuencias reportadas en el estudio realizado en Inglaterra9 y con el estudio conducido en población española, solo que con esta última población las diferencias fueron solo entre los grupos controles6. Las diferencias encontradas con Inglaterra y la India pueden ser causa de la poca o casi nula migración documentada de los ingleses y de los hindúes a México. El análisis de varios polimorfismos genéticos en poblaciones de Europa y de América, incluido Brasil, han dado evidencia de la variación en la estructura genética de las diferentes poblaciones, posiblemente como resultado de las distintas mezclas interetnias31–34. Nuestros resultados sugieren que la población mestiza mexicana tiene características genéticas particulares que la diferencian de otras poblaciones de todo el mundo.
Los factores genéticos que están implicados en el síndrome de ovario poliquístico y sus distintas expresiones podrían explicar la variación fenotípica en la presentación clínica del síndrome, la cual es fisiológicamente heterogénea y controversial en los determinantes genéticos, ya que en cada población se han encontrado resultados diferentes analizando incluso el mismo polimorfismo. Las diferencias étnicas entre los sujetos de estudio parecen estar contribuyendo a estas diferencias.
Por lo expuesto anteriormente, y debido a la etiología multifactorial del síndrome de ovario poliquístico, se debe considerar como mecanismos moleculares de este síndrome la sumatoria de factores genéticos de riesgo, ya que el efecto de un solo gen no es suficiente para favorecer el desarrollo del desbalance endocrino-metabólico que presentan las pacientes, en interacción con factores ambientales desencadenantes que contribuyen a la aparición del síndrome.
ConclusionesLa distribución de alelos, de genotipos y de haplotipos de la variante indel-19 y del SNP–63 no difieren significativamente entre las pacientes con síndrome de ovario poliquístico y las mujeres del grupo control, por lo que estos hallazgos sugieren que estas 2 variantes del gen CAPN10 no representan un factor de riesgo para el síndrome de ovario poliquístico, por lo que no pueden ser utilizados en la práctica clínica como marcadores de riesgo genético en la población estudiada. El análisis poblacional mostró que se presentan diferencias con la población de Brasil, España, Inglaterra e India, lo que indica que existe heterogeneidad en las frecuencias de las variantes del gen CAPN10 entre diferentes poblaciones.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este trabajo fue parcialmente apoyado por el Fondo de Investigación en Salud, del Instituto Mexicano del Seguro Social con número de registro 2005/1/I/064. Queremos agradecer al Dr. Sergio Alberto Sánchez Walle por el apoyo brindado para la captación de las mujeres participantes en el estudio.