Introducción
La importancia que reviste el diagnóstico microbiológico de las hepatitis infecciosas no necesita ser destacado; entre éstas, ocupan un lugar fundamental las originadas por virus. Desde el punto de vista conceptual el cuadro de afectación hepática suele consistir en inflamación y alteración de la función de los hepatocitos por virus primariamente hepatotropos (objeto de este trabajo) y otros no hepatotropos que serán abordados en otra revisión de esta serie.
Las muestras utilizadas para el diagnóstico de hepatitis víricas por virus hepatotropos combinan las técnicas indirectas con algunas directas pero casi siempre se llevan a cabo a partir de muestras de plasma o suero. En algunos casos pueden utilizarse muestras alternativas al suero (saliva, orina) que contienen cantidades variables de anticuerpos (Ac), siempre con fines de estudios epidemiológicos. Con ninguno de estos virus se emplea el cultivo como opción diagnóstica, por ello en algunos casos, el diagnóstico directo por detección de elementos estructurales del virus (antígenos o ácido nucleico) precisa además de las muestras séricas otras procedentes de biopsias. En general las muestras de plasma para diagnóstico de hepatitis víricas por métodos moleculares no deben contener heparina ya que inhibe la Taq polimerasa y deben ser conservadas a 70 °C cuando se demore su análisis más de 72 h; ya que el material nucleico de los virus ARN puede desnaturalizarse si se prolonga su conservación a temperaturas superiores.
Por las especiales implicaciones de riesgo biológico asociadas a su transmisión entre el personal de laboratorio las normas de seguridad deben ser escrupulosamente observadas. El establecimiento del diagnóstico virológico de los virus hepatotropos obliga a observar por parte del laboratorio unas normas de seguridad que se encuadran en un nivel 2. Todas las muestras deben considerarse como potencialmente infecciosas y en consecuencia manipularlas con los mismos criterios de bioseguridad 1,2. Ello conlleva emplear guantes en todos los procesos, pipeteado mecánico, utilizar contenedores de seguridad biológica para los desechos y observar aquellas normas consideradas como de buena práctica 3.
Los objetivos que se persigan con el diagnóstico van a influir en la elección de la técnica apropiada para la determinación de marcadores, en la actitud del profesional del laboratorio ante un resultado y en la estrategia de confirmación. De forma conceptual aludimos a pruebas diagnósticas a las que se emplean de forma individualizada en el suero de una persona bajo los principios clínicos de consentimiento informado 4. Cuando las pruebas se aplican en virtud de criterios de seguridad biológica, vigilancia epidemiológica o investigación y el objetivo principal no es el diagnóstico, se establece una referencia a la detección de marcadores con otras denominaciones ajustadas al ámbito de aplicación.
Las pruebas de determinación de marcadores han experimentado un considerable desarrollo y optimización desde su diseño inicial. Las mejoras han afectado tanto a los soportes antigénicos empleados para la detección de anticuerpos como a los principios técnicos en los que se fundamentan las reacciones 5. La progresiva implantación de sistemas automatizados que completan la secuencia global de la detección de marcadores y que permiten el seguimiento de la trazabilidad total de las muestras ha minimizado sustancialmente el consumo de recursos en términos de costes y cargas de trabajo 6.
En el presente trabajo pretendemos realizar una exposición estructurada de los métodos de diagnóstico virológico de los principales virus hepatotropos implicados en la etiología de las hepatitis víricas. En función de sus características epidemiológicas abordamos en primer término los virus de las hepatitis A y E, de transmisión preferente enteral y en segunda instancia los virus de las hepatitis B, D y C, de transmisión fundamentalmente parenteral.
Diagnóstico del virus de la hepatitis A
El virus de la hepatitis A (VHA) es virus desnudo de simetría icosaédrica, de pequeño tamaño (27 nm), clasificado dentro de género Hepatovirus de la familia Picornaviridae. Desde el punto de vista estructural el virión está constituido desde dentro afuera por un genoma de ARNmc lineal de polaridad positiva de 7.438 nucleótidos, que codifica una proteína de 2.227 aminoácidos de la que se derivan proteínas funcionales y cuatro proteínas estructurales (VP1 a VP4) que forman el nucleocápside. El cápside se sitúa más externamente al ácido nucleico y posee simetría isosaédrica y en él se identifica el denominado antígeno VHA (Ag VHA); careciendo de membrana de envoltura 7. Esta cualidad dota al virus de una considerable resistencia a los agentes externos y a las condiciones ambientales naturales superior a cualquier otro picornavirus, lo que explica su capacidad infecciosa. Las cepas son indistinguibles serológicamente y se integran en un mismo serotipo, a pesar de ello existe una variación de hasta el 20% en la secuencia de aminoácidos entre diferentes aislados víricos. En las cepas humanas se han descrito cuatro genotipos (I, II, III y VII), siendo los restantes genotipos simios.
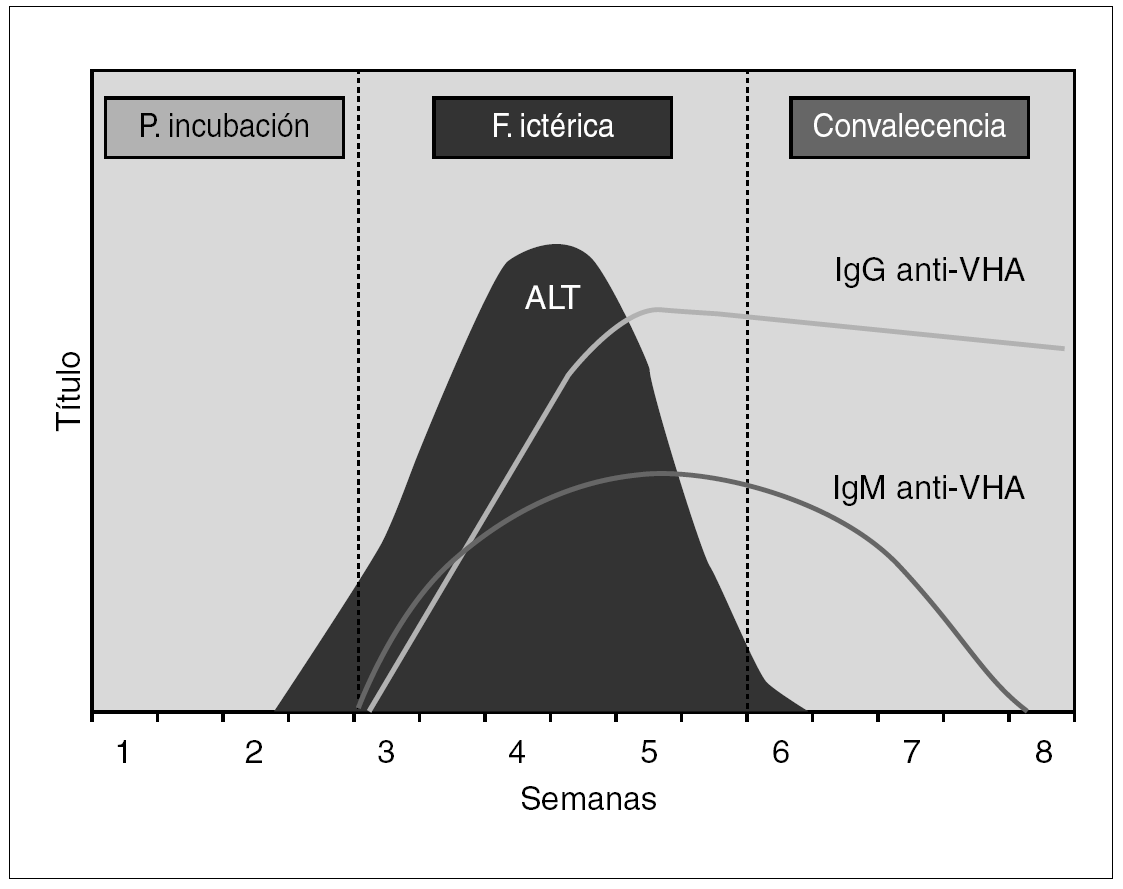
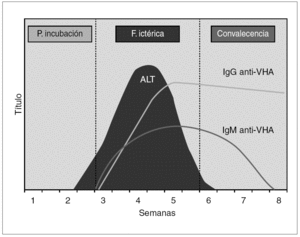
La estrategia de diagnóstico virológico habitual asienta en la detección de marcadores serológicos de infección por VHA, cuya cronología se ilustra en la figura 1. Los individuos infectados desarrollan de forma precoz y en el contexto del primer mes anticuerpos de la clase IgM frente al Ag VHA, que pueden ser detectables durante los 3 a 12 meses posteriores a la curación de la enfermedad. En segundo término se elevan los anticuerpos de la clase IgG frente al Ag VHA, que persisten indefinidamente y confieren una inmunidad permanente 8. En el momento actual las técnicas de enzimoinmunoanálisis (EIA) se han consolidado como las de mayor implantación, por su versatilidad en cuanto a su rendimiento e integración en protocolos eficientes de diagnóstico. En general el diagnóstico de hepatitis A se establece por la presencia de inmunoglobulina (IgM) anti-Ag VHA, que coincide con los síntomas clínicos de la hepatitis y tiende a declinar e incluso a desaparecer a partir de los 3 a 6 meses posteriores. La presencia de IgG perdura toda la vida y no distingue entre infección actual o pasada.
Figura 1. Curso serológico de la infección por VHA.
El VHA se puede detectar en las heces de los pacientes hasta 10 días antes de la aparición de la ictericia, persistiendo en ellas hasta 3 meses después; incluso tras la normalización de la bioquímica hepática, habiéndose empleado para ello la microscopia electrónica. La detección de Ag VHA o de viriones en materia fecal al inicio del cuadro clínico, si bien es posible, tiene poco interés práctico y su implantación casi nula en nuestro entorno. La búsqueda de ARN genómico se reserva a laboratorios de investigación y se efectúa mediante técnicas de amplificación basadas en la reacción en cadena de la polimerasa (PCR) en distintas versiones o en hibridación 9. En función de su transmisión enteral estos métodos diagnósticos se han aplicado con éxito a la documentación de la circulación del VHA en alimentos y muestras del medio ambiente 10.
Diagnóstico del virus de la hepatitis E
El virus de la hepatitis E (VHE) mantiene semejanzas morfológicas y epidemiológicas con el VHA. Posee un tamaño que oscila entre 32 y 34 nm y se clasifica dentro de género de los calicivirus 11. Desde el punto de vista estructural el virión está constituido desde dentro afuera por un genoma de ARNmc lineal de polaridad positiva de unos 7,6 kb, que presenta tres marcos de lectura abierta. De ellos el mayor codifica la síntesis de proteínas funcionales, el intermedio lo hace para proteínas estructurales del nucleocápside y el más pequeño codifica proteínas frente a las cuales aparecen anticuerpos en el suero de los individuos infectados. En este sentido se admite la existencia de un único serotipo de VHE, a pesar de que existe una diversidad genómica de hasta el 25% entre diferentes aislados 12.
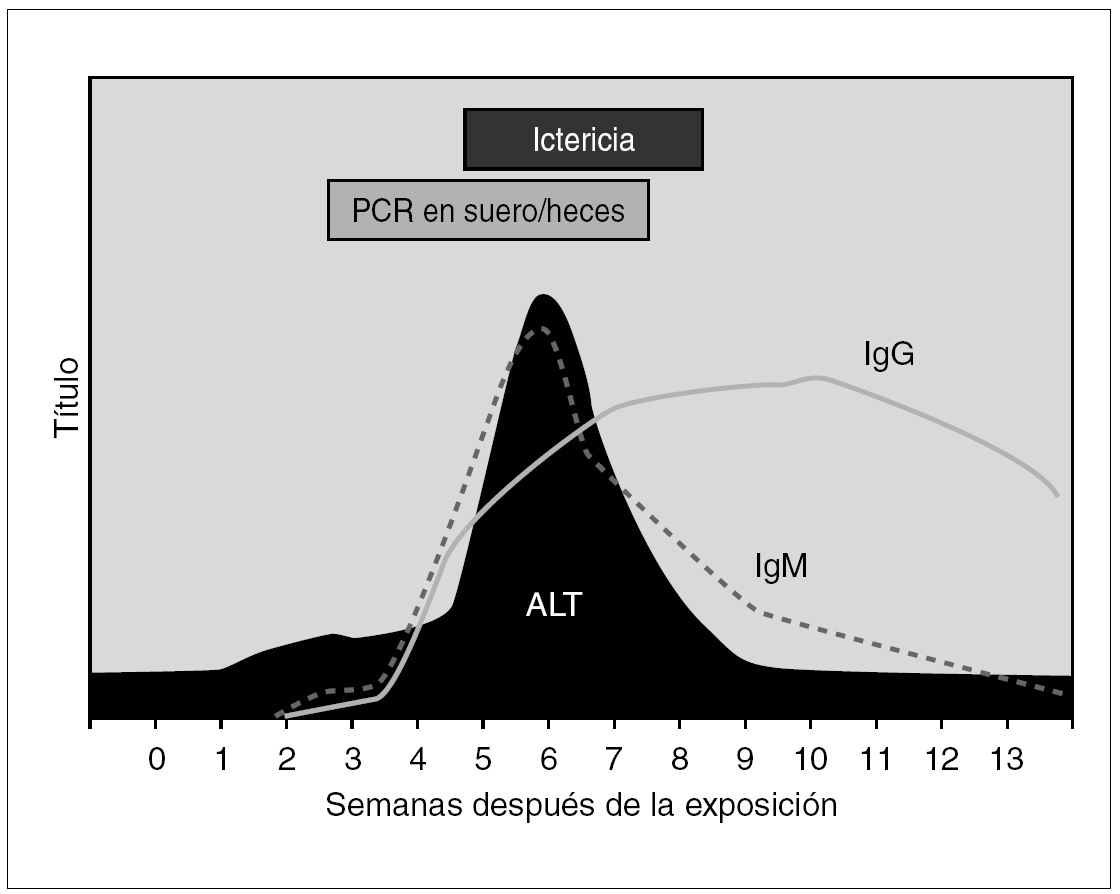
El diagnóstico virológico no está disponible en la oferta asistencial habitual de los laboratorios de nuestro entorno, debido a que se considera una enfermedad importada 13 y cuando se efectúa asienta en la detección de marcadores serológicos de infección por VHE. Los individuos infectados, al igual que se ha avanzado para el VHA, desarrollan de forma precoz anticuerpos de la clase IgM frente al VHE, que disminuyen rápidamente tras la infección aguda y declinan hasta niveles muy bajos a lo largo de los 9 a 12 meses posteriores. Los anticuerpos de la clase IgG adoptan un patrón de elevación progresiva con un cierto retraso para lo expuesto para los de la clase IgM y sus títulos se mantienen elevados durante períodos más prolongados y tanto su determinación aislada como la seroconversión en términos de cuadruplicación de su título pueden comportarse como indicadores fiables de infección aguda 14.
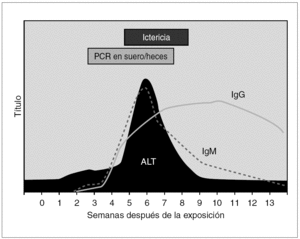
Desde el punto de vista patogénico es posible documentar una fase inicial de viremia o de eliminación fecal del VHE, coincidiendo con la infección aguda. Los métodos virológicos de detección genómica obligan a optimizar la especificidad de los promotores empleados en las diferentes reacciones de amplificación mediante PCR, que aunque prometedoras, distan mucho de ser aplicables a la asistencia clínica ordinaria 15. La cronología de los diferentes marcadores de infección se ilustra en la figura 2.
Figura 2. Curso serológico de la infección por VHE.
Diagnóstico del virus de la hepatitis B
El virus de la hepatitis B (VHB) perteneciente a la familia Hepadnaviridae infecta actualmente a alrededor de 400 millones de personas. La infección, distribuida por todo el mundo con distintos grados de endemia; tiene una elevada mortalidad debido a sus dos principales complicaciones, cirrosis y hepatocarcinoma 16. Las zonas del mundo con mayor endemicidad son Asia, África Subsahariana y el Pacífico. España se considera una región con prevalencia moderada de la infección cifrándose en el 2-7% los portadores crónicos.
El VHB es un virus ADN, envuelto y con cápside de simetría icosaédrica. Su genoma está formado por ADN bicatenario circular y asimétrico (L o cadena negativa con 3.200 nucleótidos y S o cadena positiva con una longitud variable de unos 2.000 nucleótidos) 17. Ambas cadenas están ancladas entre sí por las llamadas regiones de cohesión (DR1 y DR2). Las características genómicas de este virus son únicas dentro de los virus ADN, puesto que a la asimetría de sus cadenas se une el que adquiere una forma circular y el que los genes que codifican para sus proteínas son parcialmente solapantes entre sí. De hecho podemos distinguir 4 regiones abiertas de transcripción: S, C, P y X solapándose en los 3.200 nucleótidos del genoma que codifican 7 proteínas, de las cuales cuatro son estructurales (antígeno del core del virus de la hepatitis B [HBcAg] antígeno de superficie del virus de la hepatitis B [HBsAg], gp36 y gp42) y tres no estructurales (ADN polimerasa, antígeno X del virus de la hepatitis B [HBxAg] y antígeno e del virus de la hepatitis B [HBeAg]). El HBxAg se considera actualmente involucrado en los procesos de replicación y transcripción viral. Además, la replicación viral es mediada por ARN, gracias a la actividad retrotranscriptasa de su ADN polimerasa. Todas estas circunstancias contribuyen a la gran variabilidad genómica del virus, que a menudo presenta no sólo mutaciones puntuales, sino también deleciones e inserciones a lo largo de su genoma. La variabilidad se considera del orden del 10% y se traduce en la existencia de una gran variedad de genotipos, subtipos antigénicos y otras variantes estables 18. Hasta la fecha se han descrito 8 genotipos diferentes: A, B, C, D, E, F, G y H distribuidos por todo el mundo generados a partir de una misma estirpe, probablemente debido entre otros a fenómenos de recombinación 19. Además se definen diferentes subtipos antigénicos dependiendo de la combinación de los siguientes determinantes: el determinante "a", común para todos, y los determinantes mutuamente excluyentes d/y y w/r. En España, todos estos subtipos y genotipos están representados, aunque predominan las asociaciones D/ayw2 (30,1%), D/ayw3 (28,6%) y A/adw2 (21,2%) 20. Se han descrito variantes estables de las 4 regiones abiertas de transcripción, sin embargo entre ellas algunas tienen más relevancia para la salud pública, como son los mutantes del HBsAg (sobre todo mutantes de la región denominada determinante "a"), las variantes pre-core defectivas y las variantes del gen X. La proporción de estas variantes en la población de pacientes infectados no se conoce con exactitud, aunque algunos estudios apuntan a frecuencias altas y crecientes de los mutantes del HBsAg 21-24. La explicación de por qué estas variantes van en aumento, parece estar en el uso creciente de tratamientos antivirales cuya diana es la polimerasa 25. Los genes que codifican para la polimerasa viral y para la proteína del HBsAg están parcialmente superpuestos, con distintas fases de lectura, lo que motiva que presión biológica sobre uno de ellos provoque alteraciones sobre el otro.
El efecto de los mutantes en la evolución clínica y en el diagnóstico de la infección debe ser considerado. La progresión de la enfermedad es variable dependiendo de factores virales y factores del huésped. Entre los primeros destaca la variabilidad del curso clínico para los diferentes genotipos, así como variantes genéticas más agresivas, como las variantes pre-core defectivas o los mutantes del gen X17. El diagnóstico de VHB se centra fundamentalmente en la identificación de casos agudos de infección primaria, persistencia e infección crónica y la existencia de patrones atípicos.
Como norma que se debe tener en cuenta los distintos antígenos y componentes estructurales del VHB (excepto el HBcAg) aparecen en el suero o plasma del paciente durante la infección aguda y algunos en los estados de cronicidad; los anticuerpos contra ellos generados forman parte de la respuesta natural del huésped en el intento natural de eliminar la infección, por ello su aparición secuencial obliga a un seguimiento en el tiempo.
Infección primaria. Después de un período de incubación, que puede llegar hasta las 10 semanas, se desarrolla la infección aguda sintomática o no, que puede resolverse o bien cronificar. Durante el período de incubación, puede detectarse el genoma del VHB como única manifestación viral. La caracterización del estado de la infección se realiza mediante el seguimiento de parámetros virales referidos a la expresión o no de sus antígenos, fundamentalmente el HBsAg y el antígeno "e" (HBeAg) o la detección y cuantificación del ADN viral. Los ensayos para detección de HBsAg pueden ser confirmados mediante ensayos de neutralización en los que se produce una reducción de la señal superior al 50% entre muestras duplicadas ensayadas en paralelo, utilizando en una de ellas un suero que contiene HBsAc. La sensibilidad de los ensayos de HBsAg se aproxima hasta menos de 0,1 ng/ml en los más recientes, sin merma de la especificidad, aunque dada la gran cantidad de dicho antígeno que aparece en el suero y plasma de los individuos infectados, el cambio de test antiguos por alguno de los modernos no se traduce necesariamente en un aumento de la detección de individuos HBsAg positivos.
Además, también debe valorarse la respuesta del huésped de anticuerpos específicos, como son los generados frente a los antígenos HBsAg, HBeAg y HBcAg denominados respectivamente HBsAc, HBeAc y HBcAc. El diagnóstico del estadio de la enfermedad se completa mediante la puesta en evidencia de la lesión hepática con pruebas de laboratorio como la transaminemia y anatomopatológicas.
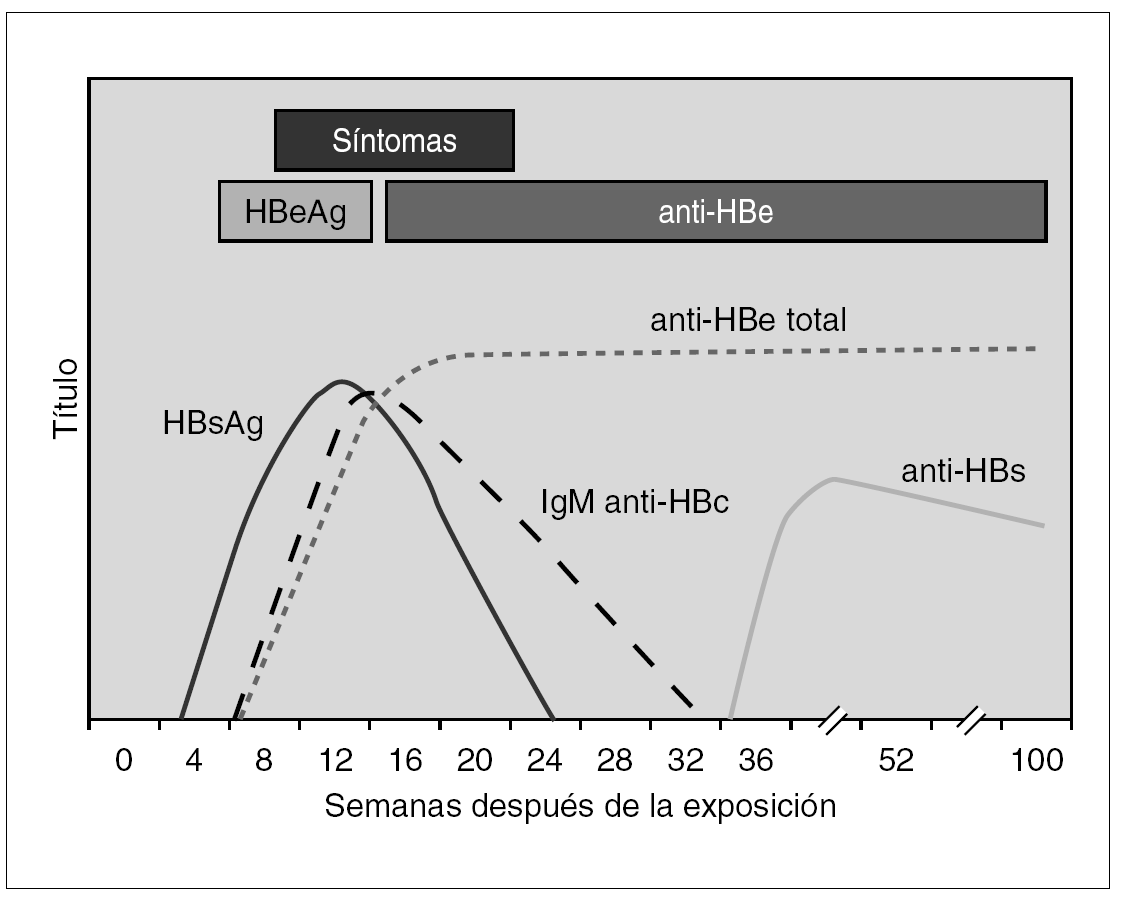
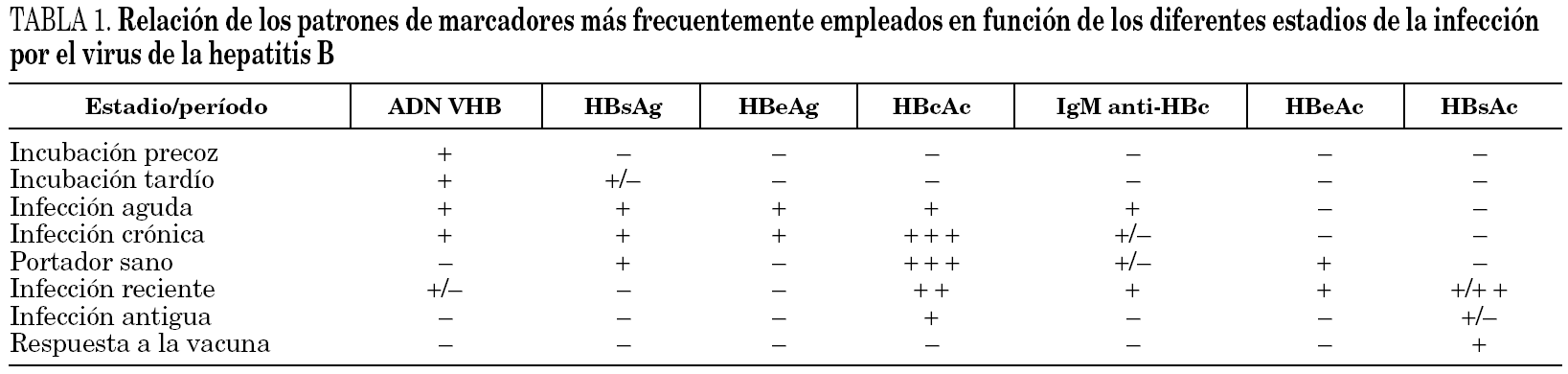
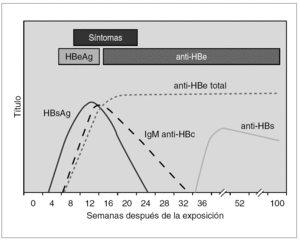
En la infección primaria aparecen los marcadores HBsAg, HBeAg e IgM-HBc, en los casos en que no aparezca IgM-HBc puede ser de ayuda la detección de ADN y en todo caso la obtención de sueros posteriores para verificar el diagnóstico (fig. 3). En este momento diagnóstico se debe descartar la presencia de coinfección VHD como será expuesto más adelante. Las distintas combinaciones de estos marcadores durante el período de incubación precoz y tardío aparecen reflejadas en la tabla 1. La resolución clínica condiciona de una parte la desaparición de HBsAg y de otra la aparición de HBcAc y HBsAc.
Figura 3. Curso serológico de la infección por VHB aguda sin complicaciones.
Infección persistente y crónica. En el caso de que no se produjese seroconversión de HBsAc y se mantuviese el HBsAg durante más de 6 meses se establece un estado de persistencia crónica del VHB. En este contexto la monitorización virológica se verá beneficiada por la determinación del ADN vírico y de los marcadores del sistema HBeAg y HBeAc, tal y como se recoge en la referida tabla. Los pacientes con variantes pre-core defectivas mostrarán presencia de HBeAc y de viremia. Justamente la realización de determinaciones cuantitativas de esta última puede lograrse mediante métodos de hibridación molecular directa; más reproducibles y precisos o bien con técnicas de amplificación genómica dotadas de mayor sensibilidad. No obstante, cualquier método con sensibilidad superior a 10 3 copias/ml es adecuado para identificar este estadio clínico en casos de infección persistente con replicación activa del virus.
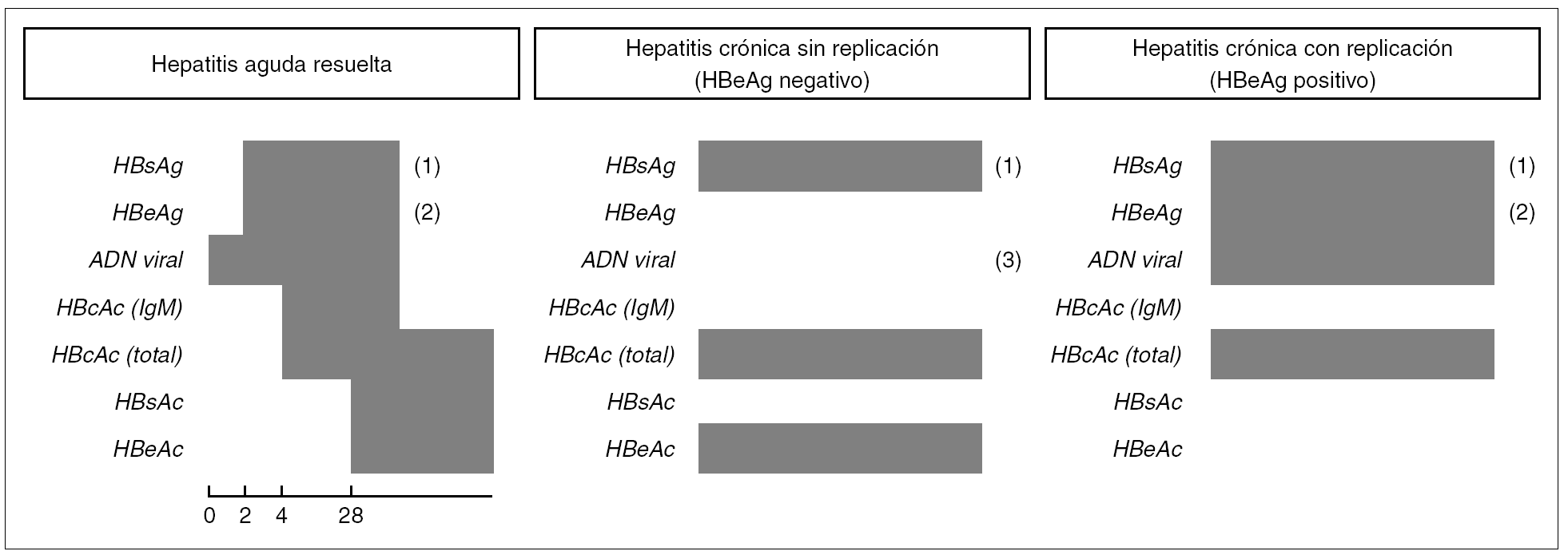
Existen patrones serológicos (resumidos en la fig. 4), que se relacionan con otros estadios de la infección, además de la infección primaria (aguda o de forma asintomática) y de la hepatitis crónica con replicación viral o sin ella.
Figura 4. Cronología de los marcadores virológicos en patrones de infección por VHB. (1) Podría ser negativo en el caso de mutaciones en el determinante "a" del HBsAg. (2) Podría ser negativo en el caso de mutantes pre-core defectivas. (3) Niveles bajos.
Patrones atípicos. En determinadas circunstancias se documentan patrones "atípicos" de los antedichos marcadores. En primer término cabe aludir a la reactividad aislada del HBsAg, en ausencia de HBcAc. En este caso es preceptivo confirmar la especificidad de la reactividad a través de una prueba de neutralización con HBsAc, detección de HBeAg, presencia de IgM anti-HBc y de ADN vírico; todo ello en sueros pareados obtenidos con un intervalo de 2-3 semanas. Si se confirma la reactividad específica cabe interpretar situaciones en las que la reactividad desaparece (contaminación) o en las que se mantiene (infección crónica sin respuesta de anticuerpos, infección por variantes no caracterizadas). En segundo lugar cabe asistir a patrones con presencia simultánea de HBsAg y HBsAc que cuando se ponen de manifiesto conjuntamente con HBcAc y presencia de HBeAg y/o viremia apunta generalmente a una reactivación de la infección en el contexto de una inmunosupresión. En tercera instancia puede evidenciarse una reactividad aislada con HBcAc en ausencia de HBcAc y con negatividad para HBsAg; lo cual a punta hacia la necesidad de determinar marcadores complementarios tales como los del sistema "e" y ADN (permitiendo etiquetar unos casos como HBsAg no detectable por fenómeno prozona y otros como infección resuelta con pérdida de HBcAc) y descartar que se trate de una reacción inespecífica.
A pesar de ello los patrones a veces son incompletos y, por tanto, difíciles de interpretar. En el origen de estos patrones incompletos se encuentran distintos factores como son, títulos de anticuerpos bajos, el fenómeno conocido como VHB tipo 2, que parece estar relacionado con mutaciones en el gen X26 o la existencia de variantes virales en las regiones C o S generadas como consecuencia de la alta variabilidad genómica del virus 24. Las variantes que principalmente influyen en defectos en los parámetros diagnósticos son las siguientes: a) variantes pre-core defectivas: que dan lugar a proteínas truncadas debido a codones de parada a distintos niveles del gen y por tanto déficit de expresión del HBeAg; b) mutantes del gen X: dan lugar a un HBxAg mutado, por lo que el virus se replica y expresa de manera anómala dando lugar a débil expresión del resto de los antígenos y estimulación inmune insuficiente; c) mutantes del HBsAg: que producen déficit de expresión del mismo 27 o bien, expresándose en cantidad teóricamente detectable, las mutaciones producen alteración de las proteínas del virión, que escapan a los tests diagnósticos diseñados 28-33. Comisiones de expertos reunidas recientemente aconsejan identificar la frecuencia de estas variantes así como su impacto real en los tests diagnósticos 34. En España se ha evaluado la frecuencia y se ha estimado que el 12,5% de los portadores crónicos están infectados con alguna de dichas variantes 24. Sin embargo, el impacto real sobre los tests diagnósticos actuales parece ser mucho menos dramático, y sobre todo asociado con algunos cambios como los que involucran a las posiciones 120 (Pro120Gln), 126 (THr/Ile126Ser), 143 (Thr/Ser143Leu), 144 (Asp144Ala) o 145 (Gly145Arg/Ala), o con los virus que tienen acumulación de cambios de aminoácido a lo largo del determinante "a". Otro hecho relevante es en qué proporción está representado el mutante en el contexto de toda la población viral. Muestras en las que predomina un determinado mutante, pueden ser perfectamente detectadas gracias a la población minoritaria de virus no mutados. También se conoce la distinta capacidad de los diferentes tests comerciales para detectar cada uno de los mutantes 24,35.
La vacunación frente a VHB incluida desde hace unos años en los calendarios vacunales de España influirá en la presencia de HBsAc de forma aislada en gran número de personas. El uso del sistema de HBsAc se ha extendido para verificar la protección alcanzada tras la vacunación, la Organización Mundial de la Salud (OMS) y los Centers for Disease Control and Prevention (CDC) han aceptado que un nivel de estos anticuerpos de 10 mU/ml tiene carácter protector. Existen distintas aproximaciones para la cuantificación de estos anticuerpos basadas en paneles de sueros estándar que generan una curva y otros que calibran un umbral situado en las 10 mU/ml.
La vacunación junto con el cribado virológico de hemoderivados y la inmunoprofilaxis maternofetal mediante inmunoglobulina específica, ha disminuido significativamente la incidencia de la infección. Sin embargo se han descrito variantes con cambios de aminoácidos en el HBsAg que pueden escapar a la protección de la vacuna 29,36-41. Algunos de estos cambios descritos son en las posiciones 120 (Pro120Ser/Glu), 126 (Thr/Ile126Asn/Ser/Ala), 129 (Gln129His/Arg), 133 (Met133Leu), 141 (Lys141Glu), 142 (Pro142Ser), 144 (Asp144Ala) y 145 (Gly145Arg/Ala). Algunas de esas sustituciones en las posiciones 114, 118, 120, 121, 123, 124, 126, 129-131, 133, 134, 137, 139-142 y 144-147 condicionan una resistencia a la inmunoterapia específica para prevenir la infección en neonatos de madres infectadas 21,29,42-49. La frecuencia real de los mutantes asociados a fallos en la protección de la vacuna o en resistencia a la inmunoterapia específica se desconoce, pero en España se estima que en portadores crónicos podrían alcanzar el 6,6 y el 9,2% respectivamente 24. Además los virus con determinadas mutaciones en el determinante "a", podrían no ser detectables en el cribado de hemoderivados debido a falsos negativos en la detección de HBsAg.
Diagnóstico del virus de la hepatitis D
El virus de la hepatitis D (VHD) o agente delta es un virus defectivo que requiere al VHB para su replicación y posterior expresión. El virión es una partícula con un tamaño que oscila entre 36 y 43 nm y desde el punto de vista estructural desde dentro hacia fuera está constituido por un genoma de ARNmc de 1,7 kb, con cinco marcos de lectura, de los que sólo uno es responsable de la síntesis de las 2 proteínas principales del cápside (p24 y p27) que posee simetría icosaédrica y se asocia con el antígeno delta (Ag D) 50. En la parte más externa posee una membrana de envoltura similar a la del VHB, el VHD es un virus quimérico que alberga estructuras derivadas de 2 genomas diferentes.
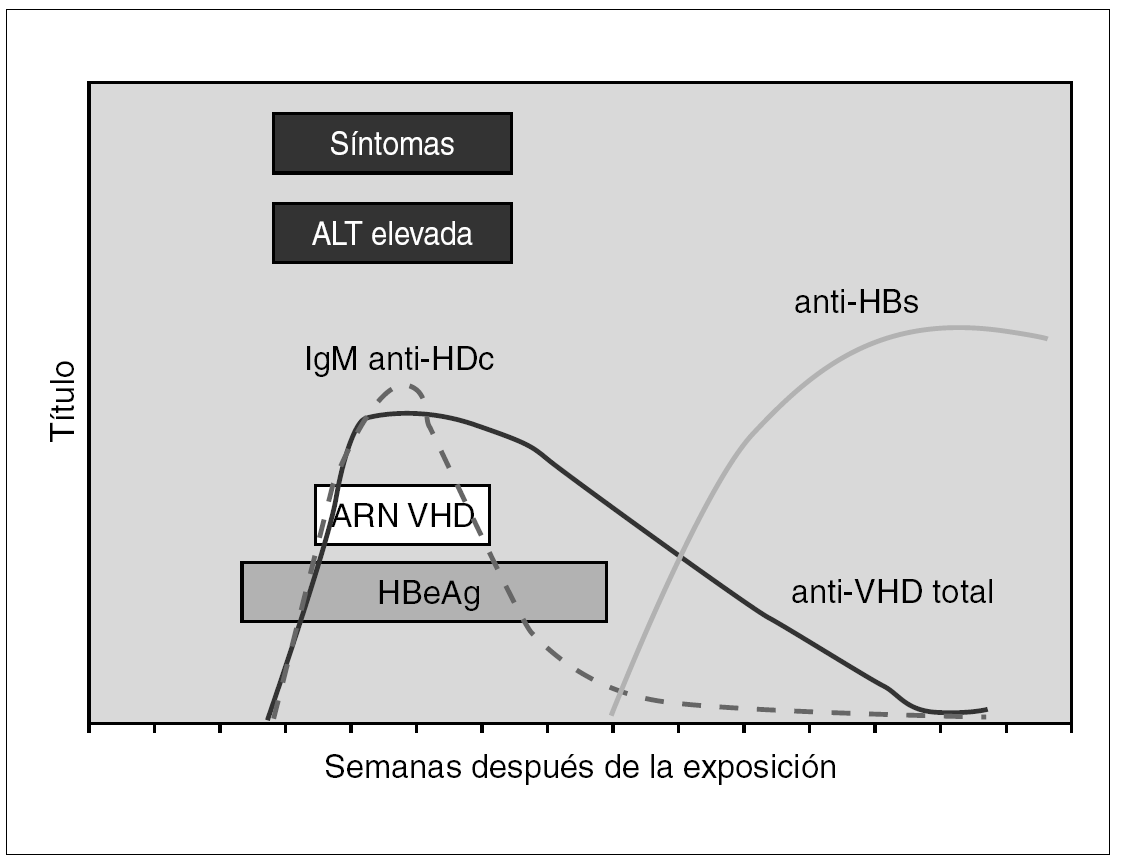
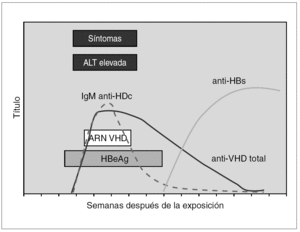
El diagnóstico de la infección por el VHD se realiza en el contexto de una infección por VHB como coinfección o como sobreinfección 51,52. En la coinfección aguda la viremia del VHD suele ser breve y por ello la detección de Ag VHD en suero tiene un rendimiento bajo. Resulta sin embargo de utilidad la demostración de anticuerpos de la clase IgM frente VHD, que suelen aparecer precozmente y de anticuerpos de la clase IgG, que son de aparición un poco más tardía, pero cuya demostración resulta de utilidad para diagnosticar o excluir la coinfección o sobreinfección por este agente en la inmensa mayoría de los casos 53 (fig. 5). La importancia del diagnóstico deriva de la posibilidad de que una infección crónica por VHB de evolución benigna o paucisintomática sufra un agravamiento inesperado o una evolución fulminante y fatal debido a una superinfección por VHD.
Figura 5. Curso serológico de la coinfección por VHD y VHB.
Diagnóstico del virus de la hepatitis C
El virus de la hepatitis C (VHC) tiene una distribución a escala mundial desigual calculándose que infecta al 3% de la población del planeta, con una amplia variabilidad en función de las zonas. En España se estima que su prevalencia en población general es del 1-2%. El 80% de los individuos infectados evolucionan a un modelo de cronicidad y entre éstos entre el 10-20% pueden desarrollar una cirrosis en las 2 décadas siguientes 54.
Es un virus ARN de polaridad positiva cuyo genoma contiene 9.600 nucleótidos, rodeado de un cápside proteico y una membrana de envoltura y un tamaño que oscila entre 55-65 nm. Desde el punto de vista taxonómico se clasifica dentro de la familia Flaviviridae en el género Hepacivirus 55. El genoma contiene un gran marco de lectura abierto de unos 3.000 aminoácidos, flanqueado por 2 regiones no codificantes, 5'y 3'UTR, altamente conservadas. De ellas la región 5' y la porción inicial del gen del core viral son las más conservadas entre diferentes cepas, contienen unos 340 aminoácidos y su principal segmento está constituido por 300 aminoácidos, y confirman el dominio de entrada al ribosoma en el inicio de la replicación. Ésta puede ser la razón por la cual el VHC inicia la traducción de sus proteínas mediante un mecanismo único y parecido al de los procariotas 56. La región 3' consta de varios segmentos, de ellos el más conservado es el final que se corresponde con el sitio de reconocimiento de la replicasa durante la iniciación de la síntesis de la cadena negativa del ARN.
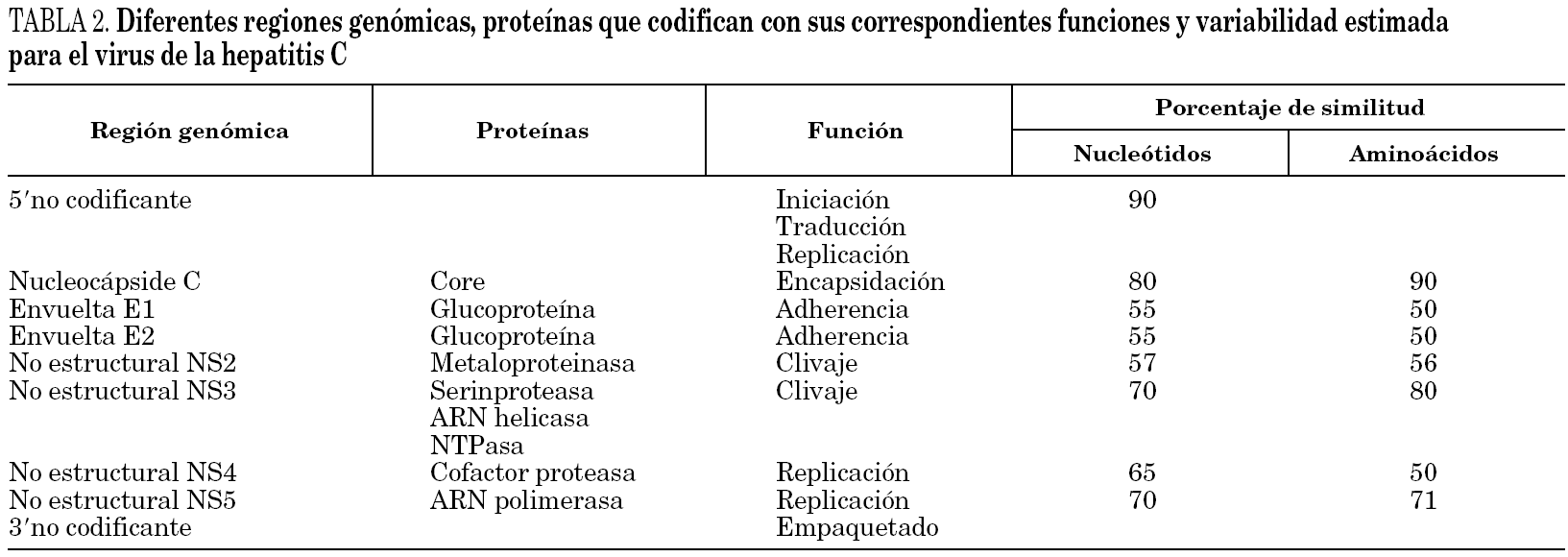
El genoma se traduce en un único marco de lectura que codifica una gran poliproteína de 3.000 aminoácidos que es hidrolizada por proteasas del huésped inducidas por UHC en proteínas estructurales (E) y no estructurales (NS). Dichas proteínas incluyen la del nucleocápside viral (p21), las 2 proteínas de envoltura E1 (gp31) y E2 (gp70) y una proteína transmembranaria (p7). Entre los genes E1 y E2 se sitúan unas regiones denominadas hipervariables (HVR1 y HVR2) que permiten al VHC establecer un sistema de escape al sistema inmunitario y condiciona la aparición de infecciones persistentes 57. La región no estructural, codifica 6 proteínas implicadas en la replicación de ARN viral que se asimilan a diferentes enzimas entre los que destacan proteasas, helicasa, nucleotidotrifosfatasa, ARN polimerasa dependiente de ARN (tabla 2).
El VHC presenta un elevado grado de diversidad genética, que afecta de una parte al genoma de cada cuasiespecie existente (variabilidad intragenoma) y de otra a las diferentes genotipos y subtipos (variabilidad intergenómica). En el proceso de replicación viral la cinética y la baja fidelidad de la ARN polimerasa condicionan una elevada variabilidad genética 58. La población de viriones que infecta a un paciente es una mezcla heterogénea de genomas emparentados entre sí, consideradas cuasiespecies 59. Éstas no representan sino una pequeña porción de la variabilidad del genoma, que alcanza mayores cotas de expresión en la denominada variabilidad intergenómica. En este ámbito se encuentran los denominados genotipos, que están representados por genomas cuyo grado de homología se sitúa entre el 66-69%. Su notación se efectúa mediante números arábigos y aunque su designación es un campo sometido a continuos cambios se acepta la existencia de al menos 6 genotipos distintos 60. Dentro de un genotipo cuando el grado de homología sube al 77-80% se establece un subtipo, cuya designación se realiza con una letra minúscula que sigue al número del genotipo, y de los que se han descrito más de un centenar. En un mismo subtipo pueden a su vez diferenciarse distintas cepas, cuyo grado de homología es del 91-95% 61. El grado de diversidad dentro de cada genotipo y subtipo es variable y está condicionado por la región del genoma a la que afecta. Las cepas que infectan a un individuo tienen también un grado de variabilidad, de tal forma que se establecen clones predominantes o variantes mayores frente a otros clones minoritarios. Este concepto es importante, ya que cuando la infección tiende a resolverse disminuye la variabilidad genética, mientras que en los que se establece una infección crónica aquélla aumenta.
La estrategia inicialmente empleada para el diagnóstico virológico de la infección por el VHC es la detección de anticuerpos frente a péptidos recombinantes. El progresivo diseño de sistemas de determinación de los mismos mediante EIA se ha efectuado en "generaciones" de acuerdo con el soporte antigénico empleado. Los EIA de última generación 62,63 incluyen antígenos de las diferentes regiones estructurales y no estructurales y sus características operacionales de sensibilidad y especificidad ofertan valores cercanos al 98% en sensibilidad y los anticuerpos son detectables al cabo de 6 semanas que siguen a la infección 64. De manera complementaria pueden emplearse sistemas de detección de antígeno del core, mediante EIA, que optimizan la detección de marcadores en el período ventana, si bien su sensibilidad se relaciona con el grado de viremia 65.
Por la trascendencia clínica de la infección por VHC, se emplean métodos de confirmación de anticuerpos, entre los que destacan por su amplia implantación los que tienen como fundamento el inmunoblot con antígenos recombinantes (RIBA) 66,67.
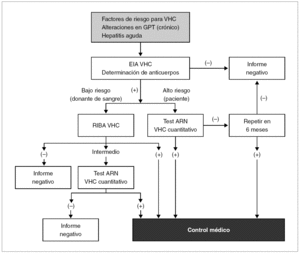
El diagnóstico de infección activa por VHC exige detectar ARN del mismo en suero o plasma. La determinación de viremia circulante se efectúa mediante detección cualitativa de ARN o a través de su cuantificación. Los métodos más empleados se fundamentan en la amplificación genómica mediante PCR o a través de amplificación isotérmica 68,69. Existen numerosos sistemas comercializados con buenas características operacionales que alcanzan sensibilidades de detección para menos de 100 copias ARN/ml.
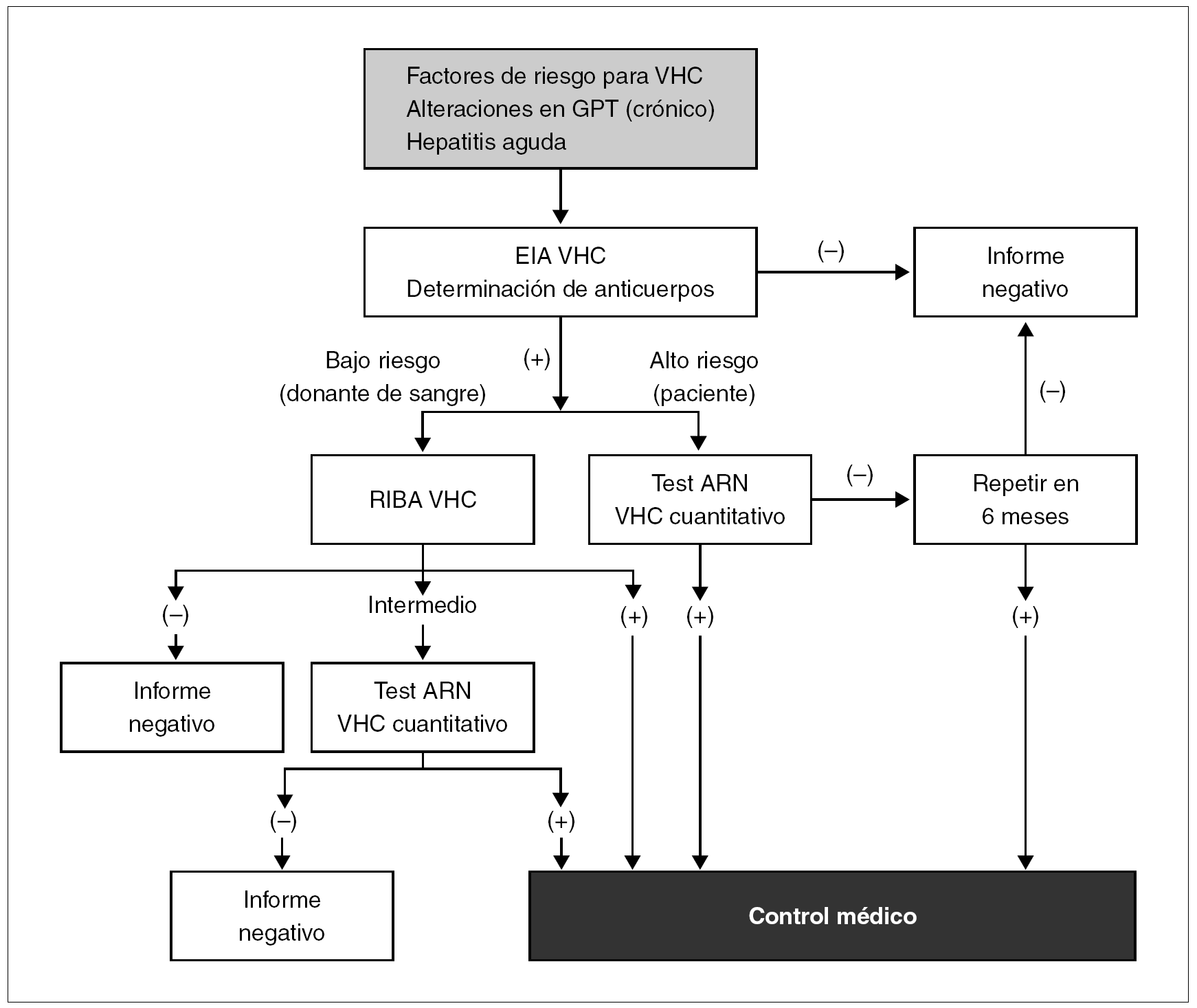
La cuantificación del ARN se puede realizar a través de una amplia gama de metodologías que oscilan desde la amplificación mediante PCR convencional, la amplificación de señal mediante ADN ramificado 70 o la PCR en tiempo real 71. Su aplicación se efectúa fundamentalmente en la monitorización de la respuesta a la terapia antivírica. En la figura 6 se representa un algoritmo para seguir los pasos de la determinación de marcadores en el cronograma diagnóstico en un paciente con infección por VHC.
Figura 6. Algoritmo para el diagnóstico de la infección por VHC. Fuente: Modificada de Shuhart C, Gretch DR. Hepatitis C and G viruses. En: Murray PR, Baron EJ, Jorgensen JH, Pfaller MA, Yolken RH, editors. Manual of Clinical Microbiology 8th ed., 2003.
Mediante análisis filogenéticos se reconoce la existencia de 6 genotipos del VHC, derivados de otras tantas ramas o agrupamientos en los árboles de comparación de secuencias obtenidas de las diferentes cepas identificadas en el mundo. En esta revista se ha revisado 72 la diversidad de los mismos y su distribución a nivel mundial, así como la evolución de la prevalencia de genotipos del VHC en España 73. Aunque el genotipo 1b sigue ocupando un lugar prevalente en la referida serie se ha podido documentar la existencia de recientes episodios de importación y diseminación de cepas de los genotipos 1a y 3, así como de la introducción en nuestro medio del genotipo 4. La metodología seguida para estos estudios suele consistir en la amplificación por PCR de un fragmento de la región 5' no codificante y posterior determinación del genotipo por hibridación reversa o secuenciación. La identificación de cuasiespecies es mucho más compleja y difícil de aplicar a la rutina. Sin embargo, el genotipado es clínicamente importante porque predice la respuesta al tratamiento, siendo muy favorable para los genotipos 2 y 3, que desafortunadamente son menos prevalentes; por ello el genotipado se dirige sobre todo cuando hay indicación de tratamiento.
El genotipado se puede realizar por secuenciación y análisis filogenético de los genes E1 o (NS)5B. Sin embargo, esta técnica es de difícil implementación en laboratorios hospitalarios y, por otra parte, no distinguiría infecciones mixtas por genotipos diferentes. Existen otros ensayos que combinan la amplificación genómica del VHC y su posterior digestión con enzimas de restricción que cortan el genoma por puntos específicamente reconocidos por las enzimas, dando lugar a patrones de fragmentos que sirven para la caracterización definitiva. Otras aproximaciones al genotipado usan la hibridación con sondas tipoespecíficas para amplificar las regiones 5'UTR y el genotipado mediante detección de anticuerpos específicos de genotipo. Las ventajas de estas últimas son su simplicidad, mejor facilidad en la conservación de las muestras, ya que los anticuerpos frente a genotipo son mucho más estables que el genoma vírico.
Conclusiones finales
En el momento actual se están produciendo avances en las técnicas de diagnóstico de hepatitis basadas en la biología molecular; entre las aplicaciones más importantes se deben identificar las mutaciones asociadas a resistencias en el tratamiento, así como implementar mejoras en el diagnóstico que afecten a la mayor automatización de un volumen tan importante de analítica. Es necesario también recordar de forma general, que dado el curso clínico evolutivo de estos síndromes clínicos y la patogenia de algunos de los virus responsables, los laboratorios encargados de este diagnóstico deben advertir al clínico en muchos casos, de la necesidad expresada al inicio de este artículo, de obtener sueros pareados a lo largo del tiempo 74 que por las razones de tiempo y temperatura requeridos; necesitarán de un almacenamiento y conservación que supera los estándares habituales.
Agradecimientos
Al doctor J. M. Echevarría por su continuo apoyo y ayuda.
NOTA
Los artículos publicados en la sección "Formación Médica Continuada" forman parte de grupos temáticos específicos (antibiograma, antimicrobianos, etc.). Una vez finalizada la publicación de cada tema, se irán presentando al Sistema Español de Acreditación de la Formación Médica Continuada (SEAFORMEC) para la obtención de créditos.
Una vez concedida la acreditación, esta se anunciará oportunamente en la Revista y se abrirá un período de inscripción gratuito para los socios de la SEIMC y suscriptores de la Revista, al cabo del cual se iniciará la evaluación, durante un mes, que se realizará a través de la web de Ediciones Doyma.
Correspondencia: Dr. R. Ortiz de Lejarazu.
Microbiología. Facultad de Medicina.
Avda. Ramón y Cajal, 7. 47005 Valladolid. España.
Correo electrónico: lejarazu@med.uva.es
Manuscrito recibido el 7-2-2006; aceptado el 7-2-2006.