La noción de conectividad cerebral es un aspecto clave para entender el funcionamiento cerebral. Las metodologías para detectar y cuantificar los diferentes tipos de conectividad con técnicas de neuroimagen son en la actualidad un área de estudio fundamental en la comprensión de la fisiopatología de muchos trastornos, tanto neurológicos como psiquiátricos.
Con este artículo se pretende realizar una revisión crítica de las técnicas con resonancia magnética para medir la conectividad cerebral dentro del actual contexto del proyecto Conectoma. Las técnicas revisadas se dividen en: a) conectividad estructural b) conectividad funcional (análisis de componentes principales, análisis de componentes independientes, vóxel semilla, meta-análisis) y c) conectividad efectiva (interacciones psicofisiológicas, modelo dinámico causal, modelos autorregresivos multivariantes y modelo estructural de ecuaciones).
Estos tres enfoques permiten combinar y utilizar distintas técnicas matemático-estadísticas cuyos resultados proporcionan modelos para intentar predecir la funcionalidad cerebral. Es necesario validar los hallazgos de estas técnicas con otras formas de análisis de la conectividad estructural y funcional. Esta información se integra dentro del proyecto Conectoma donde este conjunto de técnicas convergen para ofrecer una representación de todos los modelos de conectividad.
Brain connectivity is a key concept for understanding brain function. Current methods to detect and quantify different types of connectivity with neuroimaging techniques are fundamental for understanding the pathophysiology of many neurologic and psychiatric disorders.
This article aims to present a critical review of the magnetic resonance imaging techniques used to measure brain connectivity within the context of the Human Connectome Project. We review techniques used to measure: a) structural connectivity b) functional connectivity (main component analysis, independent component analysis, seed voxel, meta-analysis), and c) effective connectivity (psychophysiological interactions, causal dynamic models, multivariate autoregressive models, and structural equation models).
These three approaches make it possible to combine and use different statistical techniques to elaborate mathematical models in the attempt to understand the functioning of the brain. The findings obtained with these techniques must be validated by other techniques for analyzing structural and functional connectivity. This information is integrated in the Human Connectome Project where all these approaches converge to provide a representation of all the different models of connectivity.
Santiago Ramón y Cajal demostró que el sistema nervioso central (SNC) estaba formado por células, al igual que el resto de los tejidos vivos1. Esas células, las neuronas, y las conexiones entre ellas, las sinapsis, son fundamentales para su funcionamiento y comunicación. Mucho de lo que ocurre en el SNC puede describirse como un entramado de corrientes eléctricas y reacciones bioquímicas entre neuronas.
A partir del siglo XIX la observación y estudio de diferentes síndromes y enfermedades debidas a lesiones cerebrales jugaron un gran papel en el desarrollo de las neurociencias. Por primera vez fue posible establecer algunas correlaciones entre determinadas áreas del cerebro y ciertas funciones mentales superiores como el lenguaje o la memoria. Sin embargo, hace tiempo que se ha superado ese modelo. Hoy se asume que las funciones cognitivas no están localizadas en un área cerebral específica, sino que se basan en el funcionamiento de complejos sistemas funcionales2. Gracias a la resonancia magnética funcional (RMf) podemos relacionar una tarea concreta con un determinado patrón de activación cerebral 3, es decir, un conjunto de áreas coactivadas. Consolidar este conocimiento de los patrones de actividad cerebral es uno de los grandes retos de las neurociencias en la actualidad.
Muchas de las enfermedades del SNC, tanto neurológicas como psiquiátricas, no obedecen a una lesión focal o a la alteración de una sola área cerebral. Diferentes trastornos como la esquizofrenia o el autismo se conceptualizan en la actualidad como desordenes complejos de la conectividad neural4.
El estudio de la conectividad neural ‘in vivo’ es uno de los principales objetivos de las técnicas de neuroimagen. Se pretende con esta aproximación mejorar de forma directa los diagnósticos y tratamientos de diversas enfermedades neurológicas y mentales.
El propósito de este artículo es describir las diferentes técnicas basadas en la resonancia magnética (RM) que se han desarrollado recientemente para estudiar la conectividad cerebral. Analizaremos las ventajas, las limitaciones y las líneas futuras de las mismas. Esta información se integra dentro del proyecto Conectoma donde este conjunto de técnicas convergen para ofrecer una representación de todos los modelos de conectividad, modelos que pretende recopilar este artículo.
Conectividad cerebral: Conceptos básicosEn el cerebro concurren dos principios complementarios de organización funcional: la segregación y la integración 2.
El concepto de segregación funcional parte de la base de que determinadas funciones cognitivas se encuentran ubicadas en regiones cerebrales concretas. La caracterización de la actividad cerebral en términos de especialización funcional de las áreas del cerebro ha sido el enfoque principal de la RMf. Sin embargo, la actividad cerebral caracterizada en estos términos no revela nada sobre cómo las diferentes áreas cerebrales se comunican entre sí. Esta especialización funcional proporciona una visión limitada del sustrato neuronal de procesos que son mucho más complejos.
A partir de múltiples investigaciones se ha podido determinar que estas regiones no trabajan de forma independiente, sino que interactúan entre ellas creando circuitos dinámicos neuronales responsables de las funciones cognitivas. Consecuentemente, estos procesos cognitivos no pueden entenderse como específicos de una región, ya que el cerebro no actúa de forma modular aislada 5. Este concepto es lo que se conoce como la integración funcional.
La transferencia de información es la influencia del estado de un sistema sobre el estado de otro. Es importante el conocimiento de las conexiones directas entre las áreas cerebrales, incluyendo conocer cómo se transmite la información entre esas estructuras. Toda neurona está interconectada de forma dinámica y compleja con multitud de neuronas a través de una gran variedad de receptores y neurotransmisores sinápticos. El análisis de la conectividad neural se puede abordar con multitud de técnicas. Por una parte, se pueden realizar mapas histológicos y citoarquitectónicos. También se pueden utilizar técnicas neurofisiológicas en modelos animales a través de la implantación de microelectrodos que recojan cómo se trasmite la actividad bioeléctrica entre grupos específicos de neuronas cerebrales. A su vez resulta factible analizar las diferencias y las similitudes en los patrones cuantitativos y temporales de expresión génica, pues es desde los genes donde empieza a programarse la conectividad. Pero es el estudio in vivo e incruento, disponible ampliamente con la RMf, la técnica que más interés despierta en biomedicina. Los nuevos desarrollos tecnológicos y especialmente la mayor implantación de equipos de alta intensidad de campo (1,5 y 3 Teslas) con una mayor señal y resolución espacial de la imagen adquirida, junto con los nuevos modelos computacionales de ingeniería biomédica, hacen de esta técnica una de las mejores herramientas para estudiar la conectividad del SNC.
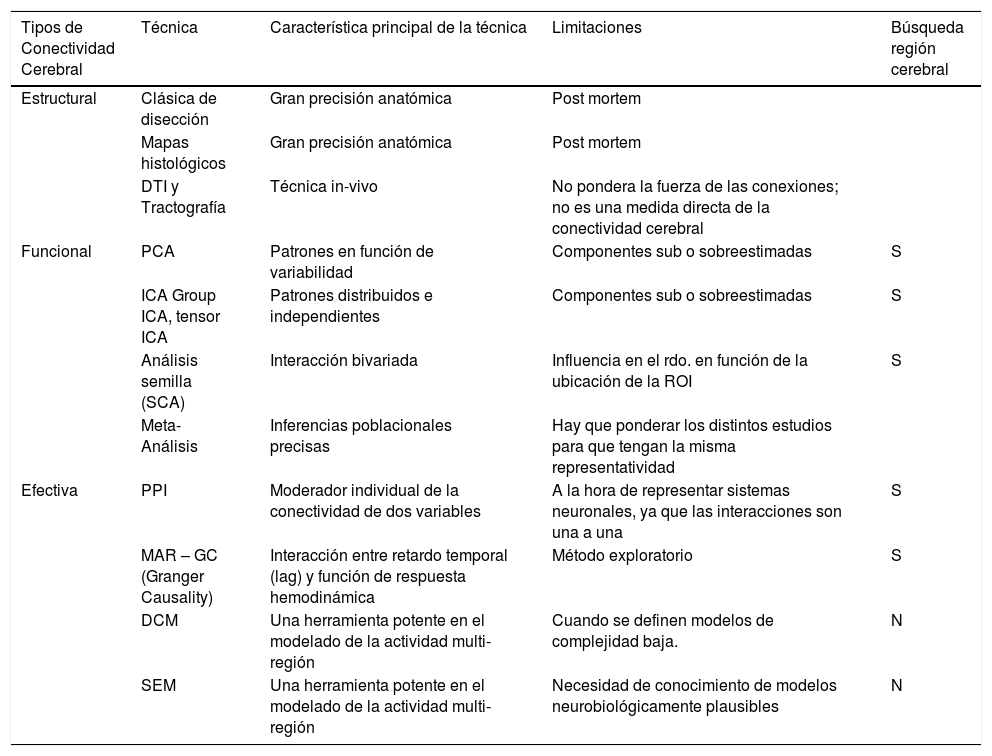
Esta revisión se centra en las diferentes formas de estudiar la conectividad a partir de la RM cerebral. Estas técnicas se resumen en la tabla 1.
Cuadro resumen que recopila las técnicas actuales que abordan la conectividad cerebral. Abreviaciones: DTI, Imagen de tensor de difusión. PCA, análisis de componentes principales. ICA, análisis de componentes independientes. ROI, región de interés. PPI, Interacciones psico-fisiológicas. SCA, análisis vóxel semilla. MAR, modelo autorregresivos multivariante. GC, causalidad de Granger. DCM, modelo dinámico causal. SEM, modelado estructural de ecuaciones.
| Tipos de Conectividad Cerebral | Técnica | Característica principal de la técnica | Limitaciones | Búsqueda región cerebral |
|---|---|---|---|---|
| Estructural | Clásica de disección | Gran precisión anatómica | Post mortem | |
| Mapas histológicos | Gran precisión anatómica | Post mortem | ||
| DTI y Tractografía | Técnica in-vivo | No pondera la fuerza de las conexiones; no es una medida directa de la conectividad cerebral | ||
| Funcional | PCA | Patrones en función de variabilidad | Componentes sub o sobreestimadas | S |
| ICA Group ICA, tensor ICA | Patrones distribuidos e independientes | Componentes sub o sobreestimadas | S | |
| Análisis semilla (SCA) | Interacción bivariada | Influencia en el rdo. en función de la ubicación de la ROI | S | |
| Meta-Análisis | Inferencias poblacionales precisas | Hay que ponderar los distintos estudios para que tengan la misma representatividad | ||
| Efectiva | PPI | Moderador individual de la conectividad de dos variables | A la hora de representar sistemas neuronales, ya que las interacciones son una a una | S |
| MAR – GC (Granger Causality) | Interacción entre retardo temporal (lag) y función de respuesta hemodinámica | Método exploratorio | S | |
| DCM | Una herramienta potente en el modelado de la actividad multi-región | Cuando se definen modelos de complejidad baja. | N | |
| SEM | Una herramienta potente en el modelado de la actividad multi-región | Necesidad de conocimiento de modelos neurobiológicamente plausibles | N |
La conectividad estructural o anatómica se define como el conjunto de conexiones físicas entre las unidades neuronales 6. El modelo físico de conexiones anatómicas es relativamente estable en escalas de tiempo cortas (segundos o minutos), pero a escalas mayores de tiempo (días), es probable que esté sujeto a cambios morfológicos significativos debido a la plasticidad de los patrones estructurales de conectividad. En la actualidad sólo los estudios de seguimiento invasivos son capaces de demostrar de manera fehaciente las conexiones axonales directas, ofreciendo un mayor nivel de detalle que las técnicas de exploración in vivo.
En el análisis de patrones estructurales de conexión neuronal, el tipo de análisis a realizar dependerá del nivel de la escala espacial que se utilice. En la actualidad el avance de las técnicas de RM de alta resolución en equipos de alta intensidad de campo hace posible examinar el córtex humano in vivo a resoluciones de 300 micras 7. Estas escalas se están aproximando a las resoluciones obtenidas mediante análisis histológico, lo cual permite contrastar la información obtenida con mapas citoarquitectónicos para lograr una mejor parcelación de las imágenes obtenidas mediante RM.
Imagen de RM potenciada en difusión. TractografíaLas técnicas de imagen de RM basadas en difusión son técnicas no invasivas que permiten estudiar la conectividad estructural del cerebro humano 8. Muchos grupos de investigación han demostrado que, analizando los principales circuitos de la sustancia blanca, es posible reconstruir de manera tridimensional los haces de fibras de forma significativamente similar a los obtenidos en la disección post-mortem. Sin embargo, mediante el trazado de tractos y la modelización mediante tensores (entidad algebraica que representa la dirección preferente de la difusión en cada vóxel), no es posible todavía reconstruir de manera totalmente detallada las principales rutas neuronales, especialmente cuando las fibras se ramifican o se cruzan 9. Los métodos desarrollados para resolver el problema del cruce de fibras mejoran parcialmente las limitaciones del modelado por medio de tensores, aunque todavía quede mucho trabajo por hacer. Un único haz de fibras que cambia su dirección dentro de un vóxel, introduce múltiples orientaciones posibles de fibras que pueden reconstruirse a través de diversos métodos. Por tanto, aunque estas técnicas son una mejora de la información ofrecida por el modelo de tensores, no son una solución completa. Parece necesario un mayor desarrollo de estos métodos para que puedan resolverse de manera unívoca las ambigüedades topológicas presentes en la sustancia blanca del cerebro humano. En términos generales, la tractografía no acaba de ofrecer la posibilidad de ponderar la fuerza de las conexiones entre redes de neuronas.
Sorprendentemente, a pesar de la gran cantidad de trabajo realizado para desarrollar y refinar estos algoritmos, hay pocos estudios que comparen las reconstrucciones de tractos obtenidas mediante los métodos de difusión no invasivos, con los obtenidos con la disección neuroanatómica clásica (una excepción son 10,11). Las comparaciones directas entre la disección virtual in vivo y las disecciones clásicas neuro-anatómicas, tal como ha realizado Lawes, son esenciales para saber la validez de técnicas de neuroimagen como la tractografía.
En resumen, el establecimiento de los caminos de sustancia blanca a partir de métodos de imagen basados en difusión ha recorrido un largo camino desde que se observó por primera vez la difusión anisotrópica 12. Sin embargo, la tractografía no ofrece una medida directa de la conectividad del cerebro humano, aunque puede ofrecernos información que está relacionada con la conectividad cerebral estructural.
Conectividad FuncionalConviene recordar primero que, desde el punto de vista de los datos que obtenemos en la RM, la conectividad funcional es fundamentalmente un concepto estadístico. En general, la conectividad funcional relaciona redes neuronales espacialmente remotas que muestran cierta interrelación. Dicha interrelación se determina a través de la dependencia estadística, que se puede calcular a partir de medidas de correlación o covarianza. La conectividad funcional se calcula a menudo entre todos los elementos de un sistema, independientemente de si estos elementos están conectados por vínculos estructurales directos. A diferencia de la conectividad estructural, la conectividad funcional es altamente dependiente en el dominio temporal. Cabe señalar que la conectividad funcional no hace ninguna referencia explícita a los efectos direccionales específicos (causa-efecto) o a un modelo estructural implícito 13–15.
El análisis de la conectividad funcional se realiza generalmente a través de métodos estadísticos dirigidos por los datos de neuroimagen (métodos no inferenciales), los cuales hacen pocas suposiciones acerca de la biología subyacente.
La conectividad funcional se apoya principalmente en las técnicas de RMf tradicional, mediante la cual es posible medir las variaciones en la imagen de la intensidad de la señal asociadas a los cambios hemodinámicos que acompañan a la activación celular, desencadenada por un estímulo determinado a través del contraste dependiente del nivel de oxigenación en sangre (BOLD, Blood Oxygen Level-Dependent Contrast). Por otra parte, cabe resaltar que además existe una actividad intrínseca neuronal que corresponde a fluctuaciones espontáneas de baja frecuencia. Generalmente se trata de un grupo de zonas vinculadas a la organización de la actividad interna intrínseca del cerebro. La mayoría de los estudios de estas fluctuaciones se llevan a cabo durante el estado de reposo. Se trata de la adquisición de dinámicos de RMf en los que no se aplica estímulo alguno. Las redes funcionales generadas bajo tales condiciones se denominan redes ‘resting-state’. Aproximadamente entre un 60-80% del consumo metabólico del cerebro se debe a la actividad intrínseca de estas redes. Es por ello que su estudio resulta de especial importancia para llegar a comprender los circuitos neuronales.
Los primeros análisis para estudiar la integración funcional utilizaban el Análisis de Componentes Principales (PCA) para descomponer los datos de RMf en un conjunto de componentes no correlacionadas mutuamente ni en el espacio ni en el tiempo. Más recientemente, el análisis de componentes independientes (ICA) se ha utilizado para identificar componentes que describen la actividad en una red ampliamente dispersa 16. Los métodos basados en PCA/ICA son especialmente útiles cuando no se conoce qué regiones están implicadas en una tarea determinada o no se conoce la conectividad estructural implícita.
A continuación se describen las principales técnicas que emplean este paradigma.
Análisis de Componentes PrincipalesEl método PCA descompone una matriz de imágenes en componentes 17. Una matriz es una tabla bidimensional de números ordenados en filas y columnas que se utilizan, entre otros usos, para describir sistemas de ecuaciones lineales. El PCA se puede aplicar a un sujeto, a través de una secuencia temporal de RMf, o bien a varios sujetos promediando las componentes, tales como los cambios en el valor medio de la señal BOLD. El objetivo de aplicar PCA es obtener la dirección de máxima variación. Se considera la primera componente, donde la variación espacial de la señal se representa mediante una imagen llamada imagen propia y la variación temporal a lo largo de un conjunto de sujetos se representa con un vector asociado, llamado vector propio. Las siguientes direcciones de varianza, en orden decreciente, se recogen en las subsiguientes componentes. El método PCA consiste en una transformación de los datos a un nuevo sistema de coordenadas definido por las direcciones de mayor varianza de los datos. Las imágenes propias pueden parecer similares a los mapas de activación RMf estándar, pero el análisis está dirigido por los datos y no dependen de ninguna hipótesis previa como sucede con el modelo lineal general. Por el contrario, las imágenes propias capturan características intrínsecas de los datos que se manifiestan en múltiples regiones que constituyen una amplia red funcional 14. El vector propio de cada componente muestra cómo la actividad de la red varía a través del tiempo, dependiendo de las condiciones experimentales, el sujeto o el grupo.
Análisis de Componentes IndependientesEl ICA 18,19 se puede definir mediante un modelo estadístico de variables latentes, es decir, que no pueden ser directamente observadas, por lo que se considerará que se observan n combinaciones lineales de n fuentes independientes. Cada variable observada, así como cada componente independiente, es una variable aleatoria. El modelo ICA se puede expresar de forma matricial:
donde la matriz x recopila los datos observados, en nuestro caso las imágenes obtenidas por RMf, la matriz A es la matriz de mezcla y la matriz s representa las fuentes originales.
El ICA es un modelo generativo 20,21, lo cual implica que describe cómo los datos observados son generados por una combinación de las componentes s. Cuando se aplica el modelo, sólo la matriz x es conocida y a partir de ella deberemos estimar A y s. El modelo considera que las componentes son estadísticamente independientes y tienen una distribución no-gaussiana.
El primer autor en utilizar ICA para el análisis de datos de RMf fue McKeown 22. Su aplicabilidad ha abierto las puertas a nuevas posibilidades en el diseño de estudios y de análisis de medidas en este tipo de imagen, aunque esta aplicabilidad presenta algunas restricciones y limitaciones 22. La metodología de la técnica ha sido analizada por diferentes autores y existen diferentes variaciones del método 23,24.
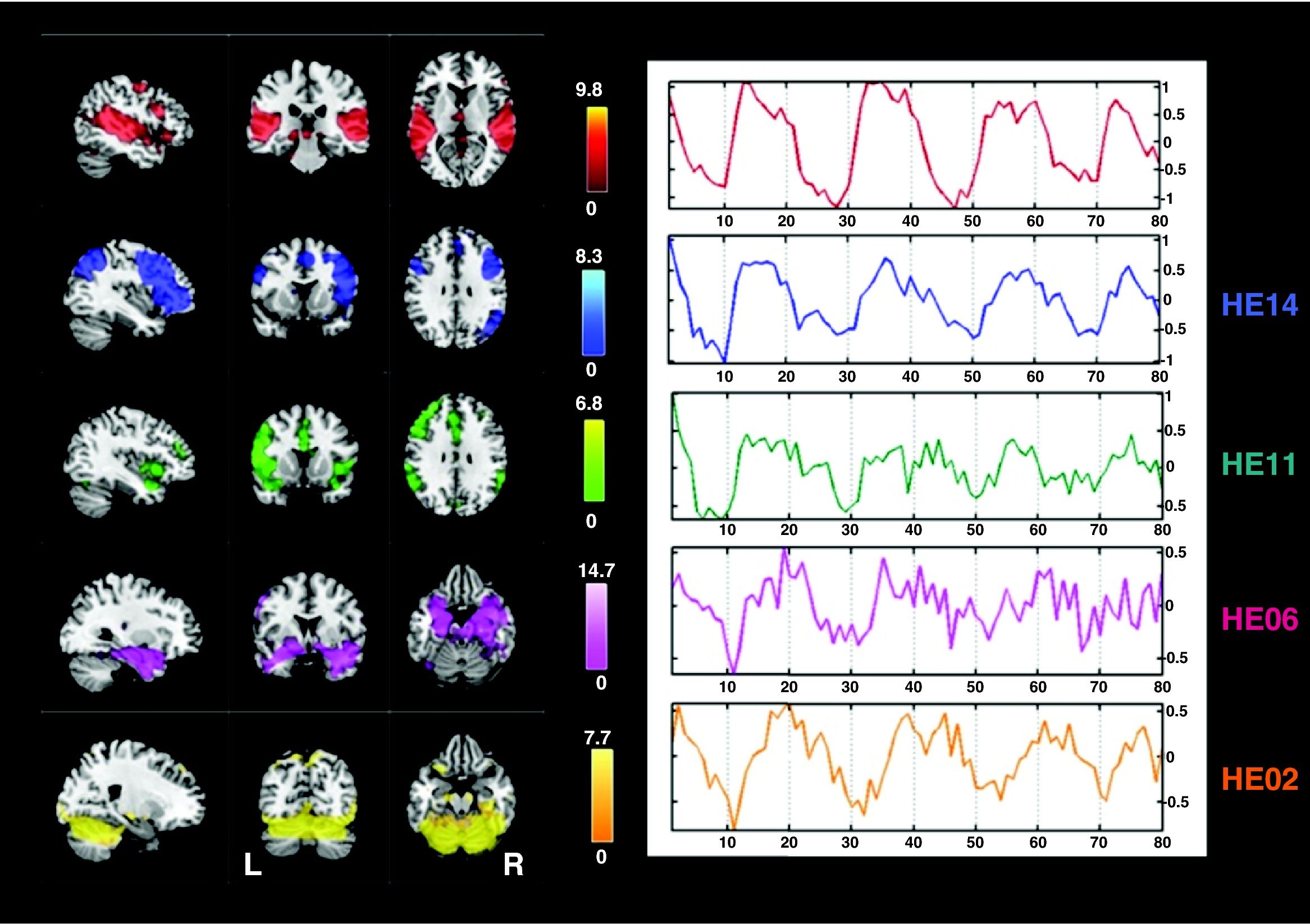
Esta técnica resulta especialmente apropiada a la hora de obtener las redes de conectividad intrínsecas anteriormente descritas (figura 1) y en particular la red de estado por defecto (DMN, Default Mode Network) que se asocia a un conjunto de regiones caracterizadas por estar anticorrelacionadas con la red asociada a una tarea determinada.
Componentes de Interés identificadas en un grupo de pacientes esquizofrénicos en un estudio de RMf con paradigma auditivo de contenido emocional. Estas componentes representan los mapas espaciales medios representativos del grupo. La conectividad funcional viene definida con un estadístico. A la derecha se representa la serie temporal promedio de cada una de las componentes obtenida con ICA.
El método de análisis de correlación basado en vóxel semilla (SCA, seed-based correlation analysis) parte de la idea de que existe una coherencia en las fluctuaciones espontáneas de la señal BOLD para bajas frecuencias 25. Este método requiere de la selección a priori de un vóxel, o grupo de vóxeles o región de interés (ROI), de donde se extraen las series temporales. Estos datos se utilizan posteriormente como un regresor en un análisis de correlación lineal a fin de calcular en todo el cerebro los mapas de la conectividad funcional entre vóxeles (comparándolos dos a dos) que covarían con la región semilla. La técnica SCA se ha utilizado por muchos grupos 26–28. La principal ventaja de SCA respecto a otros métodos es que este enfoque proporciona una respuesta directa a una pregunta directa: qué regiones de la red están más vinculadas funcionalmente al vóxel semilla. Esta posibilidad de interpretación directa, en comparación con otros métodos, hace del método SCA un enfoque muy atractivo para muchos investigadores.
Meta-AnálisisEste método pretende resumir los resultados de muchos estudios de neuroimagen para poder extraer conclusiones comunes entre los distintos resultados en un campo determinado de estudio. Una propuesta de esta metodología es la utilización de esta técnica para establecer un consenso sobre la ubicación de regiones funcionales y desarrollar nuevas hipótesis sobre la correspondencia entre estructura y función.
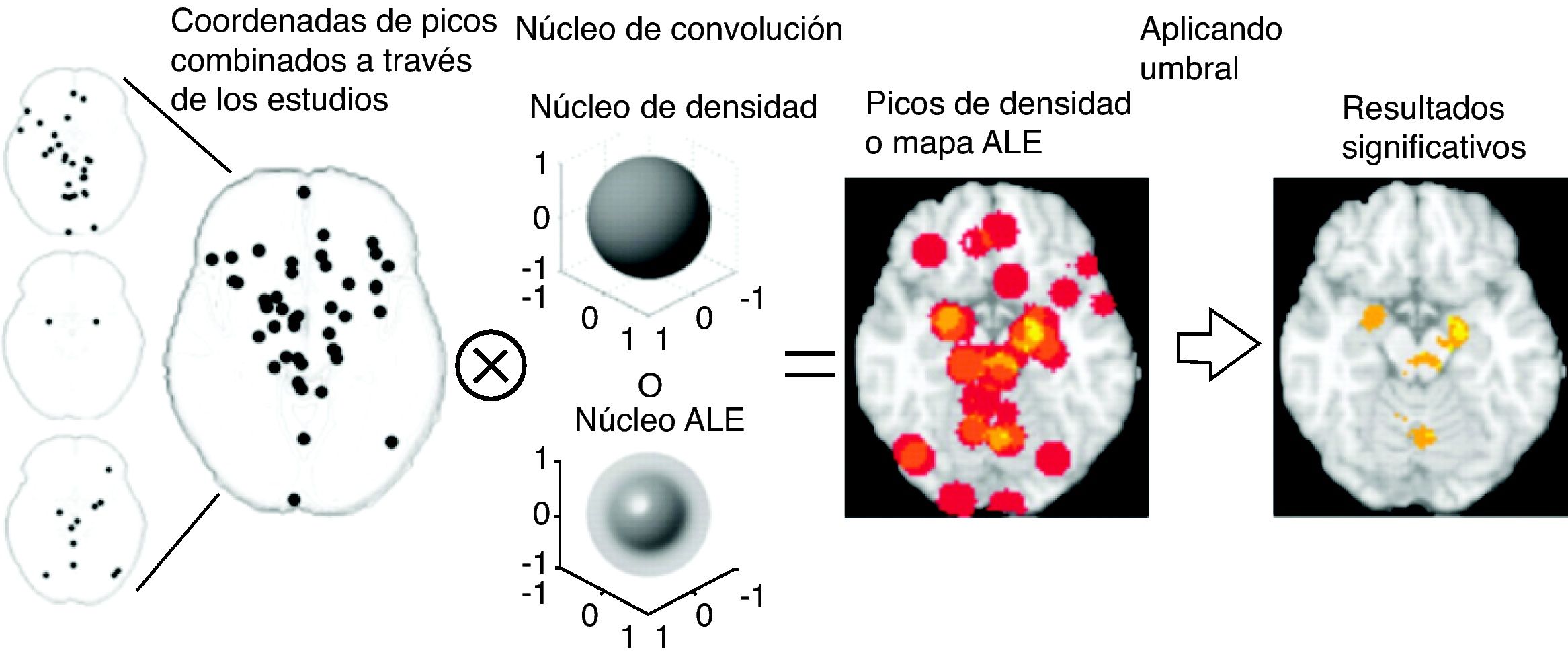
El objetivo de estos análisis es localizar regiones consistentemente activadas (si existen) en un conjunto de estudios relacionados con el mismo estado psicológico 29. Los métodos funcionan básicamente por conteo del número de picos de activación presentados en los estudios y comparando el número de picos observados con una distribución de hipótesis nula para establecer un criterio de significancia. Los dos métodos más representativos (figura 2) para realizar este tipo de análisis son: análisis de densidad de núcleos (KDA, kernel density analysis) y estimación de la verosimilitud de la activación (ALE, activation likelyhood estimate).
El metanálisis consiste en un análisis grupal que combina los picos (zonas de activación reportadas) obtenidos en varios estudios. Estos picos se convolucionan con un núcleo determinado (dependiendo de método) para obtener un mapa de picos de densidad en el que se busca resultados significativos. Modificado de Wager T D et al. Soc Cogn Affect Neurosci 2007;2:150-158. Resumen de las técnicas de Meta Análisis (ALE y KDA).
Cabe resaltar que una limitación de todas las técnicas funcionales es el gran número de variables de confusión que pueden provocar cambios en la actividad estudiada sin que tengan relación directa con la patología.
Conectividad EfectivaLa conectividad efectiva trata de la influencia directa de una región del cerebro en la actividad fisiológica que se registra en otras regiones del mismo 30. Este paradigma se ha utilizado, por ejemplo, para el estudio de atención frente a un estimulo visual 31. El análisis de la conectividad efectiva se realiza utilizando modelos estadísticos que hacen suposiciones fundamentadas en la anatomía del cerebro, lo cual restringe las características de las redes que se vayan a inferir a un subconjunto de regiones de interés que previamente se hayan seleccionado. Por lo tanto, se trata de métodos dirigidos por hipótesis y no por los datos, y se aplican cuando es posible especificar un conjunto completo de las áreas funcionales que previamente hemos considerado relevantes para la tarea especificada.
La conectividad efectiva se puede calcular basándose en el análisis de series temporales tanto en estudios con paradigma de estimulación como en redes de «resting state» 32. Cabe resaltar el hecho de que la extracción de la causalidad a partir de los datos de estas series temporales es sensible a decisiones de tipo técnico, como pueden ser la elección de la velocidad de muestreo o la amplitud de la ventana temporal 33. Una lista del software disponible para realizar este tipo de análisis puede encontrarse en www.nitrc.org 34. A continuación se describen las principales técnicas que emplean este paradigma.
Interacciones psico-fisiológicasLas interacciones psico-fisiológicas (PPI) son uno de los modelos más simples de los que se dispone para determinar las interacciones funcionales a partir de datos de RMf 35. Dada una serie temporal de referencia escogida (obtenida a partir de un vóxel o región de referencia), la técnica PPI calcula mapas de conectividad del cerebro completo del vóxel tomado de referencia con respecto al resto de vóxeles de acuerdo con una ecuación de regresión.
A pesar del hecho de que se trata de un modelo no dinámico, éste contiene los elementos fundamentales para describir un sistema. Sin embargo, dado que solo contempla interacciones una a una de manera independiente y no como un todo, este modelo está muy limitado a la hora de representar sistemas neuronales 35. Aunque la técnica PPI no es propiamente una herramienta para modelar un sistema, se ha mostrado especialmente útil a la hora de analizar las interacciones funcionales de una determinada región cerebral con el resto del cerebro para determinar la causalidad.
Modelado estructural de ecuacionesEl modelado estructural de ecuaciones (SEM) es una técnica estadística muy consolidada en las ciencias sociales, pero comenzó a utilizarse aplicada a neuroimagen a principios de los noventa por McIntosh y González-Lima 36. Se trata de una técnica multivariante dirigida por hipótesis que está basada en un modelo estructural en el que se representan las hipótesis de las relaciones causales entre las variables36–39. En el contexto de RMf estas variables son las series temporales BOLD y1...yn de n regiones cerebrales y las hipotéticas relaciones causales se basan en conexiones anatómicamente plausibles entre las regiones. La intensidad de cada conexión y1→yn se determina por un ‘coeficiente de camino’ que, por analogía con un coeficiente de regresión parcial, indica cómo la varianza de yj depende de la varianza de yi cuando todas las demás influencias sobre yj permanecen inalteradas.
En el caso especifico de RMf, los ‘coeficientes de camino’ del SEM describen la conectividad efectiva del sistema a lo largo de toda la sesión experimental. En cualquier caso, sería preferible conocer cómo cambia el acoplamiento entre determinadas regiones en función del contexto controlado experimentalmente, es decir, diferencias de acoplamiento entre dos tareas diferentes. Cabe resaltar que el SEM no tiene en cuenta el orden temporal, ya que si todas las series temporales regionales fueran permutadas del mismo modo, los parámetros estimados no cambiarían.
Una limitación del SEM es la restricción a la hora de utilizar modelos estructurales de complejidad relativamente baja, dado que los modelos con conexiones recíprocas y bucles con frecuencia no son identificables40.
Modelos Autorregresivos MultivariantesLos modelos autorregresivos tratan explícitamente los aspectos temporales de causalidad en las series temporales BOLD. Los modelos autorregresivos multivariantes (MAR) tienen en cuenta la dependencia causal del pasado en el presente: cada punto de una serie temporal de un área concreta se puede expresar como combinación lineal de los puntos precedentes al tiempo actual de una misma área. Estos MAR representan influencias directas entre un conjunto de regiones cuyas interacciones causales son inferidas por su mutua predecibilidad a partir de puntos de tiempo de instantes anteriores. Aunque los MAR constituyen una técnica estadística consolidada, las implementaciones específicas para RMf solo han sido propuestas recientemente.
Un enfoque complementario de los MAR basado en la idea de ‘Granger Causality’ 41, fue propuesto por Goebel y cols. 42. En este contexto, dadas dos series temporales y1 e y2, se considera que y1 es causada por y2 si se puede realizar una mejor predicción de su dinámica cuando se emplean valores pasados de y1 e y2 que cuando se emplean valores pasados únicamente de y1. Este método está limitado porque no tiene en cuenta las interacciones múltiples.
Modelo Dinámico CausalLa idea básica del modelo dinámico causal (DCM) es emplear una ecuación de estado bilineal para modelizar un sistema cognitivo a nivel neuronal, tarea que no podemos abordar utilizando RMf, y con este planteamiento la dinámica neuronal modelada se transforma en señales BOLD específicas de una región mediante un modelo hemodinámico predictivo. Esta idea es equivalente a especificar el modelo lineal general en un análisis estándar y convolucionarlo con la función de respuesta hemodinámica, pero de una forma más sofisticada. El objetivo principal del DCM es estimar los parámetros a nivel neuronal de tal manera que las señales BOLD medidas y modelizadas tengan una máxima similitud.
La arquitectura causal del sistema que sería deseable identificar se expresa a nivel de dinámica neuronal. Por lo tanto, para establecer inferencias sobre la conectividad de unidades neuronales, son necesarios modelos que combinen dos características: (i) Un modelo simple pero neurobiológicamente plausible de la dinámica de la población neuronal y (ii) un modelo predictivo biofísicamente plausible que describa la trasformación de la actividad neuronal en la señal medida. Los modelos que cumplan estas características harán posible ajustar conjuntamente los parámetros del modelo neuronal y del modelo predictivo, ya que las series temporales predichas son similares a las series temporales observadas. El DCM es el único enfoque en el que la unión entre los modelos de dinámica neuronal y los modelos predictivos biofísicos se convierte en un componente necesario. El DCM se ha implementado para datos RMf 43.
Integración de las técnicas: el proyecto ConectomaSi bien las distintas técnicas anteriormente revisadas suelen utilizarse de manera individual, resulta especialmente interesante integrar la información que ofrecen de manera conjunta para obtener resultados combinados que expliquen tanto función como comportamiento. Un proyecto fundamental en este esfuerzo de integración es el Conectoma 44,45.
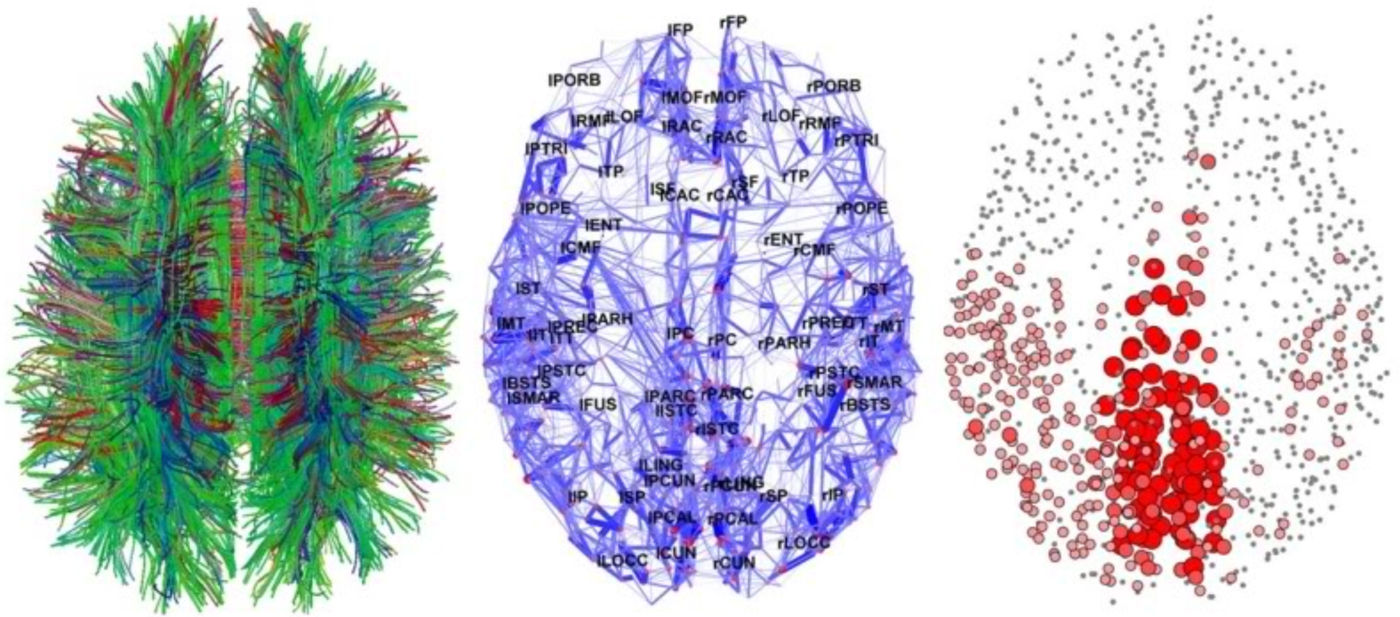
Una de las principales razones por las que estas técnicas de conectividad no se aplican en la práctica clínica en el diagnóstico es por la ausencia de un conocimiento sobre cual es el patrón de conectividad normal. En el año 2005 Olaf Sporns, profesor de neurociencias en la universidad de Indiana (EE.UU.), publicó un artículo45 haciendo notar que uno de los principales problemas de la investigación en neurociencias se debía a la falta de una descripción normativa anatómico-funcional del cerebro. Los mapas que tenemos en la actualidad sirven para detectar lesiones ocupantes de espacio, infartos cerebrales u otras lesiones groseras fácilmente identificables. Pero son insuficientes cuando el daño es más sutil y está menos localizado. Es decir, es necesario realizar un mapa cerebral mucho más detallado que los atlas cerebrales que disponemos en la actualidad. Inspirándose en el proyecto del genoma humano, Sporns propuso llamar a este mapa el «Conectoma». El proyecto Conectoma consiste en establecer un nuevo atlas sistemático y detallado de todas las conexiones cerebrales (figura 3). En la actualidad los Institutos Nacionales de la Salud de EEUU (NIH) han lanzado este proyecto con el propósito de tener en el año 2015 un mapa completo de un cerebro adulto sano. Este proyecto parte de la idea central de que las áreas cerebrales tienen diferentes características funcionales dependiendo de sus conexiones, aunando los conceptos de función y estructura. Es por ello que la estructura es fundamental para estudiar la función, dado que resulta lógico que las neuronas que se encuentren próximas en el espacio procesen el mismo tipo de información. A su vez la conectividad entre estas regiones más pequeñas nos permitirá conocer la dinámica bajo la que actúan procesos más diversificados en distintas áreas cerebrales. Del mismo modo que hace solo 10 años era impensable que se pudieran realizar estudios de todo el genoma en un corto tiempo y a un coste relativamente bajo, también es posible imaginar que en unos años los avances de la bio-informática y las neurociencias nos permitan tener esos patrones de conectividad del cerebro humano. De poder llevarse a cabo, el proyecto Conectoma permitirá sentar las bases para conocer qué microcircuitos funcionan mal en el cerebro en muchos trastornos neurológicos y psiquiátricos.
Ejemplo de imagen de tractografía (izquierda), de conectividad estructural (centro) y de conectividad funcional (derecha). Modificada de Hagmann P, Cammoun L, Gigandet X, Meuli R, Honey CJ, Wedeen VJ, Sporns O (2008) Mapping the structural core of human cerebral cortex. PLoS Biology 6, e159”.
Durante los últimos años se han realizado importantes avances en la comprensión de la estructura anatómica y funcional del sistema nervioso central, desarrollándose métodos experimentales y analíticos para la caracterización de la conectividad neural. Las técnicas de RM para el estudio estructural, funcional o efectivo de la conectividad precisan de una validación concurrente con técnicas anatómicas y neurofisiológicas. La distinción entre la conectividad funcional y efectiva es una herramienta conceptual importante para seleccionar los métodos adecuados en la caracterización de la integración funcional.
Uno de los grandes retos de las neurociencias es conocer la matriz de conexiones del cerebro humano, como se pretende en el proyecto «Conectoma». En el momento actual la red de conexiones que une los elementos neuronales del cerebro humano es todavía en gran parte desconocida y este proyecto todavía tiene un gran camino por recorrer. Su gran relevancia radicará en el establecimiento de un patrón detallado de las relaciones neurales, de tal forma que la influencia que las enfermedades tienen sobre esta red de comunicación pueda analizarse con exactitud y reproducibilidad.
Por último, todas las técnicas descritas tienen limitaciones que ya hemos comentado. Quizás las principales limitaciones son (i) que todavía deben considerarse de uso experimental por su falta de validación clínica y (ii) que analizan diferencias grupales aplicables a grandes poblaciones de sujetos, pero no a individuos aislados. Para que puedan demostrar su utilidad en el diagnóstico y en la práctica clínica se necesita que la predicción radiológica de la presencia de anomalías se realice individualmente. El objetivo es acercarse a la fiabilidad y especificidad que tiene la imagen médica en el diagnostico radiológico de un infarto cerebral o de una esclerosis múltiple. Los rápidos avances de los últimos años en este campo nos permiten ser prudentemente optimistas y pensar que podremos superar, en un futuro no muy lejano, este gran reto.
Autorías- 1.
Responsable de la integridad del estudio: MDIV
- 2.
Concepción del estudio: MDIV
- 3.
Diseño del estudio
- 4.
Obtención de los datos
- 5.
Análisis e interpretación de los datos
- 6.
Tratamiento estadístico
- 7.
Búsqueda bibliográfica: MDIV, JMM, MJEF
- 8.
Redacción del trabajo: MDIV, JMM, MJEF, JS
- 9.
Revisión crítica del manuscrito con aportaciones intelectualmente
- 10.
relevantes: MDIV, LMB, MRV, TM, EJA
- 11.
Aprobación de la versión final: MDIV, JMM, MJEF, LMB, MRV, TM, EJA, JS.
Dado que se trata de un trabajo de actualización, se han dejado en blanco los puntos 3,4,5 y 6.
FinanciaciónMinisterio de Sanidad: Instituto de Sanidad Carlos III, FIS02/0018, Red de genotipación y Psiquiatría Genética ISCIII G03/184, Red de Enfermedades Mentales y CIBER de Salud Mental (CIBERSAM).
Instituto de Salud Carlos III a través de la RETICS Combiomed RD07/0067/2001.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores desean agradecer a Cesar Tomé y Noelia Ventura su contribución a la hora de aportar información bibliográfica para realizar este artículo y a Gregorio Gómez y Rosa Valenzuela de la Agencia Valenciana de Salud por apoyar el proyecto Conectome funcional.