La obtención rápida y asequible de resultados es prioritaria. Para ello propusimos la realización de identificación y antibiograma sobre muestras de orina directa, mediante placa cromogénica CPS® y E-test (BioMérieux, Francia).
Se evaluó la concordancia con los procedimientos standard, estableciendo un protocolo paralelo a la rutina haciendo lecturas de identificación y antibiograma a las 6 y 24 horas sobre 54 muestras clínicas de orina.
La identificación concordó en un 88,88% a las 6 horas y en un 100% a las 24. El antibiograma solo detectó errores menores en amoxicilina-clavulánico y cefuroxima en la lectura a las 6 horas.
Concluimos que en el caso de infecciones urinarias causadas por bacilos Gram negativos no Pseudomonas spp. la lectura a las 6h del E-test sobre muestra directa es muy fiable como antibiograma presuntivo y para la detección de BLEE.
Getting fast and affordable laboratory results is a priority. A method for the direct identification and susceptibility testing on direct urine samples by chromogenic plate CPS ® and E-test (BioMérieux, France) is presented.
Tests were performed in parallel in order to compare the direct identification and susceptibility results at 6h and 24h on 54 samples of urine with the standard procedure.
Identification agreement was 88.88% (6h) and 100% (24h). Antimicrobial susceptibility testing detected only minor errors in amoxicillin-clavulanate and cefuroxime (6h).
It is concluded that six hours testing is a very reliable susceptibility and extended-spectrum beta-lactamase (ESBL) detection method for urinary tract infection caused by non-Pseudomonas spp Gram negative bacteria.
La realización rápida del antibiograma y la información eficaz de los resultados ha demostrado ser de extraordinario valor en el manejo clínico de los pacientes, disminuyendo las estancias medias de los mismos y el coste global del tratamiento1,2. Los métodos tradicionales no permiten obtener resultados de identificación y antibiograma hasta 48-72 horas después de que la muestra llegue al laboratorio de microbiología. En la actualidad, hay sistemas basados en tecnología de biología molecular, que permiten la identificación rápida de microorganismos directamente de muestras clínicas3. Sin embargo, esta tecnología es cara, compleja y limitada3, no accesible a todos los laboratorios de microbiología.
Se hace pues imprescindible la búsqueda de soluciones sencillas y versátiles, que permitan la realización del antibiograma en menos tiempo. En varios estudios se ha demostrado que adelantar en 24 horas la información al clínico, tiene un impacto significativo en la evolución clínica de los pacientes y en los costes asociados1,2,4–9.
El objetivo principal de este estudio consistió en comparar la lectura de la sensibilidad obtenida por E-test sobre muestra clínica urinaria a las 6 horas (ED6) y a las 24 horas de incubación (ED24), con la obtenida sobre las colonias aisladas del urocultivo siguiendo la metodología standard, tanto por E-test (ET) como por el sistema automatizado Vitek 2 (BioMérieux, Francia). Además se evaluó la concordancia entre los resultados de identificación obtenidos con la placa cromogénica CPS® (BioMérieux, Francia) con lectura a las 6 y 24 horas, sobre la muestra clínica y los obtenidos con el sistema Vitek 2 con tarjetas inoculadas a partir de colonias aisladas del urocultivo.
MétodosEl protocolo del presente estudio fue aprobado por la Comisión de Ética del Área de Gestión Sanitaria del Norte de Almería.
Los criterios de inclusión adoptados en el estudio fueron los siguientes: muestras de orina provenientes de pacientes ingresados, con más de 500 leucocitos/μl, un análisis de sedimento urinario con más de 40 leucocitos/campo y con presencia únicamente de bacilos Gram negativos en la tinción del sedimento.
Se excluyeron las muestras procedentes de pacientes sondados y aquellas en que se sospechó la presencia de un segundo microorganismo, levaduras o cocos Gram positivos mediante tinción de Gram.
Las orinas aceptadas se procesaron siguiendo los dos protocolos; por una parte, para la realización de la identificación y el antibiograma directos, se sembró para aislamiento una gota del sedimento de la orina en la placa cromogénica CPS® y por agotamiento otra gota en cada una de las placas de agar Mueller-Hinton (BioMérieux, Francia) donde se colocaron las tiras de ET descritas a continuación. Todas las placas se incubaron a 35-37°C en O2 y se leyeron por el facultativo a las 6 y a las 24 horas. De forma paralela estas mismas muestras se procesaron siguiendo el protocolo habitual del laboratorio que consiste en siembra para cuantificación de 0,001mL de orina sin centrifugar en agar CLED (BioMérieux, Francia) e incubación a 35-37°C en atmósfera aeróbica durante 18-24 horas. Tras la lectura de los urocultivos, a las colonias aisladas se les realizó identificación y antibiograma con el sistema automatizado Vitek 2 siguiendo las recomendaciones del fabricante, utilizando las tarjetas GN para la identificación, AST-113/244 y AST-114/245 para la sensibilidad. Sobre estas colonias aisladas se realizó además el antibiograma en agar Mueller-Hinton con las mismas tiras de ET siguiendo las recomendaciones de la SEIMC. Este procedimiento se consideró el gold standard. La identificación cromogénica se comparó con el resultado de las tarjetas GN.
Se usaron seis tiras de ET para el antibiograma: amoxicilina-clavulánico, AMC (0,016-256μg/ml), cefuroxima, FUR (0,016-256μg/ml), ceftazidima, TZ (0,016-256μg/ml), cefotaxima, TAX (0,016-256μg/ml), ciprofloxacino, CP (0,002-32μg/ml) y cefepime+clavulánico vs. cefepime para la detección de BLEE.
Se calcularon las concentraciones mínimas inhibitorias (CMI) para ED6, ED24 y ET, y los valores de CMI50 y CMI90. Los resultados se interpretaron como susceptible (S), intermedio (I) o resistente (R), siguiendo los criterios del CLSI (Clinical and Laboratory Standards Institute)10. Los valores de CMI sobre muestra directa a las 6 y 24 horas se compararon con los obtenidos a partir de colonias aisladas. Los criterios usados fueron los siguientes: error menor, cuando la CMI obtenida por el método de estudio clasificó al microorganismo como intermedio y la obtenida por el método de referencia como sensible o resistente, y viceversa; error mayor, cuando la CMI obtenida por el método de estudio clasificó al microorganismo como resistente y la obtenida por el método de referencia como sensible y error muy mayor, cuando la CMI obtenida por el método de estudio clasificó al microorganismo como sensible y la obtenida por el método de referencia como resistente; concordancia, cuando las CMI obtenidas con el método en estudio y el de referencia clasificaban al microorganismo en la misma categoría o cuando con respecto al valor de CMI fueron idénticas o variaban en±un log2.
ResultadosSe aceptaron un total de 54 orinas. Todas ellas con un único aislamiento en recuento significativo, como se vio en el urocultivo posterior. La concordancia del Gram del sedimento con la colonia resultante fue del 100%. Los aislamientos se distribuyeron como sigue: 39 Escherichia coli (E. coli) (72,2%), 8 Klebsiella spp./Enterobacter spp. (15%), 3 Proteus spp. (5,5%) y 4 Pseudomonas spp. (7,4%). La concordancia entre la identificación presuntiva con CPS sobre muestra directa a las 6 y 24 horas con la identificación mediante el sistema Vitek 2 fue de un 88,88% y un 100% respectivamente. Los aislamientos que no se pudieron identificar a las 6 horas fueron 6 (11%), las 4 Pseudomonas spp. y 2 de los 3 Proteus spp. Todos los aislamientos excepto un Proteus spp. y una Pseudomonas spp. mostraron un crecimiento suficiente para poder determinar de manera visual la CMI a las 6 horas.
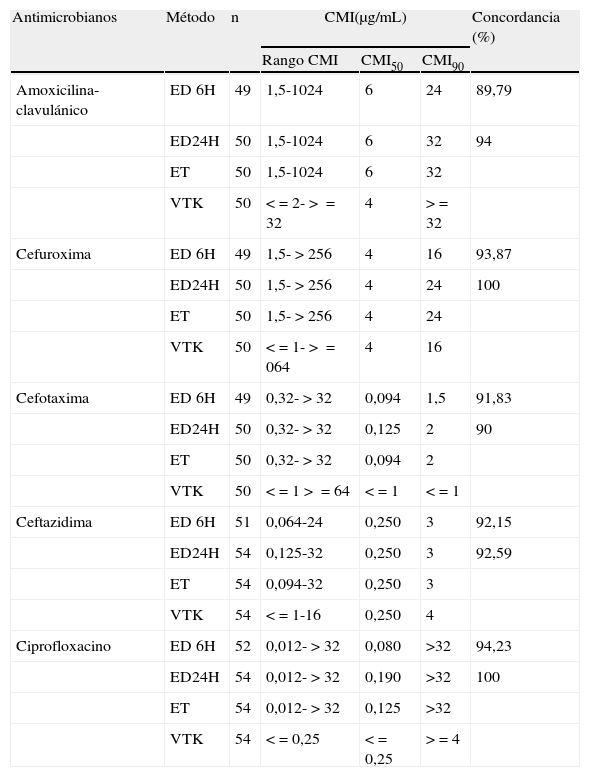
Los rangos de CMI, CMI50 y CMI90 determinados por ED6 y ED24, ET y Vitek 2 se resumen en la tabla 1, así como la concordancia (%) de cada uno de los métodos directos con el ET.
Susceptibilidad de los aislamientos procedentes de 54 muestras, resultados de CMI (rango, CMI50 y CMI 90) por los distintos métodos empleados. Porcentaje de concordancia de los métodos directos con el método standard de E-test, considerando concordantes valores menores o iguales a 1 log2 del resultado de E-test standard
| Antimicrobianos | Método | n | CMI(μg/mL) | Concordancia (%) | ||
| Rango CMI | CMI50 | CMI90 | ||||
| Amoxicilina-clavulánico | ED 6H | 49 | 1,5-1024 | 6 | 24 | 89,79 |
| ED24H | 50 | 1,5-1024 | 6 | 32 | 94 | |
| ET | 50 | 1,5-1024 | 6 | 32 | ||
| VTK | 50 | <=2->=32 | 4 | >=32 | ||
| Cefuroxima | ED 6H | 49 | 1,5->256 | 4 | 16 | 93,87 |
| ED24H | 50 | 1,5->256 | 4 | 24 | 100 | |
| ET | 50 | 1,5->256 | 4 | 24 | ||
| VTK | 50 | <=1->=064 | 4 | 16 | ||
| Cefotaxima | ED 6H | 49 | 0,32->32 | 0,094 | 1,5 | 91,83 |
| ED24H | 50 | 0,32->32 | 0,125 | 2 | 90 | |
| ET | 50 | 0,32->32 | 0,094 | 2 | ||
| VTK | 50 | <=1>=64 | <=1 | <=1 | ||
| Ceftazidima | ED 6H | 51 | 0,064-24 | 0,250 | 3 | 92,15 |
| ED24H | 54 | 0,125-32 | 0,250 | 3 | 92,59 | |
| ET | 54 | 0,094-32 | 0,250 | 3 | ||
| VTK | 54 | <=1-16 | 0,250 | 4 | ||
| Ciprofloxacino | ED 6H | 52 | 0,012->32 | 0,080 | >32 | 94,23 |
| ED24H | 54 | 0,012->32 | 0,190 | >32 | 100 | |
| ET | 54 | 0,012->32 | 0,125 | >32 | ||
| VTK | 54 | <=0,25 | <=0,25 | >=4 | ||
ED6H: lectura a las 6 horas del E-test sobre muestra directa; ED24H: lectura a las 24 horas del E-test sobre muestra directa; ET: E-test método standard; VTK: Vitek2.
Las CMI obtenidas mediante ET para los distintos antibióticos se tradujeron a las categorías de S, I o R. Se obtuvieron 9 I (18%) y 7 R (14%) para AMC, 4 I (8%) y 6 R (10%) para FUR, 5 R (10%), sin intermedios, para TAX y TZ, y 19 R (36%) también sin intermedios para CP.
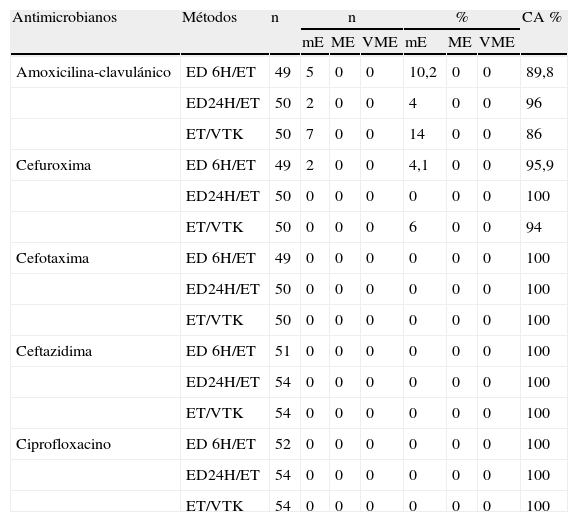
En la tabla 2 están descritas las diferencias de concordancia en forma de errores menores, mayores y muy mayores. No hubo diferencias en el caso de TAX, TZ, o CP. Con respecto a los ED6, para AMC los 5 errores menores consistieron en mostrar una mayor sensibilidad de intermedio a resistente o de sensible a intermedio. En la ED24 dos intermedios se leyeron como sensibles. Para FUR solo hubo 2 errores menores con ED6, también de sensibilidad aumentada de intermedia a sensible. Los errores encontrados entre Vitek 2 y el ET siempre fueron menores y son perfectamente aceptables por las diferencias metodológicas. El grado de concordancia para cada caso se encuentra reflejado en forma de porcentaje con respecto al ET para cada uno de los antibióticos.
Errores en número y porcentaje, concordancia en porcentaje entre los métodos directos de E-test (ED6 y ED24) y entre el standard de E-test. Errores en número y porcentaje, concordancia en porcentaje entre el método standard de E-test y Vitek 2
| Antimicrobianos | Métodos | n | n | % | CA % | ||||
| mE | ME | VME | mE | ME | VME | ||||
| Amoxicilina-clavulánico | ED 6H/ET | 49 | 5 | 0 | 0 | 10,2 | 0 | 0 | 89,8 |
| ED24H/ET | 50 | 2 | 0 | 0 | 4 | 0 | 0 | 96 | |
| ET/VTK | 50 | 7 | 0 | 0 | 14 | 0 | 0 | 86 | |
| Cefuroxima | ED 6H/ET | 49 | 2 | 0 | 0 | 4,1 | 0 | 0 | 95,9 |
| ED24H/ET | 50 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| ET/VTK | 50 | 0 | 0 | 0 | 6 | 0 | 0 | 94 | |
| Cefotaxima | ED 6H/ET | 49 | 0 | 0 | 0 | 0 | 0 | 0 | 100 |
| ED24H/ET | 50 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| ET/VTK | 50 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| Ceftazidima | ED 6H/ET | 51 | 0 | 0 | 0 | 0 | 0 | 0 | 100 |
| ED24H/ET | 54 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| ET/VTK | 54 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| Ciprofloxacino | ED 6H/ET | 52 | 0 | 0 | 0 | 0 | 0 | 0 | 100 |
| ED24H/ET | 54 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
| ET/VTK | 54 | 0 | 0 | 0 | 0 | 0 | 0 | 100 | |
CA: concordancia; ED6H: lectura a las 6 horas del E-test sobre muestra directa; ED24H: lectura a las 24 horas del E-test sobre muestra directa; ET: E-test método standard;. mE: error menor; ME: error mayor; VME: error muy mayor; VTK: Vitek2.
La producción de BLEE, se observó tanto por Vitek 2 como por ET en 4 de las cepas (7%). Las cuatro fueron detectados por ED6 y ED24 y no se registró ningún falso positivo. Los VPP y VPN fueron del 100%.
DiscusiónLa independencia del inóculo del resultado del ET abre la posibilidad de su uso con diluciones no estandarizadas7 como puede ser una muestra directa en este caso de orina, lo que supone un considerable ahorro de tiempo. Para ello debíamos asegurar un crecimiento suficiente a las 6 y 24 horas para identificar el microorganismo y hacer una lectura visual de la CMI. Observamos que esto era posible en el caso de bacilos Gram negativos, salvo Pseudomonas spp. y por lo tanto adecuado para enterobacterias. Para las bacterias Gram positivas el crecimiento fue insuficiente.
El grado de concordancia entre estos métodos con el estándar fue muy elevado, del 89,8 al 100% en el caso de variaciones de CMI y del mismo rango en cuanto a la concordancia de categorías de susceptibilidad, apreciándose solo errores menores. Las mayores variaciones se observaron en el caso de amoxicilina - clavulánico y en menor medida con cefuroxima lo que nos llevó a concluir que se debe ser cauteloso a la hora de informar sobre estos antibióticos aunque solo si la CMI se encuentra próxima al punto de corte; en cepas claramente sensibles o resistentes la lectura a las 6 horas no ofrece duda y correlaciona con la lectura final.
En el caso de enterobacterias productoras de BLEE, la correlación fue del 100%, lo que la hace una excelente herramienta para la detección precoz de este tipo de microorganismos.
Una limitación de este método es que quedan excluidos patógenos urinarios como Enterococcus spp. o Pseudomonas aeruginosa (P. aeruginosa), a pesar de lo cual, en pacientes con sepsis de origen urinario donde las enterobacterias son el principal agente causal es de gran utilidad, ya que en estos pacientes es fundamental iniciar el tratamiento de manera precoz con un antibiótico adecuado y por lo tanto, descartar o confirmar la presencia de cepas productoras de BLEE lo más rápido posible.
En conclusión, en el caso de infecciones urinarias monobacterianas por enterobacterias, los métodos de ET directo tienen muy buena correlación con el standard sobre colonia aislada del urocultivo y se pueden utilizar como un método de antibiograma preliminar, que ofrece una información muy ajustada más de 48 horas antes que el antibiograma definitivo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.