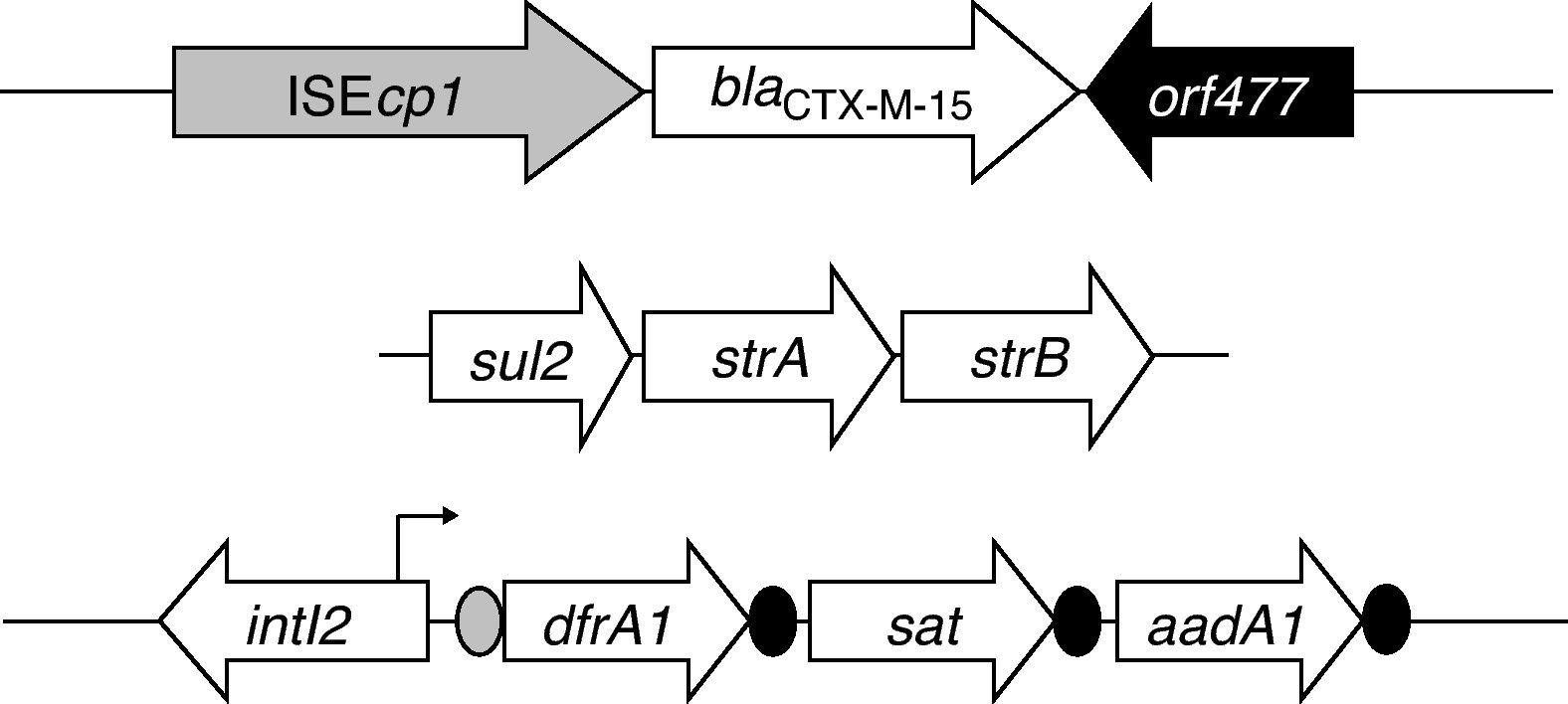
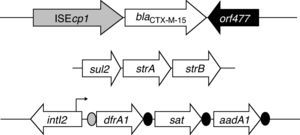
De las 107 Shigella spp. aisladas entre 2000-2010, en una cepa de Shigella sonnei se detectaron los genes codificantes de TEM-1 y CTX-M-15, identificándose la estructura ISEcp1+blaCTX-M-15+orf477. La cepa portaba un integrón de clase 2 con los genes cassettes dfrA1+sat+aadA1. Se detectó un plásmido del grupo IncI1 ST31 (CC-31) demostrando su transferencia por conjugación. Describimos la primera cepa de S. sonnei en nuestra comunidad productora de CTX-M-15 perteneciente a un paciente español que no había viajado al extranjero.
One hundred and seven Shigella spp. strains were isolated in our laboratory during the years 2000 to 2010. One Shigella sonnei harboured the genes that coded the β-lactamases TEM-1 and CTX-M-15, identifying the structure, ISEcp1+blaCTX-M-15+orf477, in their genetic environment. The strain also carried a class 2 integron with the gene cassettes dfrA1+sat+aadA1. A plasmid group IncI1 ST31 (CC-31) was detected and its mobilization by conjugation was demonstrated. We describe for the first time a S. sonnei strain producing a CTX-M-15 β-lactamase recovered from a Spanish patient who had not travelled abroad.
La shigelosis es una infección intestinal aguda de presentación esporádica y/o epidémica y de distribución mundial, si bien la prevalencia de las diferentes especies (S. dysenteriae, S. flexneri, S. boydii, S. sonnei) varía geográficamente, en relación con el grado de desarrollo socioeconómico. En España predomina S. sonnei1. La infección por aislados de esta especie resistentes a cefalosporinas de tercera generación y productora de β-lactamasas de espectro extendido (BLEE) es inusual en nuestro país. Presentamos la distribución de las infecciones producidas por Shigella spp. durante los últimos 10 años y las características de una cepa de S. sonnei portadora del gen que codifica para una BLEE identificada en nuestro medio.
MétodosSe procesaron 75.892 coprocultivos en el período 2000-2010. La identificación y el estudio de sensibilidad a 22 antibióticos se realizaron mediante el sistema WIDER (Soria-Melguizo, España). Se confirmó la presencia de BLEE en una cepa mediante test de sinergia y E-test (CT/CTL, CZ/CZL, PM/PML [bioMérieux, Marcy l’Etoile, Francia]). La interpretación de los resultados se realizó de acuerdo a las recomendaciones del CLSI2. Los genes implicados en la resistencia a β-lactámicos (blaTEM, blaSHV, blaOXA, blaCTX-M), así como sus entornos genéticos se estudiaron mediante PCR y secuenciación3. Mediante PCR4 se determinó la presencia de los genes de resistencia a tetraciclina (tet(A), tet(B), tet(C), tet(D) y tet(E)), a sulfamidas (sul1, sul2 y sul3) y a estreptomicina (aadA, strA y strB). La detección de integrones de clase 1 y 2 se analizó por PCR y posterior secuenciación4. El tipado de plásmidos se realizó mediante la asignación del grupo de incompatibilidad por el método PCR-based replicon typing (PBRT). Se designó la secuencia tipo del grupo de incompatibilidad I1 (IncI1) mediante multilocus sequence typing (pMLST) (http://pubmlst.org/plasmid/)5,6. Los experimentos de conjugación se realizaron utilizando como cepa receptora E. coli CSH26 y los transconjugantes (TCs) se seleccionaron en placas de cefotaxima (CTX) (5mg/L) y rifampicina (RIF) (100mg/L). Los TCs se confirmaron mediante REP-PCR usando los siguientes cebadores: REP-1 (5′-IIIGCGCCGICATCAGGC-3′) y REP-2 (5′-ACGTCTTATCAGGCCTAC-3′).
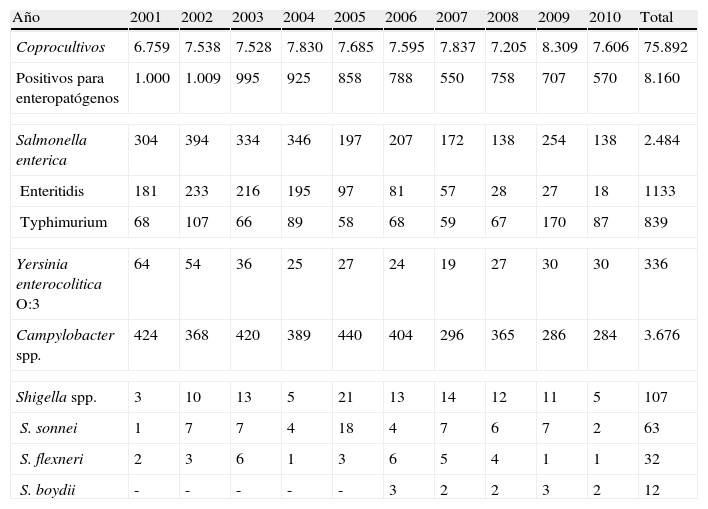
Resultados y discusiónEn el 10,8% de los coprocultivos procesados se aisló algún enteropatógeno bacteriano (tabla 1). Se identificaron 107 aislados de Shigella spp. (1,3% de los positivos). Las especies encontradas fueron: S. sonnei (63 aislados, 57,8%), S. flexneri (32, 29,3%) y S. boydii (12, 11%). Entre los porcentajes de resistencia detectados a distintos antibióticos destacan: cotrimoxazol (72,5%), ampicilina (35,8%), gentamicina (22,4%) y ciprofloxacino (2,7%). Todos los aislados fueron sensibles a fosfomicina. Un aislado de S. sonnei mostró resistencia a cefalosporinas de tercera generación (cefotaxima>8mg/L, ceftazidima>16mg/L) y de cuarta generación (cefepime>8mg/L) y, además, a tetraciclina, estreptomicina, sulfonamidas, cotrimoxazol y trimetoprim. En este aislado se detectaron los genes codificantes de TEM-1 y de una BLEE, CTX-M-15. Analizando el entorno genético del gen blaCTX-M-15, se identificó la estructura ISEcp1+blaCTX-M-15+orf477. No se detectó la secuencia de inserción IS26 truncando a ISEcp1. Además aguas arriba del gen blaCTX-M-15 se encontró la región W (48pb) y el promotor típico (-10 TACAAT) (-35 TTGAA) implicado en la transcripción de dicho gen bla (fig. 1). Este entorno del gen blaCTX-M-15 se ha descrito como el más frecuente7.
Coprocultivos procesados durante los años 2001-2010 y enteropatógenos aislados en dichas muestras
| Año | 2001 | 2002 | 2003 | 2004 | 2005 | 2006 | 2007 | 2008 | 2009 | 2010 | Total |
| Coprocultivos | 6.759 | 7.538 | 7.528 | 7.830 | 7.685 | 7.595 | 7.837 | 7.205 | 8.309 | 7.606 | 75.892 |
| Positivos para enteropatógenos | 1.000 | 1.009 | 995 | 925 | 858 | 788 | 550 | 758 | 707 | 570 | 8.160 |
| Salmonella enterica | 304 | 394 | 334 | 346 | 197 | 207 | 172 | 138 | 254 | 138 | 2.484 |
| Enteritidis | 181 | 233 | 216 | 195 | 97 | 81 | 57 | 28 | 27 | 18 | 1133 |
| Typhimurium | 68 | 107 | 66 | 89 | 58 | 68 | 59 | 67 | 170 | 87 | 839 |
| Yersinia enterocolitica O:3 | 64 | 54 | 36 | 25 | 27 | 24 | 19 | 27 | 30 | 30 | 336 |
| Campylobacter spp. | 424 | 368 | 420 | 389 | 440 | 404 | 296 | 365 | 286 | 284 | 3.676 |
| Shigella spp. | 3 | 10 | 13 | 5 | 21 | 13 | 14 | 12 | 11 | 5 | 107 |
| S. sonnei | 1 | 7 | 7 | 4 | 18 | 4 | 7 | 6 | 7 | 2 | 63 |
| S. flexneri | 2 | 3 | 6 | 1 | 3 | 6 | 5 | 4 | 1 | 1 | 32 |
| S. boydii | - | - | - | - | - | 3 | 2 | 2 | 3 | 2 | 12 |
También se detectó el gen tet(A) de resistencia a tetraciclina y los genes strA-strB que formaban parte del entorno genético del gen sul2 (fig. 1). El aislado portaba un integrón de clase 2 con la estructura genética característica del transposon Tn7 que contenía en su región variable los genes cassettes dfrA1+sat+aadA1 (fig. 1); esta estructura genética de integrón de clase 2 es muy frecuente en la especie S. sonnei y se cree que son organizaciones altamente conservadas debido a que pueden estar localizadas en superestructuras genéticas móviles8. La técnica de PBRT reveló la presencia de un plásmido del grupo de incompatibilidad IncI1 que pertenecía a la secuencia tipo ST31 (CC-31). El fragmento obtenido por PCR de los genes repI, sogS y ardA analizados para designar el pMLST del IncI1 fue el esperado, pero curiosamente los genes trbA y pndC implicados en el mantenimiento y la transferencia del plásmido, estaban truncados por el gen finQ, por lo que se obtuvo un amplicón de 2305 pb, como también sucede en la secuencia del plásmido pPWDA_03 de E. coli (número de GenBank HQ114284)6. Únicamente se aisló un tipo de TC que era resistente a ampicilina y productor de BLEE y sensible a tetraciclina, estreptomicina, sulfamidas, cotrimoxazol y trimetoprim. El estudio genotípico del TC reveló que portaba los genes de las β-lactamasas CTX-M-15 y TEM-1 así como el plásmido IncI1. El integrón de clase 2, el gen tet(A) y el entorno del gen sul2 no se detectaron en el TC.
Las β-lactamasas plasmídicas de espectro extendido de clase A de Ambler son todavía infrecuentes en el género Shigella, aunque se han difundido ampliamente en otros géneros de la familia Enterobacteriaceae. Hasta la fecha, se han descrito 5 tipos de β-lactamasas de tipo CTX-M en Shigella spp.: CTX-M-2, CTX-M-3, CTX-M-14, CTX-M-15 y CTX-M-649,10 y diversas β-lactamasas tipo TEM. En Europa (República Checa), se ha descrito recientemente un aislamiento clínico de S. sonnei en un paciente que había viajado a Asia y que portaba el gen blaCTX-M-15 localizada en un plásmido IncFII11. Por el contrario, en nuestro aislado de S. sonnei se detectó la presencia de un plásmido IncI1 que podría albergar el gen blaCTX-M-15 como se ha descrito previamente en cepas de Escherichia coli y Salmonella enterica aisladas en Francia, Reino Unido, Pakistán y Honduras12,13.
En pacientes con shigelosis, particularmente en los casos más graves, está indicado el tratamiento antibiótico porque puede ayudar a resolver la diarrea, mejorar la evolución clínica y disminuir el periodo de comunicabilidad. Los antimicrobianos de primera línea son ampicilina y cotrimoxazol, aunque no son infrecuentes las infecciones por aislados multirresistentes (ampicilina, cotrimoxazol y tetraciclinas y/o cloranfenicol), para los que pueden indicarse tratamientos alternativos, como fluoroquinolonas o cefalosporinas de tercera generación. En la actualidad se registran tasas elevadas de resistencia y/o sensibilidad disminuida a fluoroquinolonas en pacientes que han viajado a la India14, lo que ha llevado a aconsejar el uso de ceftriaxona para el tratamiento empírico de la diarrea del viajero de esta procedencia14. Aunque la resistencia a cefalosporinas de tercera generación en Shigella es anecdótica en nuestro país, describimos la primera cepa de S. sonnei encontrada en nuestra comunidad portadora del gen blaCTX-M-15 y aislada de un paciente español que no había viajado fuera de España. Este hallazgo, en un paciente autóctono, del que hay descrito un precedente análogo en Francia10, confirma la importancia de continuar vigilando la transmisión de CTX-M-15, enzima ampliamente distribuida en otras enterobacterias, particularmente en E. coli.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.