Se evaluaron 3 metodologías de extracción de proteínas para la identificación de hongos miceliales por MALDI-TOF MS en 44 aislados: la extracción con agua-ácido fórmico (E. agua), la extracción con zirconio-etanol-acetonitrilo-ácido fórmico (E. zirconio) y la recomendada por el proveedor del equipo (E. tubo). Se compararon 2 bases de datos: Bruker (BK) y BK+National Institutes of Health. Los resultados obtenidos utilizando dichas bases fueron los siguientes (en el orden citado): identificación correcta (IC) a nivel de género, 10 y 16 con E. agua; 27 y 32 con E. zirconio y 18 y 23 con E. tubo; IC a nivel de especie, 5 y 7 con E. agua; 17 y 20 con E. zirconio y 11 y 14 con E. tubo; identificaciones no confiables, 18 y 12 con E. agua y 9 y 4, tanto con E. zirconio como con E. tubo; ausencia de pico, 16 con E. agua, 8 con E. zirconio y 17 con E. tubo. La extracción con zirconio mostró el mejor rendimiento (p<0,05).
In order to optimize the identification of molds with MALDI-TOF MS, three protein extraction-methodologies were evaluated against 44 isolates: water extraction (WE), zirconium extraction (ZE) and the provider's recommended method (PRM). Two data bases were compared, Bruker (BK) and Bruker+National Institutes of Health. Considering both databases, results were respectively as follows: correct identification (CI) at gender level, 10 and 16 by WE; 27 and 32 by ZE and 18 and 23 by PRM; CI at species level, 5 and 7 by WE; 17 and 20 by ZE and 11 and 14 by PRM; non-reliable identification, 18 and 12 by WE; 9 and 4 by ZE and by PRM. No peaks were observed in 16 by WE, 8 by ZE and 17 by PRM. ZE showed the best perfomance (p<0.05).
Las infecciones fúngicas se han incrementado en los últimos años, especialmente las infecciones invasoras (IFI), que son una causa importante de morbimortalidad. Este aumento se debe a la mayor utilización de procedimientos diagnósticos invasivos y al creciente número de pacientes inmunodeprimidos13.
Los métodos de diagnóstico micológico tradicionales se basan en la identificación fenotípica o morfológica, que puede demorar de 24 a 48h para las levaduras y hasta varias semanas en el caso de los hongos miceliales. A su vez, estos métodos son dependientes de la especie y de las condiciones de cultivo, son laboriosos y requieren de personal especializado7,11,14.
Con el objetivo de mejorar el diagnóstico micológico se han desarrollado diferentes técnicas moleculares, las más utilizadas para la identificación de aislamientos se basan en el reconocimiento de especies filogenéticas por concordancia genealógica (Genealogical Concordance Phylogenetic Species Recognition [GCPSR])15. Estas técnicas requieren de una identificación presuntiva inicial, debido a la necesidad de secuenciar diferentes regiones de ADN blanco en función del agente fúngico sospechado (p. ej., región ITS, betatubulina, D1/D2, factor de elongación, calmodulina)4,8. Estas metodologías, propuestas como gold standard, tienen la ventaja de identificar una amplia variedad de especies fúngicas; sin embargo, son más laboriosas y no pueden aplicarse a la práctica clínica diaria7,11.
La espectrometría de masas conocida como matrix-assisted laser desorption/ionization time-of-flight mass spectrometry (MALDI-TOF MS) se fundamenta en la obtención de un perfil proteico de un aislamiento, que es comparado con una base de datos; en estos casos, la identificación va a depender tanto de la calidad del espectro obtenido como del tamaño de la base de datos. Por sus características, la MALDI-TOF MS es una herramienta prometedora que podría mejorar la calidad de los resultados y disminuir los tiempos de respuesta7,11. Esto contribuiría a instaurar precozmente el tratamiento apropiado, dado que la sensibilidad a los antifúngicos está íntimamente relacionada con el género y la especie.
La extracción de proteínas es un paso crítico para lograr espectros de buena calidad y, por consiguiente, una identificación correcta. Los hongos miceliales poseen una pared celular más robusta y rígida que las bacterias y levaduras, por lo que la preparación de la muestra requiere un proceso de extracción más agresivo. En la bibliografía se describen diferentes procedimientos de extracción (mecánicos y químicos) para mejorar la identificación de los hongos miceliales; sin embargo, los resultados no son concluyentes3,5,7,10,11,14. Otra condición necesaria para optimizar la identificación de hongos miceliales por MALDI-TOF MS es mejorar la cantidad y la variedad de los espectros en la base de datos provista por el proveedor7,10.
Con el objetivo de evaluar el procedimiento más conveniente para la extracción de proteínas en el laboratorio de micología clínica, se seleccionaron 3 metodologías basadas en las publicaciones de Lau et al.10, Cassagne et al.3 y Gautier et al.7 y el procedimiento recomendado por el proveedor14, a saber: 1) extracción con agua-ácido fórmico; 2) extracción con zirconio-etanol-acetonitrilo-ácido fórmico (E. zirconio)10, y 3) extracción en tubo con caldo Sabouraud-etanol-acetonitrilo-ácido fórmico (la recomendada por el proveedor Bruker) para dermatofitos y otros hongos miceliales (Guide Fungi Library MALDI Biotyper. Bruker Daltonics, EE. UU., 2012). Se analizó, además, la eficiencia de identificación utilizando dos bases de datos, Bruker (BK) y BK con la adición de la base del National Institutes of Health (BK-NIH).
Se incluyeron 29 aislamientos de hongos filamentosos provenientes de un hospital de la Ciudad Autónoma de Buenos Aires y 15 cepas provenientes de programas de control de calidad externos (CCE). Los hongos fueron identificados por sus características fenotípicas macro y micromorfológicas, siguiendo las claves del Atlas of Clinical Fungi de de Hoog et al.6. Se incluyeron 27 dermatofitos (11 Trichophyton rubrum, 3 Trichophyton tonsurans, 3 Trichophyton mentagrophytes, 5 Microsporum canis, 4 Microsporum gypseum y 1 Epidermothyton floccosum); 10 hongos hialinos y tabicados no dermatofitos (1 Fusarium solani, 4 Aspergillus fumigatus, 1 Aspergillus niger, 1 Pseudallescheria boydii, 1 Paecilomyces spp., 1 Scedosporium spp. y 1 Acremonium killiense); 6 mucorales (1 Mucor circinelloides, 1 Mucor spp., 1 Rhizopus oryzae, 1 Rhizopus spp., 1 Lichtheimia spp. y 1 Syncephalastrum spp.) y 1 hongo dematiáceo (Alternaria spp.).
Los microorganismos fueron cultivados en agar Sabouraud glucosado (ASG) a 28°C durante 5-7 días. Se consideró un desarrollo óptimo para la extracción de proteínas cuando la colonia en la placa de ASG alcanzó un diámetro aproximado de 5mm.
Se evaluaron 3 métodos de extracción de proteínas:
- –
Extracción en agua-ácido fórmico (E. agua): se realizó una suspensión acuosa del hongo (1-4 Mc Farland) con agua calidad espectrometría (Sigma-Aldrich, Francia), se agitó durante 15min, posteriormente se colocaron 2μl de la suspensión en un pocillo con 1μl de ácido fórmico (AF) 70% (Sigma-Aldrich, Francia) y 1μl de matriz alfa-ciano-4 hidroxi-ácido cinámico (HCCA) (Bruker Daltonics, Alemania).
- –
E. zirconio: se disgregó el hongo en 250μl de etanol absoluto (Sigma-Aldrich, Lyon, Francia) con 50μl de perlas de zirconio de 0,1mm de diámetro (Cole-Palmer Instrument Co., Vernon Hills, EE. UU.), se agitó durante 15min, se centrifugó a 13.000 rpm (2min) y se descartó el sobrenadante. Se adicionó al pellet 50μl de AF al 70%, se agitó durante 5min, luego se añadieron 50μl de acetonitrilo al 100% (Sigma-Aldrich, Lyon, Francia). Se agitó nuevamente durante 5min y se centrifugó a 13.000 rpm (2min); finalmente, se colocó en el pocillo 1μl del sobrenadante y 2μl de matriz HCCA10.
- –
Extracción en tubo con caldo Sabouraud-etanol-acetonitrilo-ácido fórmico, recomendada por el proveedor Bruker (E. tubo): se inoculó una porción del cultivo en caldo Sabouraud glucosado (Biokar Diagnostics, Francia) y se incubó durante 48h en agitación con rotor. Se dejó reposar el caldo durante 10min y se centrifugó un volumen de 1,5ml del sedimento a 13.000 rpm (2min). Se realizó un lavado con agua agitando durante 5min, se centrifugó y el pellet se resuspendió en 300μl de agua. La suspensión se agitó durante 5min y se agregaron 900μl de etanol absoluto, luego se agitó otros 5min y se centrifugó a 13.000 rpm (2min); se descartó todo el etanol y se dejó secar el pellet hasta evaporación total. El pellet se resuspendió en 50μl de AF al 70% y se agitó durante 10min; posteriormente, se adicionó 50μl de acetonitrilo al 100% y se agitó 10min más, luego se centrifugó a 13.000 rpm (2min). Finalmente, se colocó en el pocillo 1μl del sobrenadante y 1μl de matriz HCCA (Guide Fungi Library MALDI Biotyper. Bruker Daltonics, 2012, EE. UU.).
Todos los aislamientos fueron procesados para extracción de proteínas por los 3 métodos propuestos.
Análisis por MALDI-TOF MS. Se realizó por duplicado en las placas recomendadas por el proveedor (MSP 96, Bruker Daltonics, Alemania). Se calibró siguiendo las instrucciones del fabricante con 1μl de estándar bacteriano (Bruker Daltonics, Alemania) y 1μl de matriz HCCA, por duplicado.
Lectura e interpretación de los resultados obtenidos por MALDI-TOF MS. Los resultados se leyeron y analizaron por el sistema Bruker Microflex LT versión 3.1. (Bruker Daltonics, Alemania) utilizando la base de datos del proveedor Bruker (BK) (MBT_DB_5627_ MSP list, Filamentous Fungi Library 1.0, Bruker Daltonics). Se realizó un segundo análisis incorporando la base de datos del NIH10 (BK-NIH).
La interpretación de los resultados se realizó sobre la base de los siguientes criterios: identificación aceptable a nivel de especie y género, cuando el score fue ≥ 1,6. Los scores menores de 1,6 y mayores o iguales a 1,5 se consideraron como identificación aceptable solo a nivel de género, y scores<1,5 se tomaron como identificación no confiable. Para establecer un género o especie diferente, se consideró necesaria una diferencia mínima de 10% entre el score superior y el más próximo.
Para el análisis de los resultados se tuvieron en cuenta las siguientes definiciones:
- –
Identificación correcta solo a nivel de género (IC género): la identificación por MALDI-TOF MS coincidió con la identificación morfológica a nivel de género.
- –
Identificación correcta a nivel de especie (IC especie): la identificación por MALDI-TOF MS coincidió con la identificación morfológica a nivel de género y especie.
- –
Identificación incorrecta (No ID), identificación no confiable (score menor que 1,5).
- –
Sin pico: en la identificación por MALDI-TOF MS no se obtuvieron picos (no peaks found).
Métodos estadísticos: se aplicó el análisis de la varianza no paramétrico de Friedman mediante el programa estadístico InfoStat versión 2014 (Grupo InfoStat, FCA, Universidad Nacional de Córdoba, Argentina, http://www.infostat.com.ar) y la prueba de concordancia kappa, utilizando el programa estadístico EpiDat 4.1 versión 2014 (Consellería de Sanidade, Xunta de Galicia, España; Organización Panamericana de la Salud; Universidad CES, Colombia; http://dxsp.sergas.es) para comparar el desempeño de los diferentes métodos de extracción con las 2 bases de datos utilizadas.
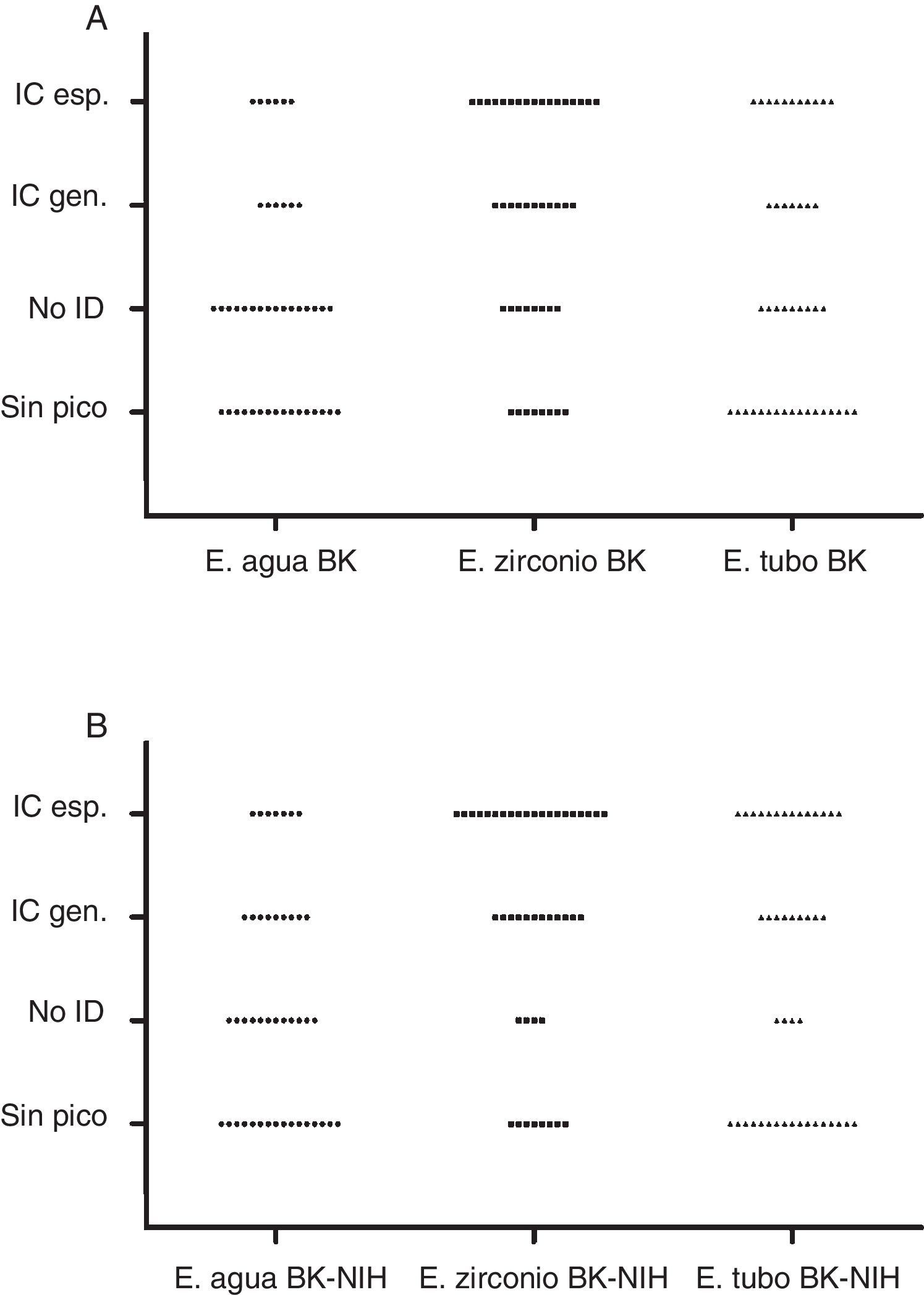
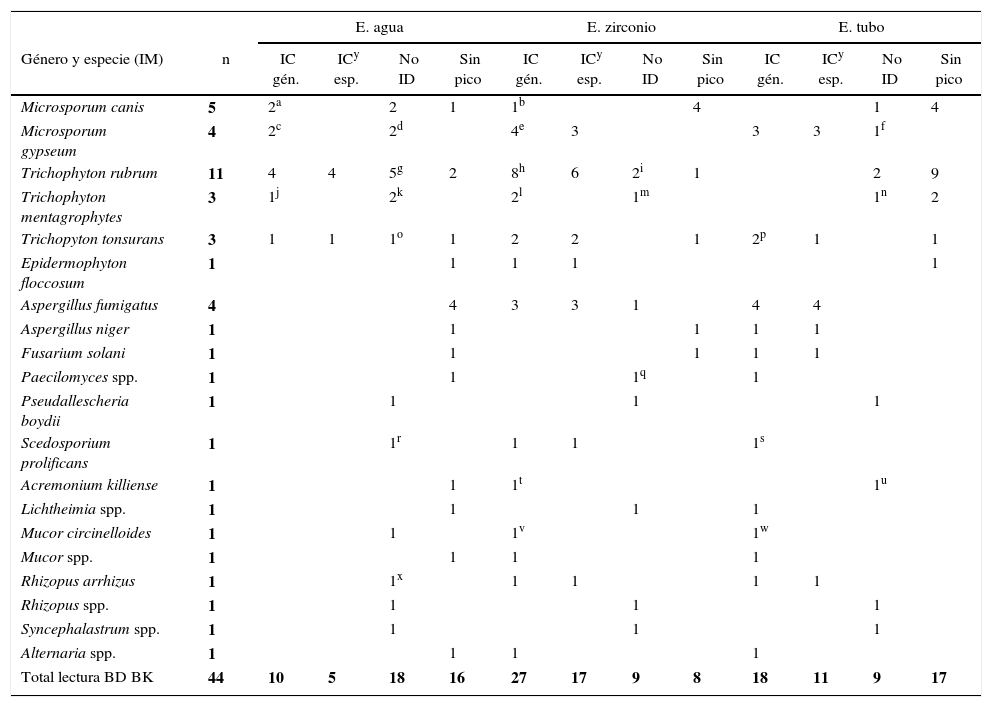
El análisis con la base de datos de BK mostró los siguientes resultados: IC género en 22,72% (10/44) con E. agua, en 61,36% (27/44) con E. zirconio y en 40,90% (18/44) con E. tubo. Se consideraron para el análisis 36 de 44 hongos identificados a nivel de especie: IC especie en 13,89% (5/36) con E. agua, en 47,2% (17/36) con E. zirconio y en 30,56% (11/36) con E. tubo. No ID, en 40,91% (18/44) con E. agua, 20,45% (9/44) con E. zirconio y con E. tubo. No se obtuvieron picos en 36,36% (16) con E. agua, 18,18% (8) con E. zirconio y 38,64% (17) con E. tubo (tabla 1 y fig. 1A).
Análisis de los 44 aislamientos estudiados por el sistema Bruker Microflex con la base de datos BK
| E. agua | E. zirconio | E. tubo | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Género y especie (IM) | n | IC gén. | ICy esp. | No ID | Sin pico | IC gén. | ICy esp. | No ID | Sin pico | IC gén. | ICy esp. | No ID | Sin pico |
| Microsporum canis | 5 | 2a | 2 | 1 | 1b | 4 | 1 | 4 | |||||
| Microsporum gypseum | 4 | 2c | 2d | 4e | 3 | 3 | 3 | 1f | |||||
| Trichophyton rubrum | 11 | 4 | 4 | 5g | 2 | 8h | 6 | 2i | 1 | 2 | 9 | ||
| Trichophyton mentagrophytes | 3 | 1j | 2k | 2l | 1m | 1n | 2 | ||||||
| Trichopyton tonsurans | 3 | 1 | 1 | 1o | 1 | 2 | 2 | 1 | 2p | 1 | 1 | ||
| Epidermophyton floccosum | 1 | 1 | 1 | 1 | 1 | ||||||||
| Aspergillus fumigatus | 4 | 4 | 3 | 3 | 1 | 4 | 4 | ||||||
| Aspergillus niger | 1 | 1 | 1 | 1 | 1 | ||||||||
| Fusarium solani | 1 | 1 | 1 | 1 | 1 | ||||||||
| Paecilomyces spp. | 1 | 1 | 1q | 1 | |||||||||
| Pseudallescheria boydii | 1 | 1 | 1 | 1 | |||||||||
| Scedosporium prolificans | 1 | 1r | 1 | 1 | 1s | ||||||||
| Acremonium killiense | 1 | 1 | 1t | 1u | |||||||||
| Lichtheimia spp. | 1 | 1 | 1 | 1 | |||||||||
| Mucor circinelloides | 1 | 1 | 1v | 1w | |||||||||
| Mucor spp. | 1 | 1 | 1 | 1 | |||||||||
| Rhizopus arrhizus | 1 | 1x | 1 | 1 | 1 | 1 | |||||||
| Rhizopus spp. | 1 | 1 | 1 | 1 | |||||||||
| Syncephalastrum spp. | 1 | 1 | 1 | 1 | |||||||||
| Alternaria spp. | 1 | 1 | 1 | 1 | |||||||||
| Total lectura BD BK | 44 | 10 | 5 | 18 | 16 | 27 | 17 | 9 | 8 | 18 | 11 | 9 | 17 |
IM: identificación morfológica; E. agua: extracción en agua-ácido fórmico; E. zirconio: extracción con zirconio-etanol-acetonitrilo-ácido fórmico; E. tubo: extracción en tubo con caldo Sabouraud-etanol-acetonitrilo-ácido fórmico, recomendada por el proveedor; IC gén.: identificación correcta a nivel de género; IC esp.: identificación correcta a nivel de género y especie; No ID: identificación incorrecta; BD: base de datos.
Un aislamiento S. prolificans con score mayor que 1,6, pero una diferencia menor del 10% con el siguiente score, que fue S. apiospermun.
Un aislamiento M. circinelloides que dio M. ramosissimus con score mayor que 1,6, pero con una diferencia menor del 10% con el siguiente score.
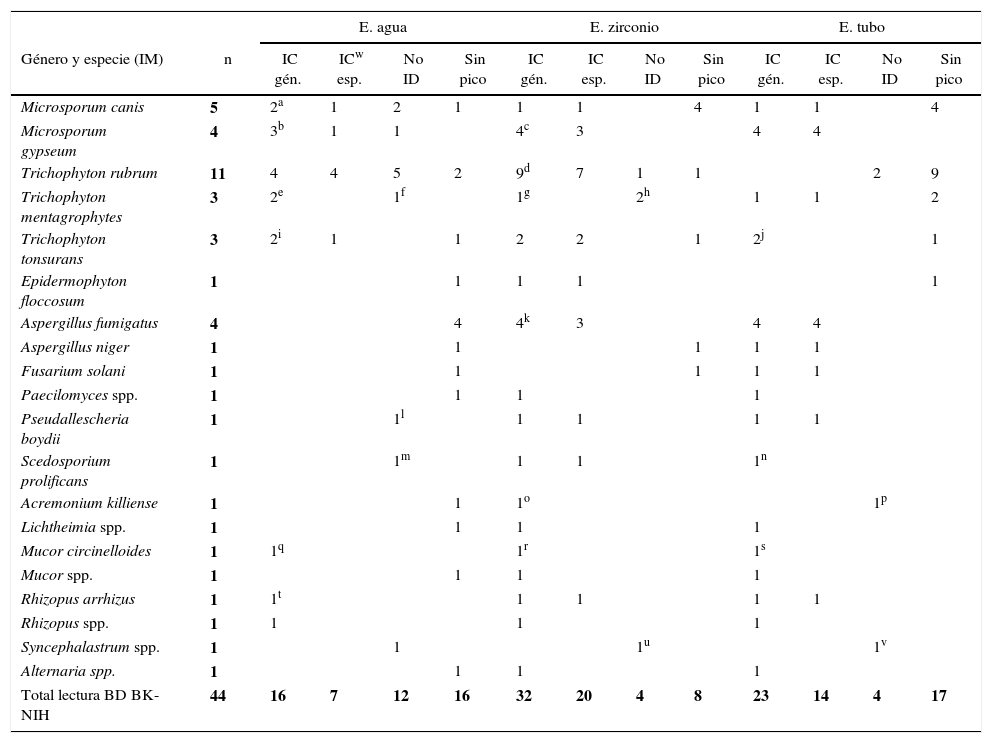
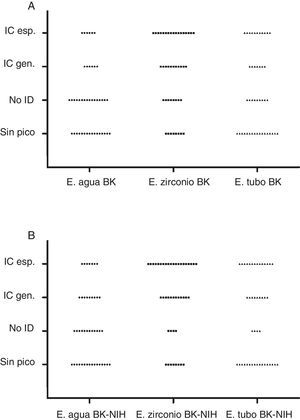
Distribución de los resultados obtenidos por MALDI-TOF MS en función del método de extracción y la base de datos utilizada. Identificación correcta a nivel de especie (IC esp.), identificación correcta a nivel de género (IC gen.), sin identificación (No ID), extracción con agua (E. agua), extracción con zirconio (E. zirconio) y extracción en tubo (E. tubo). A) Análisis con la base de datos de BK. B) Análisis con la base de datos de BK+NIH.
En segunda instancia, se analizaron los espectros con una base de datos más extensa, BK-NIH, para evaluar el rendimiento del MALDI-TOF MS. Las principales diferencias se observaron con respecto a las IC a nivel de género, que se incrementaron al 72,72, el 52,27% y el 36,36% con la E. zirconio, E. tubo y E. agua, respectivamente. A su vez, las identificaciones no confiables se redujeron a 4 aislamientos (9%) con E. zirconio y E. tubo y a 12 (27,3%) con E. agua; sin embargo, estas diferencias no fueron significativas (p>0,05) (tabla 2 y fig. 1B).
Análisis de los 44 aislamientos estudiados por el sistema Bruker Microflex con la base de datos BK-NIH
| E. agua | E. zirconio | E. tubo | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Género y especie (IM) | n | IC gén. | ICw esp. | No ID | Sin pico | IC gén. | IC esp. | No ID | Sin pico | IC gén. | IC esp. | No ID | Sin pico |
| Microsporum canis | 5 | 2a | 1 | 2 | 1 | 1 | 1 | 4 | 1 | 1 | 4 | ||
| Microsporum gypseum | 4 | 3b | 1 | 1 | 4c | 3 | 4 | 4 | |||||
| Trichophyton rubrum | 11 | 4 | 4 | 5 | 2 | 9d | 7 | 1 | 1 | 2 | 9 | ||
| Trichophyton mentagrophytes | 3 | 2e | 1f | 1g | 2h | 1 | 1 | 2 | |||||
| Trichophyton tonsurans | 3 | 2i | 1 | 1 | 2 | 2 | 1 | 2j | 1 | ||||
| Epidermophyton floccosum | 1 | 1 | 1 | 1 | 1 | ||||||||
| Aspergillus fumigatus | 4 | 4 | 4k | 3 | 4 | 4 | |||||||
| Aspergillus niger | 1 | 1 | 1 | 1 | 1 | ||||||||
| Fusarium solani | 1 | 1 | 1 | 1 | 1 | ||||||||
| Paecilomyces spp. | 1 | 1 | 1 | 1 | |||||||||
| Pseudallescheria boydii | 1 | 1l | 1 | 1 | 1 | 1 | |||||||
| Scedosporium prolificans | 1 | 1m | 1 | 1 | 1n | ||||||||
| Acremonium killiense | 1 | 1 | 1o | 1p | |||||||||
| Lichtheimia spp. | 1 | 1 | 1 | 1 | |||||||||
| Mucor circinelloides | 1 | 1q | 1r | 1s | |||||||||
| Mucor spp. | 1 | 1 | 1 | 1 | |||||||||
| Rhizopus arrhizus | 1 | 1t | 1 | 1 | 1 | 1 | |||||||
| Rhizopus spp. | 1 | 1 | 1 | 1 | |||||||||
| Syncephalastrum spp. | 1 | 1 | 1u | 1v | |||||||||
| Alternaria spp. | 1 | 1 | 1 | 1 | |||||||||
| Total lectura BD BK-NIH | 44 | 16 | 7 | 12 | 16 | 32 | 20 | 4 | 8 | 23 | 14 | 4 | 17 |
IM: identificación morfológica; E. agua: extracción en agua-ácido fórmico; E. zirconio: extracción con zirconio-etanol-acetonitrilo-ácido fórmico; E. tubo: extracción en tubo con caldo Sabouraud-etanol-acetonitrilo-ácido fórmico, recomendada por el proveedor; IC gén.: identificación correcta a nivel de género; IC esp.: identificación correcta a nivel de género y especie; No ID: identificación incorrecta; BD: base de datos.
Un aislamientoT. mentagrophytes con score entre 1,5-1,6 y otro que dio T. tonsurans con score mayor que 1,6.
Un aislamientoT. mentagrophytes que dio T. tonsurans con score menor que 1,5; y otro T. mentagrophytes que dio T. mentagrophytes con score menor que 1,5.
Un aislamientoT. tonsurans que dio T. interdigitale con score mayor que 1,6 y otro que dio T. rubrum con score mayor que 1,6.
Un aislamiento P. boydii que dio Scedosporium apiospermun (teleomorfo de P. boydii) con score menor que 1,5.
Un aislamiento S. prolificans con score mayor que 1,6 pero con una diferencia menor del 10% con el siguiente score, que fue S. apiospermun.
Un aislamiento M. circinelloides que dio M. ramosissimus con score mayor que 1,6, pero con una diferencia menor del 10% con el siguiente score.
El método de E. zirconio mostró ser superior a las otras metodologías de extracción, tanto al comparar con la base de datos de BK (kappa 0,09; p<0,05) como al hacerlo con la base de datos ampliada por el agregado de los espectros del NIH (kappa 0,08, p<0,05) (fig. 1).
No se obtuvieron identificaciones incorrectas a nivel de género; no obstante, se obtuvieron identificaciones incorrectas a nivel de especie, correspondientes a T. mentagrophytes seguido de T. tonsurans. A su vez, un aislamiento de A. killiense fue identificado erróneamente como Acremonium strictum, probablemente porque esta especie no se encuentra en la base datos. Una cepa de Scedosporium prolificans CCE fue identificada con un score mayor que 1,6 con E. tubo; sin embargo, fue considerada solo IC a nivel de género porque hubo una diferencia menor del 10% con el siguiente score, que correspondió a Scedosporium apiospermun. Un resultado similar se observó con otra cepa CCE, M. circinelloides, que se confunde con Mucor ramosissimus, tanto en la E. zirconio como en la E. tubo (tablas 1 y 2).
Con respecto a los 3 géneros de dermatofitos y las especies más frecuentes implicadas en infecciones superficiales, las 2 metodologías con las que se obtuvieron los mejores rendimientos fueron E. agua y E. zirconio. Si bien son esperables mejores resultados con el método recomendado por el proveedor, la extracción en tubo mostró la menor eficiencia, ya que no se obtuvieron picos en el 39% de los aislamientos. Los errores de identificación correspondieron a las especies T. mentagrophytes y T. tonsurans. Esto podría deberse a que estas especies forman parte de un complejo9 que requiere cambios taxonómicos8. Recientemente, Guarro8 ha publicado que el complejo Arthroderma vanbreuseghemii (teleomorfo) está asociado a 3 anamorfos de distribución mundial, T. tonsurans, Trichophyton equinum y Trichophyton interdigitale/mentagrophytes8,12. Esto confirma la similitud entre las especies de T. mentagrophytes y T. tonsurans, y explica la dificultad en su diferenciación por los diferentes métodos de identificación.
Para el género Trichophyton, la metodología MALDI-TOF MS presenta dificultades para la identificación a nivel de especie. De hecho, el sistema emite una alerta con respecto a la limitación del método para diferenciar las especies del género por estar estrechamente relacionadas (T. equinum/interdigitale/mentagrophytes/rubrum/tonsurans/violaceum). Por el contrario, para los géneros Microsporum y Epidermophyton, las especies son correctamente identificadas cuando se logra un espectro de buena calidad. Por lo anteriormente mencionado, es importante que las bases de datos para dermatofitos sean extensas, especialmente para el género Trichophyton, de tal manera de poder incluir la variabilidad intraespecie de este grupo de hongos9,12. El agregado de la base de datos del NIH no mejoró la identificación porque aquella solo suma 12 espectros a los 63 que tiene la base de datos de BK para los dermatofitos.
Con respecto a los hongos causantes de hialohifomicosis y mucorales, la identificación con E. zirconio y E. tubo mostraron rendimientos similares, mientras que el método de E. agua no fue de utilidad para este grupo de hongos. El agregado de la base de datos de NIH permitió mejorar la identificación, especialmente con la metodología de E. zirconio.
La capacidad de la MALDI-TOF MS para la identificación de los hongos miceliales productores de IFI coincide con lo reportado por otros autores. Las especies de Aspergillus y Fusarium fueron identificadas correctamente, así como el complejo Pseudallescheria/Scedosporium y los diferentes géneros de los hongos mucorales. La excepción fue un aislamiento de Syncephalastrum, que con el agregado de la base de datos del NIH fue identificado con un score no confiable, ya que dicha base solo agrega dos espectros al único que aportaba la base de datos BK.
De Carolis et al.5 publicaron que la metodología MALDI-TOF MS es capaz de diferenciar entre especies de Aspergillus morfológica y filogenéticamente similares, indistinguibles por técnicas genotípicas. Asimismo, Balajee et al.1 encontraron que el 24% de los aislamientos identificados por análisis morfológico como A. fumigatus correspondían a la especie Aspergillus lentulus por técnicas de secuenciación, los cuales también fueron clasificados correctamente por MALDI-TOF MS.
Más interesante aún es el gran poder de discriminación de MALDI-TOF MS de cepas pertenecientes a especies estrechamente relacionadas del complejo F. solani5. Las publicaciones que evalúan el rendimiento de MALDI-TOF MS para la identificación de mucorales demuestran la capacidad de discriminar aislamientos de este grupo tanto a nivel de género como de especies7,14.
Los hongos dematiáceos están poco representados en las bases de datos; a su vez, está descripto en la literatura que se obtienen espectros de mala calidad debido a su pigmento. Estos pueden inhibir la ionización, lo cual lleva a una mala adquisición de los espectros de masas en MALDI-TOF MS, como lo describen Buskirk et al.2. Sin embargo, en nuestro trabajo, el único aislamiento de Alternaria spp. pudo ser identificado correctamente tanto con la E. tubo como con la E. zirconio.
El promedio de los scores a nivel de género y de especie fue mayor utilizando la base extendida: 1,66; 1,97 y 2,15 para E. agua, E. zirconio y E. tubo, respectivamente, con las bases de BK-NIH (p<0,05), frente a 1,64; 1,81 y 2,01 utilizando la base de datos de BK.
La interpretación recomendada por Bruker aplica criterios más estrictos que los utilizados en este trabajo, tanto para la identificación aceptable a nivel de género y especie como la identificación solo a nivel de género, scores ≥ 2,0 y de 1,7 a 2,0, respectivamente, considerando puntajes inferiores a 1,7 como poco fiables15. Pero estos criterios de interpretación son muy exigentes para hongos miceliales, como se refleja en varias publicaciones3,5,7,10,14. En este trabajo consideramos adecuado para aceptar una identificación como correcta a nivel de género un score ≥ 1,5 y para especie un score ≥ 1,6 en función de la correlación obtenida entre la identificación convencional y el MALDI-TOF MS.
Con el agregado de la base de datos del NIH, se observó un aumento estadísticamente significativo en los scores (p<0,05), que implicó una disminución de las identificaciones no confiables. Esto nos permite hipotetizar que con un incremento de espectros en la base de datos podríamos llegar a obtener una diferencia significativa en cuanto a la identificación. Existen varias publicaciones que demuestran que se mejora el rendimiento de esta tecnología con el agregado de una base de datos in house y, mejor aún, si las bases incorporan datos provenientes de cepas de colecciones de cultivos de diferentes regiones geográficas, con el fin de reflejar la diversidad natural de las especies fúngicas5.
Finalmente, el método de E. zirconio mostró el mejor desempeño, independientemente de la base utilizada. Permitió el mayor número de identificaciones a nivel de género y de género-especie, y fue el que presentó la menor cantidad de espectros sin pico (18%) (fig. 1). Por este motivo, consideramos que este método es el más adecuado para la identificación de hongos miceliales. El rendimiento de la E. tubo fue mejor para algunos géneros, en particular para hongos con pigmento como A. niger y F. solani, pero no fue de utilidad para los dermatofitos. Debido a que este método es más laborioso y demanda más tiempo que la de E. zirconio, lo recomendamos como alternativa cuando no se obtienen picos o la identificación es no confiable para hongos productores de IFI. El método de E. agua, que es sencillo y económico, podría ser un procedimiento para utilizar solo en dermatofitos.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
AutoríaIvana Maldonado y Dolores García Ramírez contribuyeron en forma igualitaria a este trabajo.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
La Fundación Dr. Roberto B. Domecq contribuyó con los reactivos y el equipamiento necesario para la realización de este trabajo.