To evaluate the risk of progression of diabetic retinopathy (DR) using new strategies to obtain genetic information in type 2 diabetes (T2D) based on interfering ribonucleic acid (RNA).
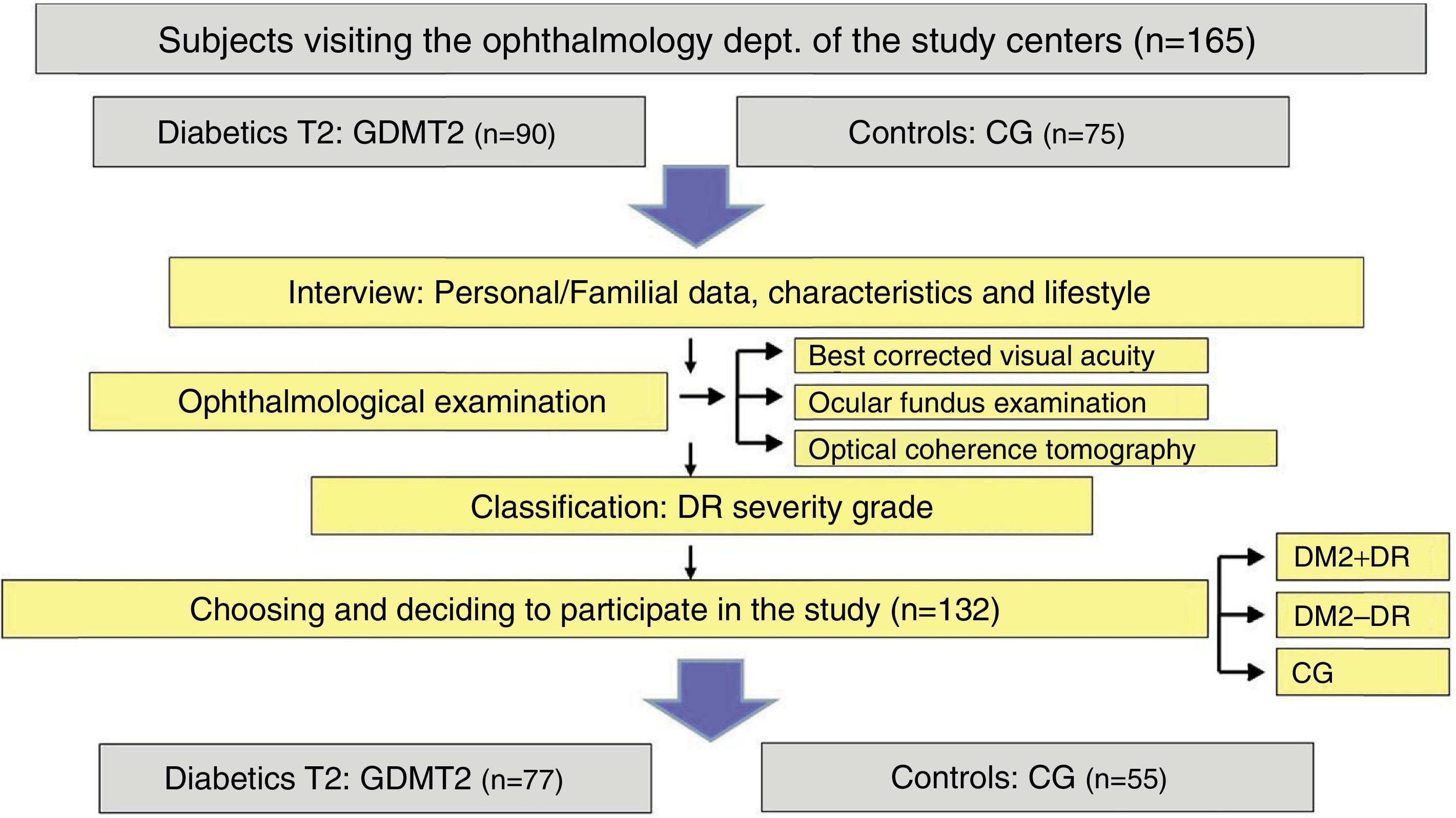
Material and methodsA prospective multicentre case–control study of 132 participants was distributed into: T2D with (+DR) or without (−DR) (T2DG; n=77), and a control group (CG; n=55). After an eye examination and personal interview, tears were collected for molecular analysis (expression of microRNAs [miRNAs] (miRCURY™ ARN Isolation Kit, Qiagen)]. Libraries, 137 vs. 140bp (GeneMapper, Applied Biosystems), were obtained in 18 samples (T2DG+DR=6; T2DG−DR=6; CG=6) by performing next-generation sequencing (NGS). SPSS 15.0 statistical program was used to perform data analysis.
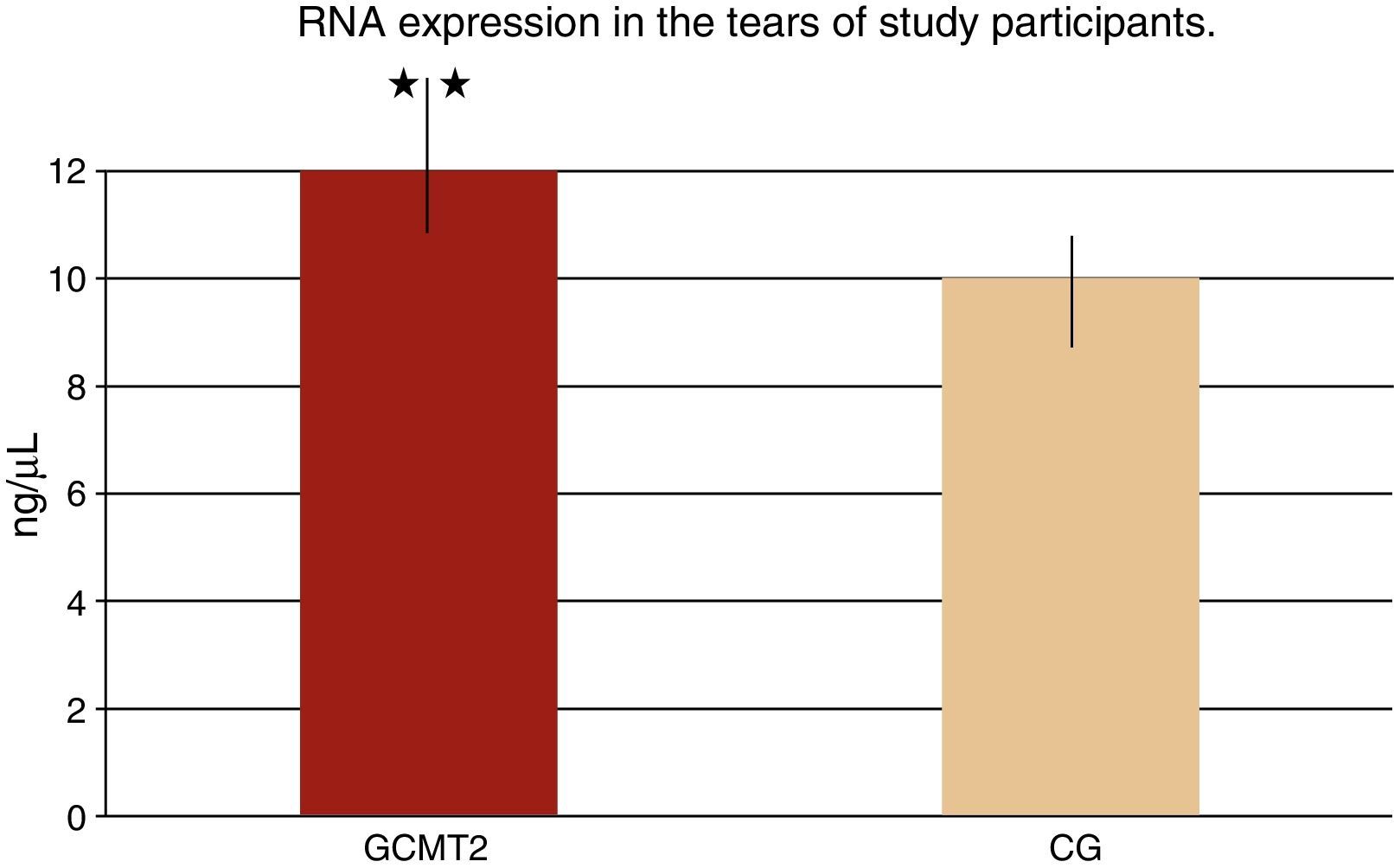
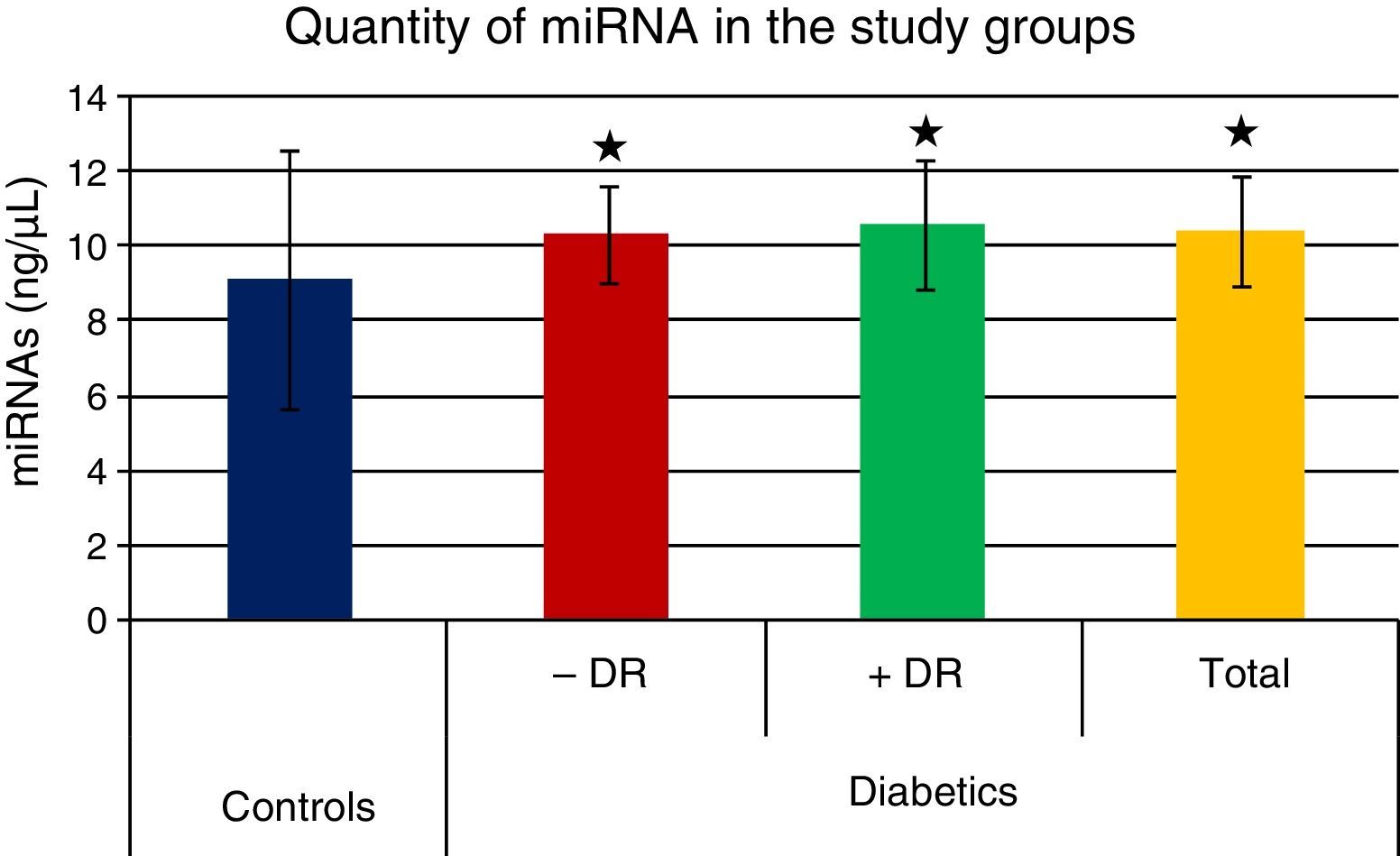
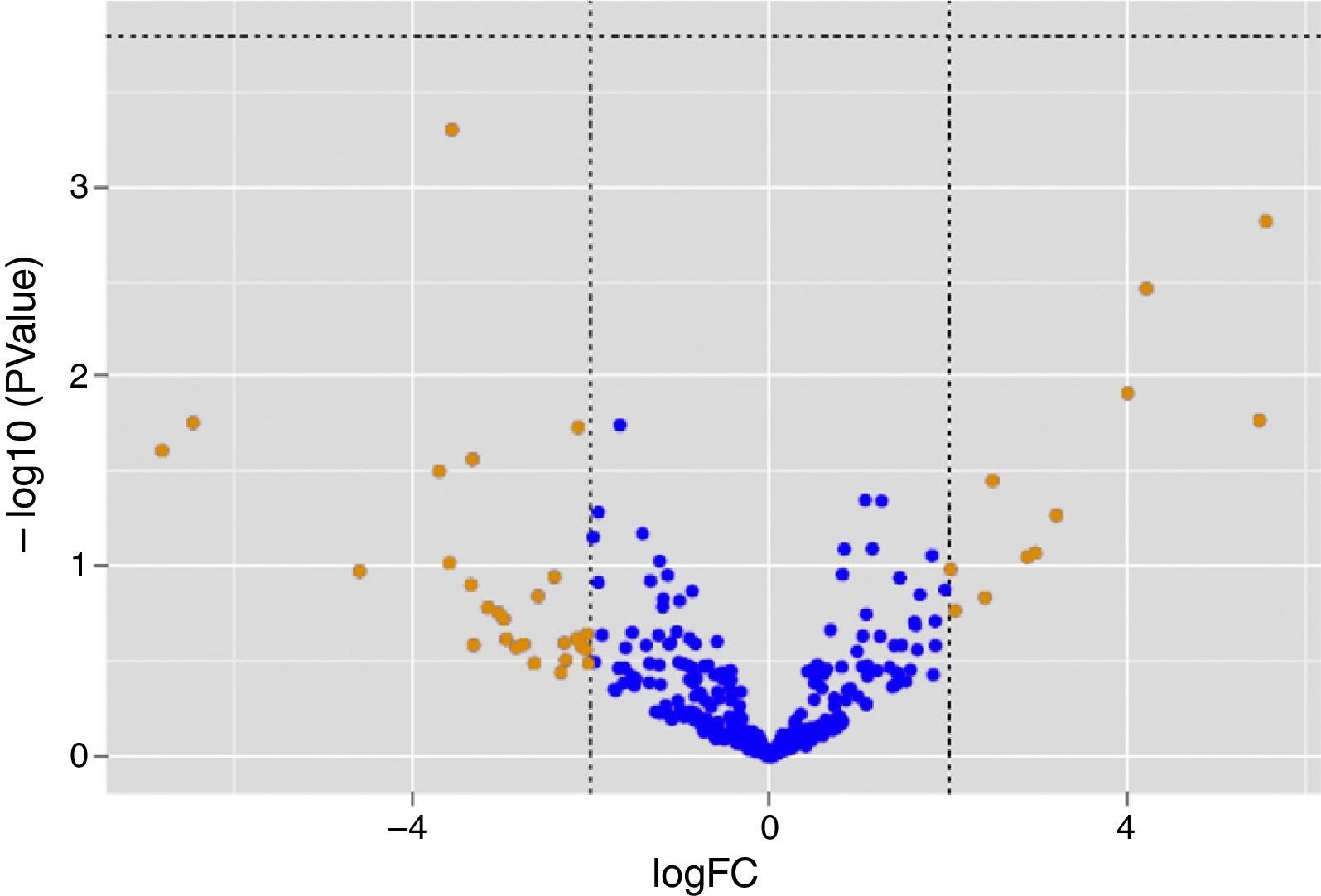
ResultsThe mean age was 67±12 years in the T2DG vs. 55±21 years in the CG. Distribution men/women: 25/30 in T2DG vs. 51/28 in CG. A family history of DM, diet compliance, smoking, drinking and exercise, showed significant differences between groups (p<0.001). A 20–25μL sample of tears contained a mean of 9.42±3.30ng/mL of purified ARN, with significant differences between T2DG/CG (p=0.002) and T2DG+RD/CG (p=0.004). Tear expression of miARNs in T2DG directly correlated with age/obesity/T2D duration (p<0.05), and indirectly with visual acuity (p<0.05). A total of 14 miRNAs related to the presence, pathogenic mechanisms and risk factors for the progression of diabetic retinopathy, were identified.
ConclusionsWe propose to use tears as a source of genetic information for DM. Specific miRNAs involved in DR development and/or progression can be used as molecular biomarkers, and based on these, for developing future biotherapies.
Evaluar el riesgo de progresión de la retinopatía diabética (RD) utilizando nuevas estrategias para obtener información genética en diabéticos tipo 2 (DT2) basadas en interferencia por ácido ribonucleico (ARN).
Material y métodosEstudio multicéntrico, prospectivo de casos–controles en 132 participantes divididos en: grupo DT2 (GDT2) con RD (+RD) y sin RD (−RD) (n=77) y grupo control (GC) (n=55). Tras entrevista personal y examen oftalmológico, se extrajeron lágrimas para análisis molecular (expresión de micro-ARN [miARN] [miRCURY™ ARN Isolation Kit, Qiagen]). En 18 muestras (GDT2+RD=6; GDT2–RD=6; GC=6) obtuvimos librerías de 137 vs. 140 pares de bases (GeneMapper, Applied Biosystems) y realizamos secuenciación de próxima generación (NGS). El programa SPSS 15.0 vehiculizó el análisis estadístico.
ResultadosEdad media: 67±12 años en GDT2 vs. 55±21 años en GC. Distribución hombres/mujeres: 51/28 en GDT2 vs. 25/30 en GC. Los antecedentes familiares de DM, cumplir dieta, fumar, beber y realizar ejercicio mostraron diferencias significativas entre grupos (p<0,001). Con 20–25μL de lágrimas extrajimos 9,42±3,30ng/mL de ARN purificado, con diferencias significativas entre GDT2/GC (p=0,002) y GDT2+RD/GC (p=0,004). La expresión lagrimal de miARN en GDT2 correlacionó directamente con: edad/obesidad/duración de DM (p<0,05), e indirectamente con: agudeza visual (p<0,05). Hemos identificado 14 miARN relacionados con la presencia, mecanismos patogénicos y factores de riesgo para la progresión de la RD.
ConclusionesProponemos utilizar lágrimas como fuente de información genética para la DM. Los miARN específicos implicados en desarrollo o progresión de la RD pueden utilizarse como biomarcadores moleculares y, a partir de ellos, desarrollar futuras bioterapias.