Las quilomicronemias generalmente se diagnostican genéticamente mediante secuenciación genómica o cribado de mutaciones en genes causales con un gran efecto fenotípico. Esta estrategia ha permitido mejorar la caracterización de estos pacientes, pero aún tenemos un 30% de ellos sin un diagnóstico genético concluyente. Es por esto que hipotetizamos que añadiendo el componente epigenético podemos mejorar el diagnóstico genético y para ello hemos explorado el grado de metilación en el ADN de pacientes hipertrigliceridémicos.
MetodologíaEl ADN de células sanguíneas fue obtenido de 16 pacientes hipertrigliceridémicos y de 16 sujetos control emparejados por edad y sexo. El grado de metilación en el ADN de todo el genoma fue determinado mediante el Illumina® Infinium MethylationEPIC Array Analysis.
ResultadosIdentificamos 31 citosinas diferencialmente metiladas al comparar los patrones de metilación que presentaban los pacientes hipertrigliceridémicos vs. los sujetos control. La cg03636183 en el gen F2RL3 estaba un 10% hipometilada en los pacientes hipertrigliceridémicos, y ha sido previamente asociada a un mayor riesgo cardiovascular. La cg13824500 está un 10% hipometilada en pacientes hipertrigliceridémicos y se localiza en VTI1A, que es un gen limitante en el tránsito de los quilomicrones en el enterocito a través del retículo endoplásmico y el aparato de Golgi. La cg26468118 en el gen RAB20 (13% hipometilada) y la cg21560722 en el gen SBF2 (33% hipermetilada) están implicadas en la regulación de vesículas del aparato de Golgi.
ConclusionesNuestros resultados evidencian que existen regiones diferencialmente metiladas relacionadas con la formación de los quilomicrones en pacientes hipertrigliceridémicos.
Chylomicronemias are generally diagnosed genetically by genomic sequencing or screening for mutations in causal genes with a large phenotypic effect. This strategy has allowed to improve the characterization of these patients, but we still have 30% of the patients without a conclusive genetic diagnosis. This is why we hypothesize that by adding the epigenetic component we can improve the genetic diagnosis, and for this we have explored the degree of methylation in the DNA of hypertriglyceridemic patients.
MethodologyBlood cell DNA was obtained from 16 hypertriglyceridemic patients and from 16 age- and sex-matched control subjects. The degree of methylation in genome-wide DNA was determined using the Illumina® Infinium Methylation EPIC Array Analysis.
ResultsWe identified 31 differentially methylated cytosines by comparing the methylation patterns presented by hypertriglyceridemic patients vs. control subjects. The cg03636183 in the F2RL3 gene was 10% hypomethylated in hypertriglyceridemic patients, and has previously been associated with an increased cardiovascular risk. Cg13824500 is 10% hypomethylated in hypertriglyceridemic patients and is located in VTI1A, which is a limiting gene in the transit of chylomicrons in the enterocyte through the endoplasmic reticulum and the Golgi apparatus. Cg26468118 in the RAB20 gene (13% hypomethylated) and cg21560722 in the SBF2 gene (33% hypermethylated) are involved in the regulation of Golgi apparatus vesicles.
ConclusionsOur results suggest that there are differentially methylated regions related to the formation of chylomicrons in hypertriglyceridemic patients.
El diagnóstico genético basado en la determinación de mutaciones causales con un gran efecto fenotípico es clave en el diagnóstico de las formas más severas de dislipidemia1. En el caso de las quilomicronemias o hipertrigliceridemias graves, encontramos que son trastornos con una etiopatogenia compleja que se definen por la presencia anormal de quilomicrones en plasma en ayunas, produciendo niveles de triglicéridos (TG) superiores a 500-1.000mg/dl. Diferenciamos 2 tipos de hipertrigliceridemias graves.
La hipertrigliceridemia primaria suele tener una causa monogénica y se presenta en forma autosómica recesiva. Actualmente hay 5 genes identificados como principales responsables que codifican para proteínas clave en el proceso de lipólisis de los TG: son LPL, APOC2, GPIHBP1, APOA5 y LMF1. Ciertas mutaciones en estos genes producen niveles elevados de TG de forma crónica. Las hipertrigliceridemias primarias tienen una prevalencia de 1:100.000-1:1.000.000; son de aparición temprana; su principal manifestación clínica es la pancreatitis, que puede llegar a ser fatal; y no suelen asociarse a un elevado riesgo cardiovascular (revisado en Dron y Hegele2).
Por otro lado, las hipertrigliceridemias secundarias tienen una mayor prevalencia (1:6.000); suelen aparecen en adultos; a nivel metabólico presentan un aumento del número de quilomicrones, junto con VLDL, y sus remanentes, y esto hace que se asocien a un elevado riesgo cardiovascular; encontramos una influencia significativa de factores secundarios (como obesidad, síndrome metabólico, diabetes o una ingesta elevada de alcohol, entre otros); y la causa genética es más compleja, con la participación de variantes genéticas raras en forma heterocigota en los 5 genes diagnóstico descritos anteriormente, junto con variantes en otros genes asociados a TG3. Cada vez parece más claro que esta forma de quilomicronemia se suele presentar como una hipertrigliceridemia relativamente moderada pero asociada a descompensaciones hiperlipémicas esporádicas que, debido a ciertas situaciones de sobrecarga lipídica, pueden multiplicar los niveles de TG hasta 10 veces o más, para volver a disminuir rápidamente4.
Por lo tanto, existen diferencias claras entre ambas formas de hipertrigliceridemia grave, y esto hace que conocer la causa genética responsable pueda ayudar a caracterizar mejor el fenotipo de cada paciente; estratificar mejor su riesgo cardiovascular; mejorar la identificación de nuevos pacientes, y facilitar el tratamiento más adecuado a cada paciente.
Hoy en día, la combinación de varias estrategias genéticas, como la identificación de nuevas mutaciones con gran efecto fenotípico gracias a la secuenciación masiva, junto con el diseño de risk-score genéticos que engloban varias variantes moduladoras de los TG, han permitido mejorar la caracterización de estos pacientes5, pero aun así, seguimos dejando cerca de un 30% de los casos de quilomicronemia sin causa genética conocida6,7. Por ello, no descartamos que otros componentes genéticos tengan un papel en el desarrollo de estas dislipidemias. En este trabajo hemos querido evaluar si la epigenética puede estar implicada en la hipertrigliceridemia y por ello hemos realizado una estrategia no dirigida, llevando a cabo un estudio de asociación del epigenoma completo en muestras de pacientes con hipertrigliceridemia severa sin causa genética conocida, para identificar nuevas regiones en el genoma diferencialmente metiladas en comparación con un grupo de sujetos normolipémicos.
MetodologíaParticipantesEl grupo de pacientes hipertrigliceridémicos está compuesto por 16 sujetos remitidos a la Unidad de Lípidos de nuestro centro para el estudio de hipertrigliceridemia severa. Son pacientes que presentan niveles de TG superiores a 1.000mg/dl y en los que no se detectó ninguna de las principales mutaciones causales en los genes responsables de las formas más severas de hipertrigliceridemia descritas en la bibliografía (7 mutaciones en LPL, 5 en APOA5, 5 en GPIHBP1, una en APOC2 y 2 en LMF1). Estos pacientes fueron comparados con sujetos normolipémicos emparejados por edad y sexo que formaron parte del proyecto europeo VITAGE8, que incluyó hombres aparentemente sanos de edades comprendidas entre los 20 y los 75 años. No se incluyeron sujetos con enfermedades crónicas hepáticas, renales, cardiopulmonares o afectos de neoplasias.
El estudio fue aprobado por el Comité de Ética e Investigación Clínica de nuestro centro y todos los participantes firmaron el consentimiento informado.
MuestrasSe obtuvieron muestras de sangre en ayunas de todos los participantes y se recogieron en tubos EDTA de plasma (Sarstedt, Ltd., Nümbrecht, Alemania), protegidos de la luz, y centrifugados inmediatamente a 1.500g durante 10min a 8°C. El plasma fue separado y dividido en alícuotas. El buffy coat fue recogido para la extracción de ADN genómico. Todas las muestras fueron almacenadas a −80°C hasta que se llevaron a cabo las determinaciones analíticas.
Determinación de los lípidos en plasmaLas concentraciones de TG y colesterol en plasma fueron medidas mediante kits enzimáticos (F. Hoffmann-La Roche, Ltd.) adaptados para un autoanalizador Cobas Mira (F. Hoffmann-La Roche, Ltd.). Mediante inmunoturbidometría medimos las apolipoproteína B y la lipoproteína (a) utilizando anticuerpos específicos (F. Hoffmann-La Roche, Ltd. y Incstar Corp., respectivamente).
El colesterol de las LDL fue calculado mediante la fórmula de Friedewald9 y no pudo ser calculado en todos los pacientes hipertrigliceridémicos.
Estudio de asociación de genoma completoFueron tratados con bisulfito sódico 500ng de ADN para convertir las citosinas en uracilos, mientras que las citosinas con un grupo 5-metil no reaccionan ante el bisulfito de sodio. El ADN fue seguidamente hibridado al array de Illumina® (Infinium MethylationEPIC BeadChip) siguiendo las instrucciones del protocolo de la casa comercial. Los datos de las intensidades de cada tipo de sonda del array (archivos idat) se procesaron utilizando el paquete R, ChAMP versión 2.9.1010,11.
Se filtraron las sondas con un valor p de detección superior a 0,01 en una o más muestras, sondas con un recuento de cuentas<3 en al menos el 5% de las muestras, sondas con SNP próximos tal como se identifica en Zhou et al.12, sondas que se alinean en múltiples ubicaciones como se identifica en Nordlund et al.13, y sondas localizadas en los cromosomas X o Y.
Después de filtrar las sondas, se realizó la normalización del tipo intracelular utilizando el método de normalización cuantílica de mezcla beta14 para evitar el sesgo introducido por el diseño de la sonda Infinium tipo 2.
Después de la normalización cuantílica de mezcla beta, la magnitud de los efectos del lote se evaluó utilizando la SVD. Para corregir las diferencias en la metilación resultantes de las diferencias en la heterogeneidad celular, se utilizó la corrección de Houseman15.
El análisis de metilación diferencial se realizó utilizando el estadístico t moderado de eBayes (para la variable de resultado) mediante el paquete limma16 del software estadístico R. El valor β en los experimentos de metilación es la estimación del nivel de metilación utilizando la relación entre la intensidad de la sonda de metilación y la intensidad general, mientras que el valor M es una transformación logarítmica del valor β. El valor M se utilizó para el análisis de metilación diferencial y el valor β para reportar los resultados, lo que proporcionó una interpretación biológica más intuitiva. Los valores de p brutos se ajustaron mediante el procedimiento de Benjamini-Hochberg, y una tasa de descubrimientos falsos de corte de 0,05 y deltaBeta≥5% en los análisis relacionados con los resultados se utilizó como umbral estadísticamente significativo.
Análisis estadísticoUtilizamos la prueba de Kolmogorov-Smirnov para ver si las variables cuantitativas seguían o no una distribución normal.
Los datos descriptivos de las variables cuantitativas se presentan como media±desviación estándar o mediana (rango intercuartílico). En el caso de las variables cualitativas, se presentan como porcentaje.
Las correlaciones entre el porcentaje de metilación de las citosinas diferencialmente metiladas con las distintas variables cuantitativas fueron ajustadas por edad, sexo e IMC.
Resultados y discusiónActualmente ya existen muchas evidencias que indican que los mecanismos epigenéticos juegan un papel importante en la regulación de fenotipos metabólicos y en enfermedades complejas como es el caso de las dislipidemias17, aunque aún no está claro si la gran mayoría de los cambios descritos en la metilación del ADN son causales o consecuencia de las dislipidemias. En el presente estudio hemos utilizado la epigenética como una herramienta para la identificación de nuevas regiones genómicas relacionadas con la hipertrigliceridemia grave, y para ello hemos querido conocer el patrón de metilación de ADN en un grupo de pacientes con hipertrigliceridemia grave que no tienen un claro diagnóstico genético, para compararlo con el que presentan un grupo de sujetos normolipémicos.
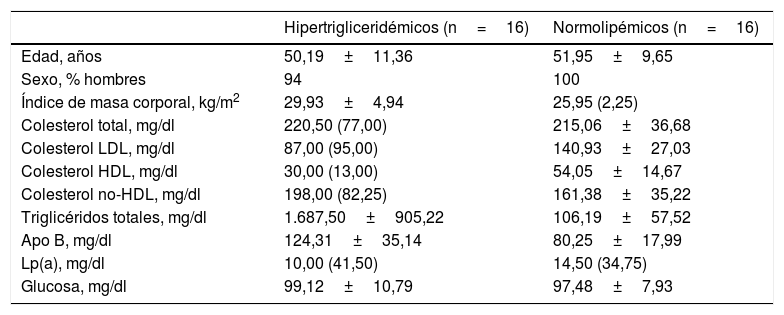
Ambos grupos estaban emparejados por edad y sexo para minimizar la variabilidad biológica entre grupos. Los participantes presentaban mediana edad y eran mayoritariamente hombres. Tal y como muestra la tabla 1, el grupo de pacientes hipertrigliceridémicos presentaban de forma estadísticamente significativa un mayor IMC, una mayor concentración de TG, una menor concentración de colesterol HDL, una mayor concentración de colesterol no-HDL y una mayor concentración de glucosa que los sujetos normolipémicos.
Características antropométricas y bioquímicas basales de los participantes
| Hipertrigliceridémicos (n=16) | Normolipémicos (n=16) | |
|---|---|---|
| Edad, años | 50,19±11,36 | 51,95±9,65 |
| Sexo, % hombres | 94 | 100 |
| Índice de masa corporal, kg/m2 | 29,93±4,94 | 25,95 (2,25) |
| Colesterol total, mg/dl | 220,50 (77,00) | 215,06±36,68 |
| Colesterol LDL, mg/dl | 87,00 (95,00) | 140,93±27,03 |
| Colesterol HDL, mg/dl | 30,00 (13,00) | 54,05±14,67 |
| Colesterol no-HDL, mg/dl | 198,00 (82,25) | 161,38±35,22 |
| Triglicéridos totales, mg/dl | 1.687,50±905,22 | 106,19±57,52 |
| Apo B, mg/dl | 124,31±35,14 | 80,25±17,99 |
| Lp(a), mg/dl | 10,00 (41,50) | 14,50 (34,75) |
| Glucosa, mg/dl | 99,12±10,79 | 97,48±7,93 |
Datos representados como media±DE o mediana (RIC) para las variables cuantitativas.
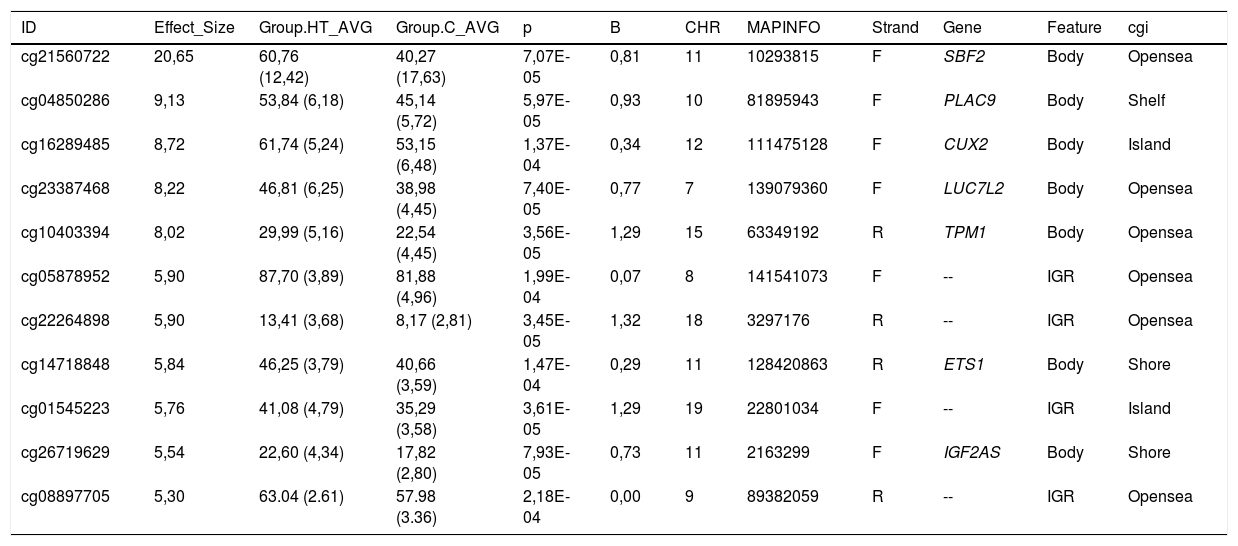
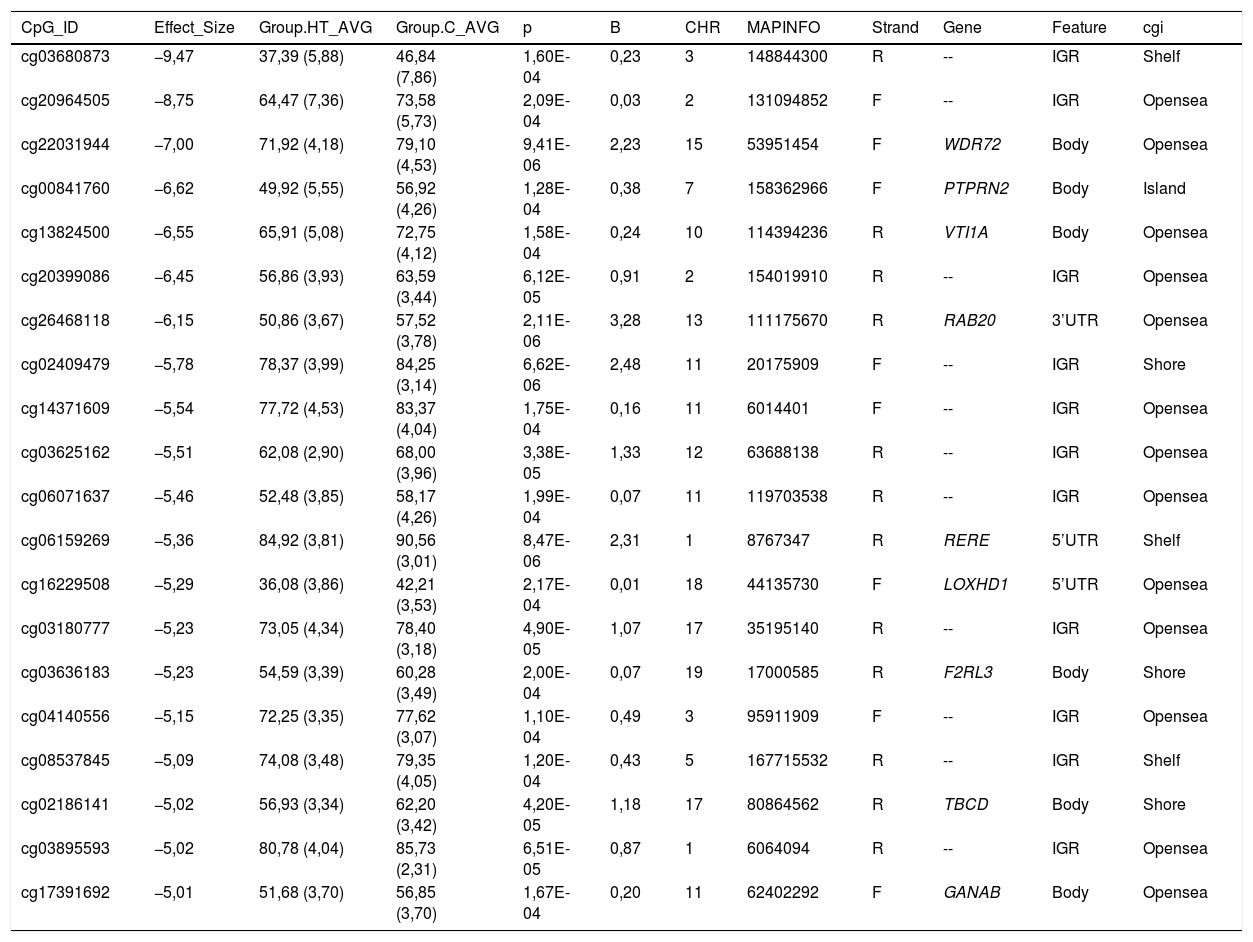
Comparamos los patrones de metilación global entre las muestras de los pacientes hipertrigliceridémicos y los sujetos control normolipémicos, e identificamos 31 citosinas diferencialmente metiladas. La mayoría de las asociaciones (75%) representan citosinas que muestran un menor grado de metilación (hipometiladas) en las muestras hipertrigliceridémicas en comparación con los sujetos normolipémicos.
Todas las citosinas diferencialmente metiladas están recogidas en las tablas 2 y 3. Algunas de estas citosinas están localizadas en regiones genómicas que corresponden a un gen conocido, y otras se localizan en regiones intergénicas y no se han podido asignar a un gen.
Citosinas diferencialmente hipermetiladas en pacientes hipertrigliceridémicos en comparación con los sujetos normolipémicos
| ID | Effect_Size | Group.HT_AVG | Group.C_AVG | p | B | CHR | MAPINFO | Strand | Gene | Feature | cgi |
|---|---|---|---|---|---|---|---|---|---|---|---|
| cg21560722 | 20,65 | 60,76 (12,42) | 40,27 (17,63) | 7,07E-05 | 0,81 | 11 | 10293815 | F | SBF2 | Body | Opensea |
| cg04850286 | 9,13 | 53,84 (6,18) | 45,14 (5,72) | 5,97E-05 | 0,93 | 10 | 81895943 | F | PLAC9 | Body | Shelf |
| cg16289485 | 8,72 | 61,74 (5,24) | 53,15 (6,48) | 1,37E-04 | 0,34 | 12 | 111475128 | F | CUX2 | Body | Island |
| cg23387468 | 8,22 | 46,81 (6,25) | 38,98 (4,45) | 7,40E-05 | 0,77 | 7 | 139079360 | F | LUC7L2 | Body | Opensea |
| cg10403394 | 8,02 | 29,99 (5,16) | 22,54 (4,45) | 3,56E-05 | 1,29 | 15 | 63349192 | R | TPM1 | Body | Opensea |
| cg05878952 | 5,90 | 87,70 (3,89) | 81,88 (4,96) | 1,99E-04 | 0,07 | 8 | 141541073 | F | -- | IGR | Opensea |
| cg22264898 | 5,90 | 13,41 (3,68) | 8,17 (2,81) | 3,45E-05 | 1,32 | 18 | 3297176 | R | -- | IGR | Opensea |
| cg14718848 | 5,84 | 46,25 (3,79) | 40,66 (3,59) | 1,47E-04 | 0,29 | 11 | 128420863 | R | ETS1 | Body | Shore |
| cg01545223 | 5,76 | 41,08 (4,79) | 35,29 (3,58) | 3,61E-05 | 1,29 | 19 | 22801034 | F | -- | IGR | Island |
| cg26719629 | 5,54 | 22,60 (4,34) | 17,82 (2,80) | 7,93E-05 | 0,73 | 11 | 2163299 | F | IGF2AS | Body | Shore |
| cg08897705 | 5,30 | 63.04 (2.61) | 57.98 (3.36) | 2,18E-04 | 0,00 | 9 | 89382059 | R | -- | IGR | Opensea |
C: sujetos control normolipémicos; CHR: cromosoma que contiene la CG (Build 38); HT: pacientes hipertrigliceridémicos; ID: identificador único de la base de datos de CG de Illumina®; MAPINFO: coordenadas de la CG (Build 38); Strand: cadena Forward (F) o Reverse (R).
Citosinas diferencialmente hipometiladas en pacientes hipertrigliceridémicos en comparación con los sujetos normolipémicos
| CpG_ID | Effect_Size | Group.HT_AVG | Group.C_AVG | p | B | CHR | MAPINFO | Strand | Gene | Feature | cgi |
|---|---|---|---|---|---|---|---|---|---|---|---|
| cg03680873 | −9,47 | 37,39 (5,88) | 46,84 (7,86) | 1,60E-04 | 0,23 | 3 | 148844300 | R | -- | IGR | Shelf |
| cg20964505 | −8,75 | 64,47 (7,36) | 73,58 (5,73) | 2,09E-04 | 0,03 | 2 | 131094852 | F | -- | IGR | Opensea |
| cg22031944 | −7,00 | 71,92 (4,18) | 79,10 (4,53) | 9,41E-06 | 2,23 | 15 | 53951454 | F | WDR72 | Body | Opensea |
| cg00841760 | −6,62 | 49,92 (5,55) | 56,92 (4,26) | 1,28E-04 | 0,38 | 7 | 158362966 | F | PTPRN2 | Body | Island |
| cg13824500 | −6,55 | 65,91 (5,08) | 72,75 (4,12) | 1,58E-04 | 0,24 | 10 | 114394236 | R | VTI1A | Body | Opensea |
| cg20399086 | −6,45 | 56,86 (3,93) | 63,59 (3,44) | 6,12E-05 | 0,91 | 2 | 154019910 | R | -- | IGR | Opensea |
| cg26468118 | −6,15 | 50,86 (3,67) | 57,52 (3,78) | 2,11E-06 | 3,28 | 13 | 111175670 | R | RAB20 | 3’UTR | Opensea |
| cg02409479 | −5,78 | 78,37 (3,99) | 84,25 (3,14) | 6,62E-06 | 2,48 | 11 | 20175909 | F | -- | IGR | Shore |
| cg14371609 | −5,54 | 77,72 (4,53) | 83,37 (4,04) | 1,75E-04 | 0,16 | 11 | 6014401 | F | -- | IGR | Opensea |
| cg03625162 | −5,51 | 62,08 (2,90) | 68,00 (3,96) | 3,38E-05 | 1,33 | 12 | 63688138 | R | -- | IGR | Opensea |
| cg06071637 | −5,46 | 52,48 (3,85) | 58,17 (4,26) | 1,99E-04 | 0,07 | 11 | 119703538 | R | -- | IGR | Opensea |
| cg06159269 | −5,36 | 84,92 (3,81) | 90,56 (3,01) | 8,47E-06 | 2,31 | 1 | 8767347 | R | RERE | 5’UTR | Shelf |
| cg16229508 | −5,29 | 36,08 (3,86) | 42,21 (3,53) | 2,17E-04 | 0,01 | 18 | 44135730 | F | LOXHD1 | 5’UTR | Opensea |
| cg03180777 | −5,23 | 73,05 (4,34) | 78,40 (3,18) | 4,90E-05 | 1,07 | 17 | 35195140 | R | -- | IGR | Opensea |
| cg03636183 | −5,23 | 54,59 (3,39) | 60,28 (3,49) | 2,00E-04 | 0,07 | 19 | 17000585 | R | F2RL3 | Body | Shore |
| cg04140556 | −5,15 | 72,25 (3,35) | 77,62 (3,07) | 1,10E-04 | 0,49 | 3 | 95911909 | F | -- | IGR | Opensea |
| cg08537845 | −5,09 | 74,08 (3,48) | 79,35 (4,05) | 1,20E-04 | 0,43 | 5 | 167715532 | R | -- | IGR | Shelf |
| cg02186141 | −5,02 | 56,93 (3,34) | 62,20 (3,42) | 4,20E-05 | 1,18 | 17 | 80864562 | R | TBCD | Body | Shore |
| cg03895593 | −5,02 | 80,78 (4,04) | 85,73 (2,31) | 6,51E-05 | 0,87 | 1 | 6064094 | R | -- | IGR | Opensea |
| cg17391692 | −5,01 | 51,68 (3,70) | 56,85 (3,70) | 1,67E-04 | 0,20 | 11 | 62402292 | F | GANAB | Body | Opensea |
C: sujetos control normolipémicos; CHR: cromosoma que contiene la CG (Build 38); HT: pacientes hipertrigliceridémicos; ID: identificador único de la base de datos de CG de Illumina®; MAPINFO: coordenadas de la CG (Build 38); Strand: cadena Forward (F) o Reverse (R).
Destacamos 4 citosinas localizadas en regiones génicas conocidas.
Los pacientes hipertrigliceridémicos presentan un descenso del 10% en el grado de metilación de la citosina cg03636183 que se localiza en el gen F2RL3 ?F2R Like Thrombin or Trypsin Receptor 3?. Este gen está implicado en procesos asociados al riesgo cardiovascular, como la activación plaquetar18, la hiperplasia de la íntima y la inflamación asociada a tabaquismo19. En concreto, la hipometilación de esta citosina cg03636183 medida en ADN de células sanguíneas ha sido asociada a un mayor riesgo de mortalidad cardiovascular más allá de los factores de riesgo tradicionales en 2 grandes estudios prospectivos20,21.
También hemos identificado 3 citosinas relacionadas con el transporte vesicular. Los pacientes hipertrigliceridémicos presentan un descenso del 10% en el grado de metilación de la cg13824500 localizada en el gen VTI1A ?Vesicle Transport through Interaction with T-SNAREs 1A?. Este es un gen limitante en la formación de los quilomicrones, y más concretamente en el tránsito de los quilomicrones a través del retículo endoplásmico hasta el aparato de Golgi en el enterocito22. En resumen, el proceso de formación de los quilomicrones tiene lugar durante la absorción intestinal de lípidos. En el retículo endoplásmico de la célula se forma el prequilomicrón con la unión de la apoB48 que se va sintetizando, a los lípidos complejos que nos llegan de la dieta. Estos prequilomicrones ahora tienen que llegar al aparato de Golgi para madurar, y este es un proceso complejo y secuencial que involucra la participación de vesículas que se fusionan e interaccionan con varios ligandos y proteínas receptoras en el complejo de Golgi. Los prequilomicrones van avanzando por las diferentes cisternas del complejo gracias a las vesículas transportadoras, y van madurando, adquiriendo proteínas esenciales y sufriendo las modificaciones necesarias hasta formar los quilomicrones que encontraremos en el torrente sanguíneo23,24. Este proceso implica la participación de proteínas como las SNARE y las RAB, entre otras.
En este mismo sentido, también encontramos que la citosina cg26468118, localizada en el gen RAB20 ?Member RAS Oncogene Family?, presentaba un grado de metilación un 13% menor en los sujetos hipertrigliceridémicos. Este gen está implicado en procesos de endocitosis apical y reciclaje de vesículas, y recientemente también se ha relacionado con el proceso arteriosclerótico. En un artículo diseñado para identificar nuevos genes implicados en el infarto de miocardio, han identificado que RAB20 está sobreexpresado en placas arterioscleróticas carotídeas25.
Finalmente, hay que destacar que la cg21560722 se encuentra hipermetilada en los pacientes hipertrigliceridémicos y está localizada en el gen SBF2 ?SET Binding Factor 2?, que regula la expresión de varios miembros de la familia de las RAB y también regula proteínas SNARE26. Este gen, además, ha sido recientemente identificado en un estudio de asociación de genoma completo asociado a la concentración de partículas de HDL determinadas mediante resonancia magnética nuclear en la cohorte del Women's Genome Health Study27.
No hemos encontrado que las otras citosinas identificadas estén localizadas en genes o regiones que a priori ya hayan sido relacionados con el metabolismo de los TG o de los lípidos en general, ni asociados a enfermedad cardiovascular o arteriosclerosis. Aunque, a falta de validar los presentes resultados, no descartamos que estas citosinas puedan tener un papel relevante a tener en cuenta en futuros estudios.
En conclusión, estos resultados apuntan a que hay nuevas regiones genómicas a considerar cuando llevamos a cabo la caracterización genética de los sujetos que presentan hipertrigliceridemias graves, destacando los genes relacionados con la formación y maduración de los quilomicrones en los enterocitos. No obstante, debemos tener en cuenta que este estudio presenta una serie de limitaciones.
Primero de todo, estos resultados necesitan ser validados en otras cohortes más grandes para confirmar su asociación y relevancia en el control de la hipertrigliceridemia.
Otra limitación a tener en cuenta es que la determinación del grado de metilación se ha llevado a cabo en ADN extraído de células mononucleares de sangre periférica y sabemos que el perfil de metilación del ADN y la regulación de la expresión génica son específicos de tejido. Sin embargo, existen estudios que demuestran que la metilación determinada en células sanguíneas, utilizada como posible marcador en un tejido de fácil acceso, tiene cierta capacidad de reflejar los efectos epigenéticos que tienen lugar en los tejidos diana. Aun así, no debemos descartar ciertas variaciones si se llevan a cabo estudios dirigidos en los órganos diana.
También debemos tener en cuenta que no conocemos qué función pueden tener estas metilaciones identificadas. Generalmente, sabemos que, a mayor metilación de citosina, mayor represión de la expresión de un gen, pero también está descrito que la metilación del ADN en diferentes regiones genómicas puede influir en la regulación génica de diferentes formas28.
FinanciaciónEste estudio ha sido subvencionado por una beca FEA 2018 para investigación clinicoepidemiológica en arteriosclerosis, y también por el Instituto de Salud Carlos III mediante el proyecto PI19/00832 (cofinanciado por el Fondo Europeo de Desarrollo Regional/Fondo Social Europeo. «Una manera de hacer Europa»/«El FSE invierte en tu futuro»). Cerca Programme/Generalitat de Catalunya.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.