The extent to which commercially available nucleic acid extraction platforms impact the magnitude of Cytomegalovirus (CMV) DNA loads measured in plasma specimens by 1st WHO standard-normalized real-time PCR assays is uncertain.
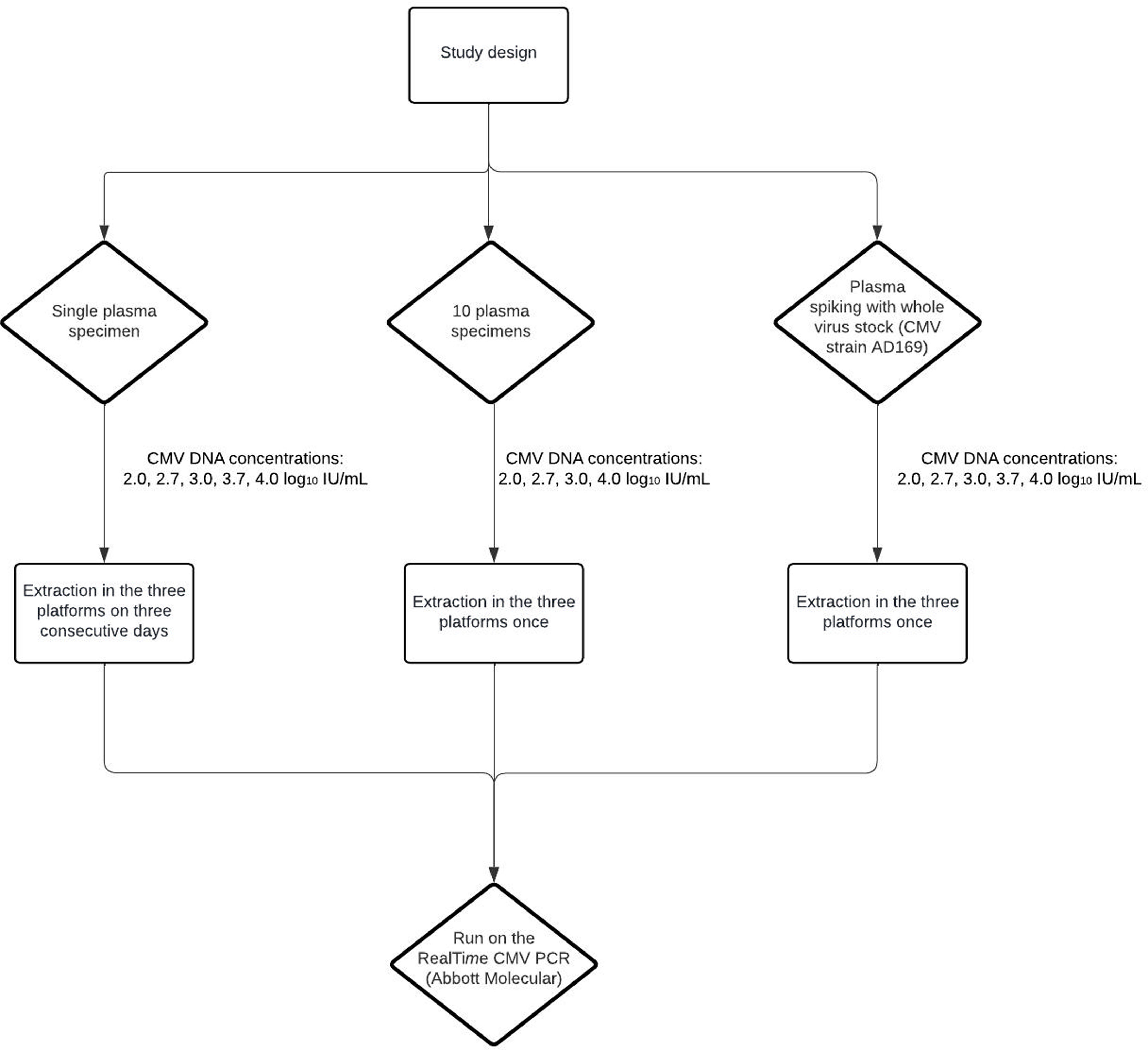
MethodsThis retrospective study compares the performance of Abbott m2000sp, Qiagen QIAsymphony SP, and KingFisher Flex platforms using plasma samples from allogeneic hematopoietic stem cell transplant recipients and plasma spiked with the CMV AD169 strain. The Abbott RealTime CMV PCR assay was used for CMV DNA quantitation.
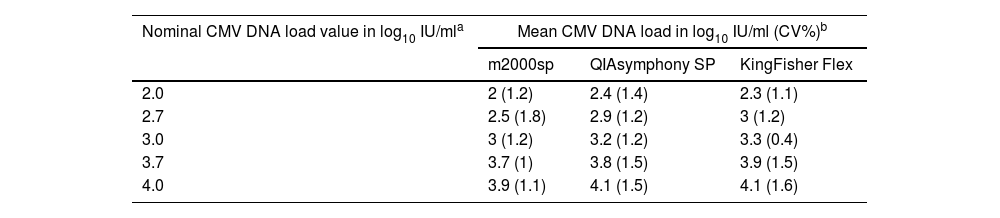
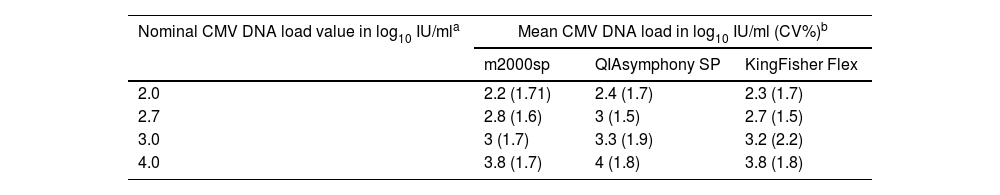
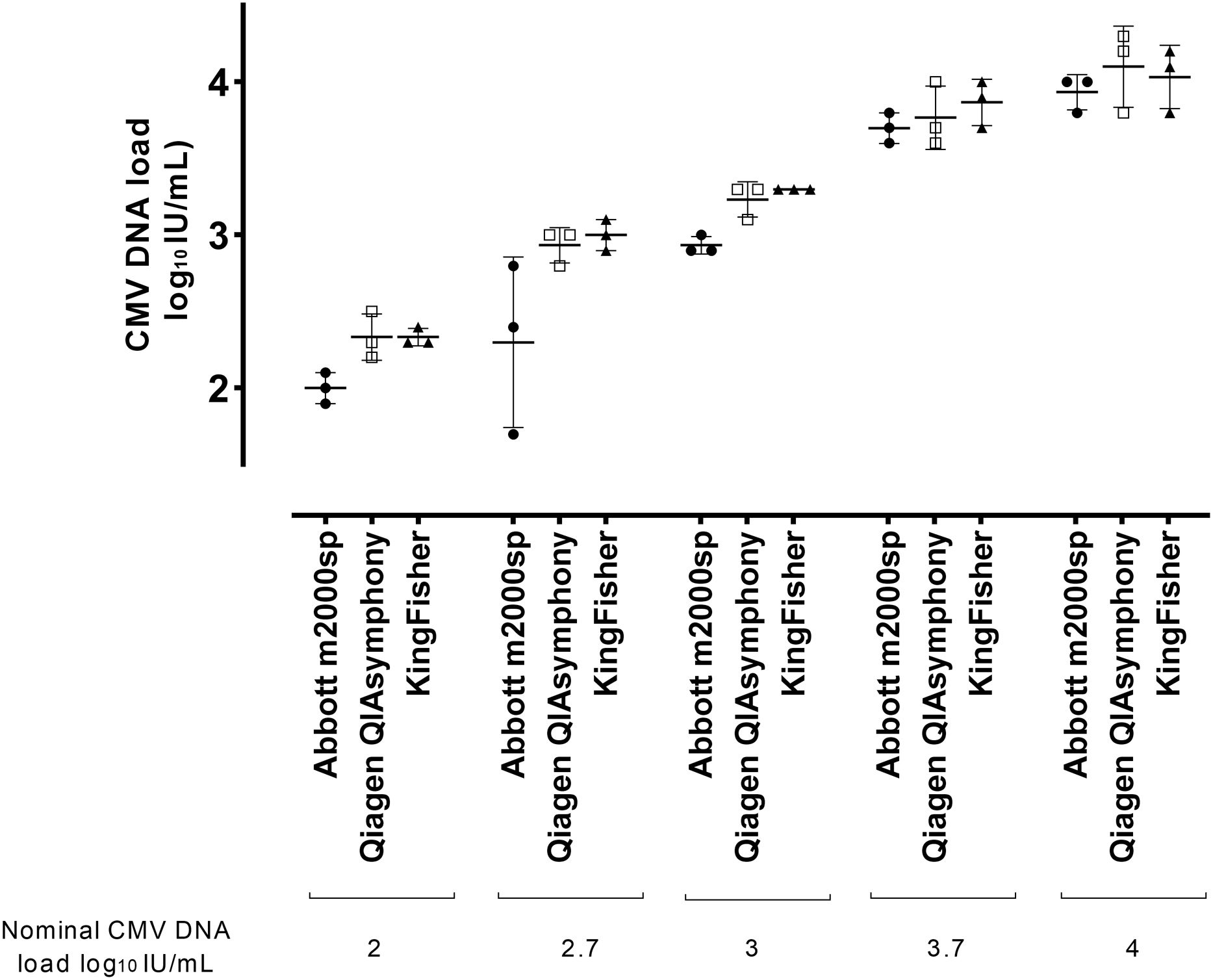
ResultsMaximum differences in CMV DNA loads quantified in plasma from 11 allo-HSCT and spiked plasma over a wide range of viral DNA concentrations (2.0–4.0 log10 IU/ml) were ≤0.5 log10 IU/ml.
ConclusionsThe CMV DNA extraction efficiency of the platforms evaluated varies. The impact of these variations on CMV DNA loads quantified in plasma may not be clinically relevant.
Se desconoce si el uso de distintas plataformas de extracción de ácidos nucleicos afecta la magnitud de las cargas de ADN de citomegalovirus (CMV) cuantificadas mediante PCR en tiempo real normalizadas al primer estándar de la OMS.
MétodosComparamos retrospectivamente las plataformas Abbott m2000sp, Qiagen QIAsymphonySP y KingFisher Flex utilizando muestras de plasma de receptores de trasplante alogénico hematopoyético (alo-TPH) y plasma inoculado con la cepa CMV AD169. Las cargas virales se cuantificaron mediante el ensayo Abbott RealTime CMV PCR.
ResultadosLas diferencias máximas en las cargas cuantificadas en plasma de 10 alo-TPH y plasma inoculado, en un rango amplio de concentraciones (2,0 a 4,0 log10 UI/ml) fueron ≤0,5 log10 UI/ml.
ConclusionesLa eficiencia de extracción de ADN de CMV de las plataformas analizadas varía; sin embargo, el impacto de estas variaciones en las cargas de ADN del CMV cuantificadas en plasma podría no ser clínicamente relevante.
Article
Socio de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica

Para acceder a la revista
Es necesario que lo haga desde la zona privada de la web de la SEIMC, clique aquí