Las infecciones respiratorias agudas son la segunda causa de morbimortalidad tanto en niños como adultos a nivel mundial en cuya etiología se implican virus, bacterias y hongos. Su diagnóstico rápido permite un mejor manejo clínico del paciente, adoptar medidas de salud pública y controlar posibles brotes. Los principales microorganismos responsables pueden diagnosticarse en las primeras horas tras el inicio del cuadro con técnicas de detección de antígeno, fundamentalmente inmunocromatografícas. Se obtienen resultados en 15–30 min, con una sensibilidad del 70–90% y especificidad superior al 95% para el diagnóstico de infecciones por Streptococcus pneumoniae y Legionella pneumophila serogrupo O1 a partir de orina, Streptococcus pyogenes en exudados faríngeos y virus respiratorio sincitial en aspirados nasofaríngeos. En infecciones por los virus de la gripe y por Pneumocystis jirovecii, los resultados con estas técnicas son peores; no obstante, existen técnicas moleculares de fácil ejecución para el diagnóstico rápido de estos microorganismos. En general, estas técnicas no deben utilizarse para control evolutivo ni para valorar respuesta al tratamiento.
Acute respiratory infections are the second cause of morbidity and mortality in children and adults worldwide, being viruses, bacteria and fungi involved in their etiology. The rapid diagnosis allows for a better clinical management of the patient, for adopting public health measures and for controlling possible outbreaks. The main etiologic agents can be diagnosed within the first hours after the onset of symptoms with antigen detection techniques, primarily immunochromatography. Results are obtained in 15–30minutes, with 70–90% sensitivity and >95% specificity for the diagnosis of Streptococcus pneumoniae and Legionella pneumophila serogroup O1 infections from urine, Streptococcus pyogenes from throat swabs and respiratory syncytial virus from nasopharyngeal aspirates. Worse results are obtained for influenza viruses and Pneumocystis jirovecii with these techniques; however, other easy-to-perform molecular techniques are available for the rapid diagnosis of these microorganisms. In general, these techniques should not be used for monitoring the outcome or response to treatment.
Las infecciones de las vías respiratorias son una de las primeras causas de morbimortalidad y uno de los principales motivos de consulta médica en todo el mundo. Como en otros procesos infecciosos, un diagnóstico rápido y preciso va a ir asociado a un tratamiento más dirigido y efectivo, una menor trasmisión de la enfermedad y en muchas ocasiones, a una disminución en la duración de la misma.
En este trabajo se ha pretendido hacer una revisión de las técnicas de diagnóstico rápido (TDR) en algunas de las infecciones respiratorias (IR) más frecuentes. Se han excluido, por ser objeto de otros capítulos, las IR de evolución crónica como tuberculosis o las que afectan a pacientes en situaciones especiales (p.ej. neumonías asociadas a ventilación mecánica).
Como TDR se han considerado aquellas cuyo resultado pueda darse en menos de 7 h (un turno habitual de trabajo) en comparación con las técnicas convencionales de cultivo bacteriano (18–24 h) o viral (48 o más h). Las técnicas serológicas de detección de anticuerpos, aun entrando en esta definición temporal, no se han incluido, dado que demostrar la seroconversión (aparición de anticuerpos en el suero) o el serorrefuerzo (aumento de 4 veces el título inicial de anticuerpos) puede tardar semanas o meses para producirse con grandes variabilidades interpersonales.
Algunas de estas TDR, dada la sencillez y rapidez de su realización, obtención e interpretación de los resultados, también podrán ser utilizadas como «point-of care test» (POCT) o «pruebas en el punto de atención» con el consiguiente beneficio para el paciente, que podría disponer del diagnóstico y en función del resultado, del tratamiento, en una misma consulta.
En general, el uso de TDR en el diagnóstico de la IR puede ayudar a:
- •
Disminuir el uso de antibióticos, dado que muchas de las IR son de etiología vírica.
- •
Utilizar terapia antivírica adecuada en casos concretos.
- •
Minimizar el uso de pruebas diagnósticas innecesarias.
- •
Disminuir el tiempo de estancia hospitalaria.
- •
Permitir, en caso necesario, una rápida implementación de medidas de aislamiento que limiten la transmisión nosocomial.
Además, en la IR de origen bacteriano, las TDR, al detectar el antígeno del patógeno diana o sus ácidos nucleicos, se ven afectadas en mucha menor medida que el cultivo en el diagnóstico en caso de que ya se haya instaurado tratamiento antibiótico.
Técnicas de diagnóstico rápido de Streptococcus del grupo A (Streptococcus pyogenes –S. pyogenes–)Las TDR de Streptococcus del grupo A (SGA) están fundamentalmente orientadas a determinar la naturaleza bacteriana de la faringitis. La faringitis es la infección más frecuente causada por los SGA, que puede acompañarse de secuelas supurativas, como abscesos peritonsilares, o no supurativas, como la fiebre reumática y la glomerulonefritis aguda, aunque hoy en día estas complicaciones son raras en la mayoría de los países industrializados. Además pueden causar con menor frecuencia infecciones graves, como la fascitis necrosante y otras infecciones como neumonías, endocarditis o meningitis.
La mayoría de las faringitis infantiles, especialmente en menores de 3 años, están causadas por virus cuya sintomatología es muy similar a las estreptocócicas. Se calcula que únicamente entre un 20–30% de las faringitis en niños y un 10% de las faringitis en adultos son debidas a SGA1 y por lo tanto un porcentaje muy bajo de ellas se beneficiará del tratamiento antibiótico. Por ello las TDR para el diagnóstico de esta enfermedad leve son unas de las más usadas y están en continua evaluación ya que ayudan a evitar un uso inadecuado de antibióticos.
La prueba gold-standard para el diagnóstico de la faringitis por SGA sigue siendo el cultivo bacteriano en agar-sangre ya sea este directo, tras enriquecimiento, o con placas selectivas con incubación de 24–48 h.
Las TDR para la detección del antígeno de SGA en frotis faríngeos aparecieron a principios de los años 1980 y desde entonces han existido varias generaciones de TDR que han utilizado diferentes metodologías. Las primeras usaban la aglutinación en látex, seguidas de los ELISA y de los test de flujo lateral e inmunocromatografías colorimétricas. Recientemente se han comercializado técnicas moleculares como sondas de DNA, PCR, e hibridación fluorescente in situ2.
Entre las TDR que actualmente se utilizan con mayor frecuencia están los test inmunocromatográficos (ICT) por su sencillez de uso y rapidez (15min) en los resultados. Existen muchos de estos test comercializados, pero todos detectan el carbohidrato C de la pared celular del SGA mediante anticuerpos específicos monoclonales o policlonales. En la mayoría de ellos hay que realizar una extracción previa del carbohidrato de la pared celular en un medio ácido capaz de solubilizarlos para poder ser detectados posteriormente en la tira inmunocromatográfica.
El mejor rendimiento de estos test se consigue en poblaciones previamente seleccionadas en base a criterios clínicos de infección faríngea3. Comparadas con el cultivo como método de referencia, los metaanálisis y estudios realizados para las TDR de SGA en faringe dan una sensibilidad media del 85% (variando del 70 al 90%) y una especificidad alrededor del 95%1,3–5. En general, las sensibilidades varían bastante entre estudios no así la especificidad, que suele ser muy elevada en todos ellos. La relativamente baja sensibilidad hace que la mayoría de los autores sigan recomendando la realización de cultivo en caso de ser el test negativo para poder así detectar un mayor número de casos6.
La gran ventaja de los test ICT es la posibilidad de realizarlos en presencia del paciente, lo que unido a la alta especificidad, hace que tras un resultado positivo no sea necesario el cultivo confirmatorio y se pueda pautar en el momento el tratamiento antibiótico específico. Salvo circunstancias especiales no está recomendado realizar un TDR o cultivo de control al finalizar este tratamiento.
A pesar de las ventajas que ofrecen los TDR conviene no olvidar que el cultivo y aislamiento del SGA va a permitir realizar posteriormente otras pruebas como los test de sensibilidad antibiótica o la caracterización genotípica de los aislamientos. Tampoco hay que olvidar que, aunque su papel aún sea controvertido, en un pequeño porcentaje de faringitis bacterianas se asilan estreptococos betahemolíticos de otros grupos (sobre todo de los grupos C y G) que no se detectan mediante los TDR para SGA ya que la composición de los hidratos de carbono de su pared celular es diferente7.
También existen comercializadas TDR moleculares de diagnóstico de la infección faríngea por SGA mediante hibridación y PCR a tiempo real. Aunque su sensibilidad y especificidad son tanto o más altas que las del cultivo y su límite de detección supera al de las técnicas de detección de antígeno8, no dejan de ser técnicas que tardan entre una y dos horas en realizarse, con la necesidad de personal cualificado y equipamientos específicos de biología molecular. Todo ello, unido a la sencillez y buen comportamiento diagnóstico de los TDR del antígeno ICT ha hecho que sean muy pocos los laboratorios clínicos que actualmente utilicen de rutina las técnicas moleculares como TDR de la faringitis por SGA.
Para las infecciones invasivas causadas por SGA también se ha comparado el comportamiento de las TDR en relación con el cultivo, siendo la sensibilidad de los test ICT de detección de antígeno semejante a la de la PCR y superior a la del cultivo8. A pesar de que no se recomienda realizar estas pruebas en presencia de tratamiento antibiótico, las TDR pueden resultar positivas en estas muestras con cultivo negativo, ya que tardan más en negativizarse.
Técnicas de diagnóstico rápido de la infección por Legionella pneumophila (L. pneumophila)Las legionelas forman un grupo de bacterias ubicuas en hábitats acuáticos que agrupan a más de 55 especies y de 70 serogrupos. La exposición a aerosoles que contienen las legionelas puede causar diferentes manifestaciones clínicas, desde la enfermedad del legionario (neumonía comunitaria grave), a la fiebre de Pontiac (síndrome febril autolimitado) o incluso a la infección asintomática. De todas ellas, la neumonía es la más importante por su frecuencia, alrededor del 2–6% de las neumonías adquiridas en la comunidad (NAC)9–11, gravedad de la enfermedad y la necesidad de un tratamiento específico con antibióticos macrólidos o fluoroquinolonas. Aunque se piensa que cualquier especie de Legionella puede causar enfermedad en el ser humano y que más de la mitad de los serogrupos se han aislado de muestras clínicas, la gran mayoría de las neumonías por Legionella están causadas por L. pneumophila serogrupo O111,12.
La dificultad y lentitud del crecimiento de las legionelas en medios de cultivo específicos y del tiempo para la seroconversión en el diagnóstico serológico han hecho que la mayoría de las técnicas diagnósticas habituales de la neumonía por L. pneumophila sean TDR. Estas se pueden clasificar entre las que utilizan anticuerpos y las técnicas de amplificación de ácidos nucleicos, fundamentalmente la PCR.
La primera TDR para la legionelosis fue la inmunofluorescencia directa (IFD) sobre muestras respiratorias utilizando anticuerpos específicos de serogrupo marcados con fluoresceína. La IFD, que puede considerarse una TDR ya que se realiza en aproximadamente una hora, presenta una sensibilidad relativamente baja (60%)13 por lo que fue desplazada por los test de detección del antígeno de Legionella en orina.
El antígeno de Legionella en orina puede detectarse desde pocos días desde el inicio de los síntomas hasta meses después de estar resuelta la infección, aunque en la mayoría de los casos se negativiza entre un mes y dos meses desde la aparición de la enfermedad. Existen diferentes formatos para la detección del antígeno de Legionella en orina: enzimoinmunoanálisis en placas de 96 pocillos (EIA), enzyme-linked immunoassay (ELISA) y los más rápidos, las ICT ya sea en formato de tarjeta o tira, también conocida como test de flujo lateral. Estas ICT y ELISA muestran sensibilidades en torno al 70–90% y especificidades del 95–100% dependiendo de los estudios11,14,15. La mayoría de los test comercializados solo detectan el antígeno urinario de L. pneumophila serogrupo O1, aunque pueden darse reacciones cruzadas con otros serogrupos. Ello implica que las neumonías causadas por otras especies de Legionella (Legionella micdadei, Legionella bozemanii, etc.) o por otros serogrupos, aunque mucho menos frecuentes, no serán normalmente diagnosticadas utilizando estos test. También existen otros test que detectan más serogrupos y especies, pero sus características de sensibilidad y especificidad son menores que para L. pneumophila serogrupo O1.
En los test de detección del antígeno urinario de Legionella, la concentración del antígeno mediante filtrado aumenta su sensibilidad. Por otro lado, hervir la orina puede aumentar la especificidad ya que reduce las interacciones no específicas. Sin embargo estas mejoras en las técnicas rápidas incrementan el tiempo y la carga de trabajo hasta la obtención del resultado. Los test ICT para la detección del antígeno urinario de L. pneumophila serogrupo O1 se consideran POCT ya que los resultados cualitativos se obtienen en min sin necesidad de personal especializado ni para la toma de muestra ni para su realización. Recientemente ha aparecido un test ICT fluorescente que aporta mayor sensibilidad a la hora de detección, eliminando además el posible sesgo atribuible a la lectura visual del resultado16. La menor especificidad observada, especialmente en orina concentrada, se mejora repitiendo los test positivos tras hervir la orina. Los EIA son algo más lentos, cuantitativos y en general utilizados en laboratorios clínicos que analizan un gran número de muestras.
En cualquier circunstancia, y a pesar de su baja rentabilidad, es importante hacer el cultivo e intentar aislar el microorganismo para posteriores estudios epidemiológicos y microbiológicos.
Entre las TDR comercializadas basadas en la ampliación de ácidos nucleicos se encuentran la amplificación isotérmica, la PCR convencional y la PCR a tiempo real en muestras respiratorias de vías bajas (esputos, aspirados o lavados broncoalveolares, …), presentando todas ellas una gran sensibilidad y especificidad. Otros beneficios de su uso son el tiempo rápido de respuesta y la generalización de su uso en la mayoría de los laboratorios de Microbiología Clínica actuales. A diferencia del cultivo, la seroconversión y la detección de antígeno en orina, una PCR positiva no se considera aún como criterio de confirmación de la infección por Legionella y debe interpretarse con prudencia.
Los genes de Legionella amplificados por la mayoría de las técnicas moleculares son los genes que codifican las subunidades de RNA ribosomal de 5S y 16S, la región espaciadora 16S-23S, y la proteína inhibidora de macrófagos (mip). Con estos genes y sondas adecuadas es posible además detectar y diferenciar entre L. pneumophila y otras especies de Legionella15,17. Los test basados en ampliación de ácidos nucleicos han demostrado mayor sensibilidad (>95%) que los test de antígeno urinario en la detección de neumonía por Legionella también con especificidades muy altas (>95%)15,17.
Técnicas de diagnóstico rápido de la infección respiratoria por Streptococcus pneumoniae (S. pneumoniae)S. pneumoniae es sin duda el patógeno que con mayor frecuencia causa NAC especialmente en las formas graves, cuando existe bacteriemia y riesgo para la vida del paciente. A diferencia de Legionella, la presencia de S. pneumoniae (neumococo) en una muestra respiratoria no es siempre indicativa de infección, ya que también es un colonizante habitual de las vías respiratorias altas lo que dificulta la interpretación de su diagnóstico.
Entre los TDR está la detección del antígeno neumocócico en orina. Estos tets detectan la presencia en la orina del polisacárido C de la pared celular del neumococo, antígeno común a los casi 100 serotipos diferentes de neumococo descritos hasta la actualidad y a otras especies relacionadas, como Streptococcus mitis y Streptococcus oralis18. Los primeros test se describieron en 1917 y se intentaron diferentes técnicas para su detección (aglutinación de látex, coaglutinación, enzimoinmunoanálisis) pero los resultados no fueron muy prometedores y se abandonaron18,19. La aparición de los test ICT con buenos resultados hizo retomar el interés en su detección20.
La sensibilidad del antígeno urinario para el diagnóstico de la neumonía neumocócica mediante ICT varía mucho según los estudios, sobre todo debido a la falta de un gold-standard que establezca de manera inequívoca la etiología de las neumonías. Estas sensibilidades varían normalmente entre el 60–75%, siendo en general más altas en estudios de neumonías neumocócicas bacteriémicas21–23. La especificidad también varía según los estudios, pero en general los resultados son más elevados y homogéneos y se sitúan entre el 95–99%21–23.
Un resultado positivo se debe interpretar con prudencia ya que puede reflejar la colonización asintomática sobre todo en niños, donde no se recomienda la realización de este test por la elevada portación nasofaríngea en edades tempranas. Además el test puede ser positivo meses después de la infección neumocócica24. Sin embargo la detección del antígeno en orina, a diferencia del cultivo, no se encuentra prácticamente influenciado por el tratamiento antibiótico previo.
Recientemente se ha utilizado otros test que detectan en orina el polisacárido capsular neumocócico en la neumonía causada por 14 serotipos (1, 3, 4, 5, 6A/C, 6B, 7F/A, 8, 9V, 14, 18, 19A, 19F and 23F) utilizando la tecnología Luminex y aunque la sensibilidad parece mayor que la de la detección del polisacárido C aún no se dispone comercialmente25. Este test demostró solo un 51% de correspondencia con los test de detección del polisacárido C y parece una alternativa más sensible cuando se incorporen más serotipos a la detección.
Otros TDR incluyen la detección del ADN de S. pneumoniae mediante PCR. La elevada sensibilidad de estas técnicas junto con la posibilidad de colonización por el patógeno hace que sea muy difícil la interpretación de un resultado positivo. Entre los genes utilizados en la detección de S. pneumoniae destacan el gen de la autolisina (lytA), el de la neumolisina (ply) y el Spn9802. Se ha intentado cuantificar mediante PCR a tiempo real la carga de S. pneumoniae en muestras respiratorias para diferenciar entre colonización e infección; sin embargo no hay criterios estándar establecidos para poder diferenciar entre ambas situaciones clínicas.
Además del diagnóstico de la neumonía, los TDR de neumococo también se han utilizado para la detección del patógeno en muestras estériles como el LCR en las meningitis26,27 o el líquido pleural en derrames paraneumónicos28. La detección del antígeno neumocócico mediante ICT en estas muestras estériles ha demostrado ser más sensible que el cultivo, sobre todo porque puede ser detectado aun en presencia de tratamiento antibiótico. Por otro lado la detección del neumococo mediante PCR en muestras estériles se ha utilizado con éxito dado que su presencia en estas muestras, a diferencia de las respiratorias, es diagnóstica de la infección.
Conviene recordar, que a pesar de las ventajas que suponen las TDR en el diagnóstico de las infecciones neumocócicas, estas no deben sustituir sino complementar al hemocultivo y al cultivo de esputo, que van a permitir aislar al microrganismo causal, conocer su susceptibilidad antibiótica además de otras características microbiológicas (serotipos, genotipos, etc.) que son de gran valor en el manejo y control de la infección29.
Técnicas de diagnóstico rápido de las infecciones respiratorias viralesLa IR aguda de etiología vírica constituye, en su conjunto, uno de los síndromes clínicos infecciosos que más afecta a la especie humana. En niños, más del 50% de las NAC, hasta el 90% de los casos de bronquiolitis y aproximadamente el 85% de las exacerbaciones asmáticas se asocian a infecciones por virus respiratorios (VR), mientras que en adultos los VR se han detectado en el 20–40% de las NAC, del 50–70% de las exacerbaciones asmáticas y entre el 30–50% de las exacerbaciones de EPOC30,31.
Hasta una veintena de virus se han implicado como agentes etiológicos de estos procesos. Los más importantes por su relevancia clínico-epidemiológica son los virus de la gripe A y B y virus respiratorio sincitial (VRS), y en menor medida: adenovirus (AD), rhinovirus (RVh), methapneumovirus y virus parainfluenza 1 a 4 y otros como enterovirus, coronavirus, bocavirus, etc.
La mayoría de las IR agudas víricas son autolimitadas y evolucionan hacia la curación sin tratamiento etiológico alguno. Solo ocasionalmente (si exceptuamos algunas virosis emergentes como coronavirus MERS o SARS o nuevas gripes) y fundamentalmente en pacientes en edades extremas de la vida o con patologías de base que favorecen las complicaciones, asistimos a cuadros clínicos que por su gravedad requieran una atención sanitaria especial. Actualmente se utilizan antivíricos específicos en infección por virus de la gripe y en situaciones clínicas concretas; para otros, como VRS, RVh o AD, aunque algunos compuestos, como ribavirina frente a VRS y AD y pleconaril para RVh han mostrado cierta actividad, su uso no está suficientemente establecido.
Como consecuencia de las IR agudas, cada año, coincidiendo con las epidemias estacionales de gripe y VRS, se produce un exceso de demanda asistencial, que se traduce en mayor frecuentación y colapso de las Urgencias, un aumento de consumo en antibióticos y una mayor generación de pruebas analíticas y de imagen.
Partiendo de muestras del tracto respiratorio, fundamentalmente aspirado o lavado nasofaríngeo, y en menor medida muestras tomadas con escobillón nasal y/o faríngeo en medio de transporte de virus, actualmente contamos con numerosos procedimientos que pueden ayudar al diagnóstico etiológico rápido de las principales IR agudas víricas.
Las diferentes técnicas podemos agruparlas fundamentalmente en dos grupos: técnicas de detección de antígeno y técnicas de amplificación de ácidos nucleicos (TAAN).
Técnicas de detección de antígenos víricosEntre ellas, se utilizan fundamentalmente las inmunofluorescencias (IF) directa e indirecta y las ICT. Otras, como las técnicas inmunoenzimáticas de membrana, muy usadas en el pasado, han quedado en un segundo plano.
Por IF se pueden detectar los principales VR. No obstante su correcta realización conlleva cierta dificultad. La necesidad de una citocentrífuga para hacer las extensiones, el fijar y teñirlas adecuadamente, el precisar la utilización de microscopio de fluorescencia para su visualización, la subjetividad en la interpretación del resultado (pericia del observador), ser muy dependiente de la calidad de la muestra (presencia de células epiteliales suficientes), y el tiempo en la obtención de resultados (entre 90–120 min) hace que esta técnica vaya siendo progresivamente menos utilizada para el diagnóstico inicial de una IR vírica en la mayoría de los laboratorios clínicos.
El uso de las técnicas clásicas de IF se puede mejorar y facilitar automatizando el proceso y realizando una lectura rápida y objetiva de la reacción. En este sentido se han desarrollado técnicas para detectar virus concretos de forma individual32 y otras que aumentan el panel de virus a detectar, que aparte de VRS y gripe, detecta AD, virus parainfluenza 1 a 4, bocavirus, methapneumovirus y bacterias como neumococo. Sus posibles ventajas, utilidades y relación coste-eficiencia se han descrito recientemente, existiendo aún muchas dudas al respecto33.
Dentro de los test ICT, los que revelan la reacción con oro coloidal son en la actualidad los más empleados. Su fácil realización (no requiere habilidades técnicas especiales) y rápida ejecución (se obtienen resultados entre 10 y 20 min) y la sencilla lectura de los resultados contribuyen a difundir su uso como procedimientos a utilizar de forma rápida a la cabecera del paciente y en áreas de Urgencias como POCT. Son menos sensibles que la IF y a diferencia de esta, que permite detectar un amplio abanico de VR, la mayoría de los dispositivos de ICT existentes están diseñados para detectar VRS y/o gripe A y B.
Con ICT se observa gran variabilidad en los resultados según el fabricante, la muestra que se utilice (se observan mucho mejores resultados de sensibilidad con aspirados/lavados nasofaríngeos que con muestras tomadas con escobillón), el tiempo de evolución de la enfermedad cuando se realiza la toma (mejor en las primeras 24 h) y sobre todo, en base a la población en la que se aplique (mucho peores resultados en población adulta que en pediátrica)34–36. En general, la sensibilidad de las técnicas ICT es mejor cuando se utilizan para detectar VRS que cuando se usan en el diagnóstico de gripe.
Técnicas de amplificación de ácidos nucleicosLas técnicas moleculares, fundamentalmente la PCR, son altamente sensibles y específicas y han sustituido al cultivo celular como método de referencia en el diagnóstico de las infecciones por VR. Además permiten detectar virus no viables o que no se han conseguido aislar en los cultivos celulares tradicionales. En general tienen un tiempo de respuesta superior a 24 h y requieren infraestructuras y equipamientos específicos y personal especializado, lo que limita su uso para diagnóstico rápido o en situaciones de urgencia37. No obstante, en los últimos años se vienen desarrollando TAAN en diferentes formatos de fácil y rápida ejecución, sin pérdida de eficiencia respecto a los procedimientos moleculares clásicos. Con una mínima manipulación de la muestra y tras dispensarla en dispositivos individuales, realizan en un mismo equipo la extracción, amplificación y detección de los VR38–40.
Como sucede con las técnicas de detección de antígenos víricos (TDA), se han desarrollado comercialmente desde pruebas diseñadas para detectar un virus concreto (VRS y/o gripe) hasta «multiplex» capaces de detectar simultáneamente la mayoría de los posibles virus implicados en estos procesos. Todo ello en un tiempo en la obtención de resultados que varía entre 15 y 90 min, con mínima laboriosidad (alrededor de 2 min) lo que permite actualmente utilizarlas como pruebas POCT, con resultados mucho más fiables que los obtenidos con las TDA31,35,41. En general estos dispositivos permiten procesar un número limitado de muestras a la vez, entre 1 y 8 según la plataforma. Otros sistemas utilizando tecnología Luminex permiten procesar hasta 96 muestras y detectar simultáneamente 22 patógenos diferentes (19 virus y 3 bacterias), obteniendo resultados en 4-5 h; pero la necesidad de utilizar tres equipos distintos en el procesamiento dificulta su uso como técnica rápida.
Utilidad de las técnicas rápidas en el diagnóstico de infección respiratoria aguda de etiología viralEn el caso de las IR agudas virales, podemos encontrar desde pacientes atendidos ambulatoriamente que requieran un diagnóstico rápido de un determinado agente, como ha sucedido con la gripe pandémica en embarazadas u obesos mórbidos, a pacientes hospitalizados con graves enfermedades de base (p.ej. oncohematológicos), en los que es importante descartar cualquiera de los posibles virus implicados. Aparte de las situaciones particulares comentadas anteriormente o en brotes en instituciones cerradas, en la actualidad existe bastante consenso en no utilizar estas pruebas fuera del ámbito hospitalario.
En las áreas de urgencias es una práctica muy extendida el utilizar TDA, sobre todo para VRS y gripe, pero ello no ha demostrado que sirva para disminuir la prescripción de antibióticos o una menor realización de pruebas de diagnóstico complementarias (analíticas o de imagen). Incluso en el caso de VRS puede llevar a una falsa sensación de seguridad a la hora de distribuir los pacientes y establecer medidas de aislamiento, con resultados a veces contraproducentes. El uso de estas técnicas en urgencias solo se ha podido asociar a un menor consumo de antibióticos, pero una vez que los pacientes vuelven a ser atendidos ambulatoriamente42.
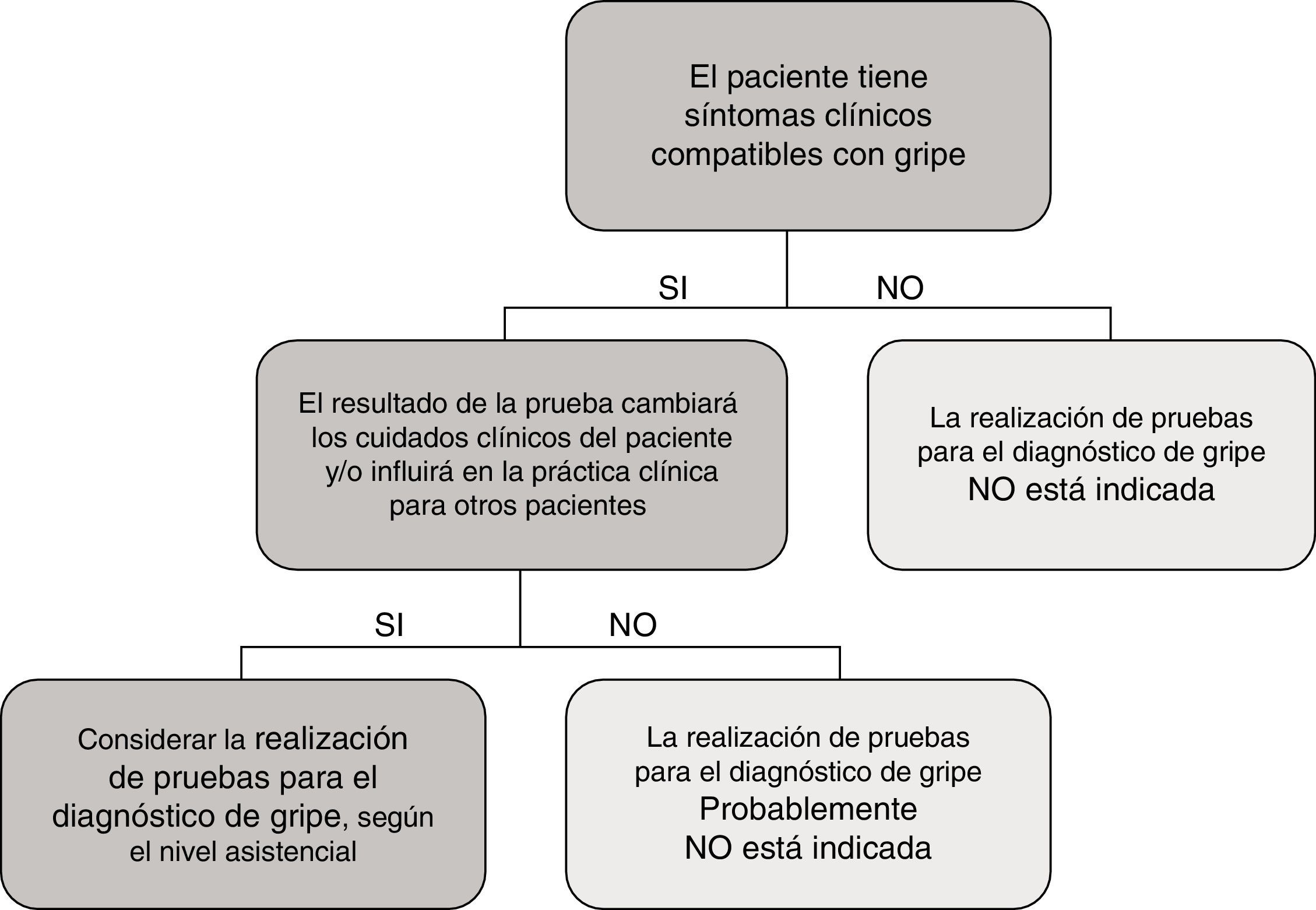
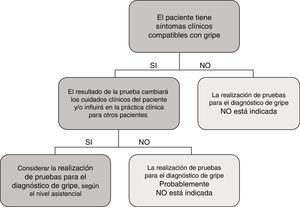
En el caso particular de las TDA de virus de la gripe, tienen en general, unos resultados de sensibilidad y un valor predictivo negativo muy deficientes, por lo que su uso en el área de urgencias, fuera de casos particulares de alto riesgo y si el paciente no requiere ingreso, está en entredicho. En la figura 1 se muestra una propuesta de algoritmo para utilización de pruebas diagnósticas de gripe durante periodos epidémicos. En general, si se considera necesario descartar virus gripal por la situación clínica del paciente, para inicio precoz del tratamiento o para implementar medidas de prevención de brotes nosocomiales de gripe, es mejor realizar directamente una TAAN, al igual que si el paciente requiere ingreso hospitalario43. Aunque la realización de pruebas para diagnóstico de gripe se recomienda en pacientes hospitalizados con clínica compatible, el tratamiento antiviral empírico, en estos casos, debe iniciarse tan pronto como sea posible, sin necesidad de esperar al resultado de las pruebas diagnósticas.
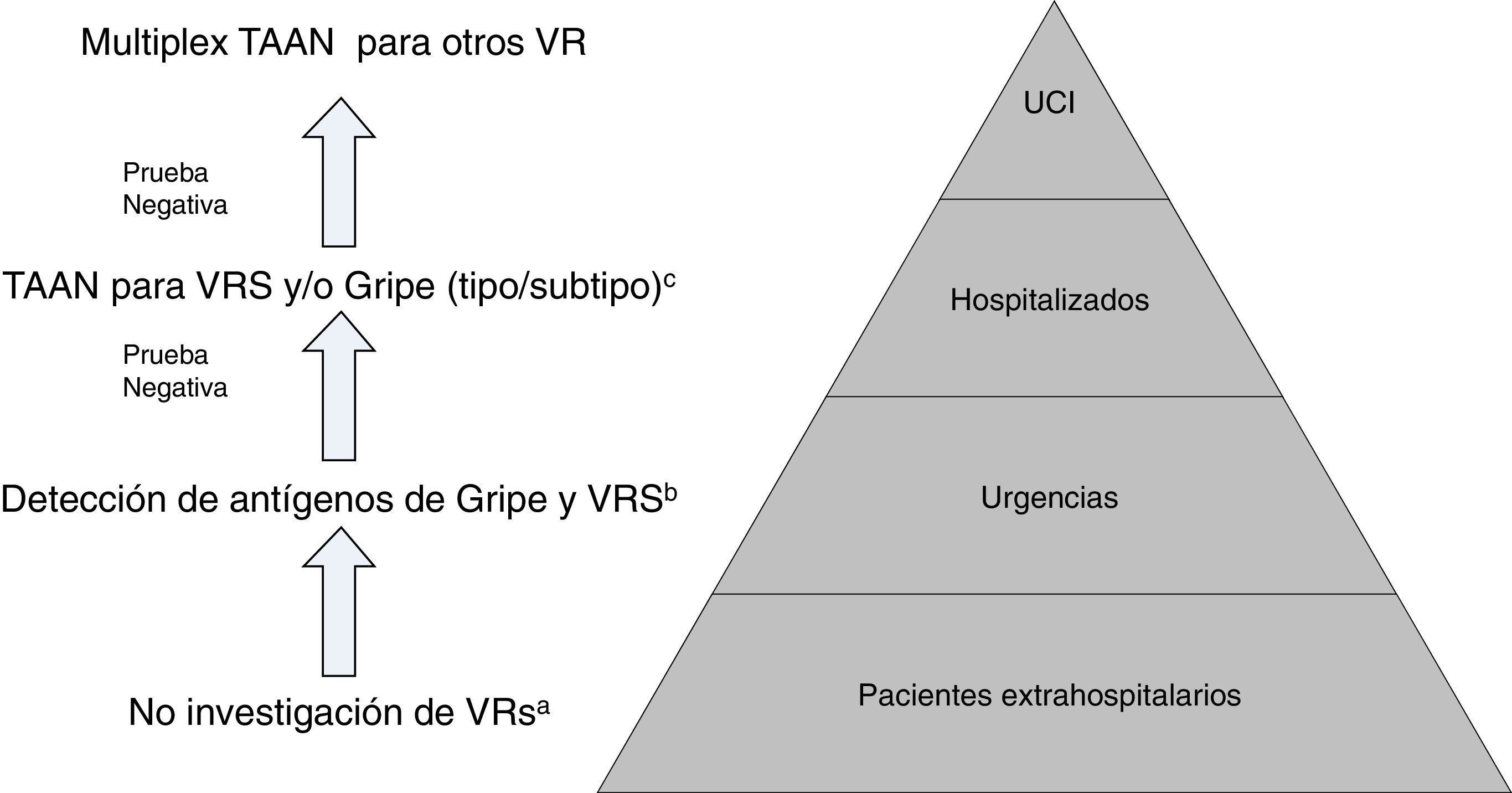
Propuesta de algoritmo de flujo de trabajo racional y costo-efectivo para la investigación de virus respiratorios (VR).
a Excepto para pacientes incluidos en programas de vigilancia (p.ej. Red Nacional de Vigilancia de Gripe).
b Excepto situaciones especiales: embarazo, enfermedad de base predisponente, etc.; en los que se hará directamente TAAN.
c TAAN para otros VR (rinovirus, metapneumovirus…), pueden ser añadidos en pacientes con alto riesgo de enfermedad respiratoria grave.
Modificada de Navarro-Marí et al.45.
Las TAAN, por su alta sensibilidad, pueden detectar excreción asintomática o cargas virales muy bajas, de dudosa significación clínica. Además, no existen datos que avalen la utilización de las TAAN para control evolutivo o del tratamiento de los pacientes44. Por otro lado, las TAAN multiplex para el conjunto de VR, que pueden utilizarse como POCT, si bien tienen una gran sensibilidad y especificidad, también tienen un alto coste que limita su uso rutinario estando normalmente reservadas para situaciones muy particulares, como pacientes graves ingresados en UCI43.
En conjunto, atendiendo al medio en que nos encontremos, situación epidemiológica y la gravedad del cuadro clínico, se hace necesario establecer algoritmos diagnósticos, que permitan un uso racional y eficiente de las diferentes técnicas diagnósticas para VR (fig. 2)45,46.
Diagnóstico rápido de la infección por Pneumocystis jirovecii (P. jirovecii)P. jirovecii es un hongo patógeno que se reproduce extracelularmente en los alvéolos pulmonares de los mamíferos sin causar enfermedad mientras el sistema inmune esté inalterado. Sin embargo, en situaciones de inmunodepresión Pneumocystis es capaz de proliferar y causar neumonía grave en ausencia de tratamiento específico. Por ello, y al igual que otros patógenos colonizantes, la detección de P. jirovecii en una muestra respiratoria es en ocasiones difícil de interpretar, ya que su detección no implica que sea necesariamente el agente responsable de la enfermedad.
Pneumocystis spp. tiene un ciclo de vida bifásico, con dos formas morfológicas muy diferenciadas: una forma trófica asexual representada por trofozoitos haploides, que constituye el estado proliferativo y que es bastante abundante y una forma quística que representa el estado reproductivo, menos abundante que las formas tróficas y que es la forma de transmisión.
La infección por P. jirovecii es difícil de diagnosticar dado que es un hongo no cultivable. Por ello, las técnicas de diagnóstico existentes las podemos clasificar como rápidas, ya que se realizan en minutos o en unas pocas horas y se dividen básicamente entre las tinciones y las técnicas moleculares, fundamentalmente la PCR, ya sea convencional o a tiempo real.
Un aspecto importante en el diagnóstico de la infección por Pneumocystis es la muestra empleada, que debe conjugarse con la sensibilidad de la técnica empleada. Dado que el Pneumocystis vive en las células alveolares, es de esperar que la carga del patógeno sea mayor en muestras recogidas de las cavidades alveolares como el lavado broncoalveolar (LBA) o el aspirado broncoalveolar, que en otras muestras como los esputos (ya sean inducidos o expectorados) y el cepillado bronquial, o incluso en muestras de vías respiratorias altas, como lavados bucales, aspirados nasofaríngeos y frotis nasales47.
Las técnicas de microscopia son en general menos sensibles que las PCR, por ello es recomendable realizarlas siempre en LBA o aspirado broncoalveolar. Estas muestras además se centrifugan antes de realizarse las tinciones para concentrar el Pneumocystis. Otras muestras respiratorias en las que se puede detectar son el fluido de aspirado traqueal, material celular obtenido por cepillado bronquial y tejido obtenido por biopsia transtraqueal.
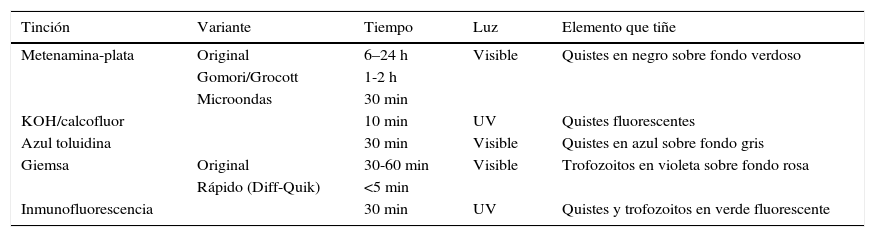
Entre las tinciones48–50, las que clásicamente se han utilizado en el diagnóstico de la infección por Pneumocystis se describen en la tabla 1. Mediante la tinción de metenamina-plata, que también tiñe otros hongos, se pueden visualizar los quistes (ascos) del Pneumocystis en negro frente a un fondo verdoso. Su mayor inconveniente es la duración de la tinción, ya que su realización puede durar entre 6 y 24 h en la tinción normal, 1-2 h en la forma abreviada de Gomori/Grocott o reducirse hasta los 30 min si se realiza en horno microondas. La tinción de KOH/calcofluor o blanco calcofluor se realiza en unos 10 min y bajo luz fluorescente se observan los hongos de azul fluorescente. La tinción modificada de azul toluidina tiñe de violeta los quistes del Pneumocystis en unos 30min.
Tinciones más comunes para el diagnóstico de la neumonía por Pneumocystis jirovecii en muestras respiratorias
| Tinción | Variante | Tiempo | Luz | Elemento que tiñe |
|---|---|---|---|---|
| Metenamina-plata | Original | 6–24 h | Visible | Quistes en negro sobre fondo verdoso |
| Gomori/Grocott | 1-2 h | |||
| Microondas | 30 min | |||
| KOH/calcofluor | 10 min | UV | Quistes fluorescentes | |
| Azul toluidina | 30 min | Visible | Quistes en azul sobre fondo gris | |
| Giemsa | Original | 30-60 min | Visible | Trofozoitos en violeta sobre fondo rosa |
| Rápido (Diff-Quik) | <5 min | |||
| Inmunofluorescencia | 30 min | UV | Quistes y trofozoitos en verde fluorescente |
Para la observación de las formas tróficas se utiliza habitualmente la tinción de Giemsa de unos 30 a 60 min o las variantes de Giemsa rápido (Diff-Quik), que se realizan en menos de 5 min. Al haber en general unas 10 veces más formas tróficas que quistes en las muestras, la sensibilidad de esta tinción puede ser mayor que las que solo tiñen los quistes.
El Pneumocystis también puede detectarse en unos 30min mediante IFD, utilizando anticuerpos monoclonales dirigidos frente a las glicoproteínas de superficie. En función del anticuerpo monoclonal elegido se pueden teñir específicamente los quistes o todas las formas del microorganismo. Dado que hay más formas tróficas, la sensibilidad de los kits que tiñen ambas formas es mayor. Los anticuerpos suelen ir marcados con isotiocianato de fluoresceína, que hace que bajo la luz ultravioleta del microscopio la pared de los quistes y los trofozoitos se observe de verde fluorescente. De las técnicas de microscopia, la más sensible es la IFD siendo la especificidad semejante entre todas las técnicas47,51,52.
Dentro de las técnicas moleculares, la más empleada es la PCR, que se considera unas 100 veces más sensible que la microscopia en el diagnóstico de neumonía por P. jirovecci53. La sensibilidad de la PCR puede verse aumentada si se utilizan dianas presentes en múltiples copias en el genoma del Pneumocystis como el gen codificante de la subunidad 23S del rRNA mitocondrial (mitochondrial large subunit rRNA) o el mayor surface glycoprotein o bien mediante la realización de una PCR anidada. Otras dianas empleadas en la detección de Pneumocystis han sido internal transcribed spacer de los genes ribosomales, 5S rRNA citoplasmático, dihidrofolato reductasa, timilidato sintetasa,…54,55.
La elevada sensibilidad de las PCR, capaces de detectar cantidades menores de 3,5 copias/μl de muestra, hace que una prueba negativa de PCR en muestra pulmonar (LBA, biopsia) sirva para descartar la implicación de P. jirovecci en la infección56.
Por otro lado, la elevada sensibilidad de la PCR hace que algunos autores la propongan como técnica diagnóstica en muestras respiratorias en las que es esperable una menor cantidad de Pneumocystis: muestras de pacientes inmunodeprimidos no infectados por el VIH o muestras respiratorias no invasivas (esputo inducido, frotis nasales,…) ya que un resultado negativo evitaría realizar técnicas invasivas. El problema es que en estas muestras no invasivas, un resultado positivo podría estar detectando una colonización subclínica en vez de una infección aguda. Se ha sugerido la realización conjunta de PCR y microscopia en esputo inducido o en muestras de vías respiratorias altas, de manera que una PCR positiva con microscopia positiva sería signo de infección, mientras que una PCR positiva con microscopia negativa representaría una colonización. Un resultado de PCR negativa excluiría la infección y la colonización por Pneumocystis57.
El inconveniente de la PCR convencional y de la PCR anidada es la falta de cuantificación, cuya determinación sería un reflejo de la carga fúngica presente y podría por ello ayudar a diferenciar entre colonización e infección aguda. Este problema se puede obviar con la PCR cuantitativa a tiempo real; sin embargo aún no se han establecido unos puntos de corte claros que distingan entre colonización y enfermedad. En general un cicle threshold o ciclo de inicio de la fase exponencial de la amplificación elevado representa una carga fúngica baja y posiblemente orienta más hacia una colonización que a una infección57. En estas situaciones de PCR positiva se pueden añadir test serológicos (como la detección en suero del 1–3 beta-D-glucano), que aun sin ser específicos de Pneumocystis aumenta en las infecciones fúngicas y pueden servir para complementar el resulto positivo de una PCR en una muestra respiratoria no invasiva (esputo o incluso saliva)58. El valor predictivo negativo del beta-D-glucano es muy elevado y puede utilizarse en pacientes a los que no se les puede realizar una broncoscopia o que tienen una sospecha muy baja de infección por Pneumocystis. La reducción en la carga del DNA de Pneumocystis mediante PCR a tiempo real también se ha utilizado como control de respuesta al tratamiento.
Tampoco es infrecuente que un paciente esté infectado con más de un genotipo concreto de P. jirovecci, lo que puede dificultar la detección de un genotipo en particular o la cuantificación en PCR cuantitativa.
Las técnicas moleculares ofrecen además otras «posibilidades» al estudio de la infección por Pneumocystis, entre ellas estudios epidemiológicos y de transmisión persona-persona mediante el «multilocus genotyping», incluso una aproximación a la resistencia a trimetoprim-sulfametoxazol analizando mutaciones en los genes codificantes de la dihidropteroato sintetasa y la dihidrofolato reductasa59.
Los test serológicos para detectar anticuerpos frente a P. jirovecci son útiles para realizar estudios epidemiológicos, pero no en el diagnóstico de la infección aguda.
ConclusiónEn general los TDR son útiles para el diagnóstico de muchas de las IR permitiendo un mejor abordaje de la infección en todos sus aspectos. Actualmente, los TDR de detección de antígenos pueden emplearse como POCT, no así la mayoría de las técnicas de amplificación de ácidos nucleicos que a su vez presentan mejores porcentajes de especificidad pero sobre todo de sensibilidad. Las mejoras en cuanto a la sencillez en el procesamiento de la muestra y en la interpretación de los resultados hacen prever que en el futuro cercano se extienda el uso de las técnicas de amplificación de ácidos nucleicos como POCT para el diagnóstico de la IR.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.