Situated in West Central Morocco, the Doukkala region carries the distinction of being one of North Africa's oldest human settlement zones. Nonetheless, it has been notably understudied in the realm of population genetics. Through allele frequency analysis and integration of forensic parameters, the research aims to gain insights into the genetic structure and neighboring affiliations of the Doukkala population.
MethodsThis study employed the AmpFlSTR Identifiler PCR system to assess the allelic frequencies and forensic parameters of 15 autosomal STRs in a cohort of 134 unrelated, healthy individuals from the Doukkala region who identify as Arab-speakers. Additionally, we explored the genetic relationships between the Doukkala population and other reference groups, considering both our dataset and previously published population data.
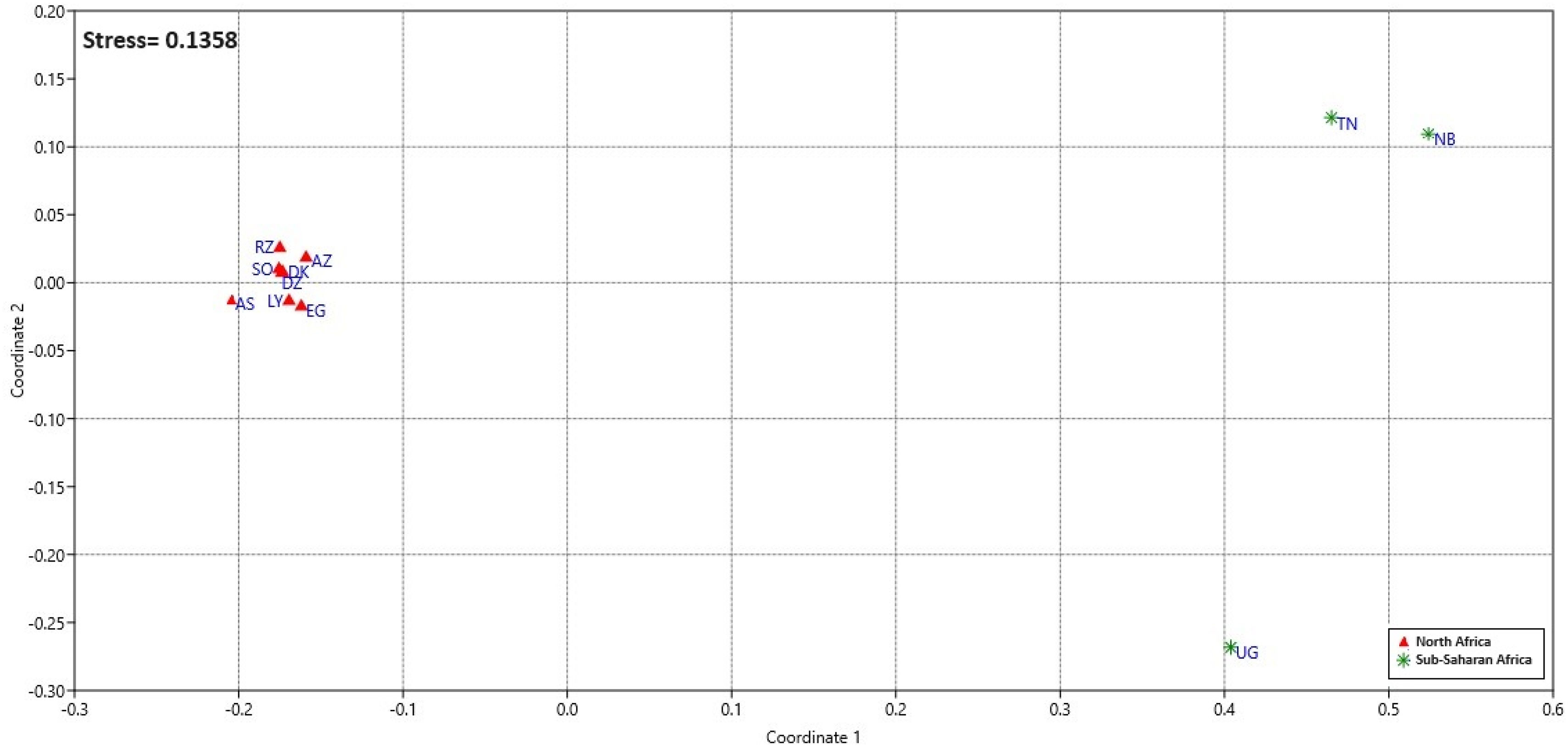
ResultsA total of 180 alleles were observed in the study population. With a count of 19 alleles, D18S51 proved to be the most diverse marker in the study. After Bonferroni's correction, 3 loci (FGA, TH01, and TPOX) deviated from Hardy–Weinberg equilibrium. The combined power of discrimination (PD) was 0.99999999999999999526 and the combined probability of exclusion (PE) was 0.99999664790900144592. The Arabic-speaking population of Rabat-Salé–Zemmour-Zaer and Southern population from Morocco exhibit the shortest genetic distance from population of Doukkala. No significant difference was observed between the Arabic-speaking population of Doukkala and all North African populations at all loci, except for the Egyptian population (North-East Africa), where a difference was observed at 2 specific loci (CSF1PO and TH01).
ConclusionsThese results indicate that the diversity found in populations from North Africa transcends geographic and linguistic barriers. The dataset's relevance to this research could stem from its capacity to offer valuable reference data for forensic, anthropological, and genetic investigations.
Situada en el centro-oeste de Marruecos, la región de Doukkala se distingue por ser una de las zonas de asentamiento humano más antiguas del norte de África. Sin embargo, ha sido muy poco estudiada en el ámbito de la genética de poblaciones. Mediante el análisis de las frecuencias alélicas y la integración de parámetros forenses, la investigación pretende profundizar en la estructura genética y las afiliaciones vecinales de la población de Doukkala.
MétodosEn este estudio se empleó el sistema de PCR AmpFlSTR Identifiler para evaluar las frecuencias alélicas y los parámetros forenses de 15 STR autosómicos en una cohorte de 134 individuos no emparentados y sanos de la región de Doukkala que se identifican como hablantes de árabe. Además, exploramos las relaciones genéticas entre la población de Doukkala y otros grupos de referencia, considerando tanto nuestro conjunto de datos como los datos poblacionales publicados previamente.
ResultadosSe observó un total de 180 alelos en la población estudiada. Con un recuento de 19 alelos, D18S51 resultó ser el marcador más diverso del estudio. Tras la corrección de Bonferroni, tres loci (FGA, TH01 y TPOX) se desviaron del equilibrio de Hardy–Weinberg. El poder de discriminación (PD) combinado fue de 0,99999999999999999526 y la probabilidad de exclusión (PE) combinada fue de 0,99999664790900144592. La población arabófona de Rabat-Salé–Zemmour-Zaer y la población meridional de Marruecos presentan la menor distancia genética con la población de Doukkala. No se observaron diferencias significativas entre la población arabófona de Doukkala y todas las poblaciones norteafricanas en todos los loci, excepto en el caso de la población egipcia (África nororiental), donde se observó una diferencia en dos loci específicos (CSF1PO y TH01).
ConclusionesEstos resultados indican que la diversidad encontrada en las poblaciones del norte de África trasciende las barreras geográficas y lingüísticas. La relevancia del conjunto de datos para esta investigación podría derivarse de su capacidad para ofrecer valiosos datos de referencia para investigaciones forenses, antropológicas y genéticas.