Estudar a patogenicidade de isolados de Escherichia coli (E. coli) nas infeções urinárias e sua relação com o hospedeiro, tendo em conta a associação de bacteriúria por E. coli a critérios definidos de patogenicidade ou a condições específicas inerentes ao hospedeiro, em mulheres com idades até 59 anos.
Materiais e métodosForam estudadas 228 estirpes de E. coli, isoladas de urinas de mulheres com idade ≤59 anos, provenientes de vários laboratórios portugueses de prestação de serviços à comunidade. A pesquisa dos genes de virulência fimH, papC, ecpA, usp, hlyA e cnf1, das ilhas de patogenicidade (PAI) ICFT073 e PAI IICFT073, e a determinação do grupo filogenético foi efetuada pela técnica de PCR.
ResultadosOs genes mais frequentemente isolados foram o ecpA, seguido de fimH, e os grupos filogenéticos mais prevalentes o B2 e D patogénicos, tanto em cITU como em ncITU.
Tanto nos isolados comensais como os patogénicos apresentaram os genes fimH, papC e ecpA de aderência e colonização; contudo, os genes usp, hlyA e cnf1, associados a processos invasivos, assim como as PAI ICTF073 e PAI IICFT073 encontraram‐se predominantemente nos isolados dos grupos patogénicos.
ConclusãoOs fatores de virulência estudados não ficaram restritos às estirpes patogénicas, tanto em ncITU como em cITU. Os fatores de risco do hospedeiro que propiciam o estabelecimento de cistites como a gravidez, recorrência e diabetes, estão associados à patogenicidade das bactérias.
To study Escherichia coli pathogenicity in urinary tract infections and their relationship with the host. Association of bacteriuria by E. coli with defined pathogenicity criteria or host specific conditions, in women aged up to 59 years.
Materials and methodsOverall 228 Escherichia coli strains were studied. These were isolated from urine of women with age ≤59 provided by various Portuguese laboratories in the community. The study of the virulence genes fimH, papC, ecpA, usp, hlyA and cnf1, pathogenicity islands (PAI) ICFT073 and PAI IICFT073, and phylogenetic group determination was performed by PCR.
ResultsThe most frequently isolated genes were ecpA, followed by fimH and the most prevalent phylogenetic groups were pathogenics B2 and D, both in cITU and ncITU.
Both commensal and pathogenic isolates showed presence of fimH, papC and ecpA genes with functions in adhesion and colonization, while usp, hlyA and cnf1 usually associated with invasive strains and PAI ICTF073 and PAI IICFT073 were found predominantly in pathogenic group strains.
ConclusionStudied virulence factors were not restricted to pathogenic strains, both in ncITU and cITU. The host risk factors which propitiate cystitis such as pregnancy, recurrence and diabetes, are associated with bacterial pathogenicity.
A Escherichia coli (E. coli) é o agente etiológico mais frequentemente isolado nas infeções do trato urinário (ITU), sendo também predominante na flora intestinal humana1. Os fatores que predispõem à infeção urinária podem ser inerentes ao hospedeiro ou específicos do microrganismo. A cistite ou infeção urinária baixa pode caracterizar‐se como complicada ou não complicada, consoante o hospedeiro apresente ou não fatores de risco, tais como internamento recente em hospital, patologias obstrutivas ou uso de cateter urinário2–6. A E. coli uropatogénica (UPEC) expressa fatores de virulência tais como adesinas, toxinas, sideróforos e proteínas de superfície responsáveis pela colonização, invasão e persistência no trato urinário1. Entre as adesinas mais frequentemente associadas à UPEC estão as fímbrias do tipo 1 e as fímbrias do tipo P ou Pyelonephritis associated pili (Pap)7,8. A adesina FimH, representativa das fímbrias do tipo 1, liga‐se especificamente à uroplaquina Ia, uma glicoproteína expressa pelas células vesicais tida como importante na formação de biofilme9,10. As Pap estão associadas a pielonefrite aguda não complicada11, encontrando‐se o recetor específico disseminado nas membranas celulares das células renais4, sendo essenciais para a colonização do trato urinário alto1. Outras adesinas têm sido descritas em estirpes de E. coli enteropatogénicas e comensais, tal como a denominada de E. coli common pilus ou ECP, codificada pelo gene ecpA, responsável pela colonização, disseminação e infeção do trato gastrointestinal12,13, como também pela formação de biofilmes em superfícies bióticas e abióticas.
Entre as proteínas excretadas pelas estirpes UPEC, a proteína uropathogenic‐specific protein (USP), codificada pelo gene usp, foi descrita recentemente como prevalente nas estirpes urinárias relativamente às estirpes fecais1,14–16. Outra proteína importante, a citotoxina ou factor necrosante citotóxico CNF1 (cytotoxic necrotizing factor 1), tem a capacidade de induzir apoptose nas células epiteliais da bexiga, estimulando a sua exfoliação e consequente invasão do epitélio. Esta intervém também na inibição da atividade fagocítica e quimiotática dos neutrófilos17. Ainda relativamente às citotoxinas destaca‐se a α‐hemolisina, codificada pelo gene hly, que promove a formação de poros com a consequente lise celular, facilitando a obtenção de nutrientes e a captação de ferro iónico, necessários ao metabolismo bacteriano17.
Os genes que codificam estes fatores de virulência podem estar localizados no cromossoma ou em plasmídeos, e podem também estar associados a elementos genéticos móveis como as ilhas de patogenicidade (PAI). Estes elementos caracterizam‐se por terem tamanho superior a 10 kilobases, o que lhes permite ter em associação genes codificadores de adesinas, toxinas, sideróforos, cápsulas e serem facilmente transferidos entre espécies bacterianas18–20. Das 8 ilhas de patogenicidade já conhecidas em E. coli destacam‐se a PAI ICFT073 e a PAI IICFT073; a primeira possui um grupo de genes que codificam para a α‐hemolisina e a Pap, enquanto a segunda transporta apenas genes codificadores da Pap18.
A fronteira entre comensalismo e patogenicidade tem suscitado aos investigadores o desenvolvimento de metodologias que permitam esclarecer se uma estirpe de E. coli considerada comensal pode ser patogénica e vice‐versa, e quais as características que as diferenciam. Clermont et al.21 desenvolveram um algoritmo baseado na deteção de 3 genes conservados, o que permite agrupar as estirpes de E. coli em 4 grupos filogenéticos – A, B1, B2, D – sendo que as estirpes extraintestinais patogénicas pertencem maioritariamente ao grupo B2 e em menor número ao grupo D, enquanto as comensais pertencem aos grupos A e B121,22.
Este trabalho surge como complemento de um projeto que visou o estudo da resistência aos antibióticos23,24, tem como objetivo caracterizar os fatores de virulência em isolados E. coli de infeções urinárias e analisar a relação bactéria‐hospedeiro. Com este estudo pretendemos verificar se a bacteriúria por E. coli, em mulheres com idades até 59 anos, está associada ou não a critérios definidos de patogenicidade da bactéria ou a fatores de risco inerentes ao hospedeiro.
Materiais e métodosEstirpes bacterianas: foram estudadas 228 estirpes de E. coli, isoladas de urinas de mulheres com idade inferior ou igual a 59 anos. As bactérias provinham de vários laboratórios portugueses de prestação de serviços à comunidade, tendo sido identificadas nos laboratórios de origem e, posteriormente, enviadas ao laboratório de Microbiologia da Faculdade de Farmácia da Universidade de Lisboa.
Pesquisa de genes de virulência e identificação dos grupos filogenéticos: a pesquisa dos genes de virulência (fimH, papC, ecpA, usp, hlyA e cnf1), das ilhas de patogenicidade PAI ICFT073 e PAI IICFT073 e do grupo filogenético (chuA, yjaA, TSPE4.C2) foi efetuada no DNA genómico extraído pela técnica de boiled25. Os produtos obtidos após amplificação pela técnica de PCR, com primers específicos para os genes em estudo12,21,26–29, foram confirmados por sequenciação, e as sequências nucleotídicas e aminoacídicas foram analisadas por software disponível no National Centre for Biotechnology Information (http://www.ncbi.nlm.nih.gov).
ResultadosCaracterização da amostra clínicaO diagnóstico de cistite foi baseado nos sintomas clínicos como ardor à micção, poliquiúria e urgência urinária, e na análise cito‐bacteriológica da urina. Este exame consiste na observação de leucocitúria, marcador de inflamação, associada à identificação de uma estirpe bacteriana em número superior a 105 unidades formadas colónias UFC/ml. Dos 228 isolados de E. coli, 170 foram identificados de urinas de mulheres sem fatores de risco, consideradas cistites não complicadas (ncITU). Os restantes 50 isolados eram provenientes de urinas de mulheres, que se diferenciaram dos critérios de inclusão das ncITU porque apresentavam um ou mais fatores como recorrência da infeção (n=25), gravidez (n=37) e diabetes (n=2), ao que se englobaram nas cistites complicadas (cITU)6.
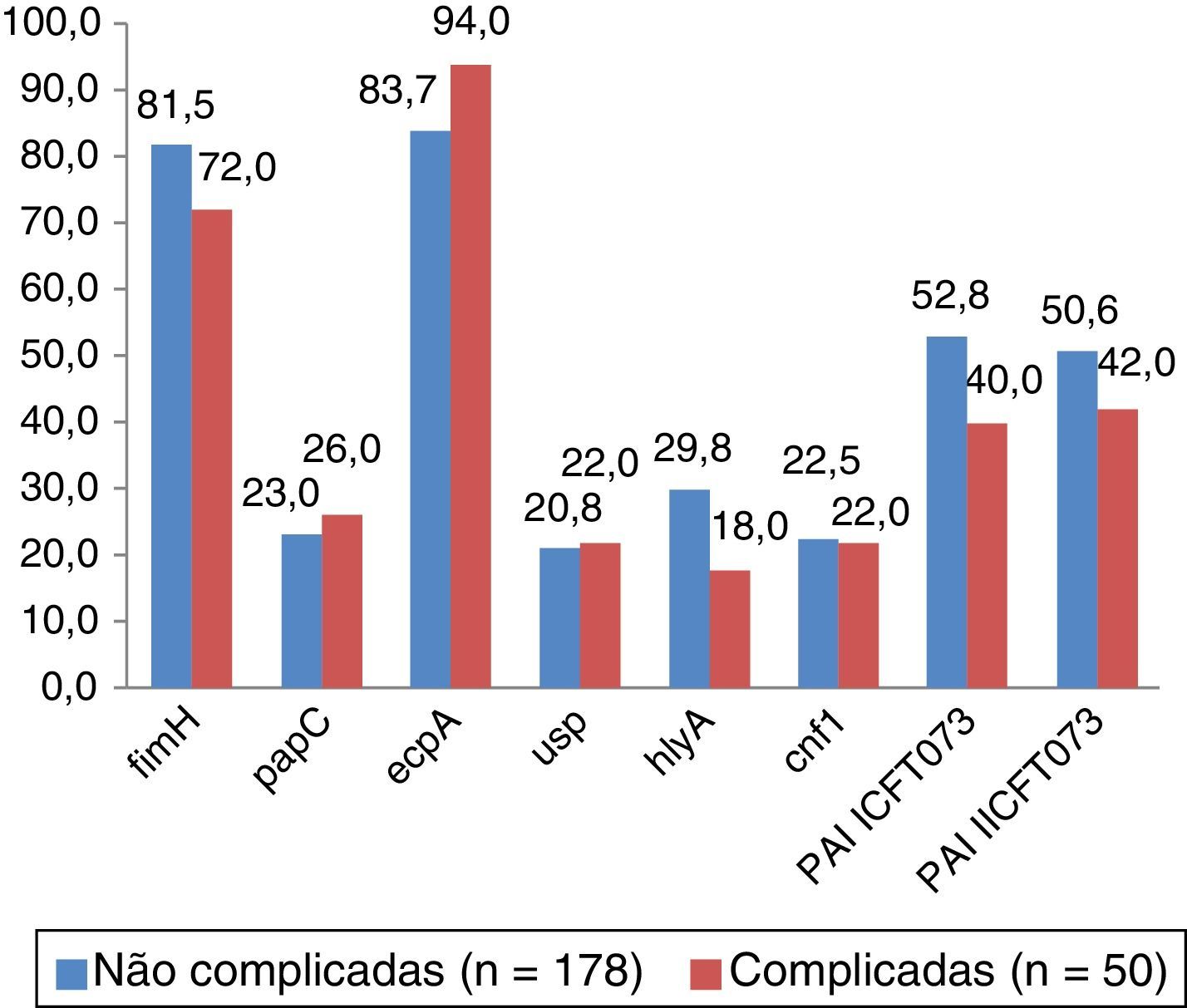
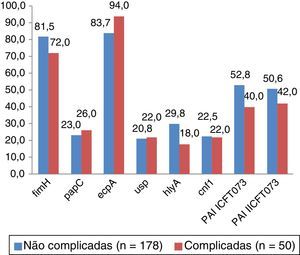
Genes de virulênciaNa pesquisa dos genes de virulência observou‐se um padrão de prevalência idêntico em todos os isolados, sendo que o gene mais frequente foi ecpA, seguido de fimH, tal como é observado na figura 1. Nos 50 isolados de cITU, os genes ecpA e fimH foram identificados em 94,0% (n=47) e 72,0% (n=36), respetivamente. Da mesma forma, nos 170 isolados de ncITU, os genes ecpA e fimH foram encontrados em 83,7% (n=149) e 81,5% (n=145). Para os restantes genes de virulência estudados (papC, usp, hlyA e cnf1), foram encontrados valores dentro da mesma ordem entre 18‐30%.
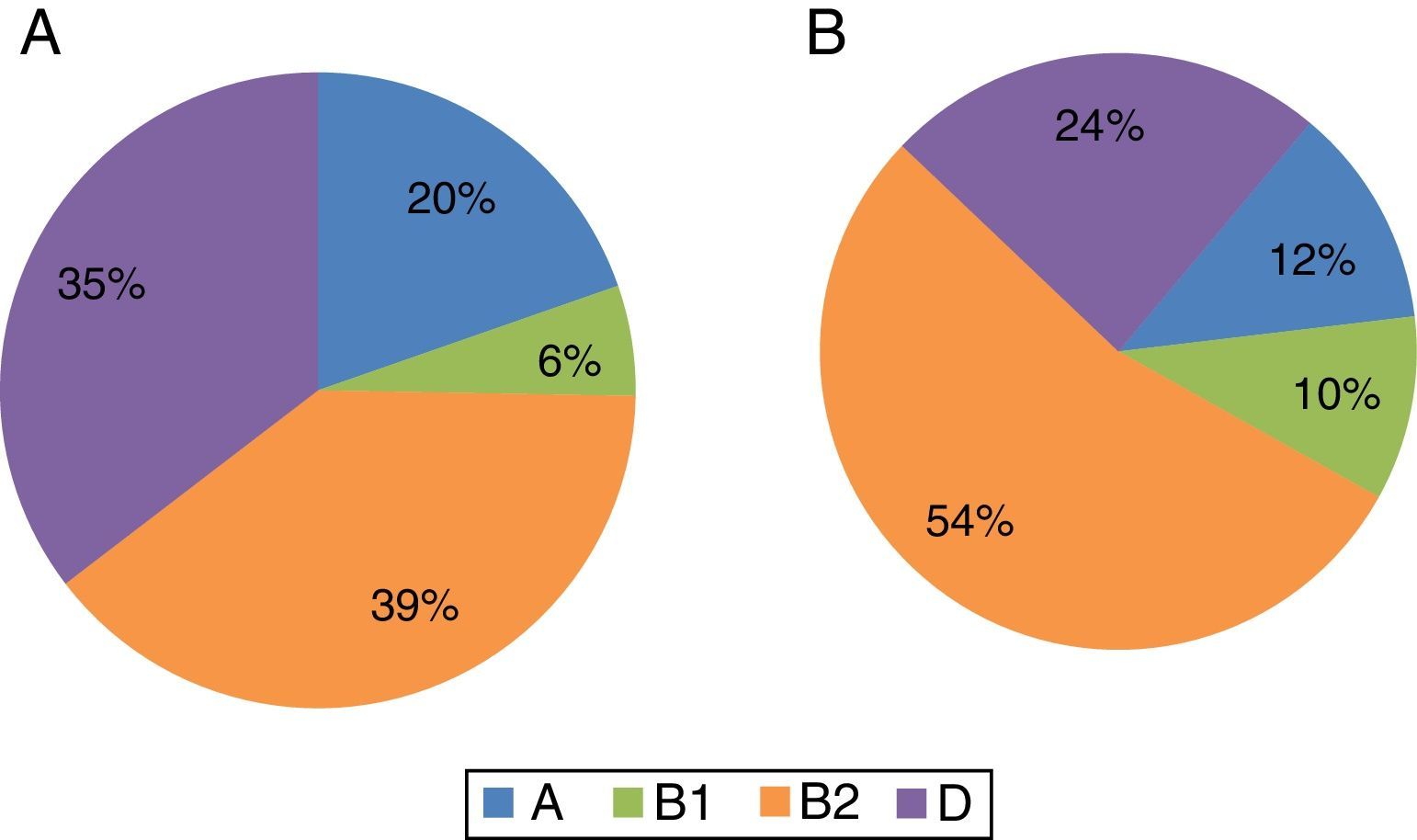
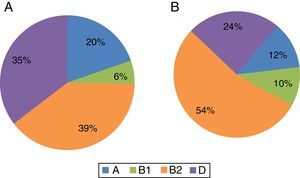
Relação dos fatores de virulência e os grupos filogenéticosOs 228 isolados de E. coli distribuíram‐se entre os 4 grupos filogenéticos, A, B1, B2, D, de acordo com a figura 2. As bactérias comensais dos grupos A e B1 foram identificadas em 45 dos 178 isolados associados às ncITU (fig. 2A) e que se distribuíram pelo grupo A (20%; n=35) e pelo grupo B1 (6%; n=10), enquanto a maioria dos isolados de ncITU (n=133) pertencem aos grupos patogénicos B2 e D em quantidade percentual semelhante de 39% (n=70) e 35% (n=63), respetivamente. De acordo com a figura 2B, dos 50 isolados provenientes de cITU, verificou‐se que 39 isolados estavam nos grupos patogénicos B2 (54%; n=27) e D (24%; n=12). Os restantes 11 isolados (12%; n=6) e (10%; n=5) distribuíram‐se entre os grupos comensais A e B1, respetivamente.
Em 8 isolados, foram identificados todos os genes de virulência em estudo; distribuíram‐se pelos grupos filogenéticos patogénicos B2 (n=3) e D (n=5). Apenas num isolado do grupo filogenético D não foram detetados quaisquer fatores de virulência em estudo.
Ao comparar os resultados expressos na tabela 1, dos isolados provenientes de cITU e ncITU, verifica‐se que, tanto os isolados comensais (A e B1), como os patogénicos (B2 e D), apresentaram todos os genes que estão implicados na aderência às células e colonização do hospedeiro (fimH, papC, ecpA). Inclusivamente, no grupo de isolados comensais e provenientes de cITU só foram detetados os genes de colonização. Quanto aos genes que codificam proteínas de proteínas responsáveis por processos invasivos, os isolados dos grupos B2 e D apresentam valores significativamente mais elevados, quando comparados com os grupos A e B1.
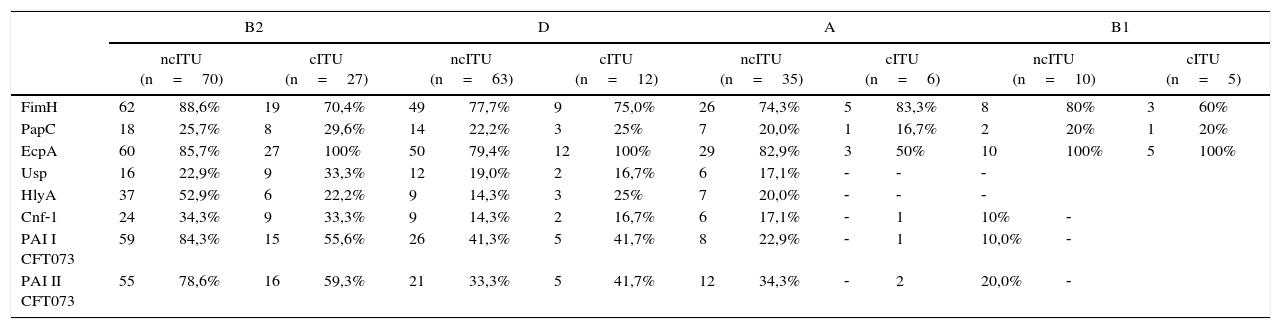
Distribuição dos isolados de Escherichia coli pelos grupos filogenéticos patogénicos (B2, D) e comensais (A, B1), de acordo com os fatores de virulência fimH, papC, espA, usp, hlyA, cnf1 e as ilhas de patogenicidade PAI ICFT073 e PAI IICFT073
| B2 | D | A | B1 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ncITU (n=70) | cITU (n=27) | ncITU (n=63) | cITU (n=12) | ncITU (n=35) | cITU (n=6) | ncITU (n=10) | cITU (n=5) | |||||||||
| FimH | 62 | 88,6% | 19 | 70,4% | 49 | 77,7% | 9 | 75,0% | 26 | 74,3% | 5 | 83,3% | 8 | 80% | 3 | 60% |
| PapC | 18 | 25,7% | 8 | 29,6% | 14 | 22,2% | 3 | 25% | 7 | 20,0% | 1 | 16,7% | 2 | 20% | 1 | 20% |
| EcpA | 60 | 85,7% | 27 | 100% | 50 | 79,4% | 12 | 100% | 29 | 82,9% | 3 | 50% | 10 | 100% | 5 | 100% |
| Usp | 16 | 22,9% | 9 | 33,3% | 12 | 19,0% | 2 | 16,7% | 6 | 17,1% | ‐ | ‐ | ‐ | |||
| HlyA | 37 | 52,9% | 6 | 22,2% | 9 | 14,3% | 3 | 25% | 7 | 20,0% | ‐ | ‐ | ‐ | |||
| Cnf‐1 | 24 | 34,3% | 9 | 33,3% | 9 | 14,3% | 2 | 16,7% | 6 | 17,1% | ‐ | 1 | 10% | ‐ | ||
| PAI I CFT073 | 59 | 84,3% | 15 | 55,6% | 26 | 41,3% | 5 | 41,7% | 8 | 22,9% | ‐ | 1 | 10,0% | ‐ | ||
| PAI II CFT073 | 55 | 78,6% | 16 | 59,3% | 21 | 33,3% | 5 | 41,7% | 12 | 34,3% | ‐ | 2 | 20,0% | ‐ | ||
ncITU: infeção do trato urinário não complicada; cITU: infeção do trato urinário complicada.
Nos 228 isolados, as 2 ilhas patogenicidade (PAI ICTF073 e PAI IICFT073) foram predominantes entre as 8 PAI pesquisadas, tanto nos isolados de ncITU como de cITU. Embora com valores semelhantes, a PAI ICTF073 foi a prevalente 51,9 e 49,3%, seguida da PAI IICFT073 com 49,7 e 46,3% para os isolados de ncITU e de cITU, respetivamente. De acordo com a tabela 1, estas PAI também foram encontradas em maior percentagem nas estirpes B2 e D, tanto nos isolados de ncITU como de cITU. Contrariamente ao que se poderia pensar, não se detetaram isolados com as 2 PAI nos grupos comensais A e B1, oriundos de cistites complicadas, tendo em conta o facto de provirem de 10 grávidas, 3 delas com recorrência, e de uma mulher com diabetes.
DiscussãoUm dos fatores principais para a ocorrência de infeções do tracto urinário é a presença de adesinas fimbriais, permitindo a aderência da bactéria ao uroepitélio. A prevalência de fímbrias do tipo 1, comparativamente às do tipo P, é um indicador de virulência importante e relevante na patogenicidade das estirpes de E. coli. Esta fímbria desempenha um papel importante na indução da inflamação, em particular nas cistites, uma vez que a adesina FimH se liga especificamente à uroplaquina, abundante no epitélio vesical. No entanto, a associação com as Pap alerta para a necessidade de um maior acompanhamento clínico, de modo a evitar infeções no aparelho urinário alto, mais concretamente, pielonefrites.
O facto de se ter verificado a presença do pilus ECP na maioria dos isolados, adesina que está implicada na colonização do intestino e que contribui para a permanência das bactérias no hospedeiro, confirma a autocontaminação por via ascendente como a principal causa de ITU. A adesina EcpA é um fator crítico para a aderência e virulência de estirpes de E. coli entero‐hemorrágicas (EHEC), porque ao mimetizar o comportamento das estirpes comensais confere a vantagem na colonização e evasão ao sistema imunitário do hospedeiro30. No nosso estudo a prevalência do gene ecpA, tanto em grupos patogénicos como comensais, permite‐nos concordar com Rendon et al.30, ao afirmar que este gene acompanha a evolução de E. coli intestinais e extraintestinais, possibilitando a colonização generalizada destas bactérias. Efetivamente, o gene ecpA foi encontrado nos isolados dos 4 grupos filogenéticos, com maior relevância nos isolados dos grupos patogénicos B2 e D, enquanto em estirpes hospitalares multirresistentes este gene foi encontrado maioritariamente em isolados do grupo A31.
Relativamente à associação do grupo filogenético e fatores de virulência, observou‐se um predomínio destes nos grupos patogénicos B2, seguido do grupo D, tanto em cistites complicadas como não complicadas, comparativamente aos grupos comensais A e B1. Esta diferença é evidenciada pela baixa percentagem dos genes usp, hly e cnf1 encontrados nestes grupos, o que poderá justificar uma menor capacidade invasiva e uma menor causalidade para a infeção por parte das estirpes comensais. A proteína USP tem sido encontrada mais frequentemente em estirpes uropatogénicas do que em comensais, e está associada às bacteriemias com origem nas infeções urinárias12. No nosso estudo, o gene usp foi detetado nos isolados de cistites complicadas nas estirpes patogénicas, o que não aconteceu nas estirpes comensais onde foi encontrado apenas nos isolados de cistites não complicadas. Se a prevalência de uma proteína como a USP, com capacidade de invadir o epitélio vesical, num grupo de isolados patogénicos é relevante, a presença desta proteína USP nos isolados comensais, em particular nos provenientes de ncITU, poderá ser um alerta pela patogenicidade que confere à bactéria, independentemente dos fatores de risco inerentes ao hospedeiro. No entanto, os isolados de cITU apresentaram percentagens elevadas de fatores de virulência, um pouco superiores aos isolados de ncITU, o que permite estabelecer um grau de patogenicidade semelhante entre os grupos, reforçando a hipótese de que a virulência inerente à bactéria poderá ser tão ou mais importante que os fatores de risco do hospedeiro. Efetivamente a presença de fatores de aderência, de invasão e de toxicidade em estirpes comensais, características inerentes à estirpe bacteriana, tem um papel determinante e justifica a dificuldade de erradicação da bactéria e da recorrência de infeção.
A prevalência das ilhas de patogenicidade PAI ICFT073 e PAI IICFT073 foi superior nos isolados patogénicos comparativamente aos comensais, sendo que neste grupo não foram detetadas nos isolados de cITU. Estas ilhas de patogenicidade possuem um grupo de genes que codificam para a Pap e α‐hemolisina, o que poderá indiciar a maior facilidade das estirpes atingirem o aparelho urinário superior e permaneceram no parênquima renal (Pap); capacidade de lisar as hemácias (α‐hemolisina), assim como células endoteliais e do epitélio renal, permitindo maior facilidade de entrada na corrente sanguínea, originando bacteriemias. No nosso estudo, não se verificou uma analogia entre o número de isolados positivos para o gene hlyA e a PAI ICFT073, dado que para a deteção da PAI a zona a amplificar por PCR não é a correspondente à do gene hlyA e o facto de poderem ocorrer deleções, mutações e rearranjos genómicos nas PAI poderá justificar a inexistência de concordância entre estes valores. No entanto, e de acordo com Schmidt et al.20, a inserção de uma PAI no cromossoma bacteriano pode converter uma bactéria não patogénica em patogénica e permitir mais facilmente a aquisição de genes de virulência.
As bactérias que transportam ilhas de patogenicidade têm maior capacidade invasiva, o que associado a fatores de aderência conduz a uma maior capacidade de lesão do epitélio, persistência, proteção do alcance do antibiótico e sistema imunitário, assim como maior capacidade de ascensão no trato urinário.
ConclusãoNeste estudo verificou‐se que tanto os isolados de ITU complicadas, como os de ITU não complicadas apresentaram um grau de patogenicidade semelhante, reforçando a hipótese de que os fatores de risco inerentes à bactéria poderão ser tão ou mais importantes que os do hospedeiro. As condições específicas do hospedeiro, tais como a gravidez, recorrência e diabetes, podem não ser determinantes para o estabelecimento da ITU, dado que os isolados nestas situações apresentaram um número elevado de fatores de virulência.
Responsabilidades éticasProteção de pessoas e animaisOs autores declaram que para esta investigação não se realizaram experiências em seres humanos e/ou animais.
Confidencialidade dos dadosOs autores declaram que não aparecem dados de pacientes neste artigo.
Direito à privacidade e consentimento escritoOs autores declaram que não aparecem dados de pacientes neste artigo.
Conflito de interessesOs autores declaram não haver conflito de interesses.