Los microRNA (miR) son RNA de pequeño tamaño involucrados en la regulación de numerosos procesos biológicos. Debido a su estabilidad y a su capacidad para detectarse en fluidos, se han convertido en objeto de estudio como posibles biomarcadores de diferentes patologías.
ObjetivoSeleccionar un número reducido de miR candidatos a ser biomarcadores de aneurisma de aorta abdominal (AAA).
Material y métodosSe determinó la expresión de 179 miR en el plasma de 7 pacientes y 7 voluntarios sanos, mediante paneles prediseñados, basados en la tecnología de RT-qPCR.
ResultadosHemos seleccionado 10 miR disregulados en pacientes como buenos candidatos para su estudio como biomarcadores debido a su implicación en diversos procesos biológicos relacionados con el desarrollo y evolución de AAA.
ConclusionesAunque son necesarios estudios adicionales y de validación, algunos de estos miR podrían ser biomarcadores no invasivos de AAA.
MicroRNAs (miRs) are small RNA molecules that are involved in several biological processes. Due to their stability and their presence in biological fluids, several studies over the last few years have focused on the use of miRs as biomarkers of different pathologies.
ObjectiveSelection of a group of miRs candidates to be validated as potential biomarkers of abdominal aortic aneurysm (AAA).
Material and methodsA total of 179 miRs were quantified in 7 patients with AAA and in 7 healthy donors, using predesigned panels based on RT-qPCR technology.
ResultsA total of 10 miRs, dysregulated in patients, were selected as good candidates for studying them as biomarkers. It has been observed that most of these miRs participate in several biological processes related to the formation and development of AAAs.
ConclusionAdditional, as well as validation, studies are needed, but some of these miRs could be non-invasive AAA biomarkers.
El aneurisma de aorta abdominal (AAA) es una patología común y potencialmente letal. Los mecanismos moleculares que subyacen a dicha patología aún permanecen inciertos y, como consecuencia, en los últimos años múltiples estudios han ido encaminados a la investigación de nuevas moléculas relacionadas con la formación y progresión del AAA. Recientemente se ha relacionado una nueva clase de RNA de pequeño tamaño, conocidos como microRNA (miR), con diferentes patologías cardiovasculares1.
Los miR constituyen un conjunto de pequeños RNA (19-24nt) de cadena sencilla, endógenos, no codificantes y altamente conservados, que regulan la expresión génica al unirse, principalmente, a la región 3’ UTR de un mRNA diana2.
Estudios recientes han demostrado la utilidad de los miR presentes en plasma o suero como marcadores de diagnóstico y pronóstico de diversas patologías3. Tanto la facilidad de obtención de los fluidos como la estabilidad de estos miR circulantes los han convertido en objeto de interés, aunque su uso como biomarcadores también presenta ciertos inconvenientes. Por un lado, la baja concentración de este tipo de moléculas en fluidos dificulta su determinación y, por otro, debido a que los estudios con miR son relativamente recientes, todavía no existe un consenso claro sobre la metodología, la normalización de los resultados y la especificidad de estos.
Los trabajos publicados hasta la fecha sobre miR como biomarcadores de AAA son escasos y los resultados son poco reproducibles o contradictorios entre los distintos grupos. Por ello es necesario ampliar los estudios al respecto4.
ObjetivoEn este estudio se pretende identificar un grupo de miR presentes en plasma que puedan ser potenciales biomarcadores de AAA en humanos.
Material y métodosDiseñoSe trata de un estudio preliminar experimental caso control.
PacientesSe obtuvo sangre periférica de 7 pacientes con AAA (grupo AAA) que iban a ser sometidos a cirugía abierta o endoprótesis y de 7 voluntarios sanos (grupo control). En el grupo control se descartó la presencia de AAA mediante eco-doppler abdominal.
No se incluyeron pacientes con efermedades graves sistémicas como cáncer, VIH o hepatitisC ni pacientes con AAA sindrómicos asociados a Marfan o Ehlers-Danlos.
El estudio contó con la aprobación del comité ético del Instituto de Investigación sanitaria La Fe y cumple con los principios de la declaración de Helsinki de 1975. Todos los pacientes firmaron el consentimiento informado correspondiente y aceptaron su participación en el estudio.
MétodosDe cada individuo, se recogieron 3ml de sangre venosa en un tubo con 5,4mg de K2EDTA como anticoagulante. Las muestras se procesaron durante las dos primeras horas desde la extracción. Los tubos se centrifugaron durante 30min a 4°C y 1.811g y el plasma obtenido se distribuyó en alícuotas de 250μl que se almacenaron a −80°C hasta su utilización.
De cada alícuota, se extrajo el RNA total mediante el miRNeasy mini-kit (Qiagen, Alemania), que permite la extracción de RNA de pequeño tamaño. A partir de 5μl de RNA se realizó la síntesis de cDNA empleando el kit Universal cDNA synthesis kit® (Exiqon, Dinamarca) que genera un único cDNA a partir del cual pueden cuantificarse múltiples miR mediante el empleo de cebadores específicos.
El estudio de expresión de miR se realizó por RT-qPCR mediante el empleo de paneles prediseñados Serum/Plasma Focus miRNA PCR 384 pocillos (Exiqon, Dinamarca) siguiendo las indicaciones del fabricante. Los paneles contienen sondas para cuantificar 179miR habitualmente presentes en suero y plasma, junto con varios miR candidatos a emplearse como miR de referencia, y 5 controles internos (spike in) que permiten evaluar la calidad de las distintas etapas de la cuantificación de expresión. Las reacciones de PCR y las lecturas de fluorescencia se realizaron en un termociclador Lightcycler 480 (Roche, Alemania).
Análisis de datosMediante la base de datos RefFinder5 se determinó el miR de referencia, que se utilizó como normalizador para estimar los niveles de los miR candidatos, calculados mediante el método del 2-ΔΔCT. Las veces de cambio se calcularon tomando como referencia los valores medios del grupo control.
Los miR candidatos para validación se determinaron estadísticamente mediante el algoritmo de clasificación de Random Forest6 con el software estadístico R (versión3.3.2).
Finalmente, se realizó una búsqueda bibliográfica exhaustiva para determinar los genes diana que podrían estar regulados por estos miR y en qué rutas conocidas podrían estar involucrados los miR candidatos, con el fin de establecer prioridades de validación.
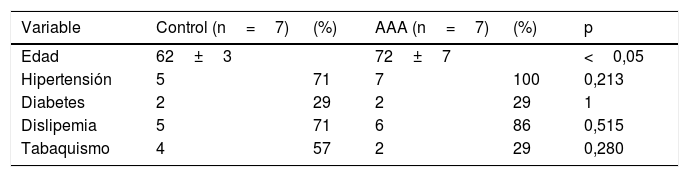
ResultadosEn la tabla 1 se muestra un resumen de los factores de riesgo de los participantes (n=14). Todos los sujetos eran varones, lo cual está en concordancia con la elevada prevalencia de esta patología en los hombres. La edad media en el grupo AAA era de 72±7 años y del grupo control de 62±3años, p<0,05 (media ±desviación estándar). El 100% de los pacientes AAA y el 71% de los controles habían sido diagnosticados en algún momento como hipertensos. El porcentaje de sujetos diabéticos fue del 29% en ambos grupos. El 86% y el 71% de pacientes AAA y controles, respectivamente, se consideraron hiperlipémicos o hipercolesterolémicos. El 29% de los pacientes y el 57% de los controles eran fumadores activos o lo habían sido en los últimos 5años.
Características clínicas de los sujetos de estudio incluidos en la etapa de cribado. Las diferencias de las variables entre los grupos se analizaron mediante t-test y tablas de contingencias χ2
| Variable | Control (n=7) | (%) | AAA (n=7) | (%) | p |
|---|---|---|---|---|---|
| Edad | 62±3 | 72±7 | <0,05 | ||
| Hipertensión | 5 | 71 | 7 | 100 | 0,213 |
| Diabetes | 2 | 29 | 2 | 29 | 1 |
| Dislipemia | 5 | 71 | 6 | 86 | 0,515 |
| Tabaquismo | 4 | 57 | 2 | 29 | 0,280 |
Como candidatos a miR de referencia se estudiaron 5miR de elevada estabilidad en plasma (miR-423-5p, miR-425-5p, miR-93-5p, miR-103a-3p y miR-191-5p), siendo este último el más adecuado según RefFinder.
Se descartaron 39 miR con un nivel de expresión-CT medio mayor de 36, ya que se consideró fuera del umbral de detección. Finalmente, se normalizó el nivel de expresión de los miR en cada una de las muestras estudiadas mediante el método 2-ΔΔCT, con respecto a la expresión del miR-191-5p.
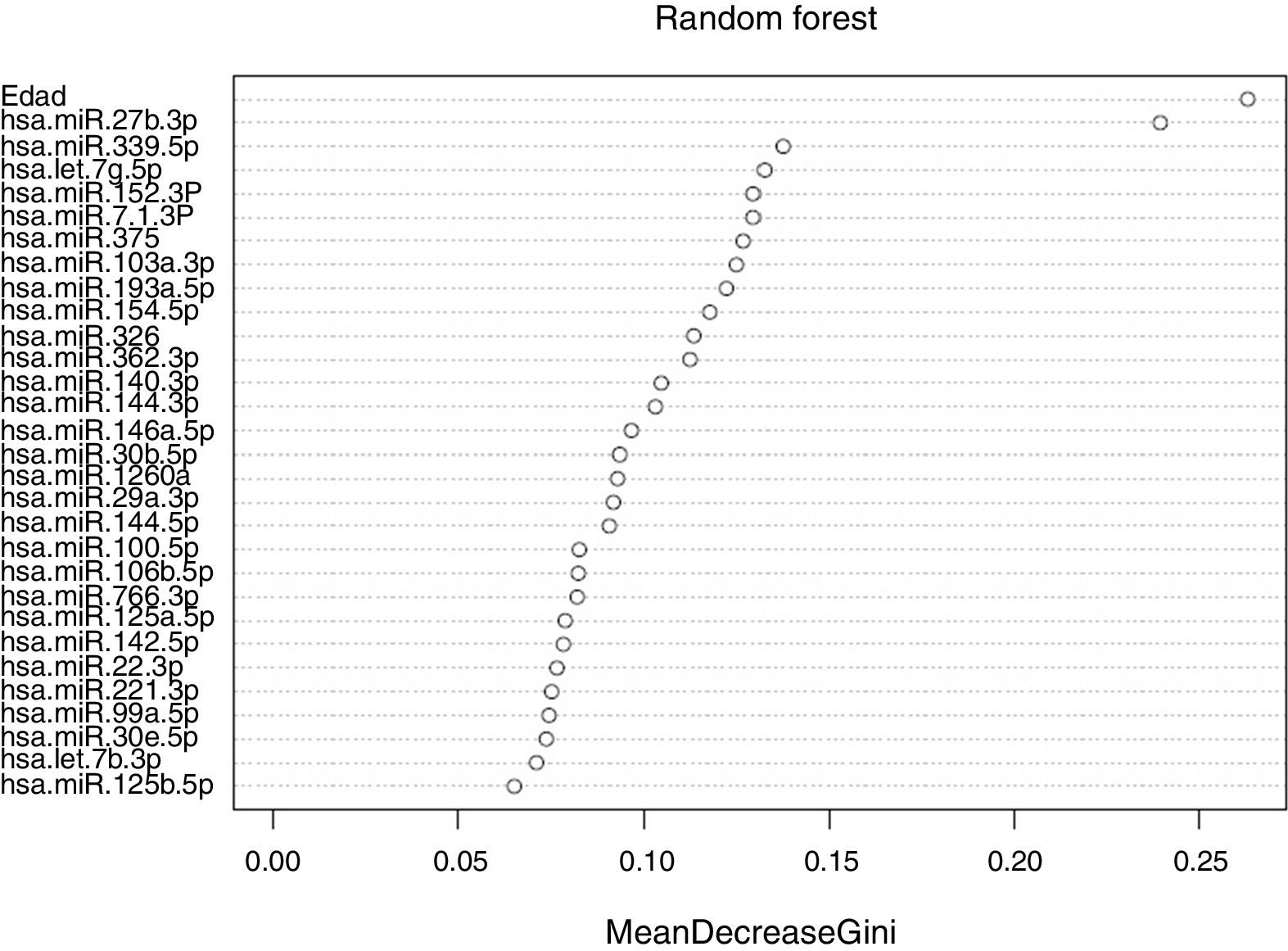
Aplicando el algoritmo de clasificación de Random Forest a los valores de 2-ΔΔCT de 140 miRs se obtuvo una lista de 29 miR como posibles candidatos a validación, ordenados por relevancia según el índice de Gini (fig. 1). Este método estableció la diferencia de edad entre los dos grupos de estudio como el factor más importante a tener en cuenta.
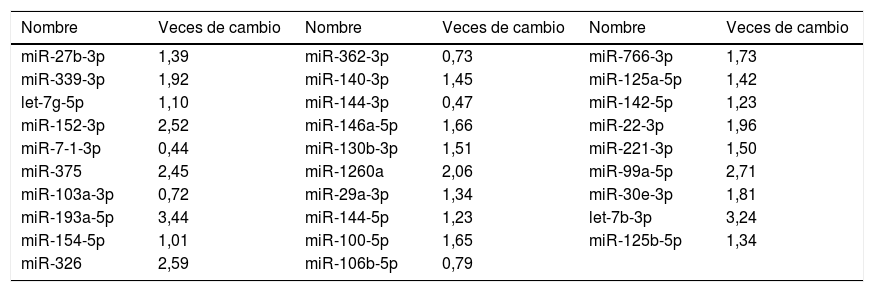
Las diferencias de expresión entre los miR más relevantes osciló entre 0,44 (infraexpresión en pacientes) y 3,44 veces (sobreexpresión en pacientes) (tabla 2).
Veces de cambio de los miR seleccionados tras el Random Forest. Las veces de cambio corresponden al cociente de las medias de 2-ΔΔCT (pacientes) frente a 2-ΔΔCT (controles)
| Nombre | Veces de cambio | Nombre | Veces de cambio | Nombre | Veces de cambio |
|---|---|---|---|---|---|
| miR-27b-3p | 1,39 | miR-362-3p | 0,73 | miR-766-3p | 1,73 |
| miR-339-3p | 1,92 | miR-140-3p | 1,45 | miR-125a-5p | 1,42 |
| let-7g-5p | 1,10 | miR-144-3p | 0,47 | miR-142-5p | 1,23 |
| miR-152-3p | 2,52 | miR-146a-5p | 1,66 | miR-22-3p | 1,96 |
| miR-7-1-3p | 0,44 | miR-130b-3p | 1,51 | miR-221-3p | 1,50 |
| miR-375 | 2,45 | miR-1260a | 2,06 | miR-99a-5p | 2,71 |
| miR-103a-3p | 0,72 | miR-29a-3p | 1,34 | miR-30e-3p | 1,81 |
| miR-193a-5p | 3,44 | miR-144-5p | 1,23 | let-7b-3p | 3,24 |
| miR-154-5p | 1,01 | miR-100-5p | 1,65 | miR-125b-5p | 1,34 |
| miR-326 | 2,59 | miR-106b-5p | 0,79 |
La búsqueda bibliográfica de las posibles vías de actuación de cada uno de los miR candidatos reveló que algunos de estos miR participan en vías de regulación relacionadas con procesos vasculares, como inflamación o metabolismo lipídico, lo que los convierte en buenos candidatos para su estudio como posibles biomarcadores de AAA. Teniendo en cuenta los resultados del Random Forest, los niveles de expresión, las veces de cambio y la revisión bibliográfica, se han seleccionado 10miR (miR-27b-3p, miR-152-3p, miR-7-1-3p, miR-375, miR-103a-3p, miR-144-3p, miR-146a-5p, miR-1260a, miR-221-3p y miR-99a-5p) como posibles candidatos para ser validados en una muestra de mayor tamaño.
DiscusiónAlgunos de los miR seleccionados como posibles candidatos se han visto previamente relacionados con mecanismos moleculares relacionados con patologías vasculares.
El miR más destacado por el algoritmo de clasificación fue el miR-27b-3p. Muchos estudios indican que la familia de miR-27 (a/b) está implicada en procesos relacionados con aterosclerosis, incluyendo angiogénesis, adipogénesis, infamación, metabolismo lipídico, estrés oxidativo, resistencia a la insulina y diabetes tipo27. Entre sus dianas se encuentran proteínas involucradas en el metabolismo lipídico y remodelado de lipoproteínas, como los receptores activados por proliferadores de peroxisomas gamma (PPARG), la proteína similar a la angiopoyetina tipo3 (ANGPTL3) o la N-deacetilasa/N-sulfotransferasa (NDST1)8. También podría estar implicado en la homeostasis del colesterol en macrófagos, actuando a través de dianas como la acetil-CoA C acetiltransferasa 1 (ACAT1), lipoproteínas de baja densidad (LDL) o el casete de unión a ATP (ABCA1)9. Su relación con la respuesta inmune e inflamatoria podría estar mediada por RXRα (receptor α del retinoideX) o nuevamente por PPARG. Una de sus dianas validadas más importantes es SPROUTY2, cuya regulación afecta a la inflamación y a la angiogénesis, por su relación con la vía de señalización MAPK y VEGFR210; además, su participación en estos procesos podría estar, también, mediada por la metaloproteasa (MMP) 13, ya que se ha visto que un aumento de esta proteína se relaciona con una disminución de la expresión de miR-27b-3p11. Su capacidad para regular estos procesos relacionados con la aterosclerosis lo ha convertido en un miR de gran interés para la patología. De hecho, sus niveles en suero se han relacionado con la aterosclerosis obliterante12. Tanto la importancia de la aterosclerosis en el desarrollo del AAA como la presencia de mecanismos comunes entre ambas patologías hacen pensar que este miR podría estar también implicado en el desarrollo del AAA.
Por su parte, el miR-152-3p es un miR implicado en la regulación de las interleucinas2 y 6 o el factor de necrosis tumoral-α (TNF-α) a través de su diana, la proteína cinasa-II-α dependiente de calcio/calmodulina (CAMKII-α)13 y, por tanto, podría estar involucrado en procesos inflamatorios. Además, se ha visto la capacidad de este miR para regular la expresión de ADAM 1714, una desintegrina y metaloproteasa implicada en el desarrollo de los AAA.
Una de las dianas conocidas de miR-144-3p es ABCA1, que, como se ha comentado, está relacionada con la homeostasis del colesterol. Estudios en animales de experimentación confirman que un aumento de miR-144-3p disminuye la concentración de ABCA1, mientras que su silenciamiento produce un aumento de ABCA1 y de HDL plasmático15. Además, se ha visto que un incremento de miR-144-3p produce un aumento de citoquinas inflamatorias tanto in vivo como in vitro16.
Estudios in vitro17 han demostrado que el miR-1260a podría estar implicado en la patología aneurismática, ya que se ha visto alteración de la expresión de este miR en células vasculares del músculo liso (CVML) procedentes de pacientes con AAA al compararlas con CVML de donantes sanos. Algunas de las dianas predichas de este miR están involucradas en aterosclerosis, en cambios de la pared arterial o en la rigidez aórtica, siendo la diana más destacada COL1A1, recientemente validada por Cheuk y Cheng17.
Por otro lado, aunque no está claro su mecanismo de acción, el miR-99a-5p también podría estar implicado en la formación de AAA, ya que se ha visto sobreexpresado en la zona de máxima expansión de la lesión18, aunque no se ha confirmado su utilidad como biomarcador en plasma.
Recientemente se ha publicado un estudio que sugiere la implicación del miR-103a-3p en el desarrollo de AAA a través de la regulación de ADAM10. En modelos murinos con AAA, una infraexpresión de dicho miR correlaciona con un aumento de la proteína19.
Entre los miR seleccionados, uno de los más estudiados es miR-146a-5p, que puede participar en múltiples procesos. Se sabe que, en células endoteliales, este miR regula procesos inflamatorios, de manera citoquina-dependiente. En respuesta a un estímulo bioquímico, como el TNF-α, se induce la expresión de miR-146a-5p regulando el receptor de TNF asociado al factor 6 (TRAF6), la proteína cinasa asociada al receptor de interleucinas (IRAK 1) o, incluso, actuar de manera indirecta sobre la expresión de óxido nítrico sintasa endotelial (eNOS)20-23. Este miR también puede actuar en macrófagos, por un lado, regulando el cambio fenotípico M1/M224 y, por otro, afectando al transporte de lípidos y liberación de citoquinas en macrófagos oxidados LDL-estimulados25. Curiosamente, se ha encontrado relación entre un polimorfismo en el locus de miR-146a-5p y el aumento de riesgo de enfermedad cardiovascular en la población china26. Algunos estudios relacionan directamente el miR-146a-5p con AAA. Así, al comparar tejido aneurismático con tejido sano, Kin et al.27 y Pahl et al.28 observaron la disregulación de miR146a-5p mediante arrays de expresión. De acuerdo con estos resultados, en un ensayo preliminar con 4 muestras de tejido aneurismático y 4 muestras de arteria mesentérica inferior sana de pacientes operados mediante cirugía abierta, nuestro grupo observó la sobreexpresión del miR-146a-5p en tejido enfermo comparado con tejido sano procedente de un mismo paciente, confirmando la desregulación a nivel local de este miR (datos no mostrados).
Finalmente, el miR-221-3p participa en procesos esenciales de la biología vascular, como angiogénesis, neoplasia intimal, reparación endotelial y envejecimiento vascular. Además, está involucrado en mecanismos vasculares patológicos como angiogénesis tumoral, inflamación aterosclerótica, remodelado vascular, fibrosis, calcificación vascular, hipertrofia cardiaca, entre otros29. El mecanismo de acción de este miR es complicado y actúa sobre múltiples dianas, tanto en células endoteliales como en células vasculares de músculo liso (CVML), afectando a su migración y proliferación. Entre sus dianas en CVML se encuentran p27 (Kip1), p57 (Kip2) o PTEN, involucradas en la diferenciación y el establecimiento del fenotipo contráctil30,31. Entre sus dianas en células endoteliales se encuentran factores inflamatorios como ICAM, VCAM o MMP, relacionadas, además, con la migración de este tipo celular, o eNOS (óxido nítrico sintasa), relacionada con proliferación celular y disfunción endotelial32,33. Algunos autores han relacionado este miR con la sintomatología e inestabilidad de la placa de ateroma carotídea34. Además, el grupo de Kin et al.27 observó una sobreexpresión de este miR en tejido de AAA frente a tejido sano.
La mayoría de los miR citados se han descrito en estudios in vitro o en tejido procedente de animales de experimentación. Sin embargo, en los últimos años se ha demostrado su presencia en fluidos, y esto, junto con su estabilidad35, los ha convertido en objeto de interés como biomarcadores diagnóstico y pronóstico de distintas patologías. En lo que se refiere a patologías cardiovasculares, la mayoría de los estudios en humanos son estudios piloto realizados en un número pequeño de muestras. Aun así, se han relacionado algunos miR en plasma, suero o sangre total con patologías como infarto agudo de miocardio, fallo cardiaco, enfermedad arterial coronaria, diabetes mellitus e infarto cerebral, entre otras3. Sin embargo, en el caso del AAA existen muy pocos trabajos publicados27,36,37 y, además, hasta el momento existe gran variabilidad y falta de reproducibilidad entre grupos, por lo que es necesario ampliar los estudios.
ConclusionesLa combinación entre el estudio de expresión diferenciada de miR en plasma de pacientes y voluntarios sanos junto con una exhaustiva revisión bibliográfica nos ha permitido seleccionar 10 miR que podrían estar relacionados con el AAA.
Algunos de los miR aparentemente disregulados se han relacionado con procesos vasculares como respuesta inflamatoria, homeostasis de lípidos y colesterol, o regulación fenotípica de CVML. Estos procesos están relacionados con el desarrollo de AAA; sin embargo, no son exclusivos de esta patología, sino que son comunes a otras patologías que implican lesión vascular. Así pues, es necesario ampliar estudios para determinar la especificidad de dichas moléculas y, por tanto, su potencial uso como biomarcadores no invasivos de AAA.
Aunque estos resultados no permiten confirmar que los miR descritos sean biomarcadores de la patología, sí permiten seleccionar un número reducido de miR a validar en una muestra mayor.
FinanciaciónEste trabajo ha recibido una ayuda de la Sociedad Española de Angiología y Cirugía Vascular para su realización. También cuenta con dos ayudas de contratación de personal: ISCII-FEDER (CPII15/00002) y GVA (PROMETEOII/2015/017).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Al Servicio de Bioestadística del Instituto de Investigación Sanitaria La Fe por su colaboración en el análisis de resultados mediante el software R.