El objetivo del presente trabajo fue determinar si Staphylococcus aureus puede ser identificado por la presencia de los genes nucA y femB.
MetodologíaSe analizó la presencia de los genes nucA y femB mediante PCR en 196 aislados de Staphylococcus previamente identificados por métodos microbiológicos, incluyendo la prueba de coagulasa.
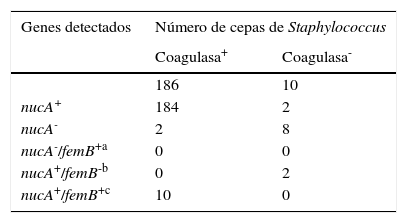
ResultadosEntre los 196 aislados, 186 fueron coagulasa positivos. De estos, 184 presentaron el gen nucA. De los 10 aislados coagulasa negativos dos presentaron el gen nucA. De los dos aislados coagulasa positivos nucA negativos y de los dos aislados coagulasa negativos nucA positivos, ninguno resultó portador del gen femB.
ConclusionesLa detección de los genes nucA y femB identifica a Staphylococcus aureus, pues solo S. aureus porta ambos genes.
The aim of this work was to determine whether Staphylococcus aureus can be identified by the presence of the genes, nucA and femB.
MethodsStaphylococcus isolates (n=196) previously identified by microbiological methods, including coagulase test, were analysed for the presence of the nucA and femB genes by PCR.
ResultsOf the 196 isolates used, 186 were coagulase positive. Among these, 184 had the nucA gene. Of the 10 coagulase negative strains, two had the nucA gene. The femB gene was not detected in the two coagulase positive nucA negative strains or the two coagulase negative nucA positive isolates.
ConclusionsDetection of nucA and femB genes identified Staphylococcus aureus, as only S. aureus carries both genes.
Staphylococcus aureus causa una amplia variedad de enfermedades infecciosas tales como infecciones cutáneas, septicemia, neumonía e intoxicación alimentaria, entre otras1,2. El principal impacto de este microorganismo se debe a las cepas de S. aureus resistentes a la meticilina (MRSA), que tradicionalmente se encontraban limitadas al ámbito nosocomial (cepas MRSA adquiridas en el hospital), a nivel mundial. Sin embargo, en años recientes las cepas MRSA también se han encontrado en la comunidad (cepas MRSA adquiridas en la comunidad) y estas han ido incrementando sustancialmente su incidencia3.
Se sabe que alrededor del 30% de las personas son portadoras sanas de S. aureus1,4, estos portadores sanos pueden transmitir a la bacteria a personas inmunodeficientes, con heridas o con implantes quirúrgicos, por lo que pueden llegar a provocar infecciones nosocomiales5. Se ha descrito la colonización nasal de S. aureus entre miembros de la misma familia sin producir la enfermedad, sin embargo, pueden dar lugar a infecciones6,7, por lo que los portadores sanos de S. aureus son un factor de riesgo importante en la infección por este microorganismo.
Entre las especies de Staphylococcus clínicamente importantes se destacan S. aureus, S. epidermidis y S. saprophyticus, aunque no hay que descartar a las demás especies de estafilococos coagulasa negativos, que contribuyen a infecciones sobre todo a nivel intrahospitalario8.
Los estafilococos producen varias enzimas que pueden contribuir a su virulencia, S. aureus tiene la capacidad de producir coagulasa, por esta razón la detección de esta enzima es el criterio más utilizado para diferenciarlo de las otras especies9.
Actualmente la identificación de cepas de S. aureus se puede llevar a cabo mediante la reacción en cadena de la polimerasa (PCR). Diferentes protocolos de PCR se han propuesto para detectar e identificar S. aureus, se detectan genes como nucA, coaA, femA o femB entre otros10.11. Si además se requiere saber si son cepas MRSA se detecta el gen mecA que es un marcador molecular de resistencia a la meticilina12,13.
Las cepas de S. aureus producen una termonucleasa extracelular (TNasa) con una frecuencia similar a las cepas que producen coagulasa. El gen nucA que codifica a la termonucleasa es producido por casi todas las cepas de S. aureus y es usado como criterio de diagnóstico de esta especie10.
Los genes fem (del inglés factors essential for methicillin resistance), como su nombre lo indica están involucrados en la resistencia a la meticilina. El gen femB es parte del operón femAB, involucrado en la síntesis del interpéptido de pentaglicina, importante en el entrecruzamiento del peptidoglucano de la pared celular de Staphylococcus11,14.
El objetivo de este trabajo fue explorar si S. aureus puede ser identificado por la presencia de los genes nucA y femB en la misma bacteria.
Materiales y métodosCepas y diagnóstico microbiológicoSe utilizaron 196 aislados de estafilococos, obtenidos en un estudio previo15, de la nariz y de la faringe de una población de estudiantes universitarios de ambos sexos de entre 17 y 22 años de edad y personal de ambos sexos de entre 25 y 65 años de edad que laboraban en una empacadora de carnes frías de la Ciudad de México. Las cepas se encontraban conservadas a -70°C. Las 196 cepas se habían identificado como Staphylococcus por pruebas microbiológicas incluyendo la prueba de coagulasa en tubo, de las cuales 186 eran coagulasa positivas y 10 coagulasa negativas. Todas las cepas eran sensibles a la meticilina. Las cepas se activaron inoculando 100μL de la suspensión descongelada en caldo soya tripticaseína y se incubaron toda la noche a 37°C. Posteriormente se sembraron en agar Sal Manitol (Bioxon) y se dejaron incubando toda la noche a 37°C. Una vez transcurrido el tiempo se verificó la morfología colonial y microscópica, se les realizaron las pruebas de catalasa y coagulasa en tubo siguiendo los protocolos de Koneman16.
Extracción de DNALa extracción del DNA de las 196 cepas se realizó utilizando el equipo comercial Wizard Genomic DNA Purification kit (Promega), siguiendo las indicaciones del fabricante.
Amplificación de los genes nucA y femB por PCRLa detección del gen nucA mediante PCR se llevó a cabo siguiendo la metodología de Brakstad et al.10, con algunas modificaciones. Se utilizaron los primers nucA1 5’ GCG ATT GAT GGT GAT ACG GTT 3’ y nucA2 5’ AGC CAA GCC TTG ACG AAC TAA AGC 3’. Las condiciones de amplificación fueron las siguientes: una desnaturalización inicial 94°C durante 5min, 10 ciclos de 94°C por 40 s, 68°C por 40 s, 72°C por 1min, seguido de 25 ciclos de 94°C por 1min, 58°C por 1min, 72°C por 2min y una extensión final de 10min a 72°C, obteniéndose un amplicón de 270 pb. Se utilizaron S. aureus ATCC 43300 y S.aureus ATCC 29213 como controles positivos y S. epidermidis ATCC 35983 como control negativo.
La amplificación del gen femB mediante PCR se realizó siguiendo la metodología descrita por Jonas et al.11, con modificaciones. Se utilizaron los primers FemB1 5’ TTA CAG AGT TAA CTG TTA CC 3’ y FemB2 5’ ATA CAA ATC CAG CAC GCT CT 3’. Las condiciones de corrida de la PCR fueron las siguientes: una desnaturalización inicial de 94°C por 5min, seguido de 35 ciclos de 94°C por 45s, 50°C por 45s y 72°C por 60s, con una extensión final de 72°C por 5 min, se obtuvo un producto de PCR de 651 pb. Se utilizaron las mismas cepas de la reacción anterior como controles.
En ambos casos la amplificación se realizó con una mezcla de reacción compuesta de 1X de buffer de reacción, 1.5mM de MgCl2, 1 U de Taq DNA polimerasa, 10nM de los primers forward y reverse, 200μM de dNTP's, <1μg de DNA de muestra y H2O grado biología molecular para completar 25μL de volumen.
Los productos de amplificación se corrieron en geles de agarosa al 1% a 80V y se tiñeron con bromuro de etidio (10mg/mL) y se visualizaron en un transiluminador Bio Rad.
ResultadosSe analizaron 196 aislados de las cuales 186 se habían identificado presuntivamente como S. aureus, por métodos microbiológicos por morfología microscópica y colonial en agar sal manitol, además de haber dado positivas las pruebas de catalasa y coagulasa en tubo. Diez cepas de las 196, resultaron ser coagulasa negativas. Para determinar la especie de las cepas analizadas se procedió a realizar la identificación molecular.
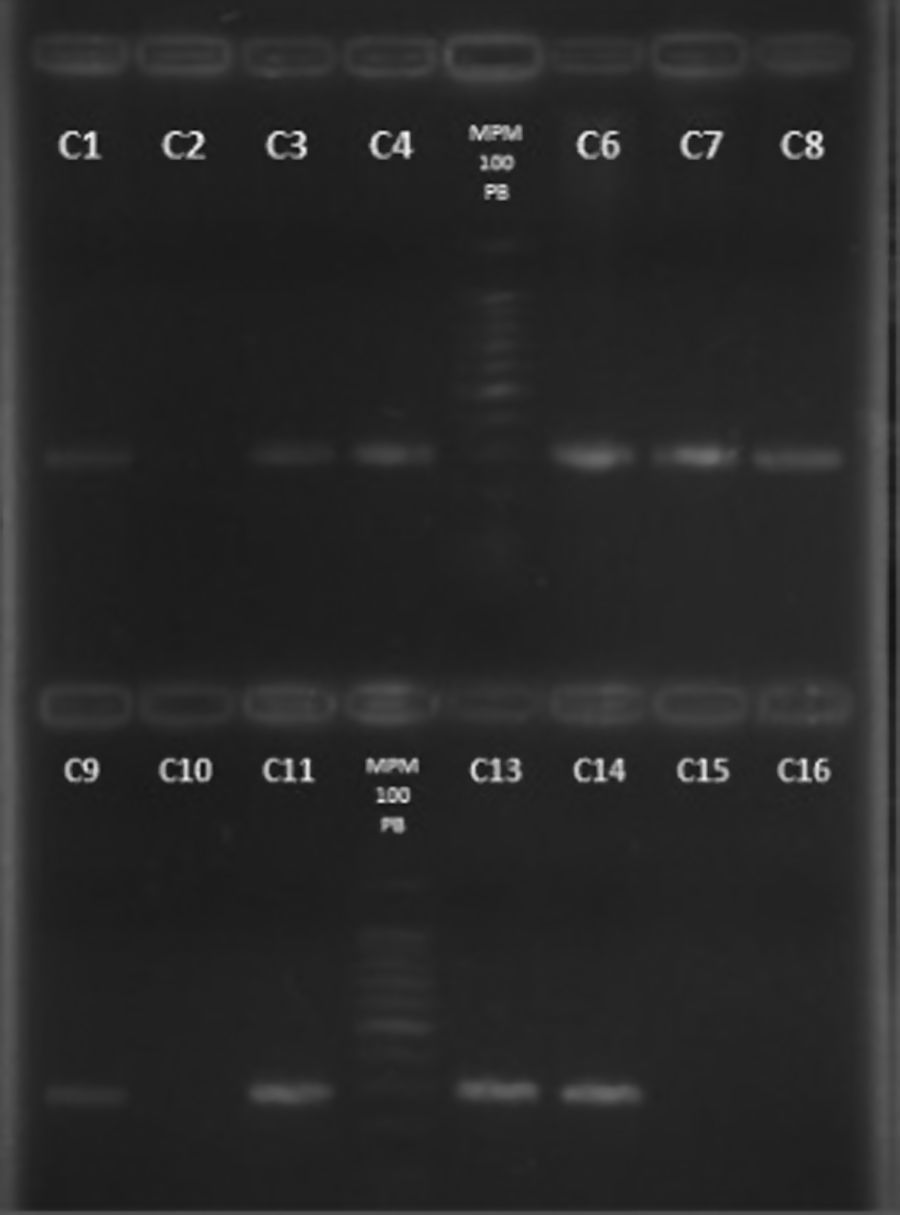
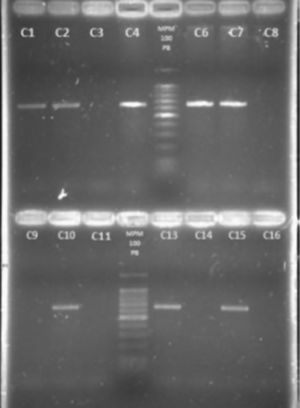
Se extrajo DNA cromosomal de todos los aislados. Mediante la técnica de PCR se detectó primeramente la presencia del gen nucA, se encontró que casi todas las cepas coagulasa positivas (184) presentaron este gen y solo dos de estas cepas coagulasa positivas no lo presentaron (67F y 527F), como se muestra en la tabla 1 y figura 1. Ocho de las diez cepas coagulasa negativas no presentaron el gen nucA, sin embargo, las otras dos cepas coagulasa negativa (12F y 287N) presentaron la presencia de este gen.
Detección del gen nucA por PCR. Electroforesis en gel de agarosa al 2%. Carriles: 1. cepa 6F; 2. cepa 67F; 3. cepa 105N; 4. cepa 106N; 5. Marcador de peso molecular 100pb; 6. cepa 139N; 7. cepa 211N; 8. cepa 217N; 9. cepa 220N; 10. cepa 527F; 11. cepa 637F, 12. Marcador de peso molecular 100pb; 13. cepa 22N; 14. S. aureus ATCC 29213; 15. S. epidermidis ATCC 35983, 16. Control de reactivos.
Para corroborar estos resultados se determinó la presencia del gen femB en las cepas coagulasa positivas (67F y 527F) y coagulasa negativas (12F y 287N), así como en otras cepas coagulasa positivas (10 cepas), que presentaron el gen nucA (tabla 1).
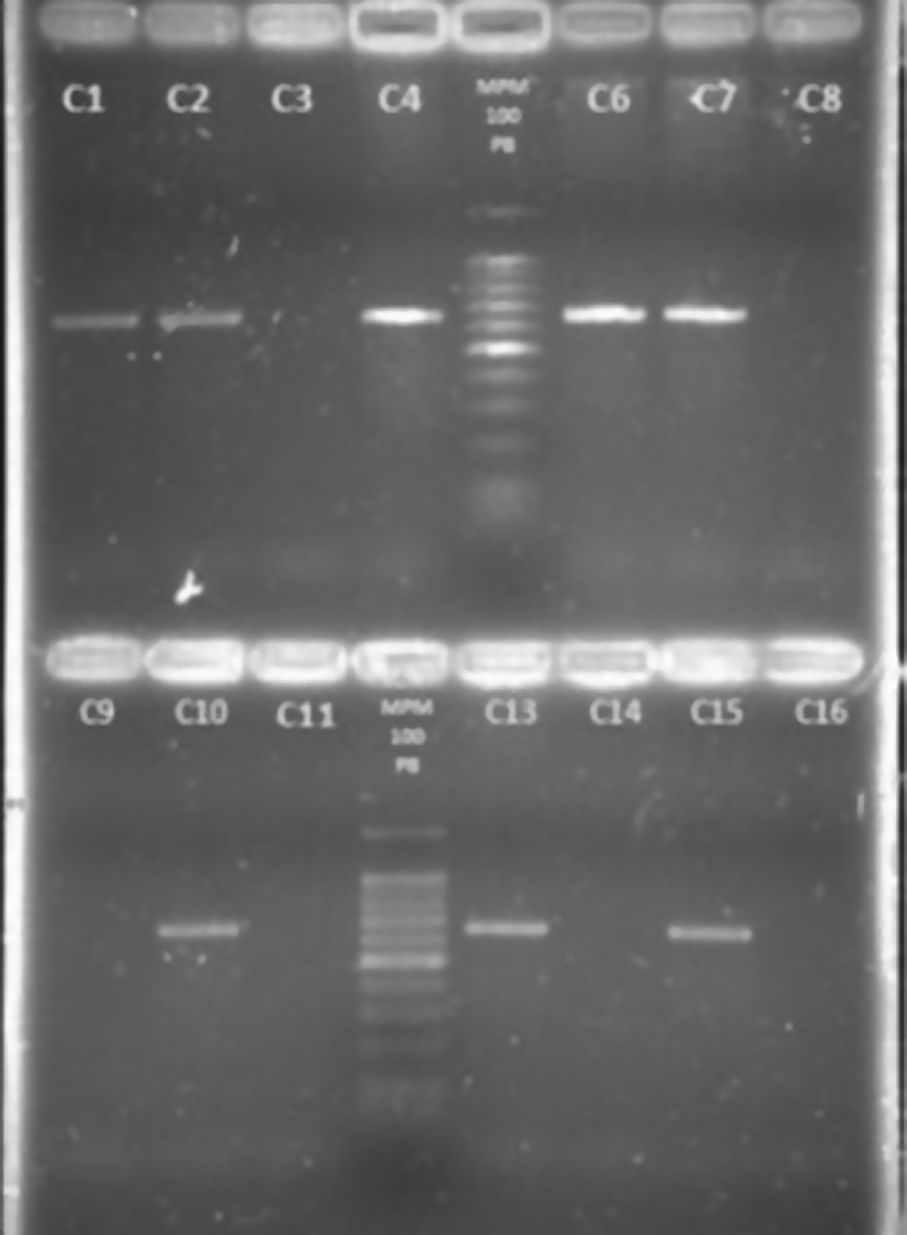
Las cepas coagulasa positivas que presentaron el gen nucA también presentaron el gen femB y las dos cepas coagulasa positivas que no presentaron el gen nucA (67F y 527F), tampoco presentaron el gen femB. Las cepas coagulasa negativas que presentaron el gen nucA (12F y 287N), no presentaron el gen femB (tabla 1 y fig. 2). En ambos ensayos se puede apreciar la amplificación de los controles ATCC (figs. 1 y 2).
Detección del gen femB. Electroforesis en gel de agarosa al 2%. Carriles: 1. cepa 211N; 2. cepa 6F; 3. cepa 67F; 4. cepa 106N; 5. marcador de peso molecular 100pb; 6. cepa 139N; 7. cepa 637F; 8. cepa 287N; 9. cepa 527F; 10. cepa 220N; 11. cepa 12F; 12. Marcador de peso molecular 100pb; 13. cepa 105N; 14. S. epidermidis ATCC 35983; 15. S. aureus ATCC 29213; 16. Control de reactivos.
La identificación de cepas de S. aureus es de gran importancia médica en los ámbitos hospitalarios y ahora también en la comunidad. Para esto se necesita contar métodos confiables y rápidos para identificar a este microorganismo.
Los medios de cultivo no son 100% seguros para identificar S. aureus, se necesitan siempre pruebas complementarias17. La prueba microbiológica más utilizada para la identificación de S. aureus es el ensayo para coagulasa que se puede realizar en portaobjeto o en tubo, sin embargo es recomendable hacerlo en tubo puesto que de este modo se detectan, tanto la coagulasa libre como la unida16. Se ha reportado que alrededor del 98% de las cepas de S. aureus dan positivo a la prueba de coagulasa. Entre un 10 y un 15% de las cepas de S. aureus pueden dar un resultado falso negativo. Algunas cepas de S. aureus producen fibrinolisina, la que puede lisar los coágulos que se han formado durante las primeras h. Si los tubos no son leídos durante las primeras cuatro h puede obtener un resultado falso negativo por lisis del coágulo. Si se utilizan kits comerciales de diferentes marcas para realizar la prueba de coagulasa se reportan alrededor de entre 10 y 30% de falsos positivos9,16. Además, también existen otras especies de estafilococos coagulasa positivos como S. intermedius, S. pseudointermedius y S. hyicus que pueden ser confundidos con S. aureus por la reacción de coagulasa18.
La termonucleasa (TNasa) es una proteína con un peso molecular de 17 Kda. Se trata de un endonucleasa, que degrada tanto al ADN como al ARN y la actividad enzimática puede resistir 100oC durante al menos 1h. La TNasa es una proteína bien caracterizada y el gen que la codifica es nucA. Una prueba enzimática de producción de TNasa es utilizada en muchos laboratorios para la identificación de S. aureus19. Sin embargo, la actividad TNasa no es específica de S. aureus, al igual que la coagulasa, S. intermedius, y S. hyicus son productores fuertes de esta enzima20.
Por otro lado, a pesar de que la proteína FemB se ha caracterizado en S. aureus, existen genes que codifican para proteínas parecidas a FemAB y que se han identificado en otros tipos de estafilococos, como S. epidermidis y S. haemolyticus con una función similar a las proteínas de S. aureus14.
Como se puede ver no existe una prueba cien por ciento segura que por sí sola nos permita identificar sin errores a S. aureus.
En nuestro caso se encontraron dos cepas coagulasa positivas que no presentaron el gen nucA, esto nos indica en principio que no se podrían clasificar en la especie S. aureus. Para corroborar los resultados se les detectó la presencia del gen femB y tampoco lo presentaron, por lo que de esta manera se infiere que estas cepas no eran de la especie S. aureus, a pesar de ser cepas coagulasa positiva, por lo que son otro tipo de estafilococos coagulasa positivos.
Entre las cepas coagulasa negativas analizadas se encontraron dos que presentaron el gen nucA, por lo que podrían tratarse de cepas de S. aureus que por alguna razón dan una prueba de coagulasa negativa, para confirmar esto se realizó la detección del gen femB, el cual no se detectó en estas cepas. Lo que nos indica que estas no son cepas de la especie S. aureus, por lo que son cepas coagulasa negativa que amplificaron para el gen nucA con los primers utilizados.
En las cepas analizadas no se encontraron cepas coagulasa negativas que presentaran el gen femB.
Con lo anterior podemos decir que bajo las condiciones estudiadas no existe una cepa coagulasa negativa, ni una cepa coagulasa positiva que no sea S. aureus que presenten tanto el gen nucA como el gen femB. Podemos decir que la detección de los dos genes en la misma cepa nos da una certeza de que se tiene una bacteria de la especie S. aureus, por lo que una detección de S. aureus implica la detección de ambos genes en el mismo microorganismo.
El siguiente paso es validar este método como una prueba diagnóstica rápida, realizando un análisis comparativo de su confiabilidad contra cepas ATCC para obtener valores predictivos positivo y negativo. También comparar costo/beneficio contra otros métodos ya establecidos entre los que se encuentran las galerías API y los agares cromogénicos21,22, así como realizar ensayos de campo en muestras biológicas.
FinanciaciónEste trabajo se realizó con apoyo de la UAM-Xochimilco.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores agradecen el apoyo técnico de Isaac Arteaga Larumbe.