Desde que en 1997 se demostrara la presencia de ADN fetal en sangre periférica materna, son numerosos los grupos que se dedican a investigar en este campo para intentar desarrollar e incorporar a la rutina clínica el diagnóstico prenatal no invasivo.
De la mano de la constatación de la presencia del ADN fetal en el torrente materno surgió el diagnóstico no invasivo del sexo fetal. Este y el estudio del RhD fetal han sido los únicos análisis incorporados a la rutina clínica.
Aunque existen grupos investigando en el campo del diagnóstico de las enfermedades mendelianas, los esfuerzos se han centrado en el diagnóstico de las aneuploidías fetales. En un principio, el alcance de los diagnósticos estuvo limitado por la presencia mayoritaria de ADN materno coexistiendo con el ADN fetal. Sin embargo, el desarrollo reciente de tecnologías mucho más sensibles, está permitiendo un avance vertiginoso de este campo.
Since the presence of foetal DNA in maternal peripheral blood was demonstrated in 1997, several research groups have developed their activity in this field in order to promote non-invasive prenatal diagnosis into clinical routine.
By demonstrating the presence of foetal DNA in the maternal bloodstream, the non-invasive assessment of the sex of the foetus was achieved. This test, together with the foetal RhD determination, has been the only ones incorporated into clinical routine.
Although there are research groups working on the diagnosis of Mendelian diseases, efforts have focused on the diagnosis of foetal aneuploidies. At first, the diagnostic scope was limited by the higher presence of maternal DNA co-existing with the foetal DNA. However, recent advances in more sensitive technologies is enabling dramatic progress to be made in this field.
El descubrimiento en 19971 de la presencia de ADN fetal circulante en sangre periférica materna abrió un nuevo campo de investigación aplicada, generando enormes expectativas acerca de la posibilidad de realizar diagnósticos prenatales sin necesidad de utilizar técnicas obstétricas invasivas para la obtención de material genético fetal.
La demostración de la existencia del ADN fetal se basó en la detección de secuencias del cromosoma Y en la sangre de la madre2. Fue de mano de esta constatación como surgió el primer test diagnóstico que se incorporó a la rutina clínica: el diagnóstico del sexo fetal en sangre materna. Este test, basado en un criterio de presencia/ausencia, se ha convertido en la actualidad en una gran herramienta para el manejo de embarazos en los que existe un riesgo de que el feto esté afecto de una enfermedad ligada al cromosoma X. Gracias a este análisis, a priori en el 50% de las gestaciones se evitarían las pruebas invasivas necesarias para llegar a un diagnóstico genético3.
Otro test, aunque de aplicación más orientada a la obstetricia, que se ha incorporado a la rutina clínica es el del diagnóstico del factor RhD fetal en aquellas gestantes con Rh(−) cuyas parejas son Rh(+). El conocimiento del factor Rh puede evitar la administración innecesaria de la inmunoprofilaxis en un 34% de los casos de gestantes Rh(−) (datos de laboratorio).
Son, sin embargo, muchas las potenciales aplicaciones diagnósticas que ofrece el estudio de esta material genético fetal que se encuentra en el torrente materno. La intención de los grupos que investigan en este campo siempre ha sido que este diagnóstico prenatal que se realiza de manera no invasiva (DPNI) llegara a sustituir el diagnóstico prenatal convencional invasivo4.
Una de las principales limitaciones para la incorporación definitiva del DPNI en la rutina clínica ha sido la coexistencia de ADN fetal y materno en el plasma de la madre. Estimaciones recientes, para las que se han empleado nuevas tecnologías mucho más sensibles, cifran la presencia de ADN fetal en tan solo un 9-20% del ADN libre circulante siendo el 80-90% restante de origen materno5. Considerando que la indicación mayoritaria para la realización del diagnóstico prenatal convencional es la sospecha de un feto con síndrome de Down, más aún con la incorporación del cribado combinado del primer trimestre, el enmascaramiento del ADN fetal por el ADN materno dificulta el diagnóstico de las aneuploidías de origen materno. Es en los últimos años, con el desarrollo de las nuevas tecnologías mucho más sensibles, cuando parece que estos diagnósticos están más cercanos6–10.
Menor atención se ha prestado al diagnóstico de las enfermedades de herencia mendeliana, aunque esta aplicación no debe ser subestimada dado que aproximadamente el 10% de los diagnósticos prenatales del primer trimestre se realizan por esta causa (datos de laboratorio). Y aunque, tal y como ocurre con el diagnóstico de las aneuploidías, la aplicación de las nuevas tecnologías ofrece un futuro prometedor para el diagnóstico de estas enfermedades independientemente del origen de la mismas; el diagnóstico de enfermedades con herencia paterna es una realidad dentro de la rutina de un laboratorio de genética11.
A lo largo de este trabajo se dará una visión sobre la situación actual del campo, así como del futuro, de la mano de las nuevas tecnologías.
Biología del ADN fetal circulante en sangre maternaLa presencia de ADN libre y circulante en sangre fue descubierta por primera vez en 1940 en pacientes con cáncer12. Basado en este hallazgo y en que la placenta durante el embarazo actúa como un tejido invasivo, el grupo de Lo et al. hipotetizó que el ADN fetal podría ser encontrado circulando en sangre materna1.
Para probar esta hipótesis la mejor estrategia fue buscar en sangre de la madre, con gestaciones de feto varón, secuencias del cromosoma Y cuya presencia solo podría estar justificada por tener un origen fetal2.
El ADN fetal circulante en sangre materna tiene dos características fundamentales:
- 1)
Su presencia en forma de fragmentos de pequeño tamaño (menor de 313pb). Esta peculiaridad está estrechamente relacionada con su origen. El ADN procede de las células apoptóticas placentarias y este proceso implica la fragmentación del ADN para así ser liberado al torrente sanguíneo13.
- 2)
Su coexistencia con ADN materno y baja proporción y aunque se aprecia un aumento en el ADN circulante a medida que progresa la gestación, el porcentaje de ADN fetal se cifró inicialmente entre un 3 y 6%2. Sin embargo estimaciones más recientes, basadas en resultados obtenidos con tecnologías más sensibles, consideran que las cantidades son de 9-20%5.
Diversos autores han centrado sus esfuerzos en desarrollar diferentes estrategias que permitieran distinguir, y por tanto aislar, el ADN fetal del materno.
Una de las primeras aproximaciones se basó en la peculiaridad del tamaño (diferencia de tamaño del ADN materno y fetal), e intentó separar el ADN fetal del materno en geles de electroforesis14. Sin embargo, ninguno de los grupos trabajando en el campo ha tenido éxito a la hora de reproducir estos resultados.
Otra estrategia utilizada para enriquecer la muestra fue la adición de formaldehído al tubo en el que posteriormente se recogía la muestra de sangre15. La base biológica de esta metodología es que el formaldehído mantiene intactas las membranas de las células, de manera que se disminuye la liberación del ADN de las células maternas al torrente sanguíneo y, por tanto, la proporción del ADN fetal no disminuye.
Sin embargo, la falta de reproducibilidad de estos resultados por otros grupos (incluido el nuestro) ha alejado estos métodos de su posible incorporación a la rutina clínica. En lo que sí parece existir un consenso es en el método de extracción del ADN presente en el plasma, siendo los kits QIAamp DNA Blood Mini Kit o QIAamp DSP Virus Kit (QIAGEN) los empleados mayoritariamente. Ambos métodos son sistemas de extracción manuales, lo que conlleva ciertas desventajas frente a los métodos automatizados como son mayor riesgo de, baja reproducibilidad y la dependencia de la experiencia/habilidad del manipulador16,17.
Una vez extraído el ADN y dependiendo del estudio a realizar, se pueden seguir distintas estrategias empleando distintas técnicas de análisis.
Diagnósticos que forman parte de la rutina clínicaDeterminación del sexo fetalLa determinación del sexo fetal se ha basado en la amplificación de secuencias del cromosoma Y. A pesar de la tendencia actual a establecer guías de buena praxis, en este campo todavía no hay ninguna publicada, de manera que son varios los protocolos que siguen los distintos servicios que han incorporado estos test diagnósticos. Así, son dos las regiones del cromosoma Y que se utilizan para el diagnóstico, secuencias de los genes SRY y DYS14, siguiendo un criterio de presencia/ausencia. La presencia en sangre materna de la secuencia del cromosoma Y se asocia a un feto varón por lo que el riesgo de que el feto esté afecto cambiaría del 25 al 50% y la ausencia, a un feto hembra3,18.
El test se realiza en el primer trimestre de gestación dado que en el caso de ser el feto varón, la gestante se podrá someter a un diagnóstico prenatal mediante biopsia corial. No existe, sin embargo, un consenso respecto a la semana de gestación en la que realizar esta prueba y cada uno de los servicios que ofrecen este análisis tiene establecido su propio protocolo. En nuestro servicio se estableció un protocolo de extracción de dos muestras, la primera en la semana 7-8 de gestación y la segunda dos semanas más tarde (semanas 9-10). Este protocolo fue el resultado de un estudio de validación en el que se observó que en muestras recogidas antes de las semana 7 de gestación la fiabilidad de la técnica era del 80% debido a la existencia de falsos negativos. Sin embargo, entre la semana 7 y la 12 la eficacia de la técnica resultó ser superior al 99%. Frente a la falta de uniformidad en cuanto a la edad gestacional en la que realizar el estudio sí que existe un consenso en cuanto a la técnica empleada, la PCR a tiempo real. El estudio incluye, junto con el estudio de presencia/ausencia del cromosoma Y, la amplificación en paralelo con un control interno que certifique la presencia de ADN en la muestra3.
Todas las enfermedades ligadas al cromosoma X son susceptibles de ser indicación clínica para el test de determinación del sexo fetal. Así, desde que en 2008 se incorporó en nuestra unidad este estudio, se han analizado 194 muestras de gestantes englobando un total de 26 enfermedades diferentes (fig. 1). En aproximadamente el 50% de los casos se evitó la necesidad de una biopsia corial para llegar a un diagnóstico fetal.
Determinación del factor RhD fetalLa determinación del factor RhD fetal ha tenido más proyección en el campo de la Obstetricia y Ginecología que en el campo de la Genética.
La inclusión de esta prueba en el manejo de los embarazos de gestantes Rh(−) permite la supresión del tratamiento de inmunoprofilaxis en aquellas gestaciones en las que se diagnosticaba el feto como Rh(−) y en las que, por tanto, ya no existe riesgo de desarrollar la enfermedad hemolítica del recién nacido (HDN).
La técnica utilizada en este caso es también la PCR a tiempo real y el protocolo de análisis incluye la amplificación de los exones 5, 7 y, ocasionalmente, exón 10 del gen RhD. Se utiliza la combinación de estos exones por la posibilidad que ofrecen de detectar algunas de las variantes Rh existentes en población caucásica19–23.
En un estudio realizado en el Servicio de Genética de la Fundación Jiménez Díaz, en población mayoritariamente caucásica, este test mostró una sensibilidad del 100% y una especificidad cercana al 98%. En el total de las 141 muestras estudiadas en el estudio de validación no se observó ningún falso negativo, aunque hubo un caso de falso positivo. Frente a la grave repercusión clínica de desarrollar HDN que supondría un falso negativo, los «falsos positivos» no suponen riesgo alguno para la gestación ya que, la gestante recibiría la inmunoprofilaxis de forma innecesaria pero sin que ello representara ningún riesgo para ella ni para el feto.
Estudio de enfermedades monogénicasEl estudio en enfermedades monogénicas se ha encontrado limitado hasta la fecha a las enfermedades de herencia paterna, debido a la presencia mayoritaria de ADN materno en la muestra de plasma. Sin embargo, este es un diagnóstico que no ha de ser subestimado ya que puede mejorar el manejo de las gestaciones en la que hay un riesgo de transmisión de una enfermedad genética.
Enfermedades de herencia dominanteEn aquellas parejas en las que el varón es portador de una mutación asociada a una enfermedad de herencia dominante, la ausencia de dicho defecto en el genoma materno permite realizar el diagnóstico fetal en sangre materna. La presencia o ausencia del alelo mutante paterno en sangre materna nos permitirá concluir si el feto es afecto o sano respectivamente para dicha enfermedad.
El abordaje diagnóstico se puede realizar tanto por método directos como indirectos, por lo que el abanico de técnicas que se pueden utilizar es amplio.
La enfermedad de Huntington (CH)24–27, distrofia miotónica28 y distonía de desarrollo precoz29 son ejemplos de enfermedades de herencia dominante estudiadas en plasma materno.
Como centro de referencia para el diagnóstico de CH, nuestro grupo ha realizado hasta la fecha un total de 13 DPNI de enfermedad de Huntington (6 de ellos publicados) mediante estudio directo del alelo expandido y/o mediante estudio indirecto por haplotipos. Ambas aproximaciones pueden realizarse en el primer trimestre de gestación con una alta precisión. Solo en dos de los casos estudiados no pudo llegarse a un diagnóstico dado que no se detectó ni el alelo sano ni el afecto. Uno de los casos puede explicarse por el alto, e infrecuente, número de repeticiones que presentaba el alelo expandido(>400pb) y por tanto de mayor longitud que los fragmentos de ADN fetal libres circulantes en sangre materna.
Para todos estos diagnósticos de CH se han utilizado los mismos abordajes que se encuentran disponibles en un laboratorio de rutina: estudio directo de la expansión e indirecto por marcadores microsatélite mediante QF-PCR. Se ha buscado con esta aproximación que la incorporación del diagnóstico no invasivo a la rutina clínica fuese lo más sencilla posible.
Otra opción en la que el DPNI en sangre materna puede suponer una alternativa al DP es en el caso de gestaciones con sospecha ecográfica de una enfermedad dominante ex novo, por ejemplo acondroplasia30–33. La acondroplasia es una enfermedad que cuando surge ex novo está asociada a la edad paterna y además existe una mutación prevalente (G1138A). En la gran mayoría de los casos se descubre la afectación fetal en un reconocimiento ecográfico en el tercer trimestre de gestación y la confirmación genética se suele hacer al nacimiento.
El estudio en plasma materno puede permitir la confirmación del diagnóstico ecográfico. Aunque el manejo del embarazo no se va a ver afectado, este diagnóstico tiene la ventaja de que, en caso de detectarse la mutación, la pareja puede contar con un periodo de adaptación psicológica antes del parto.
Son numerosos los artículos en los que, aplicando distintas metodologías, se puede detectar esta mutación en plasma materno34–37.
Enfermedades de herencia recesivaEl número de casos de herencia recesiva que se han publicado es algo mayor. En este caso, para el abordaje en sangre materna parten de una premisa importante: la mutación portada por ambos progenitores ha de ser distinta para que sea posible identificar la mutación de origen paterno en la sangre de la madre.
Al igual que los estudios anteriormente detallados, estos se basan en un criterio de presencia/ausencia del defecto paterno. Sin embargo, al contrario que en el caso de enfermedades de herencia dominante, en este caso no podremos llegar a un diagnóstico completo de la condición fetal al no poder conocer su carácter de sano o portador respecto a la mutación materna. A pesar de ello, el estudio de la mutación paterna pueda evitar la necesidad de un DP convencional en los casos en los que el DPNI en sangre materna revele que el feto no ha heredado el defecto paterno. Por el contrario, en caso de detectar la mutación de origen paterno, será necesario el DP invasivo para conocer el estatus fetal respecto a la mutación materna y, por tanto, si es afecto o únicamente portador de la enfermedad de estudio.
Ejemplos de estas enfermedades son: hiperplasia adrenal congénita35, β-talasemia38–48, fibrosis quística49–51, amaurosis congénita de Leber52 y acidemiapropiónica53.
Aunque la hiperplasia adrenal congénita tiene un carácter recesivo, es una enfermedad ligada al sexo, por lo que el primer estudio no invasivo que se suele hacer es el estudio del sexo fetal. Esta enfermedad se manifiesta con un exceso de andrógenos que no afectan a los fetos masculinos pero sí a los femeninos. Así, la determinación previa del sexo fetal indica si debe haber tratamiento prenatal o no. Solo un grupo ha publicado el estudio de la mutación paterna.
Para el diagnóstico de la fibrosis quística se han documentado tres estrategias diferentes: análisis de fragmentos de restricción, PCR alelo específica y secuenciación de una única base (minisecuenciación). La ventaja de esta última técnica frente a las demás es su fácil manejo así como su alta sensibilidad, lo que ha permitido diagnósticos más tempranos.
La amaurosis congénita de Leber y la acidemia propiónica pertenecen a un grupo de enfermedades con un amplio espectro mutacional. Esto hace de ellas enfermedades potencialmente diagnosticables en sangre materna, ya que hay una alta probabilidad de que ambos progenitores sean portadores de mutaciones diferentes.
Estudios confirmatorios tras diagnóstico genético preimplantacionalEsta es una opción que está cobrando cada vez más importancia. Tanto es así, que en las últimas guías publicadas por la ESHRE en 2010 se considera el diagnóstico prenatal no invasivo (punto 3.28) como una opción para gestantes tras un proceso de diagnóstico genético preimplantacional54.
Perspectiva de los pacientesLos pacientes han mostrado siempre gran interés en el DPNI por la posibilidad que les ofrece de evitar las técnicas invasivas. En nuestra experiencia, perciben este diagnóstico como una opción real (especialmente aquellos que han participado previamente en estudios de investigación) y están solicitando la realización de este test para tomar una posterior decisión con respecto a la realización de DP en función del resultado.
Estudio de aneuploidías fetalesAunque el diagnóstico en ADN fetal en sangre materna se conoce como DPNI, es interesante remarcar que en los últimos años se está introduciendo la terminología de cribado no invasivo de aneuploidías (screening). Sin embargo, no conviene olvidar que diagnóstico prenatal implica la determinación de una anomalía genética en el feto, mientras que el cribado permite una estimación de riesgo de que un feto presente una anomalía generalmente cromosómica.
Desde que se empezó a estudiar el material genético fetal presente en sangre materna, han sido muchos los esfuerzos empleados en intentar desarrollar un método diagnóstico para la trisomía 21. El primer hito que supuso un paso de gigante en estas investigaciones fue un descubrimiento biológico, la presencia de ADN fetal en sangre materna. Y el segundo y más reciente es el desarrollo de las nuevas tecnologías más sensibles, mereciendo especial atención la secuenciación masiva.
No cabe ninguna duda de que gracias a la aplicación de la secuenciación masiva se pueden detectar las trisomías 13, 18 y 21 en sangre materna. Son numerosos los artículos que describen estudios poblacionales donde se valida la técnica con estos fines diagnósticos. Sin embargo, cabe destacar que ninguno de los artículos publicados hasta el momento defienden el uso de esta técnica como herramienta diagnóstica, sino que defienden su utilización como herramienta de cribado6–10.
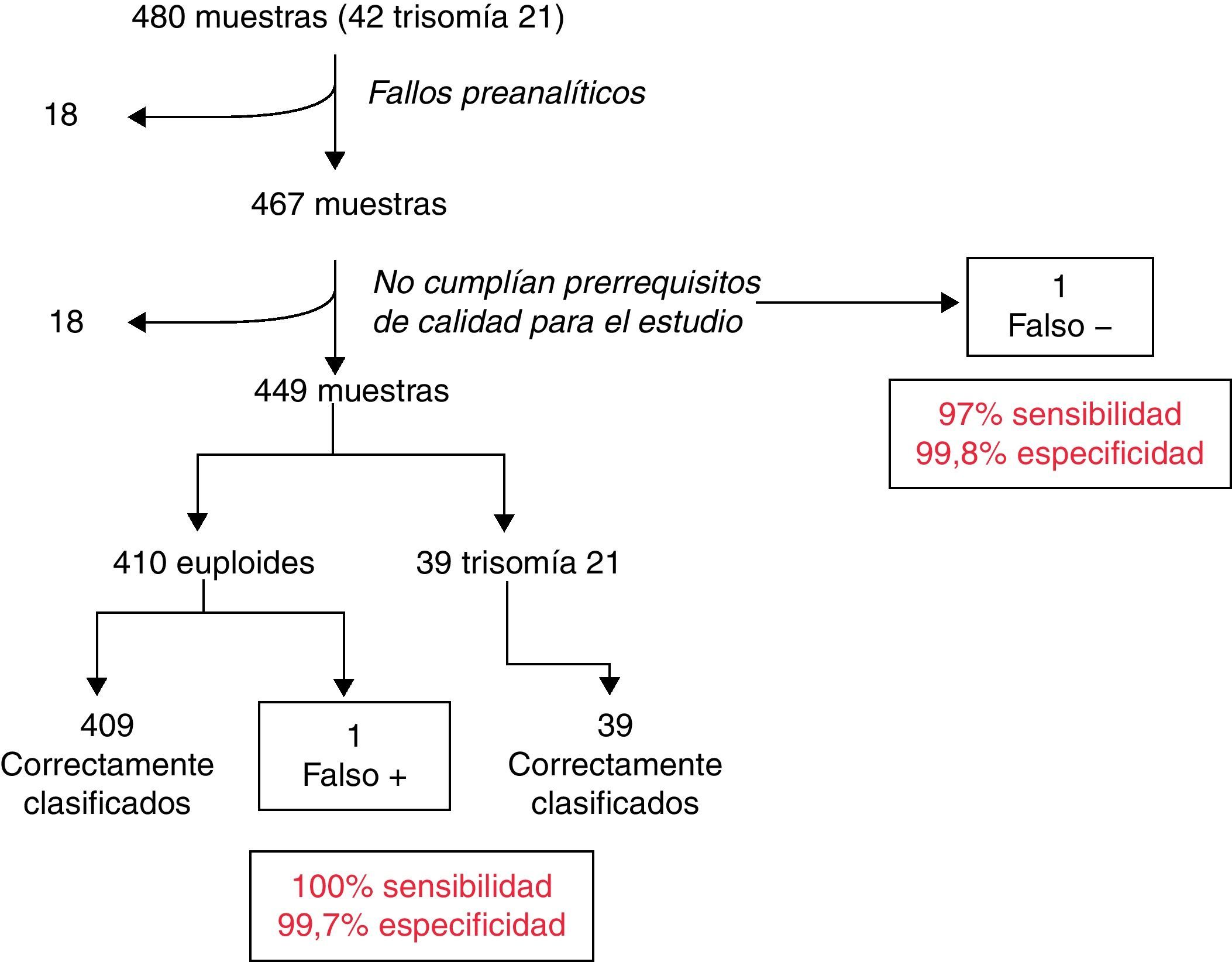
Entre todos los artículos recientes, merecen especial atención y análisis los publicados por el grupo de investigadores de la empresa Sequenom, empresa que ya ha comercializado el test8–10. En el primer estudio publicado por Ehrich et al.8 se selecciona una población de 480 muestras de gestantes con riesgo elevado de trisomía 21 fetal, analizándose un total de 449. Todas las muestras fueron recogidas entre las semanas 8 y 36 de gestación. Trece de las muestras se descartaron de forma previa al análisis por problemas fundamentalmente de cantidad, lo que en la rutina no supondría un problema ya que se puede solicitar una nueva muestra a la gestante. Posteriormente se descartaron otras 18 muestras debido a que no cumplían con unos criterios de calidad establecidos para el estudio (fig. 2). Independientemente de la aplicación de los criterios de calidad en las muestras, los resultados obtenidos de sensibilidad y especificidad se sitúan por encima del 97%. Estos valores son, indiscutiblemente, muy buenos para una técnica que se quiere utilizar como herramienta de cribado. Sin embargo, el estudio no se ajusta a la tendencia actual que se aplica a las herramientas de cribado, que es su aplicación temprana en el embarazo, ya que los resultados se han obtenido a lo largo de toda la gestación y sería necesario saber si los datos en muestras únicamente del primer trimestre serían igualmente buenos. Es de especial interés la defensa de los autores de que esta técnica se aplique a gestaciones en riesgo, cuando actualmente la prevalencia de fetos con síndrome de Down nacidos vivos se concentra fundamentalmente en el grupo de bajo riesgo. Además, el éxito de las herramientas de cribado se ha basado en la universalidad de las mismas y no en su aplicación en un grupo poblacional concreto. Se defiende en el estudio, además, que este debe estar complementado con los reconocimientos ecográficos de rutina. Este reconocimiento rutinario han permitido en la Unidad de Diagnóstico Prenatal de la Fundación Jiménez Díaz (datos de la Unidad) la sospecha ecográfica de un 80% de las anomalías cromosómicas diagnosticadas en el primer trimestre de gestación mediante vellosidad corial, correspondiendo solo la mitad de ellas a fetos portadores de trisomía 21 (fig. 3).
Datos del estudio poblacional realizado por Ehrich et al.8.
En un estudio posterior de este mismo grupo9 se restringe el periodo gestacional en el que se realiza el estudio: el 97% de las muestras (4.664 en total) se recogió entre las semanas 11 y 20 de gestación, correspondiendo un 37% de las mismas a muestras del primer trimestre de gestación. La tasa de detección alcanzada en este estudio es de un 98,6% con una tasa de falsos positivos de 0,2%, y concluye el estudio que ante un resultado positivo el riesgo de tener un feto con síndrome de Down se multiplica por 490, mientras que ante un resultado negativo, el riesgo se reduce en un múltiplo de 72. Nuevamente, estos resultados son de indudable valor para una herramienta de cribado, sin embargo, siguen planteando ciertas limitaciones. En el material on line asociado a este artículo, se presentan concienzudos estudios de valoración de la cantidad de ADN fetal en gestaciones euploides y gestaciones de fetos con síndrome de Down frente a distintos factores que podrían influir en la obtención de un diagnóstico. Así, se observa una relación significativa e inversamente proporcional entre la cantidad de ADN fetal y el peso de la gestante, dificultando por tanto los diagnósticos. Este factor se ha observado igualmente con las herramientas de cribado que se usan actualmente, hasta el punto de que se utilizan factores de corrección en función del peso de la gestante. Otro de los factores que influyen en la detección del cromosoma 21 es la edad gestacional. Estudios comparativos entre gestaciones de fetos euploides y fetos con trisomía 21 revelaron que no había relación significativa entre la fracción de ADN fetal en la muestra y la semana gestacional en ninguno de los grupos. Sin embargo, sí que existía una relación significativa entre la edad gestacional y el recuento del cromosoma 21 (z-score) en gestaciones con feto con síndrome de Down, no en gestaciones euploides, siendo mayor el recuento a lo largo de la gestación. Esto se traduce en una más fácil o menos difícil detección de los fetos con trisomía 21 a medida que avanza el embarazo (fig. 4).
Figura tomada de los datos disponibles on line del estudio de Palomaki et al.10.
El último artículo referente al diagnóstico de aneuploidías10 incluye, junto a la trisomía 21, el análisis de las trisomías 13 y 18.
Futuro de los estudios no invasivos en sangre maternaNo cabe duda de que, tras el descubrimiento de la presencia de ADN fetal en sangre materna, el reciente desarrollo de la secuenciación masiva está suponiendo un nuevo hito dentro del campo del DPNI.
Estudios del grupo de Lo11 han demostrado mediante la aplicación de la secuenciación masiva que todo el genoma fetal se encuentra representado en el plasma materno a pesar del grado de fragmentación que presenta en él. Este hecho implica que a priori cualquier defecto genético del feto puede ser estudiado en sangre materna.
Este avance es más significativo para las anomalías cromosómicas numéricas, ya que mayoritariamente estas son de origen materno; mientras que en el caso de las enfermedades monogénicas, viene a ampliar el estudio a las enfermedades de origen materno, ya que las de origen paterno ya estaban siendo diagnosticadas.
Sin embargo, parece existir un cierto giro en cuanto al modo de utilización de estos diagnósticos. Cuando surgió este campo de investigación, el fin era que llegara a sustituir los diagnósticos invasivos, para lo cual debe llegarse a niveles de sensibilidad y especificidad del 100%. Al no alcanzarse estos valores, se está empezando a hablar del diagnóstico no invasivo, ya no como una herramienta diagnóstica, sino de cribado.
Sin embargo, es importante que en paralelo a la publicación de estos nuevos diagnósticos se desarrolle un adecuado consejo genético. En ningún caso parece que, por el momento, este diagnóstico vaya a sustituir a las pruebas obstétricas invasivas ya que siempre encontraremos casos en los que haya marcadores ecográficos y el feto no tenga síndrome de Down, por lo que será necesario realizar estudios exhaustivos posteriores. Sería posible la sustitución si el estudio del genoma completo fetal fuera factible, sin embargo, y considerando que solo la secuenciación dirigida (target sequencing) ha demostrado su validez clínica en estudios postnatales, no parece que a corto plazo esto vaya a ser posible.
Por otro lado al estar hablando de técnicas que se van a incorporar en la rutina diagnóstica hay que tener en consideración los costes de las pruebas. A día de hoy, el coste de la secuenciación masiva es elevado y en el caso concreto del diagnóstico de aneuploidías en sangre materna, hay unos costes añadidos derivados de la patente que Sequenom tiene para este diagnóstico concreto.
Un nuevo futuro está en el horizonte dentro del campo del diagnóstico no invasivo en sangre materna, aunque aún hay obstáculos que salvar.
FinanciaciónSara Perlado posee una beca de la Fundación Conchita Rábago de Jiménez Díaz. Este proyecto está financiado por el Instituto de Salud Carlos III (PI08/1456).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los datos del grupo son derivados de los sucesivos proyectos de investigación subvencionados por el ISCIII (PI081456). Parte de los datos han sido hechos públicos con anterioridad, todos debidamente referenciados a lo largo del texto.