Los adenomas hipofisarios son tumores infrecuentes de diagnóstico complejo, cuya heterogeneidad y baja incidencia dificultan estudios a gran escala. El Registro Molecular de Adenomas Hipofisarios (REMAH) nació en 2008 en el seno de la Sociedad Andaluza de Endocrinología y Nutrición (SAEN), como estrategia de cooperación clínico-básica y multicéntrica, para mejorar el diagnóstico y tratamiento de tumores hipofisarios mediante la combinación de información clínica, anatomopatológica y molecular. En 2010, la Sociedad Española de Endocrinología y Nutrición (SEEN) lo extendió a nivel nacional, estableciendo 6 nodos con protocolos y métodos comunes de recogida de muestras y datos clínicos, análisis molecular y anotación en un mismo registro (www.remahnacional.com). El registro combina datos clínicos con el fenotipado molecular del adenoma intervenido, mediante PCR cuantitativa en tiempo real de la expresión de 26 genes: hormonas hipofisarias (GH-PRL-LH-FSH-PRL-ACTH-CGA), receptores (somatostatina, dopamina, GHRH, GnRH, CRH, arginina-vasopresina, ghrelina), otros marcadores (Ki67, PTTG1) y genes de control. Hasta 2015 se ha obtenido información molecular de 704 adenomas, de los 1.179 pacientes registrados. Esta estrategia permite abordar análisis comparativos y relacionales entre el perfil molecular de los distintos tipos de adenomas y el fenotipo clínico del paciente, lo que puede ofrecer un mejor conocimiento de la enfermedad y, potencialmente, ayudar en la selección del tratamiento. El REMAH constituye una red única, multicéntrica e interdisciplinar, cimentada en una base de datos compartida, que aporta un enfoque traslacional de gran proyección potencial para el manejo de los adenomas hipofisarios y abre el camino para estudios conjuntos clínico-básicos innovadores con un elevado número de pacientes.
Pituitary adenomas are uncommon, difficult to diagnose tumors whose heterogeneity and low incidence complicate large-scale studies. The Molecular Registry of Pituitary Adenomas (REMAH) was promoted by the Andalusian Society of Endocrinology and Nutrition (SAEN) in 2008 as a cooperative clinical-basic multicenter strategy aimed at improving diagnosis and treatment of pituitary adenomas by combining clinical, pathological, and molecular information. In 2010, the Spanish Society of Endocrinology and Nutrition (SEEN) extended this project to national level and established 6 nodes with common protocols and methods for sample and clinical data collection, molecular analysis, and data recording in a common registry (www.remahnacional.com). The registry combines clinical data with molecular phenotyping of the resected pituitary adenoma using quantitative real-time PCR of expression of 26 genes: Pituitary hormones (GH-PRL-LH-FSH-PRL-ACTH-CGA), receptors (somatostatin, dopamine, GHRH, GnRH, CRH, arginine-vasopressin, ghrelin), other markers (Ki67, PTTG1), and control genes. Until 2015, molecular information has been collected from 704 adenomas, out of 1179 patients registered. This strategy allows for comparative and relational analysis between the molecular profile of the different types of adenoma and the clinical phenotype of patients, which may provide a better understanding of the condition and potentially help in treatment selection. The REMAH is therefore a unique multicenter, interdisciplinary network founded on a shared database that provides a far-reaching translational approach for management of pituitary adenomas, and paves the way for the conduct of combined clinical-basic innovative studies on large patient samples.
Los tumores hipofisarios representan para la Medicina actual un reto complejo y relevante, cuyo abordaje requiere una aproximación conjunta e integrada, coordinadas desde la Endocrinología, Neurocirugía, Anatomía Patológica, Radiología y otras disciplinas de apoyo. En este contexto, la información generada en las últimas décadas por la Biología Celular y Molecular sobre los adenomas hipofisarios está ofreciendo un aporte sustancial para avanzar en el conocimiento de este conjunto de enfermedades y en el diseño de nuevos marcadores de diagnóstico y pronóstico, así como de nuevas dianas y estrategias terapéuticas. Sin embargo, existe aún una brecha importante entre los estudios moleculares de investigación básica y el manejo clínico de los pacientes con tumores hipofisarios, que dificulta una repercusión más rápida y efectiva de los descubrimientos científicos sobre la salud del paciente. Con objeto de acortar esta distancia, se ha generado una iniciativa de colaboración clínico-básica en el estudio de tumores hipofisarios, cuyo punto de partida es la creación de un registro de tumores que incluye, junto con los datos clínicos de los pacientes, el análisis molecular de la pieza del tumor hipofisario obtenido tras la intervención, en el que se evalúan los niveles de expresión de un conjunto de genes seleccionados por su potencial valor diagnóstico y por la posible utilidad clínica de la información que proporcionan. En este artículo se describen las líneas generales que han permitido generar esta iniciativa, denominada Registro Molecular de Adenomas Hipofisarios (REMAH), así como las principales características del fenotipado molecular.
Actualmente se acepta que los adenomas hipofisarios se generan por expansión clonal de un tipo concreto de célula adenohipofisaria1–3; los síndromes clínicos resultantes derivan de la producción de una o varias hormonas, o bien son consecuencia de su crecimiento local4,5. Así, entre los principales tipos de adenomas hipofisarios se incluyen los somatotropinomas, productores de hormona del crecimiento (GH)6, prolactinomas, productores de prolactina (PRL)7, corticotropinomas productores de adrenocorticotropina (ACTH)8, tirotropinomas, productores de tirotropina (TSH)9, gonadotropinomas, productores de hormona luteinizante (LH) u hormona foliculoestimulante (FSH)10 y los adenomas hipofisarios no funcionantes11, derivados mayoritariamente del linaje gonadotropo, productores de subunidad alfa de las glicoproteínas (CGA).
A primera vista, la clasificación de los adenomas hipofisarios parece relativamente simple y genérica, lo que podría indicar que el diagnóstico de estos tumores es sencillo; sin embargo, no es así. De hecho, con frecuencia aparecen células hipofisarias con secreción mixta de 2 o más hormonas, o distintas poblaciones celulares, que provocan síndromes combinados6,7,9–13. Además, pueden presentarse adenomas silentes (distintos de los no funcionantes mencionados anteriormente), con poca o nula expresión hormonal, y otros con funcionalidad cíclica, todos ellos de muy difícil detección, por lo que resulta inestimable disponer de un detallado análisis anatomopatológico para un mejor diagnóstico de estas enfermedades14,15. Todo ello, a su vez, subraya la necesidad de mejorar las herramientas disponibles actualmente para mejorar el diagnóstico y fenotipado clínico de los adenomas hipofisarios.
La secreción hormonal de las células hipofisarias y muchas de sus funciones tróficas (supervivencia, proliferación y mantenimiento de patrones específicos de expresión génica) están reguladas de forma primaria por hormonas hipotalámicas hipofisiotrópicas, un conjunto de péptidos neuroendocrinos de naturaleza estimuladora, como GHRH, GnRH, CRH y TRH, o bien inhibidora, como la somatostatina. Existen además reguladores hipotalámicos primarios no peptídicos, como el potente inhibidor dopamina4,5,16–18. Todas estas hormonas actúan sobre sus células diana a través de receptores de membrana específicos, modulando rutas de transducción de la señal y proteínas de la ruta secretora. Aunque la presencia de estos receptores está alterada en muchos adenomas4,5,16–21, la mayoría de los tumores expresan en mayor o menor medida receptores de las hormonas inhibidoras somatostatina y dopamina, que han servido como diana para el uso de fármacos en el tratamiento de varias enfermedades hipofisarias19,22–24.
Junto con los reguladores primarios mencionados, existen otros factores con capacidad para regular la función hipofisaria, como la ghrelina, cuyo receptor, GHSR1a, se expresa abundantemente en la hipófisis25–29. El papel de AVP y sus receptores (AVPR1a, AVPR1b y AVPR2) a nivel hipofisario es clave para el mantenimiento de la homeostasis basal y en respuesta al estrés30; de hecho, recientes estudios han demostrado el valor clave de AVPR1b en la enfermedad de Cushing31,32. La securina (PTTG1) participa en la transformación celular de hiperplasia a adenoma, y su presencia se relaciona con angiogénesis33,34; el biomarcador Ki67, se ha empleado como marcador inmunohistoquímico de proliferación celular, si bien su uso y utilidad real son aún controvertidos33,34. Profundizar en el conocimiento de este conjunto de moléculas y sus alteraciones en adenomas hipofisarios puede ayudar a entender mejor la enfermedad y a tomar decisiones más adecuadas con respecto a su tratamiento y seguimiento. Asimismo, la identificación en estos tumores de nuevas moléculas reguladoras permitiría ensayar su efecto y el de nuevos análogos en dichos tumores, e incrementar así el espectro de adenomas que pueden ser tratados farmacológicamente.
A la luz de la heterogeneidad de los tumores hipofisarios y de la importancia de conocer su perfil molecular, en 2008 se inicia el proyecto REMAH en el seno del grupo trabajo de Neuroendocrinología de la Sociedad Andaluza de Endocrinología y Nutrición (SAEN), concebido y desarrollado entre investigadores básicos y clínicos, con el objetivo de generar una información y un servicio útil para el endocrinólogo y su equipo neuroendocrino en el manejo de pacientes con tumores hipofisarios. En 2010, este proyecto suscitó el interés del grupo de trabajo de Neuroendocrinología de la Sociedad Española de Endocrinología y Nutrición (SEEN), que decidió apoyar y hacer suyo el proyecto desde su Fundación (FSEEN) y con el respaldo del patrocinio de Novartis. De este modo, el REMAH tomó un alcance nacional y se organizó en 6 nodos, coordinado cada uno de ellos por un investigador básico con experiencia molecular y un investigador clínico con experiencia en la atención de pacientes con enfermedad hipofisaria neuroquirúrgica. A cada nodo se adscriben los investigadores responsables de la atención y seguimiento de los pacientes, así como los investigadores responsables del fenotipado molecular. La distribución de los nodos es la siguiente: Andalucía (Córdoba), Comunidad de Madrid (Madrid), Comunidad Valenciana (Alicante), Galicia (Santiago de Compostela), Cataluña (Barcelona) y País Vasco (Bilbao). El estudio molecular que se realiza en cada nodo comprende los mismos 26 genes, cuyos niveles de expresión son analizados mediante un método sistematizado y estandarizado de registro y medida. La medida de la expresión génica aporta un mejor conocimiento de la enfermedad a nivel molecular y una posible ayuda para mejorar la decisión terapéutica. Además, como objetivo complementario, el proyecto REMAH plantea recoger los resultados moleculares de cada tumor hipofisario, junto con los datos clínicos del paciente, en un registro común, que proporcionará a todos los investigadores participantes una plataforma de investigación con una casuística importante de una enfermedad de baja prevalencia, todo lo cual puede ayudar a que la Neuroendocrinología española mejore su nivel de competitividad científica internacional.
Desde una perspectiva científica, el objetivo general del REMAH consiste en determinar la expresión en tumores hipofisarios de diferentes genes correspondientes a receptores, hormonas y otras proteínas reguladoras para su mejor diagnóstico. Ello permite el fin adicional de estudiar la relación de dichos parámetros con las características clínico-patológicas de los pacientes y analizar así su posible papel en el desarrollo y respuesta patológica de los adenomas hipofisarios. En concreto, el análisis de proteínas relacionadas en el control de la secreción hormonal, supervivencia o apoptosis celular que puedan afectar estas condiciones, hace concebible que se pueda establecer el valor potencial y la relevancia de las moléculas y señales analizadas como dianas de tratamiento, así como obtener unos marcadores tempranos de diagnóstico de estas enfermedades. De esta forma, se pretende poner en valor dichos datos para los investigadores clínicos y básicos implicados en el estudio.
Para alcanzar este objetivo general, se han planteado los siguientes objetivos específicos: 1) Evaluar la expresión de las hormonas hipofisarias y de los principales receptores de hormonas hipotalámicas, dando prioridad a los relativos a la enfermedad evaluada: somatostatina (sst1, sst2, sst3 y sst5), dopamina (DRD1, DRD2T, DRD2L, DRD4 y DRD5), GHRH (GHRH-R), CRH (CRH-R1), AVP (AVPR1b) y ghrelina (GHSR1a). 2) Estudiar la expresión de otras proteínas reguladoras de la secreción o de la proliferación y muerte celular, que pueden ser usadas como marcadores o como nuevas dianas de tratamiento, PTTG1 y Ki67. 3) Determinar la posible relación entre los niveles de expresión de estos receptores y moléculas de interés con la respuesta de los pacientes a tratamientos farmacológicos específicos para los diferentes tipos de tumores hipofisarios, y con las características clínicas y la evolución de dichos pacientes. En concreto, el objetivo de la presente publicación es detallar el funcionamiento general del REMAH, su metodología y la muestra tumoral recogida hasta la fecha.
Materiales y métodosReclutamiento de pacientes. Consentimiento informadoEl estudio REMAH se lleva a cabo de acuerdo con los principios de la Declaración de Helsinki y cuenta con la aprobación de los comités de ética de los hospitales participantes. Previamente a la inclusión de cada paciente, el investigador ha obtenido su consentimiento informado.
La metodología y diseño del estudio REMAH consta de 4 fases:
Preparación del casoDiagnósticoSe han tenido en cuenta los siguientes factores:
- •
Identificación funcional del tumor y de sus características clínicas específicas.
- •
Evaluación del tipo de respuesta a estímulos, potenciadores o supresores de la secreción hormonal en tumores funcionales.
- •
Identificación de secuelas del tumor relativas al resto de la estructura hipotálamo-hipofisaria.
- •
Evaluación de la respuesta farmacológica preoperatoria (análogos de somatostatina, inhibidores de la esteroidogénesis adrenal, etc.), tipo de fármaco, duración y efecto en la secreción hormonal (si es aplicable).
- •
Terapia de sustitución de déficits asociados previamente al tratamiento quirúrgico.
- •
Evaluación de la necesidad del uso de terapia con esteroides durante la cirugía.
- •
Toma de una muestra de sangre antes del tratamiento farmacológico o quirúrgico.
- •
Decisión y programación de los procedimientos quirúrgicos.
- •
Admisión hospitalaria.
- •
Recogida de datos de la historia clínica, siguiendo la hoja de datos que acompaña al protocolo REMAH, que se incluye en la correspondiente base de datos. En concreto se recogen los niveles, medidos en sangre circulante en ayunas, de: glucosa (mg/dL), HbA1c (%), colesterol (mg/dL), LDL (mg/dL), HDL (mg/dL), GH basal (ng/mL), PRL (mU/ml), ACTH (pg/ml), LH (mU/ml), FSH (mU/ml), TSH (μU/ml), CGA (mU/mL), IGF-I (ng/mL), T4 libre (ng/l) y cortisol (μg/dL). Informe tras cirugía: curación tras la intervención y control farmacológico en caso de no haber curación. Datos de imagen: tamaño de adenoma (<1cm, microadenoma o >1cm, macroadenoma), extensión extraselar, supraselar e infraselar, características en secuencias T1 y T2 de la RNM.
- •
Cabe mencionar que el registro permite, y tiene entre sus objetivos de futuro, desarrollar un seguimiento detallado del paciente después del tratamiento quirúrgico o bajo tratamiento médico, incluyendo la recogida de muestras de sangre a los 6 y 12 meses.
La toma de muestras se organiza según un procedimiento estandarizado. Cada centro participante aplica un sistema de coordinación propio que asegura una recogida adecuada de muestras de todos los tumores intervenidos en dicho centro, según los requisitos del procedimiento acordado. La coordinación y supervisión de los procedimientos depende de los investigadores responsables tanto de la recogida de muestras como de los datos clínicos correspondientes.
- 1.
La pieza quirúrgica se trata como una muestra «intraoperatoria» para Anatomía Patológica, evitando cualquier tipo de retraso en la recogida. En el quirófano de neurocirugía, la muestra del adenoma se pondrá inmediatamente en suero fisiológico frío, nunca en formol, y sin que pasen más de 30min desde su extracción hasta su procesamiento.
- 2.
La pieza debe valorarse en el Servicio de Anatomía Patológica, que proporciona, tras reservar la porción necesaria para su diagnóstico, un fragmento representativo, que es transferido a un criotubo con solución estabilizadora RNAlater (Life Technologies, Carlsbad, CA, EE. UU.); si el fragmento es mayor de 0,5cm, debe dividirse para que penetre la sustancia estabilizadora. El criotubo es identificado con un código REMAH, generado en la aplicación/registro informático, que será vinculado al código correspondiente de la historia clínica del paciente y al informe del Servicio de Endocrinología. El criotubo con la muestra se almacena a 4°C hasta su traslado al nodo de referencia seleccionado.
- 3.
Envío de la muestra de adenoma al nodo de referencia. El transporte de la muestra tumoral se realiza en el propio criotubo, bien en frío o a temperatura ambiente.
Se detallan a continuación los parámetros analizados a lo largo del desarrollo del proyecto global:
- •
Datos demográficos: sexo y edad.
- •
Niveles de expresión molecular, medidos por PCR cuantitativa en tiempo real (qPCR): GH, PRL, POMC, subunidades β de las hormonas luteinizante, folículo estimulante y estimulante del tiroides (LHB, FSHB y TSHB, respectivamente), subunidad α de las glicoproteínas (CGA), sst1, sst2, sst3, sst5, DRD1, DRD2T, DRD2L, DRD4, DRD5, GnRH-R, GHRH-R, CRH-R1, GHSR1a, AVPR1b, Ki67, PTTG1, y los genes de control de la β actina (ACTB), gliceraldehído-3-fosfato deshidrogenasa (GAPDH) y hipoxantina-guanina fosforribosiltransferasa (HPRT).
- •
Niveles bioquímicos en sangre, informe tras cirugía y datos de imagen indicados.
Los fragmentos de tejido tumoral se procesan de forma estandarizada y protocolizada para la evaluación de la expresión génica. Dependiendo del tipo de tumor, de las características de la enfermedad estudiada y del tamaño de la muestra disponible, se realiza una priorización de los genes que serán analizados, con atención preferente a las necesidades de información clínica de la dolencia. La extracción de ARN de la muestra se realiza, como se ha descrito previamente29, utilizando AllPrep RNA/DNA/Prot suplementado con RNase-Free DNase Set (#80004 y #79254, respectivamente, Qiagen, Limburg, Países Bajos) siguiendo las instrucciones del fabricante. La pieza tumoral se homogeneiza en frío usando buffer RLT suplementado con beta-mercapto-etanol, utilizando un vástago accionado por una pistola eléctrica (#749515-0000 y #749540-0000, respectivamente, Kontes, Sigma-Aldrich, Madrid, España), tras lo cual se realiza el aislamiento de ARN por el sistema de columnas incluido en el kit. Seguidamente, se eluye el ARN en 30-50μL de agua DEPC, dependiendo del tamaño de la muestra. La transcripción inversa del ARN se lleva a cabo utilizando RevertAid FirstStrand cDNA synthesis kit (#K1622, Fermentas, Hanover, MD, EE. UU.) según las instrucciones del fabricante, usando hexámeros aleatorios. En concreto, se ha considerado como óptima la utilización de 0,5μg de ARN por reacción, con una reacción doble (1μg de ARN) para obtener un volumen final de 40μL de ADN copia, que se guarda a −20°C hasta la medida por PCR cuantitativa en tiempo real (qPCR).
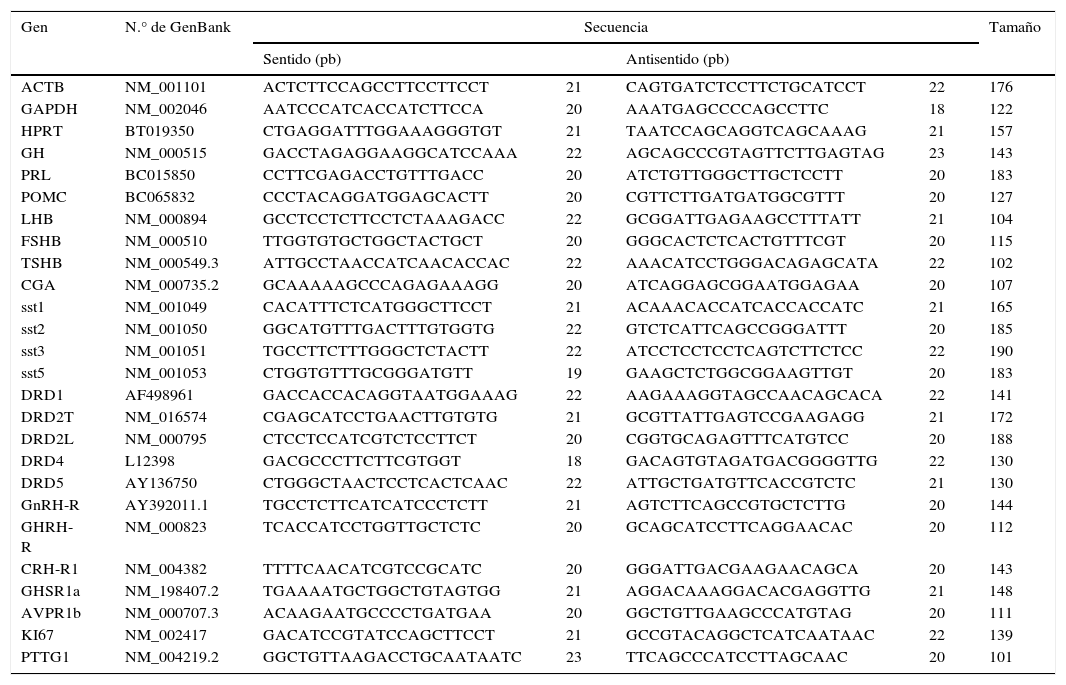
Selección de cebadores (primers)Todas las parejas de cebadores se han diseñado utilizando las secuencias genómicas de GenBank (National Center for Biotechnology Information) y el programa Primer335,36, como se ha descrito anteriormente17, utilizando los siguientes criterios: a) la diferencia en la temperatura de unión entre ambos cebadores no es mayor de 0,2°C, b) se han excluido los cebadores que generan dímeros de cebadores y 3) amplifican un producto entre 100 y 200 pares de bases. Las secuencias de los cebadores fueron comprobadas mediante BLAST (National Center for Biotechnology Information) para evitar la potencial homología con otras secuencias. La secuencia de los cebadores, los tamaños esperados y los números de GenBank se muestran en la tabla 1.
Secuencia de los primers, tamaños esperados y números de GenBank para los genes de estudio
| Gen | N.° de GenBank | Secuencia | Tamaño | |||
|---|---|---|---|---|---|---|
| Sentido (pb) | Antisentido (pb) | |||||
| ACTB | NM_001101 | ACTCTTCCAGCCTTCCTTCCT | 21 | CAGTGATCTCCTTCTGCATCCT | 22 | 176 |
| GAPDH | NM_002046 | AATCCCATCACCATCTTCCA | 20 | AAATGAGCCCCAGCCTTC | 18 | 122 |
| HPRT | BT019350 | CTGAGGATTTGGAAAGGGTGT | 21 | TAATCCAGCAGGTCAGCAAAG | 21 | 157 |
| GH | NM_000515 | GACCTAGAGGAAGGCATCCAAA | 22 | AGCAGCCCGTAGTTCTTGAGTAG | 23 | 143 |
| PRL | BC015850 | CCTTCGAGACCTGTTTGACC | 20 | ATCTGTTGGGCTTGCTCCTT | 20 | 183 |
| POMC | BC065832 | CCCTACAGGATGGAGCACTT | 20 | CGTTCTTGATGATGGCGTTT | 20 | 127 |
| LHB | NM_000894 | GCCTCCTCTTCCTCTAAAGACC | 22 | GCGGATTGAGAAGCCTTTATT | 21 | 104 |
| FSHB | NM_000510 | TTGGTGTGCTGGCTACTGCT | 20 | GGGCACTCTCACTGTTTCGT | 20 | 115 |
| TSHB | NM_000549.3 | ATTGCCTAACCATCAACACCAC | 22 | AAACATCCTGGGACAGAGCATA | 22 | 102 |
| CGA | NM_000735.2 | GCAAAAAGCCCAGAGAAAGG | 20 | ATCAGGAGCGGAATGGAGAA | 20 | 107 |
| sst1 | NM_001049 | CACATTTCTCATGGGCTTCCT | 21 | ACAAACACCATCACCACCATC | 21 | 165 |
| sst2 | NM_001050 | GGCATGTTTGACTTTGTGGTG | 22 | GTCTCATTCAGCCGGGATTT | 20 | 185 |
| sst3 | NM_001051 | TGCCTTCTTTGGGCTCTACTT | 22 | ATCCTCCTCCTCAGTCTTCTCC | 22 | 190 |
| sst5 | NM_001053 | CTGGTGTTTGCGGGATGTT | 19 | GAAGCTCTGGCGGAAGTTGT | 20 | 183 |
| DRD1 | AF498961 | GACCACCACAGGTAATGGAAAG | 22 | AAGAAAGGTAGCCAACAGCACA | 22 | 141 |
| DRD2T | NM_016574 | CGAGCATCCTGAACTTGTGTG | 21 | GCGTTATTGAGTCCGAAGAGG | 21 | 172 |
| DRD2L | NM_000795 | CTCCTCCATCGTCTCCTTCT | 20 | CGGTGCAGAGTTTCATGTCC | 20 | 188 |
| DRD4 | L12398 | GACGCCCTTCTTCGTGGT | 18 | GACAGTGTAGATGACGGGGTTG | 22 | 130 |
| DRD5 | AY136750 | CTGGGCTAACTCCTCACTCAAC | 22 | ATTGCTGATGTTCACCGTCTC | 21 | 130 |
| GnRH-R | AY392011.1 | TGCCTCTTCATCATCCCTCTT | 21 | AGTCTTCAGCCGTGCTCTTG | 20 | 144 |
| GHRH-R | NM_000823 | TCACCATCCTGGTTGCTCTC | 20 | GCAGCATCCTTCAGGAACAC | 20 | 112 |
| CRH-R1 | NM_004382 | TTTTCAACATCGTCCGCATC | 20 | GGGATTGACGAAGAACAGCA | 20 | 143 |
| GHSR1a | NM_198407.2 | TGAAAATGCTGGCTGTAGTGG | 21 | AGGACAAAGGACACGAGGTTG | 21 | 148 |
| AVPR1b | NM_000707.3 | ACAAGAATGCCCCTGATGAA | 20 | GGCTGTTGAAGCCCATGTAG | 20 | 111 |
| KI67 | NM_002417 | GACATCCGTATCCAGCTTCCT | 21 | GCCGTACAGGCTCATCAATAAC | 22 | 139 |
| PTTG1 | NM_004219.2 | GGCTGTTAAGACCTGCAATAATC | 23 | TTCAGCCCATCCTTAGCAAC | 20 | 101 |
Para verificar la especificidad de los cebadores, como se ha descrito previamente17, cada pareja de cebadores se utilizó en PCR convencional (DreamTaq DNA Polymerase, Thermo Scientific, Wilmington, NC, EE. UU.) para amplificar ADN copia generado por transcripción inversa, proveniente de tejido hipofisario humano. El perfil térmico consiste en un paso de 10 min a 95°C seguido de 35 ciclos de un minuto a 95°C, un minuto a 60-64°C y un minuto a 72°C. Los productos se corrieron en un gel de agarosa teñido con bromuro de etidio para confirmar la presencia de una única banda del tamaño esperado y la ausencia de dímeros de cebadores. Una alícuota del producto de PCR se purificó mediante el kit AccuPrep Gel Purification Kit (K3035, Bioneer, Alameda, CA, EE. UU.), y se realizó la secuenciación para confirmar la especificidad de los cebadores. Se utilizó el producto de PCR convencional para la construcción de curvas estándar para PCR cuantitativa. Para confirmar la eficiencia de los cebadores y construir las curvas estándar, el primer ensayo con qPCR se realiza haciendo una dilución 1:2 de la transcripción inversa, en la que la eficiencia óptima se demuestra por una diferencia de una Ct entre ambas diluciones. Los detalles de reactivos para qPCR se muestran en el siguiente apartado. Se determinó la concentración del producto purificado mediante PicoGreen DNA quantification kit (Molecular Probes, Eugene, OR, EE. UU.) y los productos de PCR se diluyeron de forma secuencial, obteniendo estándares de 101, 102, 103, 104, 105 y 106 copias del transcrito por microlitro. Un microlitro de cada punto de la curva se amplifica por qPCR y se genera, por tanto, una curva estándar que relaciona Ct y n.° de copias. Los valores de R2 generados de la recta se encontraron entre 0,997 y 1,003; se aceptaron las parejas de cebadores con una eficiencia entre 90 y 110%; una eficiencia del 100% indica que se amplifican todos los transcritos en cada ciclo. Si todos los parámetros de validación eran adecuados, se seleccionó la pareja de primers y sus condiciones de reacción se utilizaron para amplificar el ADN copia de las muestras tumorales.
PCR cuantitativa en tiempo real (qPCR)La medida de expresión génica se hace mediante PCR cuantitativa en tiempo real. Se ha utilizado MasterMix Brilliant III ultrafast SYBRGreen QPCR (#600882, Agilent, La Jolla, CA, EE. UU.), placas de 96 pocillos y tapas (#B70501 y #B79791B, respectivamente, Bioplastic, Landgraaf, Países Bajos). La reacción es la siguiente: 10μL de MasterMix, 1μL de ADN copia, 150nM de cada uno de los cebadores y hasta 20μL de agua DEPC. El perfil térmico consiste en un paso de 3 min a 95°C, 40 ciclos de 20 s a 95°C, seguidos de 20 s a 60°C y un paso final de disociación, un minuto a 95°C, 30 s a 55°C y 30 s a 95°C. El resultado de la qPCR es un valor de Ct que, al introducirse en la ecuación de la curva estándar, genera un valor de expresión en número de copias. La extrapolación se realiza utilizando un gráfico de dispersión con los exponentes de dilución 1, 2, 3, 4, 5 y 6 y los correspondientes valores de Ct obtenidos en la qPCR, generándose así una recta y su subsiguiente ecuación de la recta: y=mx+q, donde m es la pendiente de la recta, que debe aproximarse a 3,3, que es la diferencia de Ct entre las distintas diluciones, y q es el punto de intersección con el eje y, el valor de Ct de la muestra es el valor de x; así, al sustituir se obtiene el valor en número de copias.
Correlación de la expresión con las características clínicas y patológicas, el comportamiento y la progresión tumoralLos resultados obtenidos en las secciones descritas anteriormente se agrupan por tipo de tumor y de acuerdo con los tratamientos y tests desarrollados y, sobre esta base, los registros médicos de los pacientes se revisaron evaluando sus principales características.
Análisis estadísticoLa diferente naturaleza de las variables de estudio requiere un análisis detallado de los datos. Se han realizado tests de Komogorov-Smirnov para determinar la similitud de los diferentes conjuntos de datos con una distribución normal. Para la comparación de variables con distribuciones paramétricas se ha realizado el test de t de Student, y en el caso de las no paramétricas, la prueba de Mann-Whitney. Para más de 2 grupos se han realizado tests de Kruskal-Wallis. El análisis de las correlaciones se ha realizado mediante el coeficiente de correlación de Spearman.
ResultadosCasos registradosEl proyecto REMAH ha ido incorporando desde su inicio un número creciente de investigadores con acceso autorizado a la base de datos. Al finalizar la primera fase del proyecto, se cuenta con un total de 141 investigadores, cuya distribución geográfica se detalla en la tabla 2.
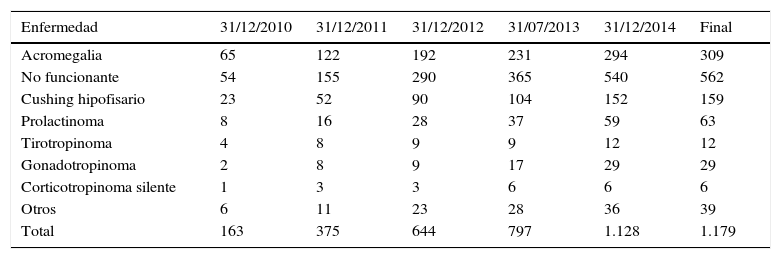
El registro acumulado en la base de datos del REMAH en esta primera fase cuenta con un total de 1.179 entradas, correspondientes a enfermedad hipofisaria registrada con código REMAH (tabla 3). Destacan por su abundancia los adenomas hipofisarios no funcionantes (562), seguidos de acromegalias (309) y enfermedad de Cushing (159). En general, la distribución de la prevalencia de las distintas enfermedades se mantiene con frecuencias similares en los distintos nodos.
Registro acumulativo de muestras y prevalencia de las enfermedades correspondientes según su fenotipado clínico. Por año, desde 2010 hasta 2015
| Enfermedad | 31/12/2010 | 31/12/2011 | 31/12/2012 | 31/07/2013 | 31/12/2014 | Final |
|---|---|---|---|---|---|---|
| Acromegalia | 65 | 122 | 192 | 231 | 294 | 309 |
| No funcionante | 54 | 155 | 290 | 365 | 540 | 562 |
| Cushing hipofisario | 23 | 52 | 90 | 104 | 152 | 159 |
| Prolactinoma | 8 | 16 | 28 | 37 | 59 | 63 |
| Tirotropinoma | 4 | 8 | 9 | 9 | 12 | 12 |
| Gonadotropinoma | 2 | 8 | 9 | 17 | 29 | 29 |
| Corticotropinoma silente | 1 | 3 | 3 | 6 | 6 | 6 |
| Otros | 6 | 11 | 23 | 28 | 36 | 39 |
| Total | 163 | 375 | 644 | 797 | 1.128 | 1.179 |
Otros: corresponde a otros tumores y lesiones hipofisarios que no concuerdan con las demás categorías o que debido a su baja incidencia no se han categorizado aparte; entre ellos se encuentran apoplejías hipofisarias, gonadotropinomas silentes, cordomas, oncocitomas y adenomas plurihormonales. Además se han incluido en este apartado los craneofaringiomas y los Cushing ectópicos, que, a pesar de no ser adenomas hipofisarios, son dolencias que tienen una relación estrecha con estos.
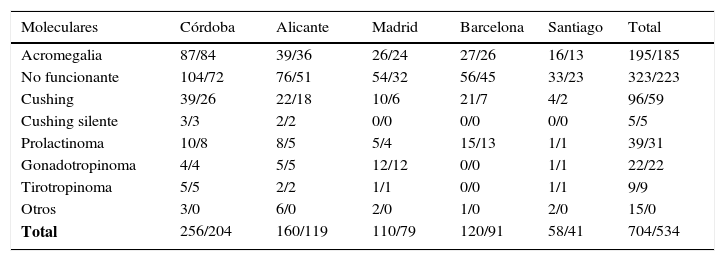
De la muestra total de 1.179 casos registrados, el número de casos con datos moleculares anotados es de 704, correspondientes a los nodos de Córdoba, Alicante, Barcelona, Madrid y Santiago de Compostela. En la tabla 4 se recoge la distribución de los tumores registrados con información molecular, según su tipología y el nodo donde se ha registrado, y los datos moleculares que se consideran inicialmente válidos para abordar un análisis estadístico, 534 en este caso (total/validados). En este registro también se han incluido craneofaringiomas y tumores secretores de ACTH ectópicos, debido a su asociación con enfermedad hipofisaria.
Distribución de las muestras registradas con fenotipo molecular disponible/validado, en función del tipo de tumor y del nodo de registro
| Moleculares | Córdoba | Alicante | Madrid | Barcelona | Santiago | Total |
|---|---|---|---|---|---|---|
| Acromegalia | 87/84 | 39/36 | 26/24 | 27/26 | 16/13 | 195/185 |
| No funcionante | 104/72 | 76/51 | 54/32 | 56/45 | 33/23 | 323/223 |
| Cushing | 39/26 | 22/18 | 10/6 | 21/7 | 4/2 | 96/59 |
| Cushing silente | 3/3 | 2/2 | 0/0 | 0/0 | 0/0 | 5/5 |
| Prolactinoma | 10/8 | 8/5 | 5/4 | 15/13 | 1/1 | 39/31 |
| Gonadotropinoma | 4/4 | 5/5 | 12/12 | 0/0 | 1/1 | 22/22 |
| Tirotropinoma | 5/5 | 2/2 | 1/1 | 0/0 | 1/1 | 9/9 |
| Otros | 3/0 | 6/0 | 2/0 | 1/0 | 2/0 | 15/0 |
| Total | 256/204 | 160/119 | 110/79 | 120/91 | 58/41 | 704/534 |
Otros: ver tabla 3.
La evaluación de la estabilidad de los 3 genes de control medidos inicialmente en el estudio, ACTB, GAPDH y HPRT, demuestra que la expresión de HPRT es notablemente más estable que la de los otros 2. Por ello, para realizar las comparaciones de niveles de expresión y las correlaciones con niveles bioquímicos, se ha decidido utilizar en adelante datos ajustados por HPRT.
Somatotropinomas causantes de acromegaliaSe han registrado 195 somatotropinomas anotados, de los que se ha validado el perfil molecular de 185. Los datos demográficos básicos (tabla 5) muestran que, en los pacientes del presente estudio, la acromegalia tiene una mayor incidencia en mujeres. La edad de los pacientes en el momento de la cirugía es significativamente menor en el caso de los hombres (p=0,005). En ambos sexos, la gran mayoría de tumores, por encima del 80%, son macroadenomas.
Adenomas hipofisarios no funcionantesEl tipo de tumores más abundante del estudio es el de los adenomas hipofisarios no funcionantes, lo que coincide con lo esperado por su mayor incidencia y prevalencia en la población general respecto al resto de adenomas. En esta primera fase del REMAH, se ha iniciado un análisis molecular de 223 adenomas no funcionantes de los 326 registrados (tabla 6). Se seleccionaron los adenomas con un perfil más compatible con el de adenoma no funcionante, y se han pospuesto para un estudio más detallado y minucioso aquellos que, pese a haber sido diagnosticados clínicamente como no funcionantes, mostraron un perfil molecular menos evidente, que pudiera corresponder a tumores silentes o a estadios no sintomáticos de una enfermedad de otra naturaleza. Los resultados obtenidos en el subgrupo seleccionado indican que los adenomas no funcionantes presentan una mayor incidencia en hombres, en los que, además, la edad en el momento de la cirugía tiende a ser superior que en las mujeres (p = 0,052). Como era de esperar, en ambos sexos se trató casi en su totalidad de macroadenomas, ya que la cirugía no está indicada en caso de microadenomas no funcionantes.
Datos demográficos básicos de los adenomas no funcionantes registrados
| No funcionante | N (%) | Edad [mediana (min-max)] | Macroadenoma (%) |
|---|---|---|---|
| Hombre | 187 (57) | 61 (16-81) | 126/126 (100) |
| Mujer | 139 (43) | 55 (19-87) | 99/102 (97) |
| Total | 323a | 58 (16-87) | 225/228 (99) |
La colección de muestras anotadas en REMAH ha permitido comenzar el análisis molecular de 59 de los 96 corticotropinomas causantes de la enfermedad de Cushing. Solo se han considerado válidos un 61% de los adenomas recogidos, ya que en muchos casos el análisis molecular reveló que la muestra analizada no se trataba de un adenoma corticotropo, sino que correspondía a un fragmento de glándula hipofisaria no tumoral, pues destacaba la expresión de GH y PRL, seguida de POMC y del resto de las hormonas adenohipofisarias: este es el perfil de expresión hormonal típico de la hipófisis normal.
En la muestra analizada (tabla 7), de la que casi 2 tercios fueron microadenomas, la incidencia es muy superior en mujeres que en hombres, mientras que no se observaron diferencias significativas entre las edades en ambos sexos.
Adenomas lactotropos causantes de hiperprolactinemiaEl cuarto tipo de tumor por su prevalencia en la serie registrada es el de los prolactinomas, causantes de hiperprolactinemia. Se ha realizado el análisis molecular en 31 de los 39 prolactinomas registrados (tabla 8). Puesto que este tipo de tumores tiene como primera línea de tratamiento la aplicación de agonistas dopaminérgicos, que con frecuencia permiten controlar e incluso resolver la enfermedad, los prolactinomas que se intervienen quirúrgicamente suelen ser casi siempre macroadenomas, que en esta serie son algo más frecuentes en hombres.
DiscusiónEl proyecto REMAH nacional ha completado satisfactoriamente su primera fase, con el establecimiento de un protocolo normalizado de trabajo, recolección de muestras, registro y anotación de pacientes y un primer análisis demográfico de la población registrada.
Desde un punto de vista práctico, los resultados obtenidos indican:
- 1.
La capacidad del proyecto para reclutar un número considerable (141) de investigadores clínicos y básicos para la recolección de pacientes y análisis de muestras, así como para su registro y estudio conjunto.
- 2.
El desarrollo de una base de datos conjunta clínico-molecular de acceso por Internet, que sirve de base para el desarrollo actual y futuro del proyecto. Su uso es ya factible para el apoyo al diagnóstico y su mejora permitirá un uso más rápido y dinámico en esa vertiente y una mayor proyección de cara al desarrollo de futuros proyectos de diversa índole.
- 3.
Se puede afirmar que el elevado número de pacientes y muestras registrado, su variedad y tipología, unidos a la caracterización molecular del tejido y al estudio clínico detallado de los casos constituyen, en su conjunto, un logro sin precedentes en el ámbito de la investigación y estudio de la enfermedad tumoral hipofisaria, no solo a nivel nacional sino también a nivel internacional.
- 4.
El sencillo análisis descriptivo de los datos demográficos de los casos analizados y de los perfiles moleculares evaluados para los principales tipos de adenomas hipofisarios ha puesto de manifiesto el considerable valor de la información generada, tanto a nivel global como, lo que seguramente será más importante, a nivel individual de cada paciente.
- 5.
El trabajo que se debe desarrollar a partir de ahora para sacar el máximo partido a los datos registrados y las medidas realizadas, y para extraer del proyecto REMAH el máximo valor de cara al futuro, es complejo y será necesariamente laborioso, pero también es de un interés extraordinario.
Los resultados obtenidos hasta la fecha, junto con la consecución de los principales hitos y objetivos marcados inicialmente, permiten afirmar que el lanzamiento y puesta en funcionamiento de este proyecto ha alcanzado un nivel adecuado de desarrollo y compleción. Así, lo logrado hasta este punto permite proponer, por una parte, la continuidad de la estrategia diseñada en una segunda fase que incluya las mejoras que se consideren pertinentes, en cuanto a revisión y reestructuración de los genes de medida y parámetros clínicos seleccionados, y, por otra, el diseño y puesta en marcha de nuevas acciones derivadas de los hallazgos realizados y, sobre todo, de los resultados obtenidos, que representan un producto de un valor considerable por la información que contienen en sí mismos los registros realizados y por su potencial como base de elaboración de nuevos estudios e iniciativas.
Conflicto de interesesMontserrat Gilabert es una empleada de Novartis Farmacéutica. El resto de autores declaran no tener ningún conflicto de intereses.
El proyecto REMAH ha sido posible gracias al patrocinio de Novartis, la Fundación FSEEN y la SAEN. Han contribuido asimismo en distinta medida los proyectos de investigación de los propios autores. Agradecemos a todos los investigadores, técnicos y personal sanitario que han contribuido al desarrollo del proyecto, así como, de forma principal y destacada, a los pacientes y sus familias por su generosa contribución.
Nodo Madrid:
Magdalena Adrados, Hospital Universitario de La Princesa
Pedro Martínez Flores, Hospital Universitario de La Princesa
Ana María Ramos Leví, Hospital Universitario de La Princesa
Miguel Sampedro-Núñez, Hospital Universitario de La Princesa
Ana Serrano-Somavilla, Hospital Universitario de La Princesa
María Paz de Miguel Novoa, Hospital Clínico San Carlos
Juan José Díez, Hospital Ramón y Cajal
Mercedes García Villanueva, Hospital Ramón y Cajal
Pedro Iglesias, Hospital Ramón y Cajal
Víctor Rodríguez Berrocal, Hospital Ramón y Cajal
Concepción Blanco Carrerra, Hospital Príncipe de Asturias
Esperanza Aguillo Gutiérrez, Hospital Clínico Zaragoza
Luciano Bances, Hospital Clínico Zaragoza
Fernando L. Calvo Gracia, Hospital Clínico Zaragoza
Fernando Comuñas, Hospital Clínico Zaragoza
Iván Quiroga López, Hospital de Talavera
Carmen Alameda Hernando, Hospital Infanta Sofía
Jesús Miguel Pérez Luis, Hospital Universitario de Tenerife
Rogelio García Centeno, Hospital Gregorio Marañón
Begoña Iza, Hospital Gregorio Marañón
Álvaro Pérez Zamarrón, Hospital La Paz
José F. Alén, Hospital 12 de Octubre
María Calatayud Gutiérrez, Hospital 12 de Octubre
Igor Paredes Sansinenea, Hospital 12 de Octubre
Álvaro Otero, Hospital Clínico de Salamanca
José María Recio Córdova, Hospital Clínico de Salamanca
Pablo Sousa, Hospital Clínico de Salamanca
José Belinchón, Hospital Virgen de la Salud
María José Herguido, Hospital Virgen de la Salud
Ángel Rodríguez de Lope, Hospital Virgen de la Salud
Almudena Vicente Delgado, Hospital Virgen de la Salud
Nodo Barcelona:
Iris Crespo, Hospital de Sant Pau
Fernando Muñoz, Hospital de Sant Pau
Eugenia Resmini, Hospital de Sant Pau
Olga Roig, Hospital de Sant Pau
Alicia Santos, Hospital de Sant Pau
Pere Tresserras, Hospital de Sant Pau
Carlos del Pozo Pico, Mutua de Terrassa
Alberto Torres, Hospital de Bellvitge
Noemí Vidal, Hospital de Bellvitge
Carles Villabona, Hospital de Bellvitge
Gemma Sesmilo, Centro Médico Teknon
Guillermo Cuatrecasas Cambra, Centro Médico Teknon
Joaquim Enseñat, Hospital Clinic Barcelona
Irene Halperin, Hospital Clinic Barcelona
Gabriel Obiols, Hospital Vall d’Hebron
Cristina Carrato, Hospital Germans Trias
Isabel Salinas, Hospital Germans Trias
Roxana Zabala, Hospital Germans Trias
Laura Cecenarro, Hospital Germans Trias
Raquel Buj, Hospital Germans Trias
Mireia Jordá, Hospital Germans Trias
Cristina Hostalot, Hospital Germans Trias
Javier Ibáñez Domínguez, Hospital Universitario Son Espases
Honorato García Fernández, Hospital Universitario Son Espases
Guillermo Serra, Hospital Universitario Son Espases
Nodo Alicante:
Pedro Riesgo, Hospital de La Ribera
Rosa Cámara, Hospital La Fe
Juan Antonio Simal-Julian, Hospital La Fe
Cristina Lamas, Hospital General de Albacete
Hernán Sandoval, Hospital General de Albacete
Javier Abarca, Hospital General de Alicante
Nieves Arias Mendoza, Hospital General de Alicante
Ruth Sánchez Ortiga, Hospital General de Alicante
Irene Monjas, Hospital General de Alicante
Teresa Pedro Font, Hospital de Denia
Nodo Santiago:
Isabel Alonso Troncoso, Complejo Hospitalario Pontevedra
Pablo Fernández Catalina, Complejo Hospitalario Pontevedra
Rosa María Álvarez San Martín, Complejo Asistencial de León
María D. Ballesteros Pomar, Complejo Asistencial de León
Rocío Villar Taibo, Complejo Asistencial de León
Sihara Pérez Romero, Universidad Santiago Compostela
Eva Fernández Rodríguez, Hospital Clínico Universitario de Santiago
Alfredo García-Allut, Hospital Clínico Universitario de Santiago
Ramón Serramito, Hospital Clínico Universitario de Santiago
Alma Prieto, Hospital el Bierzo (León)
Laura Cotovad Bellas, Complejo Hospitalario Arquitecto Marcide
Jose Ignacio Vidal Pardo, Complejo Hospitalario Xeral-Calde
Nodo Córdoba:
Juan Antonio García Arnés, Hospital Universitario Carlos Haya
Inmaculada González-Molero, Hospital Universitario Carlos Haya
Silvia María Maraver Selfa, Hospital Virgen de la Victoria
Francisco J. Tinahones Madueño, Hospital Virgen de la Victoria
María Rosa Alhambra Expósito, Hospital Universitario Reina Sofía
Paloma Moreno Moreno, Hospital Universitario Reina Sofía
Andrés de la Riva, Hospital Universitario Reina Sofía
Elena Torres Vela, Hospital Universitario San Cecilio
Miguel Ángel Japón, Hospital Virgen del Rocío
María Elena Dios Fuentes, Hospital Virgen del Rocío
Natividad González Rivera, Hospital de la Macarena
Tomas Martín Hernández, Hospital de la Macarena
Inmaculada Gavilán Villarejo, Hospital Universitario Puerta del Mar Cádiz
José Gregorio Oliva García, Hospital Universitario Nuestra Señora de la Candelaria
Judith López Fernández, Hospital Universitario de Canarias
Alberto Moreno Carazo, Complejo Hospitalario de Jaén