The identification of nontuberculous mycobacteria (NTM) by proteomic procedures has improved in the last few years. Strains from different geographical locations differ in their proteomic patterns, limiting the applicability of general databases for accurate identifications.
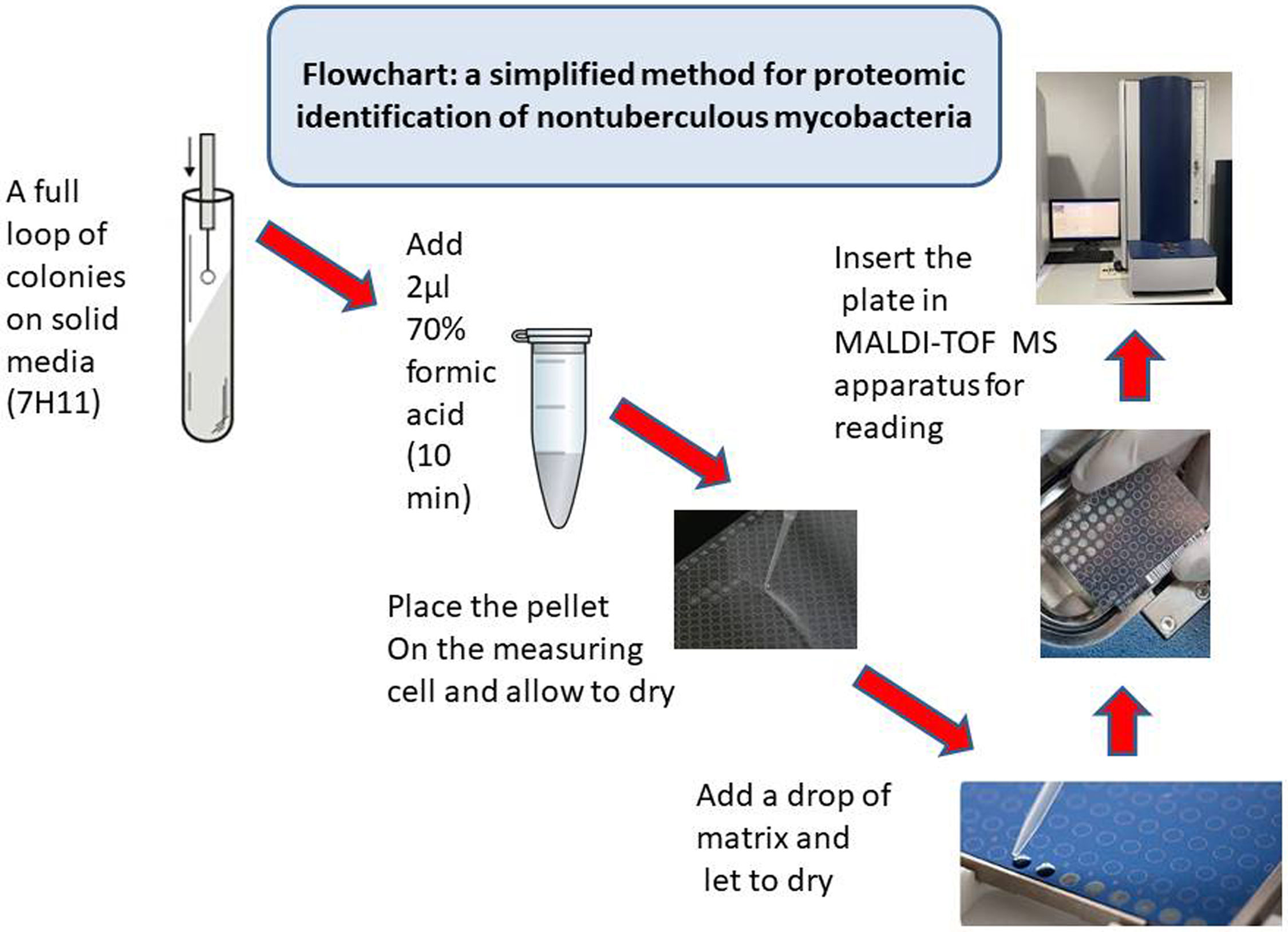
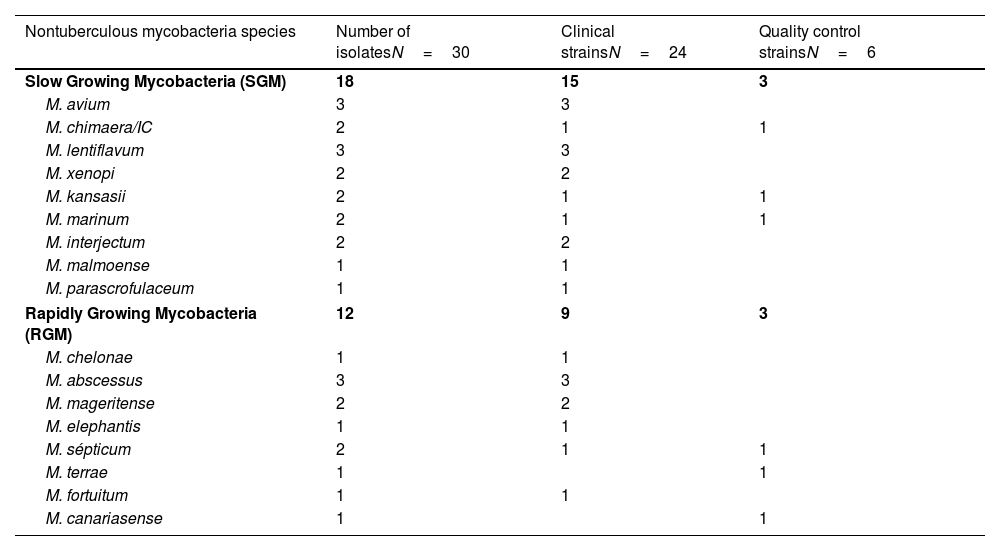
MethodsWe have optimized an alternative extraction protocol to Myco-Ex, and MBT, the protocols recommended by Bruker Daltonics, the manufacturer of MALDI Biotyper, a Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Mass Spectrometry (MALDI-TOF MS) system. 120 clinical isolates, one environmental strain (Mycobacterium canariasense) and 7 quality controls were tested from the Spanish Society of Clinical Microbiology and Infectious Diseases (SEIMC). All strains were also sent to the Laboratory of Mycobacteria belonging to the National Spanish Reference Center (Instituto de Salud Carlos III), that uses several molecular methods for mycobacterial identification. The proteomic extraction method includes a single extraction/inactivation formic acid step. 30 NTM strains were analyzed to certify the biosafety of the procedure, testing their viability after this procedure.
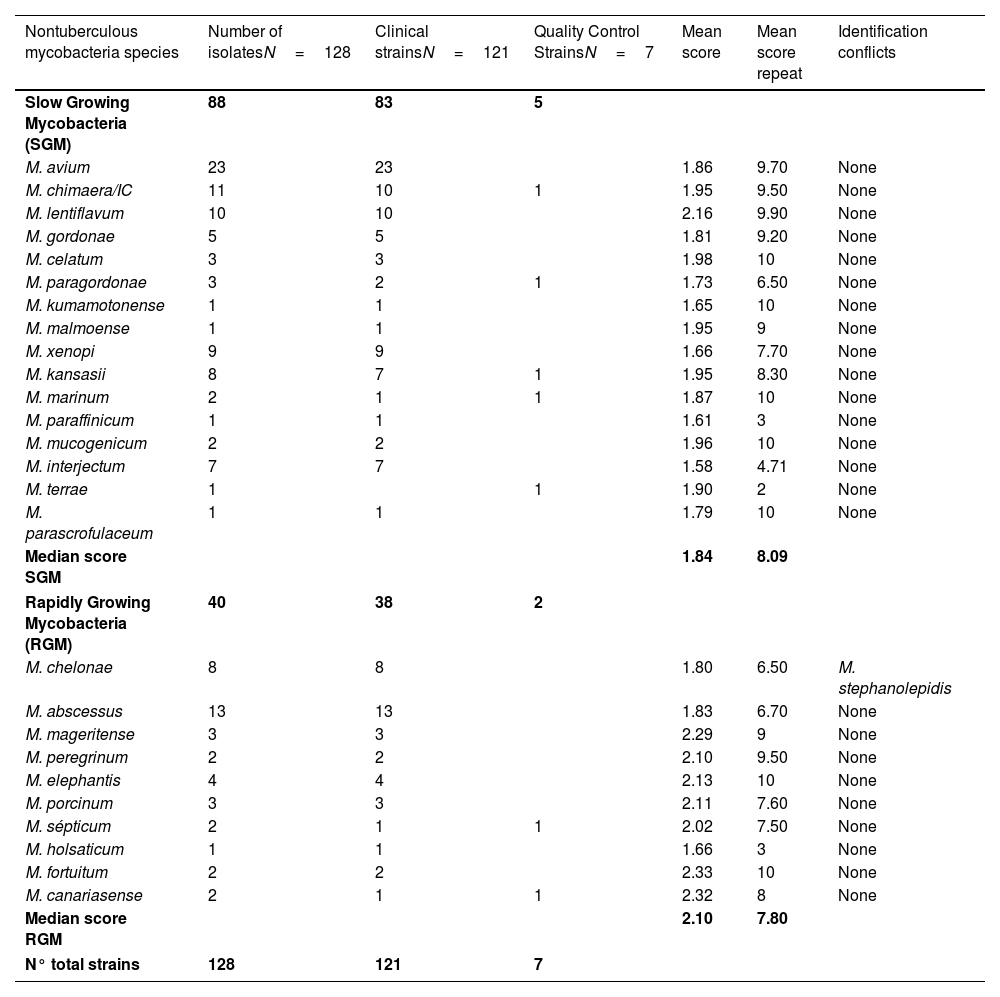
ResultsThe score ranges of the isolates were 1.61–2.16 (mean 1, 84) for the 88 isolates of the slow growing mycobacteria group (SGM) and 1.66–2.33 (mean 2.06) for the 40 isolates of the rapid growing mycobacteria group (RGM). Isolates exposed to formic acid were incubated for 8 weeks and growth was observed in neither the clinical isolates nor the controls.
ConclusionsWe have developed a simpler and faster procedure for the proteomic identification of nontuberculous mycobacteria (MALDI-TOF) retaining significant scores without compromising biosafety.
La identificación de micobacterias no tuberculosas (NTM) por técnicas proteómicas ha avanzado considerablemente en los últimos años. Aislamientos procedentes de diferentes localizaciones geográficas difieren en sus patrones proteómicos, limitando la aplicabilidad de las bases de datos y la exactitud de las identificaciones.
MétodosHemos optimizado un sistema de extracción alternativo a los recomendados por el fabricante de MALDI Biotyper, Bruker Daltonics, que son Myco-Ex and MBT. Se han probado 120 aislamientos clínicos de NTM, una cepa ambiental (M. canariasense) y 7 controles de calidad de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica (SEIMC). Todos los aislamientos se enviaron al Laboratorio español de Referencia (Instituto de Salud Carlos III), que utiliza distintos métodos moleculares para la identificación de micobacterias. El método de extracción proteómico consiste en único paso basado en extracción/inactivación con ácido fórmico. Se probó la viabilidad de 30 aislamientos de NTM sometidos a dicho procedimiento de extracción.
ResultadosLos rangos de scores obtenidos para los 88 aislamientos de micobacterias de crecimiento lento (SGM) fueron 1,61 – 2,16 (con una media de 1,84). Para los 40 aislamientos de micobacterias de crecimiento rápido (RGM) los scores obtenidos fueron 1,66 – 2,33 (con una media de 2,06). Respecto a los 30 controles de este procedimento y expuestos a ácido fórmico, a las 8 semanas de incubación no se obtuvo crecimiento en ninguno de ellos.
ConclusionesHemos desarrollado un método de extracción sencillo y más rápido para la identificación proteómica de micobacterias no tuberculosas (MALDI-TOF), manteniendo scores significativos sin comprometer la bioseguridad.
Artículo
Socio de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica

Para acceder a la revista
Es necesario que lo haga desde la zona privada de la web de la SEIMC, clique aquí