La resistencia combinada a quinolonas y β-lactámicos es frecuente en enterobacterias. La aparición en enterobacterias de metalo-β-lactamasas y determinantes de resistencia a quinolonas mediada por plásmidos constituyen un problema emergente en nuestro país.
MétodosLa sensibilidad se determinó mediante tiras de E-test. Los genes de resistencia se detectaron mediante PCR y se procedió a la caracterización de los plásmidos portadores.
ResultadosEn este estudio se describen 2 cepas (1 Klebsiella oxytoca y 1 Klebsiella pneumoniae) portadoras de los genes blaVIM-1 y qnrS2 en un plásmido transferible de 70-Kb aisladas en cultivos de vigilancia en el Hospital Universitario Virgen Macarena de Sevilla.
ConclusiónEsta es la primera asociación de blaVIM-1 y qnrS2 en un mismo plásmido no tipable en nuestro centro.
Combined resistance to quinolones and β-lactams is common in Enterobacteriaceae. The appearance in enterobacteria coding for metallo-β-lactamases and determinants of plasmid-mediated quinolone resistance are an emerging problem in our country.
MethodsThe susceptibility was determined by E-test. The resistance genes were detected by PCR and the corresponding plasmids were characterised.
ResultsThis study describes 2 strains (1 Klebsiella oxytoca, 1 Klebsiella pneumoniae) carrying the genes qnrS2 and blaVIM-1 in a transferable plasmid of 70-Kb isolated in surveillance cultures at the University Hospital Virgen Macarena in Seville.
ConclusionThis is the first combination of qnrS2 and blaVIM-1 on the same non-typeable plasmid isolated in our centre.
Los carbapenémicos son los antimicrobianos de elección para el tratamiento de las infecciones por enterobacterias productoras de β-lactamasas de espectro extendido (BLEE). Las metalo-β-lactamasas (MBLs) son enzimas pertenecientes a la clase B de Ambler capaces de hidrolizar a todos los β-lactámicos, incluidos los carbapenémicos, con la excepción de aztreonam, y su actividad resulta inhibida por EDTA. La aparición de enterobacterias productoras de MBLs empieza a ser un problema clínico y epidemiológico de importancia en España1,2.
Los miembros de la familia Qnr son proteínas pentapeptídicas capaces de proteger a las topoisomerasas bacterianas de la acción de las quinolonas. Estas presentan una distribución amplia en diferentes especies de Enterobacteriaceae como determinantes de resistencia a quinolonas mediada por plásmidos (RQMP), junto con el enzima AAC(6′)-Ib-cr, una variante de una acetiltrasferasa de aminoglucósidos capaz de inactivar quinolonas, y los sistema de expulsión activa QepA y OqxAB3. La prevalencia de estos genes en enterobacterias oscila entre el 1% y >20% dependiendo de las características de las colecciones estudiadas. Los genes qnr así como aac(6′)-Ib-cr se han asociado previamente a BLEEs o MBLs3–5.
En este trabajo se describe la asociación de los genes de resistencia qnrS2 y blaVIM-1 en dos aislados clínicos de Klebsiella spp., uno de K. pneumoniae y otro de K. oxytoca, procedentes del Hospital Universitario Virgen Macarena de Sevilla en un mismo plásmido transferible. Para nuestro conocimiento esta es la primera descripción de la asociación de los genes qnrS2 y blaVIM-1 en España.
MétodosCepas bacterianasSe estudiaron 2 aislados de Klebsiella spp. (1K. pneumoniae y 1K. oxytoca), procedentes de muestras de frotis rectal para vigilancia de infección nosocomial de 2 pacientes ingresados en la unidad de cuidados intensivos del Hospital Universitario Virgen Macarena de Sevilla. Estos aislados se recuperaron en la misma semana de placas de agar MacConkey con cefotaxima (4mg/L). La identificación se realizó mediante el sistema automático WIDER (Francisco Soria Melguizo S.A.) y se confirmó posteriormente mediante el sistema API 20E (bioMérieux). Las cepas E. coli Dh10B y E. coli J53 resistente a azida sódica se utilizaron en los ensayos de clonación y conjugación, respectivamente.
Estudios de sensibilidad antimicrobianaSe realizó mediante el sistema automático WIDER (Francisco Soria Melguizo S.A.) y mediante tiras de E-test para quinolonas y carbapenémicos. La presencia de BLEE y MBLs se estudió fenotípicamente siguiendo las recomendaciones del CLSI6.
Detección molecular de genes de resistenciaMediante PCR y secuenciación se evaluó la presencia de genes codificadores de MBLs blaVIM, blaIMP, blaGIM y blaSPM-17, así como de los genes de RQMP qnrA, qnrB, qnrS, qnrC, qnrD, aac(6′)-Ib-cr y qepA3.
Caracterización de los plásmidosLos plásmidos fueron purificados, transformados en E. coli Dh10B y seleccionados en placas con amoxicilina (100mg/L) para valorar la transmisión del fenotipo de resistencia. Se realizaron ensayos de conjugación utilizando como receptor la cepa E. coli J53 y siembra en placas con azida sódica (150mg/L) y amoxicilina (100mg/L). Los grupos de incompatibilidad plásmidica se estudiaron siguiendo el protocolo descrito por Carattoli et al.8.
Para determinar el entorno genético de los genes de resistencia, los plásmidos fueron digeridos con un enzima de restricción (BamHI, EcoRI o Sau3A), clonados en el vector pBK-CMV, electroporados en E. coli Dh10B y seleccionados en placas con kanamicina (30mg/L) y amoxicilina (100mg/L) o ciprofloxacino (0,05mg/L).
Resultados y discusiónEl número de aislados de enterobacterias portadoras de MBLs en España está aumentando, particularmente en el género Klebsiella1,2. La aparición de aislados productores de carbapenemasas y proteínas de RQMP, aunque todavía infrecuente en España, supone un importante problema de salud pues resultan difíciles de detectar en el laboratorio. Los valores de concentración mínima inhibitoria (CMI) de los aislados productores de carbapenemasas tipo VIM y KPC1,9 muestran una gran variabilidad y baja reproducibilidad en sistemas automatizados. Esto dificulta en gran medida su control epidemiológico, y el tratamiento de las infecciones que provocan resulta complicado, pues quedan muy pocas opciones terapéuticas.
Este trabajo describe la presencia en un mismo plásmido transferible de los genes de resistencia qnrS2 y blaVIM-1 en dos aislados de frotis rectal de Klebsiella oxytoca y K. pneumoniae, respectivamente, de dos pacientes ingresados en la UCI del Hospital Universitario Virgen Macarena de Sevilla en marzo de 2010. Se trataba de dos varones, uno de 72 años, diabético y cardiópata, y otro de 70 años, hipertenso con EPOC y carcinoma vesical con posible metástasis pulmonar. Uno de los pacientes presentaba una estancia prolongada en UCI (5 meses), habiendo recibido previamente tratamiento antibiótico con cefepime, meropenem, piperacilina/tazobactam y levofloxacino. En el otro paciente el aislado se detectó en una muestra de control al ingreso en UCI y no había recibido antibióticos previamente. Ambos pacientes habían sido sometidos a cirugía previa.
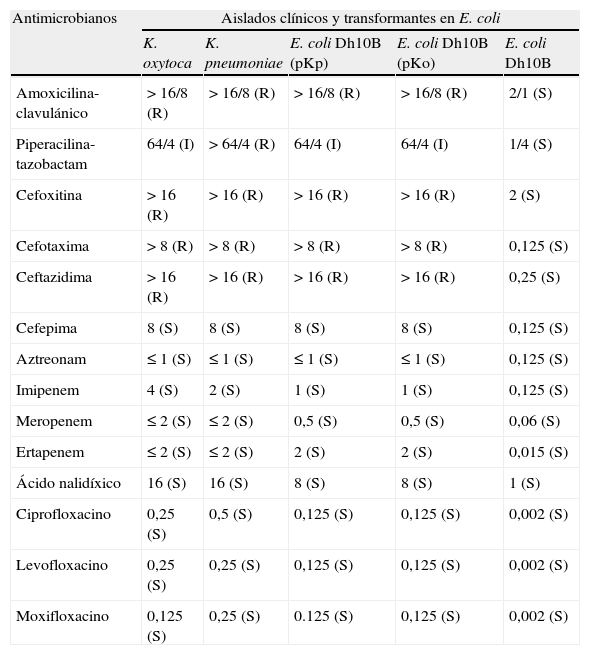
Los dos aislados de Klebsiella mostraron sensibilidad disminuida o resistencia a todos los β-lactámicos no carbapenémicos ensayados, con la excepción de aztreonam (tabla 1). Las CMIs de imipenem frente al aislado de K. pneumoniae y K. oxytoca fueron de 2mg/L y 4mg/L, respectivamente, en tanto que los valores de CMI de meropenem y ertapenem fueron de <=2mg/L frente a los dos aislados. La prueba de confirmación de BLEE fue negativa mientras que la prueba de Hodge modificada para la detección de carbapenemasas fue positiva (discos de ertapenem y meropenem). En cuanto a quinolonas, ambos aislados presentaron una CMI de ciprofloxacino de 0,5mg/L (sensible pero con sensibilidad reducida), mientras que la CMI de ácido nalidíxico fue de 16mg/L (sensible) (tabla 1). Este fenotipo es compatible con la presencia de determinantes de RQMP y descarta la presencia de mecanismos de resistencia a quinolonas por modificación de topoisomerasas de tipo II. Mediante PCR y secuenciación se confirmó la presencia de los genes blaVIM-1 y qnrS2 en ambos aislados. La PCR para el resto de genes que codifican MBLs o genes de RQMP fue negativa. En ambos pacientes se procedió a las medidas oportunas de control de infección nosocomial (aislamiento de los pacientes y limpieza de los dos boxes de UCI), no observándose posteriormente ningún otro caso, y ninguno de los dos pacientes desarrolló infección clínica por estos microorganismos.
Sensibilidad a β-lactámicos y quinolonas de los aislados de K. pneumoniae y K. oxytoca, y sus respectivos transformantes en E. coli Dh10B que contienen los plásmidos pKp y pKo que codifican ambos para VIM-1 y QnrS2
| Antimicrobianos | Aislados clínicos y transformantes en E. coli | ||||
| K. oxytoca | K. pneumoniae | E. coli Dh10B (pKp) | E. coli Dh10B (pKo) | E. coli Dh10B | |
| Amoxicilina-clavulánico | >16/8 (R) | >16/8 (R) | >16/8 (R) | >16/8 (R) | 2/1 (S) |
| Piperacilina-tazobactam | 64/4 (I) | >64/4 (R) | 64/4 (I) | 64/4 (I) | 1/4 (S) |
| Cefoxitina | >16 (R) | >16 (R) | >16 (R) | >16 (R) | 2 (S) |
| Cefotaxima | >8 (R) | >8 (R) | >8 (R) | >8 (R) | 0,125 (S) |
| Ceftazidima | >16 (R) | >16 (R) | >16 (R) | >16 (R) | 0,25 (S) |
| Cefepima | 8 (S) | 8 (S) | 8 (S) | 8 (S) | 0,125 (S) |
| Aztreonam | ≤1 (S) | ≤1 (S) | ≤1 (S) | ≤1 (S) | 0,125 (S) |
| Imipenem | 4 (S) | 2 (S) | 1 (S) | 1 (S) | 0,125 (S) |
| Meropenem | ≤2 (S) | ≤2 (S) | 0,5 (S) | 0,5 (S) | 0,06 (S) |
| Ertapenem | ≤2 (S) | ≤2 (S) | 2 (S) | 2 (S) | 0,015 (S) |
| Ácido nalidíxico | 16 (S) | 16 (S) | 8 (S) | 8 (S) | 1 (S) |
| Ciprofloxacino | 0,25 (S) | 0,5 (S) | 0,125 (S) | 0,125 (S) | 0,002 (S) |
| Levofloxacino | 0,25 (S) | 0,25 (S) | 0,125 (S) | 0,125 (S) | 0,002 (S) |
| Moxifloxacino | 0,125 (S) | 0,25 (S) | 0.125 (S) | 0,125 (S) | 0,002 (S) |
En ambos aislados de Klebsiella estos genes se localizaron en un mismo plásmido de aproximadamente 70-Kb. Este plásmido no se pudo transferir mediante ensayos de conjugación bajo nuestras condiciones experimentales, mientras que si fue transferible a E. coli mediante ensayos de transformación. Estos plásmidos pertenecieron a un grupo de incompatibilidad plasmídicas no tipable de acuerdo a protocolo de Carattoli et al.8 Los genes qnrS1 y blaVIM-1 han sido previamente asociados a los grupos de incompatibilidad plasmídicas IncN10.
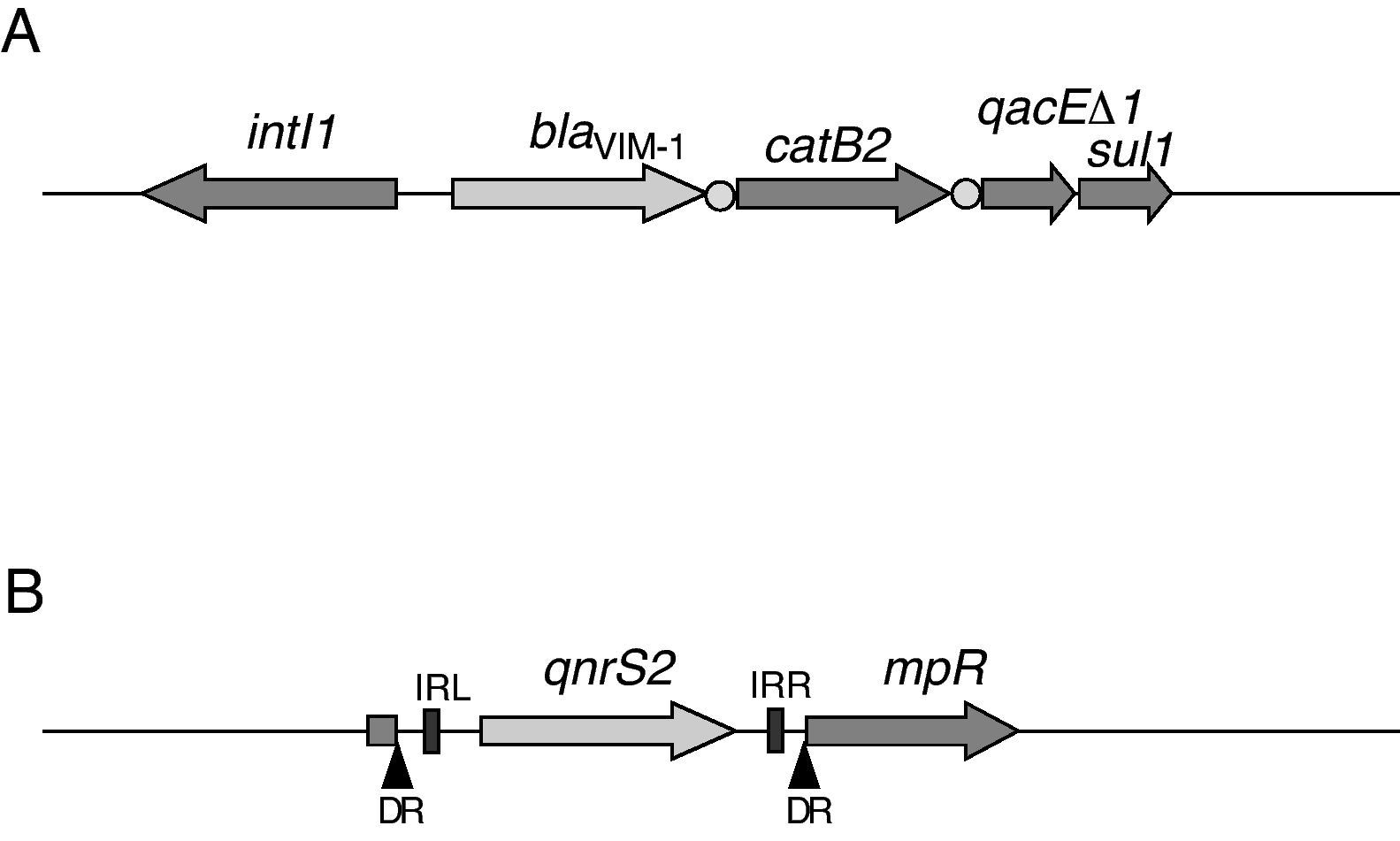
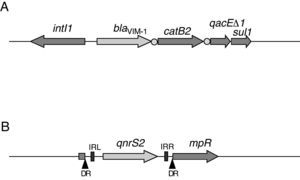
El entorno genético de los genes qnrS2 y blaVIM-1 fue analizado. blaVIM-1 se encontró como el primer gen casete de un integron de clase 1, el cual contuvo adicionalmente el gen de resistencia a cloranfenicol catB2. qnrS2 fue encontrado en una estructura idéntica a la descrita en Aeromonas punctata en el plásmido pAS37, formando parte de un elemento para la inserción de casetes (MIC)11 (fig. 1).
En conclusión este trabajo describe la presencia en un mismo plásmido transferible de los genes de resistencia qnrS2 y blaVIM-1 en dos aislados diferentes de K. pneumoniae y K. oxytoca en la UCI del Hospital Universitario Virgen Macarena de Sevilla. Para nuestro conocimiento esta es la primera descripción de la asociación de los genes qnrS2 y blaVIM-1 en España. Los sistemas de vigilancia activa son fundamentales para controlar su diseminación, dado que cuando estos microorganismos causan infecciones plantean serios problemas terapéuticos.
FinanciaciónEste trabajo fue financiado por la Consejería de Innovación Ciencia y Empresa, Junta de Andalucía (P07-CTS-02908) y por la Consejería de Salud, Junta de Andalucía (PI-0282-2010). Parcialmente financiado por el Ministerio de Ciencia e Innovación, Instituto de Salud Carlos III - cofinanciada por el Fondo Europeo de Desarrollo Regional «Una manera de hacer Europa» FEDER, Red Española de Investigación en Patología Infecciosa (REIPI RD06/0008).
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.