Se analizó la distribución epidemiological de los diferentes tipos de betalactamasas de espectro extendido (BLEE) en Escherichia coli y Klebsiella pneumoniae en distintos hospitales de España y se comparó con estudios previos.
MétodosEn 11 hospitales españoles se recogieron los 15 primeros aislamientos de E. coli y los 5 primeros de K. pneumoniae con sospecha de ser portadores de BLEE, aislados en el primer trimestre de 2004. Los estudios de clonalidad se realizaron mediante electroforesis en gel de campos pulsantes (PFGE) tras digestión del ADN total con XbaI y mediante ERIC-PCR (Enterobacterial Repetitive Intergenic Concensus Secuences-Polimerase Chain Reaction). Las BLEE se caracterizaron mediante isoelectroenfoque, PCR y secuenciación.
ResultadosSe estudiaron 124 aislamientos. El análisis de los patrones de restricción obtenidos por PFGE mostró una gran diversidad clonal entre los aislamientos de E. coli, observándose cuatro agrupaciones de dos cepas en cada una de ellas. En las 92 cepas de E. coli, la caracterización de las BLEE mostró un predominio de CTX-M-14 (45,7%), CTX-M-9 (20,6%) y SHV-12 (21,7%). En las 32 cepas de K. pneumoniae se observó una menor diversidad clonal, detectándose tres agrupaciones que incluían el 53,1% de los aislamientos. Las BLEE detectadas en estas cepas fueron del tipo CTX-M en 20 casos (62,5%) (CTX-M-1, CTX-M-9, CTX-M-14 y CTX-M-15), de tipo SHV en 11 (34,4%) (SHV-12 y SHV-5) y TEM-4 (3,1%) en una.
ConclusiónLas cepas de E. coli y K. pneumoniae analizadas en ese período presentan una mayor diversidad de BLEE que la observada en estudios epidemiológicos realizados con anterioridad. Además, el análisis de la relación clonal definió una gran diversidad en E. coli y menor en K. pneumoniae.
The epidemiological distribution of extended-spectrum beta-lactamase (ESBL) types in Escherichia coli and Klebsiella pneumoniae was evaluated in various hospitals in Spain and compared with previous studies.
MethodsA total of 11 Spanish hospitals participated in this study. Each center collected the first 15 isolates of E. coli and the first 5 of K. pneumoniae suspected of being ESBL-producers and isolated during the first quarter of 2004. Clonal study was done by PFGE after total DNA digestion with XbaI and by ERIC-PCR (Enterobacterial Repetitive Intergenic Concensus Secuences-Polimerase Chain Reaction), typing. ESBL-producers were characterized by isoelectric focusing (IEF), PCR and sequencing.
ResultsA total of 124 strains were collected. PFGE restriction patterns showed considerable diversity among E. coli strains; 4 clusters of 2 strains each were detected. ESBL characterization of 92 E. coli strains showed a predominance of CTX-M-14 (45.7%), CTX-M-9 (20.6%) and SHV-12 (21.7%). Clonal diversity among the 32 K. pneumoniae strains was less pronounced than in E. coli; 3 clusters included 53.1% of strains. The ESBL detected in these strains included a CTX-M type in 20 cases (62.5%) (CTX-M-1, CTX-M-9, CTX-M-14 and CTX-M-15); a SHV type in 11 (34.4%) (SHV-12 and SHV-5) and TEM-4 (3.1%) in 1 case.
ConclusionThe E. coli and K. pneumoniae strains analyzed in this period displayed a greater diversity of ESBL than has been observed in previous epidemiological studies. Analysis of clonal relationships revealed a greater diversity in E. coli than in K. pneumoniae.
Las betalactamasas de espectro extendido (BLEE), descritas frecuentemente en Escherichia coli, Klebsiella pneumoniae y en menor medida en otras enterobacterias, confieren resistencia a cefalosporinas de amplio espectro y constituyen un grupo heterogéneo de enzimas1–3. Las infecciones en las que están implicadas representan un reto debido a que, con frecuencia, los microorganismos que las producen son también resistentes a otros antimicrobianos, como trimetoprima-sulfametoxazol, aminoglucósidos y fluoroquinolonas, limitando las opciones terapéuticas2,4. Estas enzimas presentan una gran dispersión, lo cual parece ser dependiente del elemento móvil que vehicula el gen blaBLEE.
En un inicio, la descripción de BLEE se englobaba en el contexto de brotes hospitalarios que frecuentemente implicaban K pneumoniae y BLEE derivadas de SHV-1 o de TEM-11,5. En los últimos años se ha detectado el incremento de las BLEE del tipo CTX-M. Su difusión en bacterias de la misma o de diferente especie se ha asociado a ciertos elementos genéticos, como las secuencias de inserción, los transposones y los plásmidos1,5,6. Aunque con menor frecuencia, también se ha descrito la dispersión clonal de cepas productoras de este tipo de BLEE, principalmente de la CTX-M-157,8.
El incremento en la prevalencia de microorganismos con BLEE ha sido descrito por distintos autores, tanto en el ámbito nacional como internacional9–12. El Sistema Europeo de Vigilancia de Resistencias Antimicrobianas en patógenos invasores (EARSS) observó que la resistencia a cefalosporinas de tercera generación asociada con la producción de BLEE en E. coli aumentó en casi todos los países europeos incluidos en el estudio, y concretamente en España creció del 1,6 a 4,1% en el período 2001-200311. El estudio relaciona este incremento con el uso abusivo de antibióticos de amplio espectro11.
En un estudio multicéntrico realizado en España durante el año 2000 (proyecto GEIH-BLEE 2000) para determinar la prevalencia de E. coli y K. pneumoniae portadoras de BLEE en 40 hospitales españoles se constató que en E. coli las BLEE CTX-M-9, CTX-M-14 y SHV-12 constituían el 70% de las enzimas detectadas, mientras que en K. pneumoniae más del 50% fueron del tipo TEM13.
Ultimamente son varios los hospitales españoles que han evidenciado un cambio en la prevalencia de las diferentes enzimas detectadas en E. coli y K. pneumoniae14–17. Es por ello que en la Red Española de Investigación en Patología Infecciosa (REIPI) se planteó la necesidad de realizar el presente estudio multicéntrico, el cual permitiría conocer y actualizar la evolución y tipo de BLEE en E. coli y K. pneumoniae en distintos hospitales de España y descartaría, mediante estudios moleculares, la posible expansión clonal de los microorganismos con estas enzimas.
Material y métodosCentros participantesEn este estudio participaron un total de 11 hospitales españoles además del Centro Nacional de Microbiología (CNM) del Instituto de Salud Carlos III (ISCIII). Los hospitales participantes fueron: Corporació Hospitalaria Parc Taulí (CPT), de Sabadell; Hospital Germans Trias i Pujol (HGTP), de Badalona; Hospital Vall d'Hebron (HVH), Hospital de la Santa Creu i Sant Pau (HSP) y Hospital Clínic (HC), de Barcelona; Hospital de Bellvitge (HB), de l'Hospitalet; Hospital Son Dureta (HSD), de Palma de Mallorca; Hospital de la Ribera (HR), de Alzira; Hospital Virgen Macarena (HVM) y Hospital Virgen del Rocío (HVR), de Sevilla, y Hospital Universitario Ramón y Cajal (HRC), de Madrid.
Recolección de cepas y estudio de la sensibilidad a los antimicrobianosSe solicitó a los centros participantes el envío de los 15 primeros aislamientos de E. coli y los 5 primeros de K. pneumoniae con sospecha fenotípica de ser portadores de BLEE y aislados en el primer trimestre de 2004. Sólo se consideró una cepa por paciente.
En total se recogieron 142 aislamientos, 15 en el HGTP, HVH, HRC, HVR y HC; 14 en el HSP, CPT y HSD; 11 en el HVM; 10 en el HB; y 4 en el HR.
Posteriormente, el CNM verificó la sensibilidad a antibióticos, tanto betalactámicos como no betalactámicos, mediante técnica de microdilución18 utilizando el sistema comercial Phoenix (BD Phoenix Automated Microbiology System: Becton-Dickinson Diagnosis, Sparks, MD, Estados Unidos). La cepa de E. coli ATCC 25922 se utilizó como control.
Al mismo tiempo se recogieron los datos demográficos de los pacientes (sexo, edad, área de hospitalización y tipo de muestra). Se consideró como criterio de infección nosocomial cuando el paciente llevaba más de 48 h de hospitalización, y de infección adquirida en la comunidad cuando llevaba menos de 48 h, siendo conscientes de las limitaciones de este criterio, que puede infraestimar el número de infecciones nosocomiales.
Estructura poblacionalLos estudios de clonalidad se realizaron en el CNM mediante electroforesis en gel de campos pulsantes (PFGE) tras digestión del ADN total con XbaI según la metodología previamente descrita7. Los pulsotipos se consideraron idénticos, probablemente relacionados, posiblemente relacionados o distintos, en función de los criterios de Tenover19. Los aislamientos, cuyo ADN no pudo ser digerido con XbaI se estudiaron mediante Rep-PCR20 en comparación con aquellos que se consideraron epidemiológicamente relacionados (mismo servicio hospitalario, mismo período de tiempo).
Caracterización de las BLEELa caracterización de las BLEE se realizó mediante isoelectroenfoque (IEF)21, reacción en cadena de la polimerasa (PCR) y secuenciación15. Para la conjugación se utilizaron las cepas E. coli HB101 resistente a kanamicina y rifampicina, E. coli BM694 y E. coli BM21, ambas resistentes a ácido nalidíxico y se calculó la frecuencia de conjugación22.
Cálculos estadísticosLos resultados fueron sometidos a procesamiento estadístico con el programa STATCALC y se aplicó la prueba de la chi al cuadrado para la comparación de proporciones con un nivel de significación de p < 0,05. Los resultados se expresan en porcentajes.
ResultadosPacientes y cepasSe ha estudiado un total de 102 cepas de E. coli y 40 de K pneumoniae. Se descartaron 18 (10 E. coli y 8 K. pneumoniae) por presentar un fenotipo compatible con hiperproducción de su betalactamasa cromosómica.
Las 124 cepas restantes fueron aisladas de 54 varones (43%) y 70 mujeres (57%). En función del tipo de infección, el 51,6% fueron infecciones nosocomiales, mientras que el 48,4% fueron comunitarias.
Al desglosar estas características epidemiológicas en función del microorganismo aislado, se observó que la infección por E. coli tuvo lugar en 34 varones (37%) y 58 mujeres (63%); en 39 de los casos (42,4%) fueron infecciones nosocomiales y en 53 (57,6%) comunitarias. Por el contrario, la infección por K. pneumoniae tuvo lugar en 20 varones (62,5%) y 12 mujeres (37,5%); en 25 casos (76%) fueron infecciones nosocomiales y en 7 (25%) comunitarias.
Los servicios de los que provenían los pacientes con infección intrahospitalaria por E. coli y K pneumoniae fueron principalmente: medicina interna (48,7 y 44%, respectivamente), cirugía (25,6 y 16%), unidad de cuidados intensivos (12,8 y 20%), pediatría (7,7 y 4%), consultas externas (2,6 y 4%) y otros (2,6 y 12%). En ningún caso las diferencias llegaron a ser estadísticamente significativas. Las cepas de E. coli se aislaron de orina (68,5%), sangre (10,9%), exudados (8,7%), muestras respiratorias (3,3%), líquidos estériles (2,2%), catéteres (1,1%) y otras muestras (5,4%); y las cepas de K. pneumoniae se aislaron de orina (43,8%), exudados (21,9%), sangre (12,5%), muestras respiratorias (12,5%), líquidos estériles (3,1%), catéteres (3,1%) y otras muestras (3,1%).
El rango de edad de los pacientes en los que se aisló E. coli osciló entre 1 mes y 97 años, y la media era de 60 años. En el caso de K. pneumoniae el rango fue de 1 día a 90 años y la media de 57 años. Ocho pacientes (6,5%) presentaron edades inferiores a 1 año, cuatro (3,2%) edades comprendidas entre 2 y 14 años, tres (2,4%) entre 15 y 25 años, 12 (9,7%) entre 26 y 45 años, 36 (29%) entre 46 y 65 años, y finalmente, 61 (49,2%) mayores de 65 años.
Estructura poblacionalDe los 124 aislamientos estudiados por la técnica de PFGE, en 10 (9 E. coli y 1 K. pneumoniae) no se obtuvo ningún patrón de restricción con XbaI. Mediante el análisis por Rep-PCR se descartó la asociación clonal de estos 9 aislamientos de E. coli, entre ellos y respecto a aquellos que por fecha y lugar de aislamiento y tipo de BLEE producida se consideró que pudieran estar epidemiológicamente relacionados.
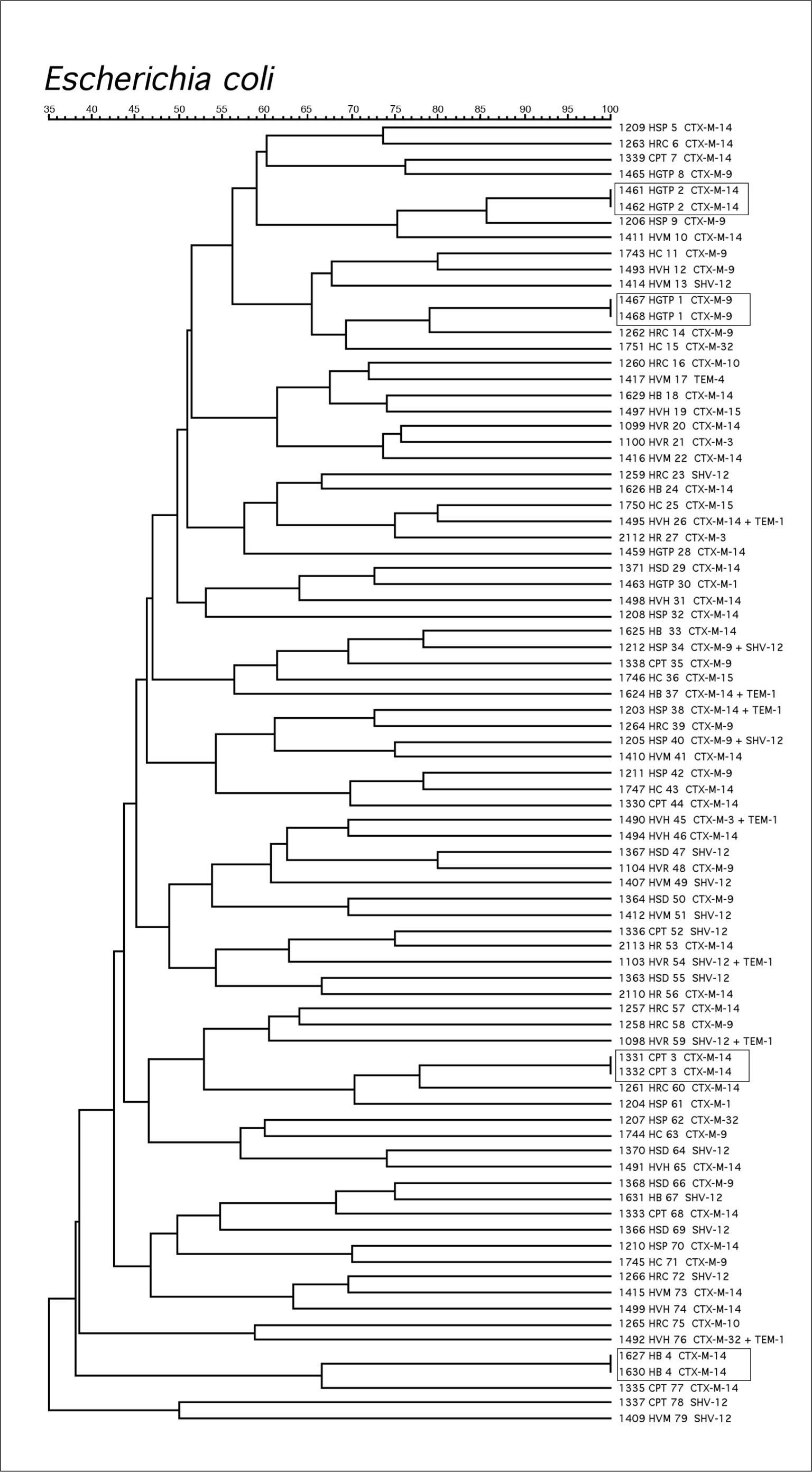
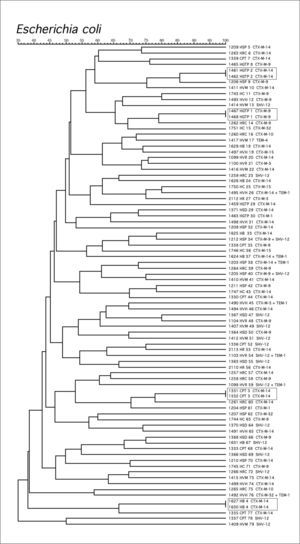
El análisis comparativo de los patrones de ADN genómico obtenidos por PFGE mostró una gran diversidad clonal entre los aislamientos de E. coli (fig. 1). Sólo se observaron cuatro agrupaciones de dos cepas cada uno con pulsotipos idénticos pertenecientes a tres hospitales distintos: dos en el HGTP (clones 1 y 2) y los otros dos en el HVH (clon 3) y el HB (clon 4). No se detectó ninguna transmisión interhospitalaria.
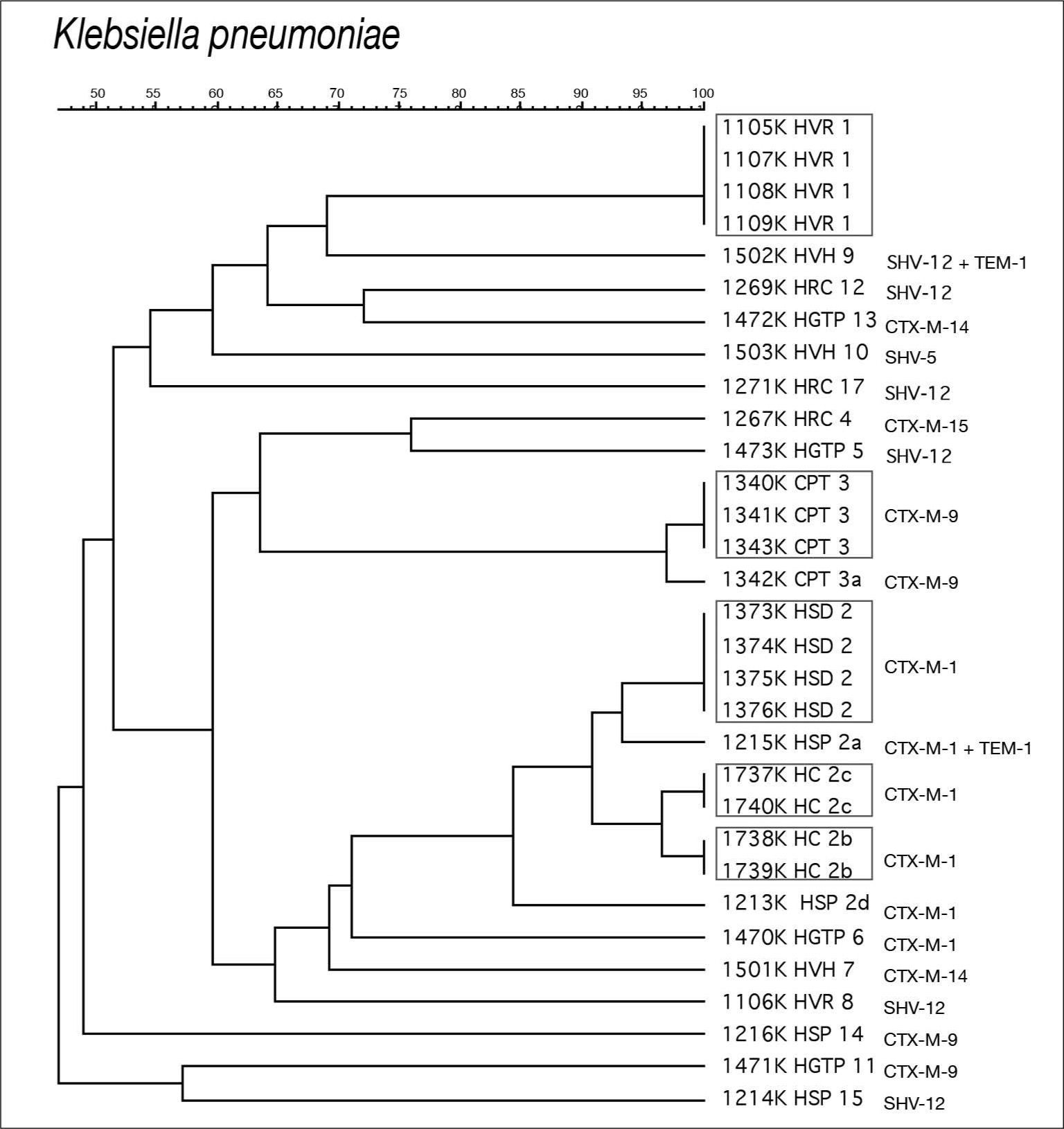
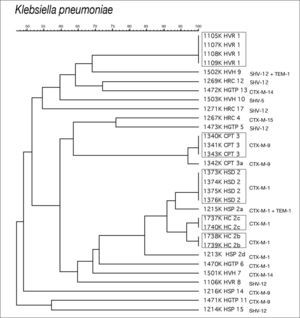
En K. pneumoniae se observó una menor diversidad clonal, detectándose tres agrupaciones que incluían 18 (53,1%) de los 32 aislamientos estudiados. La agrupación 1 estaba constituida por cuatro cepas con pulsotipos idénticos procedentes del HVR. La agrupación 2 incluía 10 cepas, genéticamente relacionadas y subagrupadas en cuatro cepas idénticas del HSD (patrón 2), una cepa del HSP probablemente relacionada (diferencia de dos bandas) (patrón 2a), y otras cuatro iguales, dos a dos, procedentes del HC probablemente relacionadas entre ellas (diferencia de una banda) y con las cepas del patrón 2 (diferencia de tres bandas) (patrón 2b y 2c). La cepa 1213 del HSP también se incluyó en la agrupación 2 (patrón 2d), considerándose posiblemente relacionada con el patrón 2. Se detectó una tercera agrupación (patrón 3) con cuatro cepas del CPT, tres idénticas y una cuarta probablemente relacionada (patrón 3a) (fig. 2).
Caracterización de BLEESe obtuvieron transconjugantes en el 72,6% de las cepas en las que se realizaron los ensayos de transferencia. La frecuencia de conjugación fue muy variable, entre 3 x 10−8 hasta > 10−1. Se obtuvieron transconjugantes en 70 de las 92 cepas de E. coli (76%) y en 20 de las 32 de K. pneumoniae (62,5%), sin que se detectara ninguna relación entre la frecuencia de conjugación y el tipo de BLEE implicada.
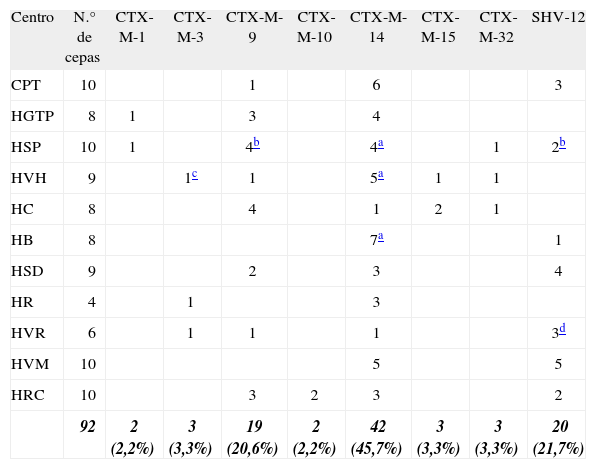
De la caracterización genética de las BLEE se obtuvo que 74 (80,4%) de las 92 cepas de E. coli presentaron una BLEE tipo CTX-M; 42 (45,7%), CTX-M-14; 19 (20,6%), CTX-M-9; dos (2,2%), CTX-M-1; tres (3,3%), CTX-M-15; tres (3,3%), CTX-M-3; tres (3,3%), CTX-M-32; dos (2,2%), CTX-M-10, y 20 (21,7%), SHV-12. Dos de estas cepas expresaron conjuntamente SHV-12 y CTX-M-9 (tabla 1).
Betalactamasas de espectro extendido caracterizadas en Escherichia coli
| Centro | N.° de cepas | CTX-M-1 | CTX-M-3 | CTX-M-9 | CTX-M-10 | CTX-M-14 | CTX-M-15 | CTX-M-32 | SHV-12 |
| CPT | 10 | 1 | 6 | 3 | |||||
| HGTP | 8 | 1 | 3 | 4 | |||||
| HSP | 10 | 1 | 4b | 4a | 1 | 2b | |||
| HVH | 9 | 1c | 1 | 5a | 1 | 1 | |||
| HC | 8 | 4 | 1 | 2 | 1 | ||||
| HB | 8 | 7a | 1 | ||||||
| HSD | 9 | 2 | 3 | 4 | |||||
| HR | 4 | 1 | 3 | ||||||
| HVR | 6 | 1 | 1 | 1 | 3d | ||||
| HVM | 10 | 5 | 5 | ||||||
| HRC | 10 | 3 | 2 | 3 | 2 | ||||
| 92 | 2 (2,2%) | 3 (3,3%) | 19 (20,6%) | 2 (2,2%) | 42 (45,7%) | 3 (3,3%) | 3 (3,3%) | 20 (21,7%) |
CPT: Corporació Hospitalaria Parc Taulí; HGTP: Hospital Germans Trias i Pujol; HSP: Hospital de la Santa Creu i Sant Pau; HVH: Hospital Vall d'Hebron; HC: Hospital Clínic de Barcelona; HB: Hospital de Bellvitge; HSD: Hospital Son Dureta; HR: Hospital de la Ribera; HVR: Hospital Virgen del Rocío; HVM: Hospital Virgen Macarena; HRC: Hospital Universitario Ramón y Cajal.
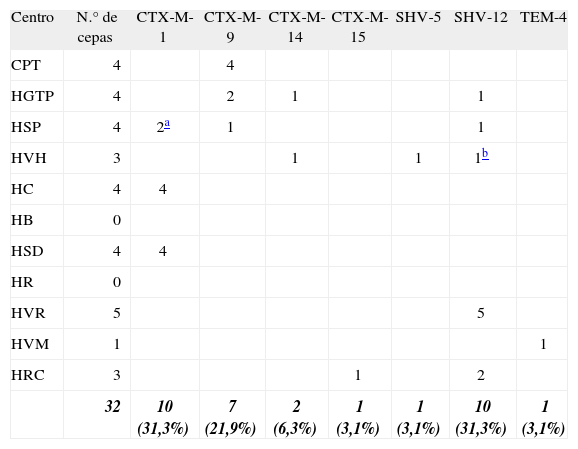
De las 32 cepas de K. pneumoniae, 20 (62,5%) presentaron una BLEE tipo CTX-M; 10 (31,3%) CTX-M-1, siete (21,9%) CTX-M-9, dos (6,3%) CTX-M-14, una (3,1%) CTX-M-15; 11 (34,4%) de tipo SHV, 10 (31,3%) SHV-12 y una (3,1%) SHV-5; y una TEM-4 (3,1%) (tabla 2).
Betalactamasas de espectro extendido (BLEE) presentes en las cepas de K. pneumoniae
De las cuatro agrupaciones de dos aislamientos cada una detectadas en E. coli, tres de ellas son productoras de CTX-M-14 y una de CTX-M-9 (fig. 1). En K. pneumoniae, la agrupación 1 produce una SHV-12, la agrupación 3 una CTX-M-9, y la agrupación 2 que afecta a tres hospitales distintos una CTX-M-1 (fig. 2).
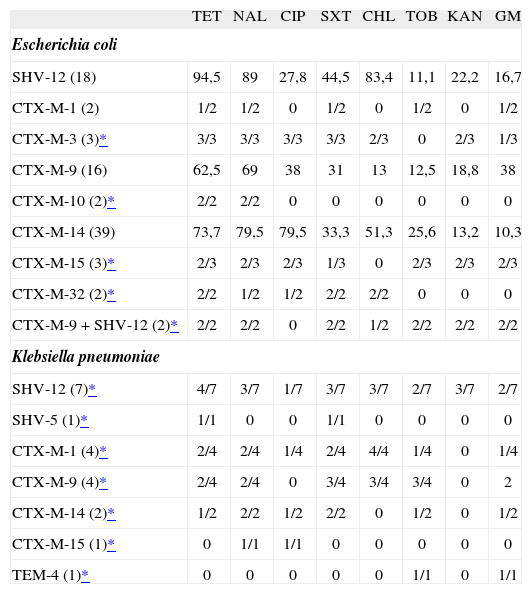
En función de la BLEE y el tipo de microorganismo, las cepas mostraron una multirresistencia a dos o más antibióticos no betalactámicos, principalmente a tetraciclina (de 62,5 a 94,5%), ácido nalidíxico (de 69 a 89%) y cotrimoxazol (de 33,3 a 44,5%) (tabla 3).
Porcentaje de resistencias asociadas en las cepas de Escherichia coli y Klebsiella pneumoniae con distintas betalactamasas de espectro extendido. Se ha incluido una cepa por clon y, en caso de electroforesis en gel de campos pulsantes y antibiogramas idénticos, sólo se ha contabilizado una cepa
| TET | NAL | CIP | SXT | CHL | TOB | KAN | GM | |
| Escherichia coli | ||||||||
| SHV-12 (18) | 94,5 | 89 | 27,8 | 44,5 | 83,4 | 11,1 | 22,2 | 16,7 |
| CTX-M-1 (2) | 1/2 | 1/2 | 0 | 1/2 | 0 | 1/2 | 0 | 1/2 |
| CTX-M-3 (3)* | 3/3 | 3/3 | 3/3 | 3/3 | 2/3 | 0 | 2/3 | 1/3 |
| CTX-M-9 (16) | 62,5 | 69 | 38 | 31 | 13 | 12,5 | 18,8 | 38 |
| CTX-M-10 (2)* | 2/2 | 2/2 | 0 | 0 | 0 | 0 | 0 | 0 |
| CTX-M-14 (39) | 73,7 | 79,5 | 79,5 | 33,3 | 51,3 | 25,6 | 13,2 | 10,3 |
| CTX-M-15 (3)* | 2/3 | 2/3 | 2/3 | 1/3 | 0 | 2/3 | 2/3 | 2/3 |
| CTX-M-32 (2)* | 2/2 | 1/2 | 1/2 | 2/2 | 2/2 | 0 | 0 | 0 |
| CTX-M-9 + SHV-12 (2)* | 2/2 | 2/2 | 0 | 2/2 | 1/2 | 2/2 | 2/2 | 2/2 |
| Klebsiella pneumoniae | ||||||||
| SHV-12 (7)* | 4/7 | 3/7 | 1/7 | 3/7 | 3/7 | 2/7 | 3/7 | 2/7 |
| SHV-5 (1)* | 1/1 | 0 | 0 | 1/1 | 0 | 0 | 0 | 0 |
| CTX-M-1 (4)* | 2/4 | 2/4 | 1/4 | 2/4 | 4/4 | 1/4 | 0 | 1/4 |
| CTX-M-9 (4)* | 2/4 | 2/4 | 0 | 3/4 | 3/4 | 3/4 | 0 | 2 |
| CTX-M-14 (2)* | 1/2 | 2/2 | 1/2 | 2/2 | 0 | 1/2 | 0 | 1/2 |
| CTX-M-15 (1)* | 0 | 1/1 | 1/1 | 0 | 0 | 0 | 0 | 0 |
| TEM-4 (1)* | 0 | 0 | 0 | 0 | 0 | 1/1 | 0 | 1/1 |
CHL: cloranfenicol; CIP: ciprofloxacino; GM: gentamicina; KAN: kanamicina; NAL: ácido nalidíxico; SXT: cotrimoxazol; TET: tetraciclina; TOB: tobramicina.
El creciente aumento de las infecciones por BLEE es un problema mundial que conlleva el incremento de la resistencia a antimicrobianos de primera elección, tanto en la comunidad como en los hospitales. Este aumento se atribuye, en general, al uso inadecuado de antimicrobianos y al intercambio de elementos genéticos de resistencia entre microorganismos23.
La prevalencia de cepas con BLEE puede ser muy variable entre diferentes países, ciudades e incluso hospitales de una misma ciudad24,25. Además, las BLEE detectadas pueden variar de una institución a otra. Son varios los estudios de seguimiento de BLEE en centros españoles donde se evidencia esta distinta distribución e incidencia9,10,21,26,27.
Las características demográficas de los pacientes portadores de BLEE en el presente estudio son similares a los estudios previamente publicados10. Es de destacar la mayor prevalencia de infecciones nosocomiales por K. pneumoniae que por E. coli (el 76 frente al 42%; p = 0,001). Alrededor del 75% de 50 estudios epidemiológicos realizados a escala mundial (donde se recogen datos de más de 3.000 pacientes) han revelado que las infecciones nosocomiales causadas por organismos productores de BLEE son en su mayoría causadas por K. pneumoniae2.
Los resultados de nuestro estudio muestran que entre las cepas de E. coli productoras de BLEE no existe un clon predominante. Se han encontrado pequeñas agrupaciones formadas por cepas que expresan mayoritariamente CTX-M-14. La caracterización de las BLEE mostró en E. coli un predominio de CTX-M-14 (45,7%), CTX-M-9 (20,6%) y SHV-12 (21,7%), aislándose también diversos tipos de CTX-M (CTX-M-1, CTX-M-3, CTX-M-15 y CTX-M-32). Estos resultados concuerdan con estudios puntuales anteriores, donde ya se describe, por un lado, una prevalencia mayor de CTX-M-14 que de CTX-M-9, así como un incremento en la diversidad de enzimas tipo CTX-M15,25,27, la sustitución de cepas portadoras de SHV-2 por cepas con SHV-1214,15 o la disminución de cepas portadoras de BLEE tipo TEM. El porcentaje de cepas portadoras de BLEE tipo TEM en el año 2000 era del 19%10, mientras que en este estudio no se ha aislado ninguna cepa. Es de resaltar la baja incidencia de cepas con CTX-M-15, ampliamente diseminada en otros países europeos25. Sólo se han detectado tres aislamientos de E. coli productores de CTX-M-15 y que no están genéticamente relacionados, al contrario de lo descrito previamente en aislamientos nosocomiales y comunitarios de distintos centros sanitarios de Madrid entre enero de 2004 y agosto de 20057. En conjunto, sólo cinco cepas de E. coli producían una variante de CTX-M de las que habitualmente generan altas CMI a ceftazidima además de a cefotaxima, tres CTX-M-15 y dos CTX-M-32.
En K. pneumoniae, al igual que en E. coli, las BLEE tipo TEM han disminuido, detectándose tan sólo TEM-4 (una cepa). De la familia SHV se encontraron las enzimas SHV-12 (31,3%) y SHV-5 (3,1%), no aislándose ninguna cepa con SHV-2. Nuestros resultados son similares a los descritos en algunos hospitales españoles13,14, pero difieren del estudio multicéntrico del año 2000, donde el 58% de las cepas presentaban BLEE tipo TEM y un 29% tipo SHV, describiéndose cepas con SHV-2 y SHV-2a10. Contabilizando una sola cepa con patrón de PFGE y antibiogramas idénticos, se observa que la enzima predominante en K. pneumoniae, dentro de la familia de las CTX-M, es CTX-M-1 y CTX-M-9, ambas con el 15,6% cada una, seguida de CTX-M-14 con el 6,2%. A diferencia de lo descrito en el estudio multicéntrico del año 2000, en el que se destacó la presencia de cepas portadoras de CTX-M-10 (13%)10 y que parece endémica en el área de Madrid26,28, en este trabajo se aislaron sólo dos cepas con esta enzima.
En el presente estudio también se evidencia la aparición de cepas de E. coli portadoras de dos BLEE (CTX-M-9 y SHV-12). Esta asociación ha sido documentada previamente15,16, aunque no se detectó en el estudio multicéntrico del año 200013.
Se ha detectado una mayor homología genética entre los distintos aislamientos de K. pneumoniae productoras de BLEE que la observada en E. coli, con la presencia de pequeñas agrupaciones producidas por cepas portadoras de SHV-12, CTX-M-9 y, principalmente, de CTX-M-1. Cabe destacar la diseminación clonal interhospitalaria de aislamientos de K. pneumoniae productores de CTX-M-1 detectada en dos centros de Barcelona (HSP, HC) y uno de Mallorca (HSD). En este último centro se había detectado previamente la existencia de un brote intrahospitalario de K. pneumoniae productora de CTX-M-129.
En conclusión, las cepas de E. coli y K. pneumoniae analizadas en este período presentan una mayor diversidad de BLEE que la observada en estudios epidemiológicos realizados con anterioridad. Además, el análisis de la relación clonal definió una gran diversidad en E. coli y menor en K. pneumoniae, aunque se detectó la presencia de cepas epidémicas de K. pneumoniae en diferentes áreas geográficas. Estos datos muestran la importancia de continuar con los estudios epidemiológicos de prevalencia y caracterización de estas enzimas. El incremento de su prevalencia, asociado al incremento de la resistencia a otras familias de antimicrobianos distintas de los betalactámicos, subraya la necesidad de establecer los factores que contribuyen a su expansión, incluyendo los de índole genética.
AgradecimientosEl presente estudio se ha podido realizar gracias a la financiación del Ministerio de Sanidad y Consumo, Instituto de Salud Carlos III-FEDER, Red Española de Investigación en Patología Infecciosa (REIPI C03/14) y Red Española de Investigación en Patología Infecciosa (REIPI RD06/0008), a la subvención económica otorgada a los proyectos FIS PI04339, PI 040162, PI040837 y PI 042012 y gracias a la participación de los grupos de investigación de los centros participantes bajo la dirección de L. Matas y M. Giménez en el Hospital Germans Trias i Pujol de Badalona, I. Sanfeliu en la Corporació Hospitalaria Parc Taulí de Sabadell, G. Prats en el Hospital Vall d'Hebron de Barcelona, M. Ruiz y J. Vila en el Hospital Clínic de Barcelona, M. Domínguez en el Hospital de Bellvitge de l'Hospitalet, A. Guerrero y J. Colomina en el Hospital de La Ribera de Alzira, A. Pascual en el Hospital Virgen Macarena y J. Aznar en el Hospital Virgen del Rocío de Sevilla.