Las técnicas de diagnóstico molecular por PCR permiten distinguir entre las diferentes especies de Cryptosporidium morfológicamente idénticas capaces de infectar a humanos. De las 23 especies actualmente reconocidas en el género, al menos 9 son capaces de infectar a humanos. Por ello, y debido a que la intensidad de las manifestaciones clínicas, la patogenicidad, la excreción de ooquistes y la incidencia varían entre ellas, la realización de estudios moleculares es crucial para una mejor comprensión de la epidemiología de la criptosporidiosis humana.
En el presente trabajo se analizan muestras procedentes de 2 estudios independientes: uno formado por 23 muestras procedentes de Madrid y otro compuesto por 72 muestras procedentes de La Coruña, todas ellas positivas para Cryptosporidium spp. por métodos microscópicos y pertenecientes a casos aislados de criptosporidiosis.
Para la identificación a nivel de especie se utilizaron las regiones de diagnóstico descritas para el ADNr 18S y las regiones de diagnóstico del gen de la COWP.
De las 95 muestras analizadas, se consiguió extraer y amplificar ADN en 77 casos, en los que las especies causantes de la infección fueron: C. parvum (40 casos: 2 Madrid y 38 La Coruña), C. hominis (30 casos: 10 Madrid y 20 La Coruña) y C. meleagridis (2 casos: uno Madrid y uno La Coruña). En otros 5 casos fue imposible detectar la especie responsable de la infección, aunque se confirmara su positividad por PCR (4 Madrid y uno La Coruña). Los genotipos aislados en estos pacientes se correlacionaron con los hallados en animales de las mismas regiones.
Molecular PCR based diagnostic techniques have enabled us to distinguish between the different, morphologically identical, Cryptosporidium species that can infect humans. Of the 23 recognized species in the genus, at least 9 are able to infect humans. As the intensity of the clinical manifestations, pathogenicity, excretion of oocysts, and incidence, are different between this species, molecular studies are crucial for a better understanding of the epidemiology of human cryptosporidiosis.
Samples form two independent studies are analyzed in this publication. One included 23 samples from Madrid, and the other, 72 samples from La Coruña. All of them positive for Cryptosporidium spp. by microscopic methods and belonging to isolated cases of human cryptosporidiosis.
For the identification of the species responsible for the infection, the 18S rDNA diagnostic region and the COWP gene diagnostic regions were used.
Out of the 95 samples tested, in 77 cases we were able to extract and amplify DNA. In those cases the species responsible for the infection were: C. parvum (40 cases, 2 Madrid and 38 La Coruña), C. hominis (30 cases, 10 Madrid and 20 La Coruña) and C. meleagridis (2 cases, 1 Madrid and 1 La Coruña). In 5 samples it was impossible to detect the species responsible for the infection, but their positivity was confirmed by PCR (4 Madrid and 1 La Coruña). The genotypes of the isolates from patients correlated well with animals from the same regions.
El género Cryptosporidium (Apicomplexa, Eucoccidiorida) incluye varias especies de coccidios parásitos que infectan células epiteliales, preferentemente del tracto digestivo, de un amplio número de vertebrados, incluyendo mamíferos, aves, peces y reptiles. Actualmente se reconocen 23 especies válidas1-7, además de haber varios genotipos pendientes de clasificación definitiva. Debido a las pequeñas diferencias morfométricas de sus ooquistes y a la baja especificidad de hospedador de las distintas especies del género, la identificación a nivel de especie requiere del uso de técnicas moleculares de amplificación por PCR1.
La criptosporidiosis humana, lejos de ser una infección poco usual, es relativamente común, con tasas de seroprevalencia que alcanzan el 25-35% en EE.UU.8 o el 20% en el Reino Unido9.
Durante años se consideró la criptosporidiosis como una antropozoonosis que implicaba principalmente a humanos y rumiantes, con C. parvum y C. hominis como únicos responsables de la infección. Sin embargo, aunque estas especies causan el 90% de los casos10, se han descrito infecciones por C. felis, C. muris, C. meleagridis, C. canis, C. suis, C. andersoni, C. ubiquitum, C. cuniculus o Cryptosporidium genotipo de mono11, entre otros. C. meleagridis, descrito tanto en individuos inmunocompetentes como en inmunosuprimidos, es considerado un parásito emergente10,12. Esta situación ha creado cierta controversia sobre el papel de los animales en la transmisión humana. Por un lado, varios estudios de subgenotipado sugieren que algunas de las infecciones por C. parvum tienen como origen a los mismos humanos. Por otro lado, otras especies y/o genotipos propios de animales afectan en menor medida a humanos, tanto inmunosuprimidos como inmunocompetentes13,14. En determinados países en vías de desarrollo, algunas especies inusuales pueden alcanzar hasta el 20% de los casos humanos estudiados14.
Debido a que la intensidad de las manifestaciones clínicas, la patogenicidad, el grado de excreción de ooquistes, la implicación en brotes hídricos y la incidencia varían entre las distintas especies14, es importante su correcta identificación para conocer el riesgo para la salud pública de la criptosporidiosis, así como el papel de las especies de Cryptosporidium presentes en animales, que pueden actuar como reservorio, y en el medio ambiente. Para ello se han desarrollado diversas técnicas de diagnóstico molecular, incluyendo técnicas de PCR-secuenciación, PCR-RFLP, RealTime-PCR, etc., que utilizan como dianas, entre otras, diversas regiones conservadas del ADN, como el ADNr 18S, ITS o el gen de la Cryptosporidium oocyst wall protein (COWP) para el diagnóstico a nivel de especie, o regiones más variables como los microsatélites ML1 y ML2 o el gen de la gp60 para la caracterización molecular a nivel de genotipo y subgenotipo1.
En España, la presencia de especies del género Cryptosporidium está bien documentada, tanto en humanos como en otros animales y el medio ambiente1. Sin embargo, los estudios moleculares llevados a cabo en muestras humanas son escasos, habiéndose demostrado la presencia en Zaragoza de C. hominis y C. parvum, además de una infección por C. meleagridis y otra por C. felis en niños15; y la presencia de C. hominis en Álava16.
MétodosSe estudiaron un total de 95 muestras fecales humanas procedentes de distintos hospitales españoles y que contenían ooquistes de Cryptosporidium sp. previamente identificados mediante métodos microscópicos (tinciones de Kinyoun y auramina) e inmunológicos (IFD Merifluor Crypto-Giardia, Meridian Diagnostics y ELISA Immnunocard STAT, Meridian Diagnostics). De ellas, 23 muestras procedían de varios hospitales de la comunidad autónoma de Madrid, todos ellos pacientes VIH+ con diarrea (13 niños y 10 adultos). Estas muestras procedían de una colección perteneciente a la Universidad San Pablo-CEU de Madrid, incluidas en un estudio de microsporidios realizado en 199617. Otras 72 muestras procedían del Servicio de Microbiología del Hospital Clínico Universitario de Santiago de Compostela (Complexo Hospitalario Universitario de Santiago, CHUS), La Coruña, pertenecientes a residentes en la provincia. De ellas, 69 pertenecían a individuos inmunocompetentes (62 niños y 7 adultos), una a un adulto VIH+ y 2 muestras de una probable infección nosocomial en lactantes. Todas ellas tomadas entre 2000 y 2003. Para la realización de las técnicas moleculares se utilizaron alícuotas de estas muestras conservadas a –80°C.
Para el proceso de rotura del ooquiste y la purificación del ADN obtenido se utilizó el kit comercial Fast DNA kit y el disruptor FP120 (QBioGene, Inc., Vista, California) según las recomendaciones de Da Silva et al.18. En todas las muestras se analizaron 3 loci diferentes: la región de diagnóstico del gen de la subunidad menor del ARN ribosomal (ADNr 18S)19 y los fragmentos codificantes de las regiones N-terminal y C-terminal de la COWP20, según las condiciones de amplificación por PCR previamente descritas19,20. La visualización de los productos de amplificación se realizó tras electroforesis en gel de agarosa y tinción con bromuro de etidio.
Estos productos de amplificación fueron purificados utilizando el StrataPrep PCR Purification kit® (Stratagene, Inc., La Jolla, California), secuenciados en ambos sentidos utilizando el BigDye Terminator kit® (Applied Biosystems, Foster City, California) y leídos en un secuenciador automático ABI-prism 3100 (Applied Biosystems).
La identificación de las especies presentes se llevó a cabo mediante el alineamiento de las secuencias de ADN obtenidas y su comparación con las secuencias publicadas en la base de datos del GenBank para las distintas especies de Cryptosporidium. Este proceso fue llevado a cabo con la aplicación informática SeqMan II program package® (DNASTAR Inc., Madison, Wisconsin).
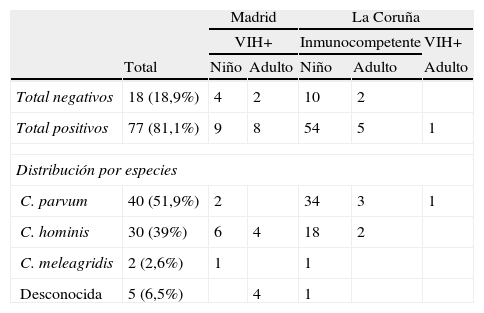
ResultadosDe las 95 muestras estudiadas, se amplificó al menos uno de los 3 loci estudiados en 77 muestras, el 81,1% de los casos (17 de Madrid y 60 de La Coruña). En las 18 muestras restantes (18,9%) no se obtuvo ningún producto de amplificación (6 de Madrid y 12 de La Coruña).
De las 77 muestras positivas, en 5 casos fue imposible determinar la especie responsable de la infección, ya que no se pudo obtener ninguna secuencia legible para ninguno de los loci analizados; en 40 se obtuvieron secuencias correspondientes a C. parvum; en 30 correspondientes a C. hominis, y otros 2 casos presentaban secuencias coincidentes con C. meleagridis (tabla 1). Los resultados obtenidos para cada uno de los 3 loci analizados se resumen en la tabla 2.
Resultados obtenidos para las 95 muestras analizadas y especies de Cryptosporidium halladas
| Madrid | La Coruña | |||||
| VIH+ | Inmunocompetente | VIH+ | ||||
| Total | Niño | Adulto | Niño | Adulto | Adulto | |
| Total negativos | 18 (18,9%) | 4 | 2 | 10 | 2 | |
| Total positivos | 77 (81,1%) | 9 | 8 | 54 | 5 | 1 |
| Distribución por especies | ||||||
| C. parvum | 40 (51,9%) | 2 | 34 | 3 | 1 | |
| C. hominis | 30 (39%) | 6 | 4 | 18 | 2 | |
| C. meleagridis | 2 (2,6%) | 1 | 1 | |||
| Desconocida | 5 (6,5%) | 4 | 1 | |||
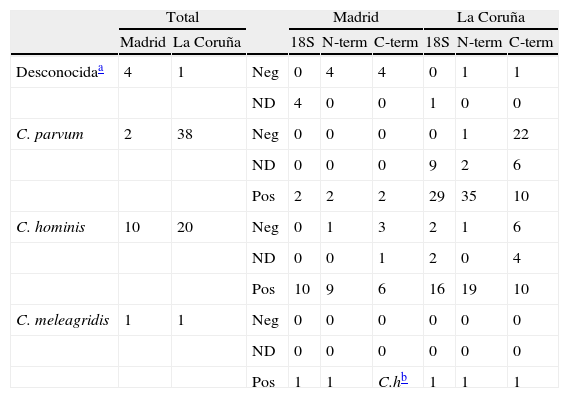
Resultados obtenidos para las muestras en cada locus analizado según la especie responsable de la infección
| Total | Madrid | La Coruña | |||||||
| Madrid | La Coruña | 18S | N-term | C-term | 18S | N-term | C-term | ||
| Desconocidaa | 4 | 1 | Neg | 0 | 4 | 4 | 0 | 1 | 1 |
| ND | 4 | 0 | 0 | 1 | 0 | 0 | |||
| C. parvum | 2 | 38 | Neg | 0 | 0 | 0 | 0 | 1 | 22 |
| ND | 0 | 0 | 0 | 9 | 2 | 6 | |||
| Pos | 2 | 2 | 2 | 29 | 35 | 10 | |||
| C. hominis | 10 | 20 | Neg | 0 | 1 | 3 | 2 | 1 | 6 |
| ND | 0 | 0 | 1 | 2 | 0 | 4 | |||
| Pos | 10 | 9 | 6 | 16 | 19 | 10 | |||
| C. meleagridis | 1 | 1 | Neg | 0 | 0 | 0 | 0 | 0 | 0 |
| ND | 0 | 0 | 0 | 0 | 0 | 0 | |||
| Pos | 1 | 1 | C.hb | 1 | 1 | 1 | |||
18S: resultados obtenidos en la región de diagnóstico del ADNr 18S; C-term: resultados obtenidos en la región de diagnóstico C-terminal del gen de la COWP; N-term: resultados obtenidos en la región de diagnóstico N-terminal del gen de la COWP; ND: PCR positiva pero no se obtuvo una secuenciada legible; Neg: PCR negativa para el locus en concreto, aunque positiva para uno de los otros 2 analizados; Pos: PCR positiva y secuencia legible comparable con las publicadas en genebank.
Para los casos de infección por C. hominis, de las 30 muestras positivas, en la región amplificada del ADNr 18S se obtuvieron 18 amplicones cuyas secuencias se correspondían con la publicada con número de GenBank AF093489 como C. parvum genotipo humano, renombrada como C. hominis. En las 2 regiones de diagnóstico del gen de la COWP todos los fragmentos obtenidos mostraron secuencias correspondientes con la publicada para C. hominis con número de GenBank XM661099, 28 amplicones en la región N-terminal y 16 en la región C-terminal.
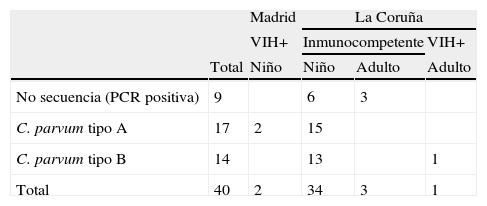
Para los casos de infección por C. parvum, de las 40 muestras positivas en el fragmento de diagnóstico del ADNr 18S se obtuvieron 2 secuencias distintas. Catorce aislados poseían una secuencia correspondiente con la publicada con número de GenBank L16996, para C. parvum genotipo bovino, que denominamos tipoA (tabla 3). Otras 17 mostraban una deleción de 3 bases en las posiciones 648 a 650 (TGA), y una substitución T a C en la posición 663 y A a T en la posición 689, y que se correspondían con la secuencia publicada como ADNr 18S tipo B de C. parvum genotipo 2 con número de GenBank AF308600 (tabla 3). En cuanto a los fragmentos que codifican para las regiones N-terminal y C-terminal de la COWP, las secuencias encontradas se correspondían con la publicada para C. parvum genotipo bovino con número de GenBank Z22537, 37 en la región N-terminal y 12 en la región C-terminal.
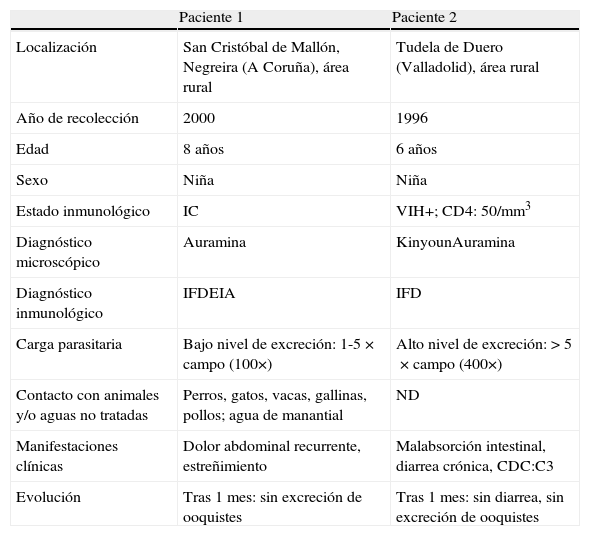
Los 2 casos de infección por C. meleagridis fueron diagnosticados en 2 niñas de 6 y 8 años procedentes de Valladolid y La Coruña (datos clínicos en tabla 4). En la región de diagnóstico del ADNr 18S se obtuvieron secuencias idénticas a la publicada para C. meleagridis con número de GenBank AF248759. En la región N-terminal de la COWP, las 2 secuencias obtenidas fueron idénticas a la secuencia de C. meleagridis con número de GenBank AY166840. Sin embargo, en la región C-terminal de la COWP se obtuvieron 2 resultados distintos. Mientras que la paciente2 presentaba una secuencia coincidente con la publicada para C. meleagridis con número de GenBank EU310392, la paciente1 presentaba una secuencia idéntica a la publicada con número de GenBank XM661099 para C. hominis, sugiriendo la presencia de una infección mixta. Con el fin de descartar una posible contaminación, se realizaron 2 extracciones y amplificaciones adicionales a partir de la muestra original, obteniendo resultados idénticos en todas ellas.
Datos clínicos de los pacientes positivos para C. meleagridis
| Paciente 1 | Paciente 2 | |
| Localización | San Cristóbal de Mallón, Negreira (A Coruña), área rural | Tudela de Duero (Valladolid), área rural |
| Año de recolección | 2000 | 1996 |
| Edad | 8 años | 6 años |
| Sexo | Niña | Niña |
| Estado inmunológico | IC | VIH+; CD4: 50/mm3 |
| Diagnóstico microscópico | Auramina | KinyounAuramina |
| Diagnóstico inmunológico | IFDEIA | IFD |
| Carga parasitaria | Bajo nivel de excreción: 1-5×campo (100×) | Alto nivel de excreción: >5×campo (400×) |
| Contacto con animales y/o aguas no tratadas | Perros, gatos, vacas, gallinas, pollos; agua de manantial | ND |
| Manifestaciones clínicas | Dolor abdominal recurrente, estreñimiento | Malabsorción intestinal, diarrea crónica, CDC:C3 |
| Evolución | Tras 1 mes: sin excreción de ooquistes | Tras 1 mes: sin diarrea, sin excreción de ooquistes |
EIA: ensayo comercial ELISA Immnunocard STAT (Meridian Diagnostics, Inc.); IC: inmunocompetente; IFD: ensayo comercial de immunofluorescencia Merifluor Crypto-Giardia (Meridian Diagnostics, Inc.); ND: no hay datos.
En España se ha informado, dentro de la Red Nacional de Vigilancia Epidemiológica, de al menos 1.812 casos de infección desde 199521-23. Además, se han publicado diversos estudios en población adulta y en niños, tanto en inmunocompetentes como en inmunosuprimidos, y asimismo se han detectado varios brotes epidémicos1. Estos estudios han sido realizados mayoritariamente en población en riesgo que presentaba sintomatología o en la que ya se sospechaba la infección. Con todo ello, los datos epidemiológicos acerca de la criptosporidiosis son escasos y sesgados, ya que son pocos los hospitales que declaran la presencia de Cryptosporidium, al no realizar su diagnóstico de forma rutinaria. Por ello los datos deben ser tomados con cautela, y cabe considerar que los datos reales de infección pueden ser mayores que los publicados, tanto por subnotificación como por la falta de universalización de los test diagnósticos en los laboratorios de microbiología. Hasta finales de los años noventa la detección se realizaba básicamente utilizando métodos microscópicos. Más recientemente se han utilizado diversas técnicas moleculares para el diagnóstico, sobre todo en muestras procedentes de animales y ambientales, habiéndose corroborado la presencia en nuestro país de C. parvum, C. hominis, C. meleagridis, C. felis, C. muris, C. bovis, C. bovis-like, C. baileyi, C. andersoni, C. varanii y los genotipos de cerdo, ratón y tortuga1.
Los resultados globales obtenidos en el presente estudio muestran una mayor prevalencia de C. parvum, como ha ocurrido normalmente en Europa. Sin embargo, un estudio publicado en humanos procedentes de Zaragoza muestra una mayor prevalencia de C. hominis frente a C. parvum15. Aunque en Gran Bretaña sigue habiendo una mayor frecuencia de C. parvum24, varios estudios publicados en otros países europeos muestran esta tendencia de aumento de la frecuencia de C. hominis, como ocurre en Estados Unidos y en Australia25. Si analizamos nuestros datos según el área, observamos que las muestras procedentes de Madrid, pertenecientes a pacientes VIH-positivos, presentan una mayor proporción de infección por C. hominis, mientras que en La Coruña la especie más abundante es C. parvum. Esta distinta distribución podría deberse a la dispersión rural y ganadera del área sanitaria que cubre el CHUS, que hace más factible un predominio de la especie zoonótica, en comparación con el área eminentemente urbana de Madrid, como se ha observado en otros estudios14. Sin embargo, es necesario un estudio más amplio y minucioso de los genotipos y subgenotipos presentes para diferenciar entre los casos de zoonosis y los casos de infección antroponótica por C. parvum.
Entre los aislados donde se detectó la presencia de C. parvum, en la región de diagnóstico del ADNr 18S se hallaron 2 secuencias distintas, nombradas como tipoA y tipoB. Este gen presenta 5 copias en el genoma del parásito, por lo que en un mismo ooquiste podría presentar ambas secuencias. Sin embargo, es difícil demostrar tanto este hecho como que una misma secuencia esté presente en las 5 copias de un ooquiste mientras que la otra lo esté en las 5 copias de otro ooquiste distinto. De este modo, aunque no podamos asegurar que todos los alelos presentes en una población de parásitos sean el mismo, sí podemos admitir, debido a la calidad de las secuencias obtenidas en cada una de las muestras, que la mayoría de las copias presentes en esa población de parásitos es el secuenciado para la muestra, independientemente de su distribución en cada ooquiste concreto, siendo por tanto representativo de la misma.
En lo referente a las muestras positivas para C. parvum que presentan la secuencia tipo B (14 casos) es importante remarcar que este es el primer estudio realizado en España que indica su presencia, ya descrita en humanos inmunocompetentes e inmunosuprimidos en Francia26. En España ya se conocía la presencia de esta misma secuencia en animales procedentes de distintas regiones, donde se ha encontrado en bovinos, ovinos y caprinos27-29. Este hecho indica la presencia de este genotipo tanto en el ciclo de transmisión zoonótico como en el antroponótico en nuestro país.
Finalmente, las 2 muestras positivas para C. meleagridis representan el 2,6% de los pacientes estudiados, datos mayores que los observados en Zaragoza (0,98%)15 y el Reino Unido, implicando a 2.411 pacientes (tanto inmunocompetentes como VIH+, adultos y niños), donde se obtuvo un valor del 0,9%24. En Portugal se han descrito prevalencias entre el 4,7630 y el 10,34%31, todas en pacientes VIH+. Por cercanía geográfica, es de suponer que la prevalencia en España debe de ser más próxima a la de Portugal que a la obtenida en el Reino Unido. Sin embargo, el bajo número de muestras estudiadas en la península Ibérica hace tomar estos datos con cautela, ya que pueden llevar a una estimación errónea de la prevalencia real de C. meleagridis en nuestro país.
El estudio de 3 loci para el diagnóstico ha permitido detectar un caso de coinfección por C. meleagridis y C. hominis en la paciente1. La coinfección con distintas especies de Cryptosporidium ha sido descrita previamente por otros autores, siendo un hecho ampliamente demostrado24.
La manifestación típica de infección por Cryptosporidium spp. es la diarrea aguda o crónica en individuos inmunocompetentes o inmunodeprimidos, respectivamente. Las manifestaciones del paciente2 (VIH+) correspondían con estos síntomas. Sin embargo, el paciente1 (inmunocompetente) presentaba dolor abdominal asociado a estreñimiento, que aun siendo un síntoma atípico, había sido informado previamente32. Tras un mes, las heces de ambos pacientes fueron negativas para Cryptosporidium sp.
Aunque la ruta de infección de C. meleagridis en humanos no está clara, se ha demostrado la presencia del parásito en mascotas y en el medio ambiente33,34. En este estudio las pacientes han mantenido un contacto frecuente con animales y/o áreas rurales (tabla 4), lo que permite suponer que la infección puede haber ocurrido por un contacto accidental con los ooquistes en el medio ambiente.
Por ello es necesario un examen más detallado de los factores de riesgo en casos de infección con especies inusuales de Cryptosporidium, para poder tener un mejor conocimiento del ciclo de transmisión en nuestro país. Esto implica la realización de nuevos estudios que permitan comprender mejor la epidemiología y las rutas de infección responsables de la criptosporidiosis humana en España. Y sobre todo, es necesario establecer la importancia en salud pública de las distintas especies, genotipos y subgenotipos pertenecientes a este género, no solo de C. parvum y C. hominis.
Conflicto de interesesLos autores declaran que no existe ningún conflicto de intereses.