Introducción
En 1961, pocos meses después de la introducción de la meticilina en la práctica clínica, se detectaron en el Reino Unido las primeras cepas de Staphylococcus aureus resistentes a meticilina (SARM)1. A principios de la década de 1960, las cepas de SARM se diseminaron rápidamente a lo largo de varios hospitales europeos, y se convirtieron, a partir de los años 1980, en uno de los principales patógenos responsables de infecciones nosocomiales, sobre todo en las unidades quirúrgicas o de cuidados intensivos2,3.
En España, el primer brote intrahospitalario por este microorganismo se describió en 1981 en un hospital de San Sebastián4,5. La mayoría de los brotes de SARM entre 1989-1995 estaban producidos por el llamado clon Ibérico, detectado por primera vez en el Hospital Universitario de Bellvitge de Barcelona en 19896. Dicho clon se diseminó a otros países, como Portugal, Escocia, Italia, Bélgica, Alemania, y a un hospital de Nueva York7,8. Las cepas de este clon se caracterizaron por presentar resistencia a múltiples grupos de antibióticos: macrólidos, tetraciclinas, aminoglucósidos y quinolonas, y ser sensibles a los glucopéptidos y al cotrimoxazol. A partir de mediados de la década de 1990, en el conjunto de España, ha ido disminuyendo el predominio del clon Ibérico, siendo sustituido por otros clones de SARM sensibles a más antibióticos, como muestran los datos de un reciente estudio de ámbito nacional realizado conjuntamente por el Grupo de Estudio de Infección Hospitalaria (GEIH), Grupo de Estudio de Mecanismos de Acción y Resistencia a Antimicrobianos (GEMARA) y la Red Española de Investigación en Patología Infecciosa (REIPI)9.
En el Hospital Universitario Son Dureta de Palma de Mallorca, el SARM fue rara vez aislado hasta junio de 1999, cuando se detectó el primer brote de SARM, seguido de una endemia moderada.
Recientemente se ha constado un aumento en la frecuencia de cepas de SARM multirresistentes, lo que motivó la realización de un estudio de epidemiología molecular de los aislados recuperados en muestras clínicas de los pacientes ingresados en nuestro hospital entre el 1 de julio de 2002 y el 30 de junio de 2003. Además, los resultados obtenidos se han comparado con datos históricos de 1999-2000.
Métodos
El Hospital Son Dureta es un hospital terciario con aproximadamente 900 camas, de referencia para toda la Comunidad de las Islas Baleares. Está compuesto por dos pabellones de hospitalización separados, aunque situados en el mismo recinto: el hospital general y el hospital materno-infantil.
A partir de los registros microbiológicos se han documentado todos los pacientes con aislamiento de SARM en muestras clínicas que han ingresado en nuestro hospital entre el 1 de julio de 2002 hasta el 30 de junio de 2003. No se incluyeron en el estudio las cepas aisladas de cultivos de vigilancia del estado de portador (exudados nasales, axilares y/o inguinales). La identificación de los aislados de S. aureus se realizó por métodos convencionales10.
El perfil de sensibilidad antibiótica (oxacilina, penicilina, eritromicina, clindamicina, ciprofloxacino, gentamicina, cotrimoxazol, vancomicina, teicoplanina, rifampicina, mupirocina y ácido fusídico) se determinó mediante el método de difusión con discos (Rosco, Taastrup, Dinamarca) siguiendo los criterios del National Committee for Clinical Laboratory Standards (NCCLS)11. Además, se realizó aglutinación con látex de los aislados de SARM para confirmar la presencia de PBP2' (Slidex MRSA Detection, bioMérieux, Marcy l'Etoile, Francia).
La relación clonal de los aislados de SARM se determinó mediante electroforesis en campo pulsado (PFGE), utilizando SmaI como enzima de restricción. La electroforesis se realizó utilizando el aparato Chef-DR® III (Bio-Rad, Richmond, EE.UU.) con las siguientes condiciones: pulso inicial de 1 s, pulso final de 30 s, tiempo de electroforesis 23 h a 6 V/cm. Los patrones de bandas de la PFGE se interpretaron según los criterios de Tenover12. Como controles se utilizaron los clones de SARM predominantes en un estudio multicéntrico nacional P1 y Q19 y los clones pandémicos Ibérico (ATTC BAA-44)7, Brasileño (ATTC BAA-43)13, Húngaro (ATTC-BAA 39)14, Nueva York/Japón (ATTC BAA-41)15 y Pediátrico (ATTC BAA-42)16. La cepa de S. aureus NCTC 832512 se utilizó como marcador de peso molecular.
Para el estudio comparativo se utilizó una colección de 161 cepas de SARM, previamente tipificadas, de pacientes ingresados en nuestro hospital durante los años 1999 y 2000 (datos no publicados). La nomenclatura de los clones de SARM de nuestro trabajo se basó en la utilizada en este estudio.
Resultados
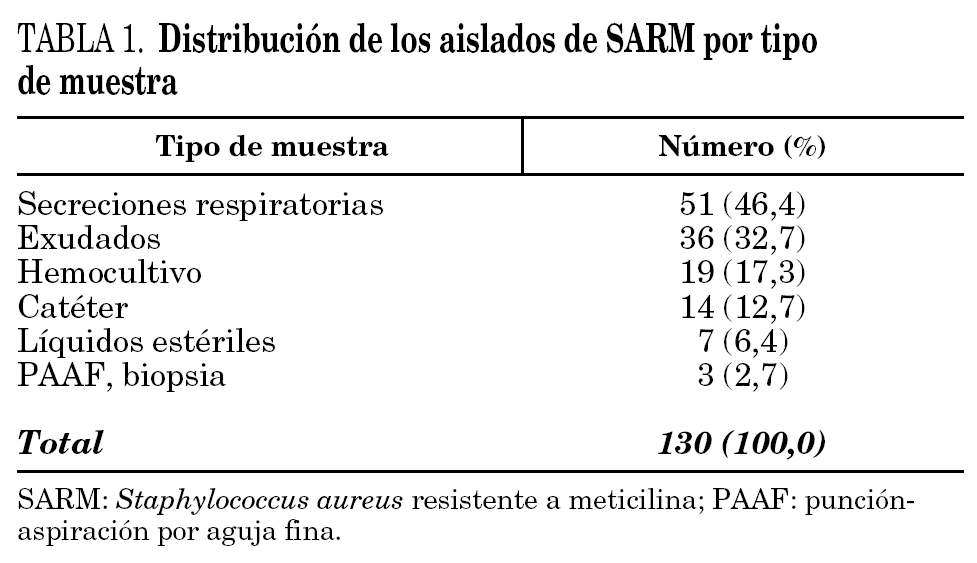
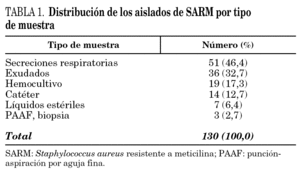
Durante el período del estudio (julio de 2002-junio de 2003), el porcentaje de aislados de SARM en muestras clínicas con respecto al total de los aislados de S. aureus fue del 38%; este porcentaje descendió al 30% al referirlo al número de pacientes distintos con S. aureus en una muestra clínica. Se aisló SARM en 110 pacientes distintos: en 86 de ellos (78%) se detectó en una única muestra, mientras que en 24 (22%) se aisló en más de una. Las muestras más frecuentes fueron las secreciones respiratorias (51 pacientes; 46%), exudados (36 pacientes; 33%), hemocultivos (19 pacientes; 17%), y catéteres (14 pacientes; 13%) (tabla 1).
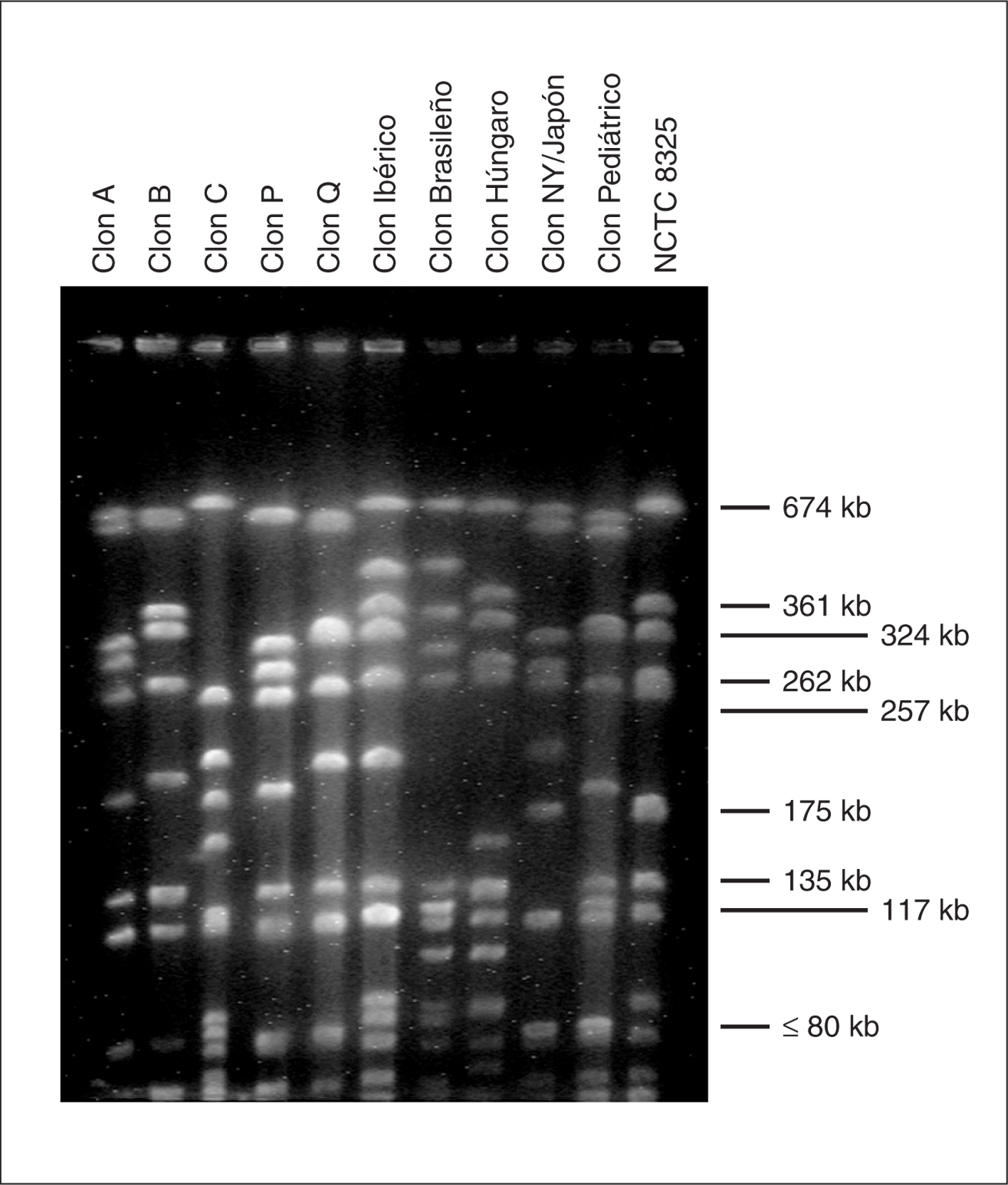
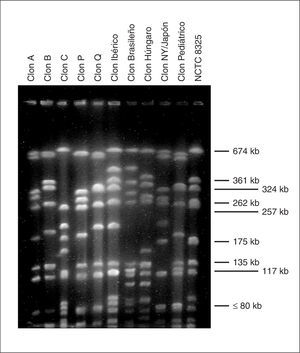
Mediante PFGE se detectaron tres clones mayoritarios denominados A, B y C, que comprendían el 93% de los pacientes, así como ocho clones minoritarios, cada uno de éstos en un único paciente. El clon A se encontró en 21 (19,1%) pacientes, el clon B en 63 (57,3%) pacientes y el clon C en 18 (16,4%) pacientes, cada uno de ellos con varios subtipos. El patrón de bandas del campo pulsado en los tres clones más frecuentes se muestra en la figura 1.
Figura 1. Comparación de los patrones de macrorrestricción con SmaI de los tres clones mayoritarios con diferentes clones de SARM representativos a nivel nacional y mundial. Carriles 1 a 11: clon A, clon B, clon C, clon P19, clon Q19, clon Ibérico7, clon Brasileño13, clon Húngaro14, clon Nueva York/Japón15, clon Pediátrico16, Staphylococcus aureus NCTC 832512 (utilizado como marcador de peso molecular).
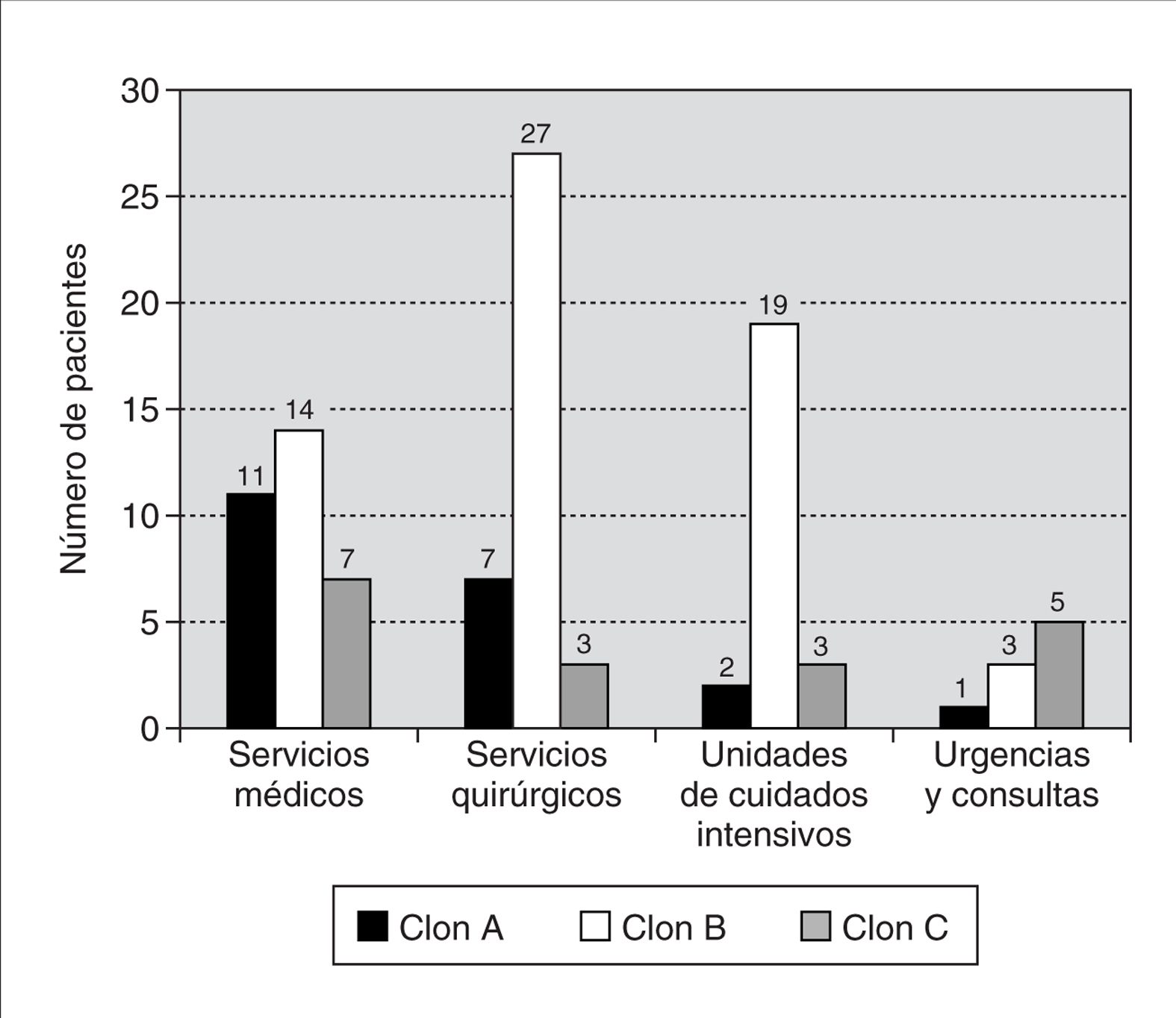
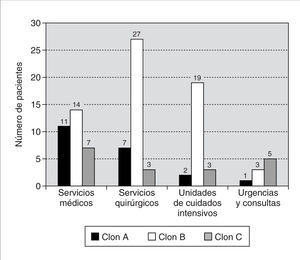
La distribución por servicios de los clones de SARM se resume en la figura 2. El clon B fue el más frecuente en pacientes de unidades de cuidados intensivos (76%) y de áreas quirúrgicas (69%), mientras que en los servicios médicos la frecuencia de los clones A y B fue similar.
Figura 2. Distribución de los distintos clones de SARM del Hospital Universitario Son Dureta por servicios.
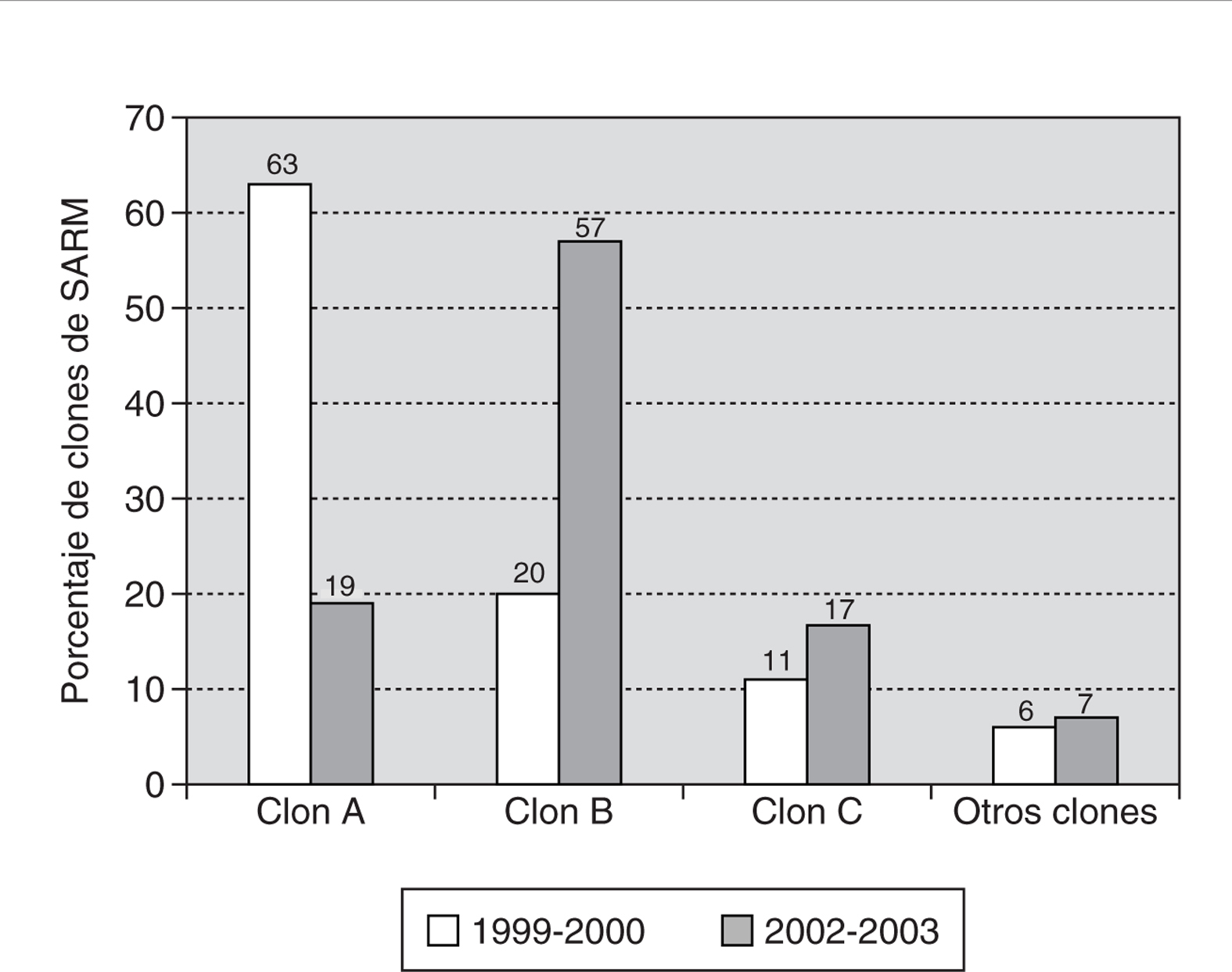
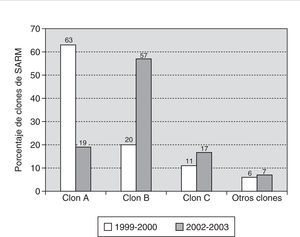
Estos tres clones mayoritarios estaban ya presentes en el estudio previo de 1999-2000, aunque en menos de 2 años ha habido un importante cambio en su distribución. Mientras que en 1999-2000 predominaba el clon A (63% de los aislados), actualmente éste ha sido desplazado por el clon B (57% de los aislados) (fig. 3). Este desplazamiento coincide con un aumento del porcentaje de resistencia a meticilina que pasó del 21% (por pacientes distintos) en el estudio previo (1999-2000) al 30% en el estudio actual (2002-2003).
Figura 3. Comparación de la distribución de los clones de SARM del HUSD en los períodos 1999-2000 y 2002-2003.
Ninguno de los tres clones mayoritarios está aparentemente relacionado genéticamente con los cinco clones de SARM pandémicos descritos17: los clones Ibérico6,7, Brasileño13, Húngaro14, Nueva York/Japón15 y Pediátrico16 (fig. 1). Asimismo, ninguno de los tres clones está directamente relacionado con los dos más frecuentemente encontrados en el estudio de ámbito nacional, aunque los clones A y B presentan cierta similitud con los clones P y Q9, respectivamente.
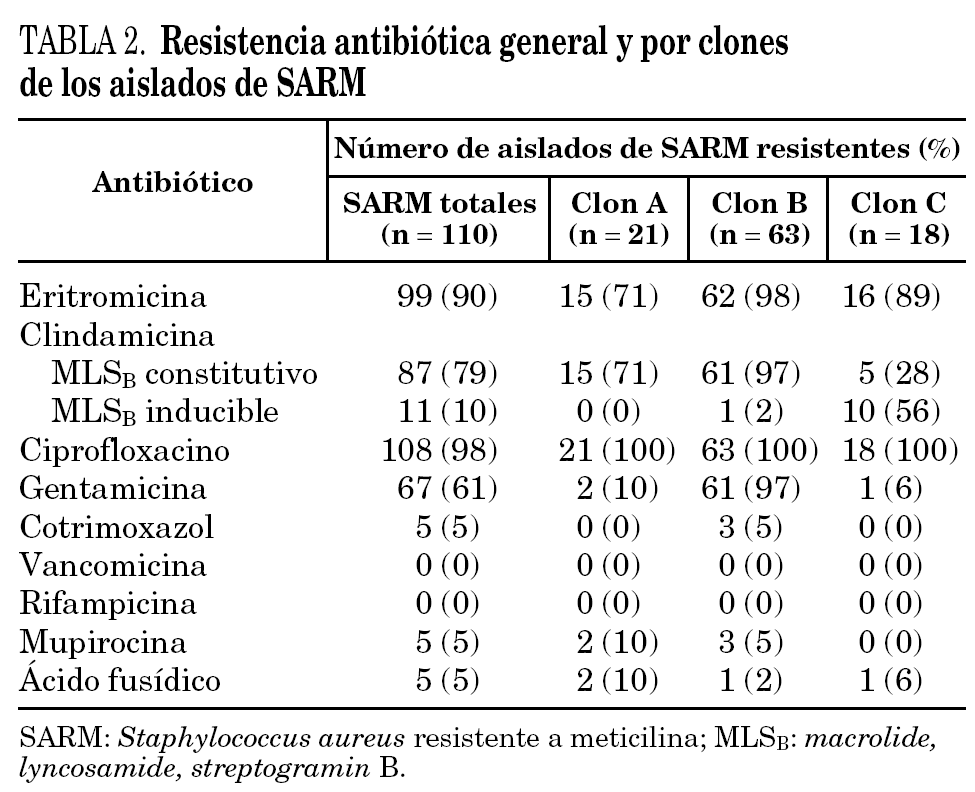
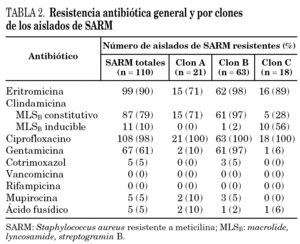
La resistencia general de todos los aislados de SARM del estudio y de los tres clones mayoritarios se muestra en la tabla 2. Ninguna cepa de SARM fue resistente a la vancomicina, teicoplanina o rifampicina. Únicamente el 4,5% fueron resistentes a cotrimoxazol, mupirocina y a ácido fusídico. La mayoría fueron resistentes a ciprofloxacino (98%) y a eritromicina (90%). La resistencia a la gentamicina fue variable (61%). Con respecto a la clindamicina, 87 aislados (79%) presentaban una resistencia MLSB(macrolide, lyncosamide, streptogramin B) constitutiva, mientras que 11 (10%) mostraban un fenotipo MLSB inducible, la mayoría de los cuales (10 de los 11) pertenecían al clon C. El nuevo clon mayoritario (clon B) fue significativamente más resistente que los otros, presentando de forma uniforme resistencia a la eritromicina, clindamicina, ciprofloxacino y gentamicina.
Discusión
Staphylococcus aureus resistente a la meticilina continúa siendo uno de los principales patógenos nosocomiales. Una vez que se introduce en el hospital, por lo general se llega a situaciones de endemicidad, a pesar de las medidas de control de la infección8. En los últimos años, el reservorio de SARM ha pasado de detectarse inicialmente en los grandes hospitales, a las residencias geriátricas y centros de larga estancia, suponiendo un riesgo de transmisión a la comunidad. Un estudio realizado por nuestro grupo en S. aureus procedentes de úlceras de decúbito de pacientes ingresados en una residencia geriátrica para la que nuestro hospital es el centro de referencia mostró el 70% de resistencia a la oxacilina en los aislados de S. aureus. El análisis de PFGE reveló, entre los 17 aislados de SARM de pacientes distintos, la presencia de cuatro clones, todos ellos encontrados también en pacientes ingresados en nuestro hospital18.
En el Hospital Son Dureta, el primer brote de SARM se detectó en junio de 1999, 10 años más tarde que en otros hospitales de Madrid y Barcelona. Inicialmente se aisló en pacientes ingresados en las unidades de cuidados intensivos y traumatología, extendiéndose después a la mayoría de las plantas del pabellón de hospitalización general. Curiosamente, hasta la fecha, no se ha encontrado ningún aislado de SARM en pacientes del pabellón materno-infantil. La situación epidémica inicial de nuestro hospital se transformó en una endemia moderada.
Durante el período del estudio (julio de 2002-junio de 2003), el porcentaje de SARM con respecto al total de los aislados de S. aureus fue del 38%, mientras que el porcentaje de SARM en relación al número de pacientes con aislamiento de S. aureus fue del 30%. Este porcentaje es superior al encontrado en 36 hospitales españoles en el año 2002 según el European Antimicrobial Resistance Surveillance System (EARSS)19, que fue del 23%, y también al del estudio español de los grupos GEIH/GEMARA/REIPI realizado en junio de 2003 en el que participaron 66 hospitales, con una frecuencia media de SARM del 21,8%20. Nuestra tasa de SARM es similar a la encontrada en otros países mediterráneos como Italia (38%) y Francia (33%) y algo inferior a la del Reino Unido (44%)19.
En este trabajo, la gran mayoría de SARM (93%) detectados recientemente pertenecían únicamente a tres clones. En este sentido, concuerda con otros que señalan que las cepas de SARM hospitalarias corresponden a unos pocos clones. Así, por ejemplo, en un estudio realizado en más de 3.000 aislados de SARM procedentes de hospitales de Europa y América, casi el 70% de los aislados pertenecía a uno de los cinco clones pandémicos17. En otro trabajo realizado en 217 aislados de SARM de 12 hospitales de varios estados de Estados Unidos, el 79% pertenecía a dos únicos clones. Por último, en el estudio español de GEIH/GEMARA/REIPI el 80% de las cepas pertenecía a dos clones mayoritarios, que llamaron clon P y clon Q9.
En el estudio de la sensibilidad antibiótica, nuestros aislados tenían una alta resistencia al ciprofloxacino (98%), eritromicina (90%), clindamicina (79%) y gentamicina (61%). La resistencia a la mupirocina (5%) fue más baja que la informada en otros centros (22% en el estudio español9) probablemente debido al uso del ácido fusídico en vez de la mupirocina como primera elección en el tratamiento de la descontaminación de los portadores de SARM. La cifra de resistencia al ácido fusídico es baja (5%), pero se desconocen los datos de resistencia a este antibiótico a nivel nacional.
Si se comparan los resultados de 2002-2003 con el estudio previo de 1999-2000, se puede observar que en los últimos años se ha producido un acusado desplazamiento del clon inicialmente predominante (clon A) por otro con mayores resistencias asociadas (clon B). El nuevo clon mayoritario presenta resistencia uniforme al ciprofloxacino, eritromicina, clindamicina y especialmente a gentamicina, característica que le diferencia del resto de los clones mayoritarios en los que el porcentaje de resistencia a este antibiótico únicamente fue del 10 y del 6% para los clones A y C, respectivamente.
Ninguno de los tres clones mayoritarios está aparentemente relacionado genéticamente con los cinco clones de SARM pandémicos descritos. No obstante, si bien el clon C es completamente distinto del resto, los clones A y B de este estudio presentan importantes similitudes con los clones P y Q, respectivamente (fig. 1), quizá reflejando la presencia de una línea ancestral común que debe evaluarse con técnicas con mayor capacidad para establecer relaciones clonales a largo plazo como el MLST (multilocus sequence typing). El clon C parece sin embargo mostrar un perfil de restricción similar al clon EMRSA-15 común en hospitales británicos21.
Mientras que la tendencia nacional es una clara disminución de las resistencias asociadas en las cepas de SARM9, en nuestro hospital ha ocurrido justamente lo contrario: el desplazamiento de un clon moderadamente resistente por otro multirresistente (no relacionado con el clon Ibérico) en un período de tiempo relativamente corto. Este desplazamiento determina el hecho preocupante de que las resistencias asociadas a SARM en nuestro medio sean muy superiores a la media nacional. En este sentido destacan las resistencias a la clindamicina y la gentamicina que doblan la media nacional (79% frente a 38% y 61% frente a 26% para la clindamicina y la gentamicina, respectivamente)9.
Agradecimientos
Agradecemos a la Dra. Herminia de Lencastre del Instituto de Tecnologia Quimica e Biologica da Universidade Nova de Lisboa, Oeiras, Portugal y al Dr. Alexander Tomasz de The Rockefeller University, Nueva York, el envío de las cinco cepas de los clones pandémicos de SARM; y también a las Dras. M.ª Ángeles Domínguez y Carmen Borraz del Servicio de Microbiología del Hospital Universitario de Bellvitge por el envío de las dos cepas de los clones P y Q. Este trabajo ha sido parcialmente financiado por la Red Española de Investigación en Patología Infecciosa (REIPI) del Fondo de Investigaciones Sanitarias (FIS C03/014).