El género Cryptosporidium, ha sido objeto de importantes revisiones en los últimos años, tanto a nivel taxonómico, con la identificación de nuevas especies y sus principales reservorios, como por la contribución de esta información al conocimiento de la epidemiología de la infección en humanos. En España, aunque son múltiples las publicaciones realizadas, todavía son pocos los estudios llevados a cabo para la identificación de las especies y genotipos circulantes. Este hecho nos ha motivado a realizar una revisión actualizando estas novedades y los estudios publicados en España, especialmente aquellos donde se utilicen métodos moleculares que permiten la caracterización de especies y genotipos presentes en nuestro país.
Genus Cryptosporidium, has undergone major revisions in recent years. The identification of new species and their major reservoirs has contributed to the knowledge of the epidemiology of human infection. In Spain, although there are many publications, few studies have been conducted to identify the circulating species and genotypes. This fact has led us to review and update these new studies published in Spain, particularly those that use molecular methods in order to characterise the species and genotypes present in our country.
Desde que en 1907 Tyzzer describiera un parásito infectando las células del tracto digestivo de ratón, que denominó Cryptosporidium1, hasta que se reconoció la importancia patogénica de este género en humanos, pasaron varios decenios. Fue en 1955, con la descripción de C. meleagridis en pavos2, y más adelante, en los años 70, con el descubrimiento de Cryptosporidium infectando ganado bovino3, cuando se empezó a apreciar su importancia en veterinaria. Los primeros casos de infección en humanos se detectaron en 1976 asociados a diarrea acuosa4,5 y 1982 en individuos VIH-positivos6, precisando el carácter oportunista de este protozoo, en aquel momento emergente7. Su importancia para la salud pública no se reconoció hasta que en 1993 causara un gran brote epidémico por contaminación de aguas de consumo que afectó a más de 400.000 personas en Milwaukee, EE.UU8. Este episodio motivó el inicio de estudios de biología básica, métodos de toma de muestras, detección, prevención y tratamiento de este parásito7.
Lejos de ser una infección inusual, la criptosporidiosis es relativamente común, con tasas de seroprevalencia que alcanzan el 25-35% en EE.UU.9 o el 20% en el Reino Unido10. Además, aproximadamente un 6,1% de los casos de diarrea en pacientes inmunocompetentes de países de baja renta podrían ser debidos a Cryptosporidium spp.11.
El género Cryptosporidium incluye varias especies de parásitos intracelulares obligados que infectan células epiteliales, preferentemente del tracto digestivo. Su estadio de resistencia y propagación son los ooquistes, que miden de 3 a 8μm de diámetro7.
Taxonómicamente, este género está incluido en la clase Coccidea, dentro del filo Apicomplexa. Sin embargo, recientes descubrimientos han reabierto el debate sobre su filiación. El hecho de: a) ser un parásito extracitoplasmático, en contraposición al resto de coccidios que son claramente intracelulares12; b) su separación filogenética del resto de coccidios, situándolos cerca de la clase Gregarinasina13–15, y c) la descripción in vivo e in vitro de estadios extracelulares presentes en estos últimos16–18 ubican a Cryptosporidium fuera del grupo de los coccidios, situando la divergencia entre gregarinos y Cryptosporidium en la base del filo Apicomplexa con anterioridad a la emergencia de los coccidios12.
La identificación presuntiva de Cryptosporidium puede llevarse a cabo mediante distintas técnicas de tinción (Kinyoun, auramina, etc.)7,19–21. Para la identificación de género se pueden utilizar técnicas de detección de antígenos como inmunofluorescencia o enzimoinmunoensayos7. Sin embargo, la identificación de especie requiere del uso de técnicas moleculares de amplificación por PCR. Esto es debido a las pequeñas diferencias morfométricas de los ooquistes y a la baja especificidad de hospedador de las especies del género Cryptosporidium. En este sentido, se han desarrollado diversas técnicas, tanto para el diagnóstico como para la caracterización molecular de las distintas especies y genotipos pertenecientes al género Cryptosporidium, que utilizan regiones del genoma con distinta variabilidad según los objetivos del estudio.
De forma general, para la determinación de especie, se han utilizado regiones de baja o moderada variabilidad. Entre los genes de baja variabilidad encontramos aquellos que codifican: el gen de la subunidad menor del ARN ribosomal (ADNr 18S)22–25, la proteína de la pared del ooquiste (COWP)26,27, la proteína de choque térmico de 70 KDa (HSP-70)28, o el gen que codifica la actina29. Entre las regiones de variabilidad moderada se han utilizado los genes de la β-tubulina30,31, genes TRAP (C1, C2 y C4)32–34 o las regiones intergénicas ITS-1 e ITS-225,35–39. Estas regiones se utilizan tanto en estudios de taxonomía como diagnóstico o epidemiología, sin embargo estas regiones únicamente identifican la especie y algunos genotipos. Por ello, para la identificación de genotipos, subtipos o linajes, se utilizan regiones más variables, como el gen de la glucoproteína de 60 kDa (GP60)40,41 y mini o microsatélites como el ML142 y ML243.
Por otro lado, y debido a la necesidad en epidemiología y salud pública de la caracterización de poblaciones y subgenotipos dentro de las distintas especies del género Cryptosporidium, se está utilizando frecuentemente el análisis de varios loci hipervariables (en inglés MLT, multilocus typing) que aumenten la resolución del subgenotipado. De este modo, se crean unos patrones de MLT dependiendo de las combinaciones de genotipo para cada loci analizado44–47. Estos estudios se pueden realizar por detección de diferencias de longitud de los fragmentos amplificados (MLFT) en gel de agarosa o por secuenciación (MLST), permitiendo el uso de marcadores con SNP (single nucleotide polymorphism) además de micro y minisatélites44.
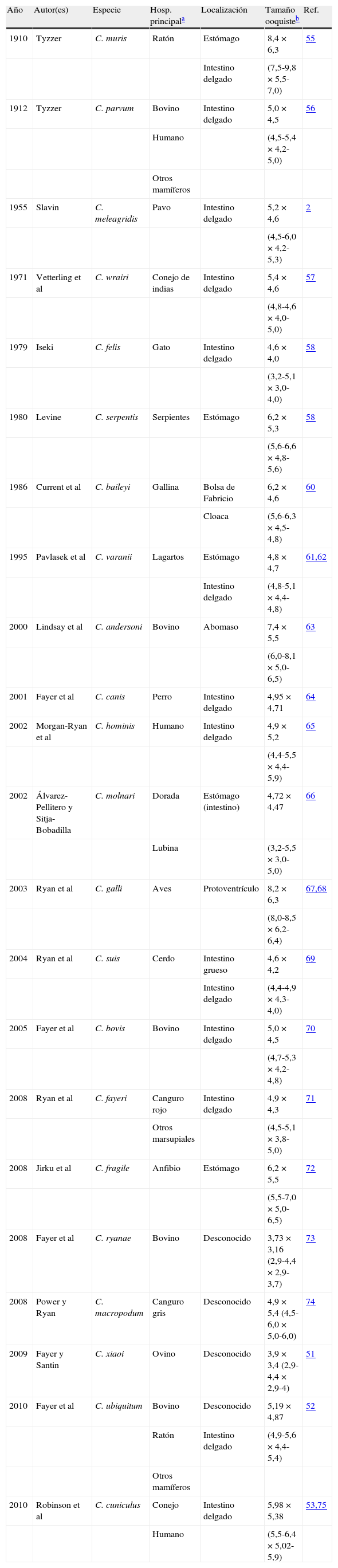
Por todo ello, durante el 6th International Meeting on Molecular Epidemiology and Evolutionary Genetics in Infectious Diseases, llevado a cabo en el Instituto Pasteur, en París, en julio de 2002, se sugirió que para la designación de especies dentro del género Cryptosporidium se deberían cumplir los siguientes requisitos básicos: a) estudios morfométricos del ooquiste; b) caracterización multigénica con genes de segregación independiente; c) estudios de especificidad de hospedador, tanto natural como experimental, y d) conformidad con el ICZN -–International Code of Zoological Nomenclature–48,49. Atendiendo a estos criterios, actualmente se reconocen 22 especies válidas50–53 (tabla 1). Además, existen varios genotipos pendientes de su clasificación definitiva49,50,54, lo que indica que el género está en continua revisión.
Especies aceptadas del género Cryptosporidium y características morfométricas de los ooquistes.
| Año | Autor(es) | Especie | Hosp. principala | Localización | Tamaño ooquisteb | Ref. |
| 1910 | Tyzzer | C. muris | Ratón | Estómago | 8,4×6,3 | 55 |
| Intestino delgado | (7,5-9,8×5,5-7,0) | |||||
| 1912 | Tyzzer | C. parvum | Bovino | Intestino delgado | 5,0×4,5 | 56 |
| Humano | (4,5-5,4×4,2-5,0) | |||||
| Otros mamíferos | ||||||
| 1955 | Slavin | C. meleagridis | Pavo | Intestino delgado | 5,2×4,6 | 2 |
| (4,5-6,0×4,2-5,3) | ||||||
| 1971 | Vetterling et al | C. wrairi | Conejo de indias | Intestino delgado | 5,4×4,6 | 57 |
| (4,8-4,6×4,0-5,0) | ||||||
| 1979 | Iseki | C. felis | Gato | Intestino delgado | 4,6×4,0 | 58 |
| (3,2-5,1×3,0-4,0) | ||||||
| 1980 | Levine | C. serpentis | Serpientes | Estómago | 6,2×5,3 | 58 |
| (5,6-6,6×4,8-5,6) | ||||||
| 1986 | Current et al | C. baileyi | Gallina | Bolsa de Fabricio | 6,2×4,6 | 60 |
| Cloaca | (5,6-6,3×4,5-4,8) | |||||
| 1995 | Pavlasek et al | C. varanii | Lagartos | Estómago | 4,8×4,7 | 61,62 |
| Intestino delgado | (4,8-5,1×4,4-4,8) | |||||
| 2000 | Lindsay et al | C. andersoni | Bovino | Abomaso | 7,4×5,5 | 63 |
| (6,0-8,1×5,0-6,5) | ||||||
| 2001 | Fayer et al | C. canis | Perro | Intestino delgado | 4,95×4,71 | 64 |
| 2002 | Morgan-Ryan et al | C. hominis | Humano | Intestino delgado | 4,9×5,2 | 65 |
| (4,4-5,5×4,4-5,9) | ||||||
| 2002 | Álvarez-Pellitero y Sitja-Bobadilla | C. molnari | Dorada | Estómago (intestino) | 4,72×4,47 | 66 |
| Lubina | (3,2-5,5×3,0-5,0) | |||||
| 2003 | Ryan et al | C. galli | Aves | Protoventrículo | 8,2×6,3 | 67,68 |
| (8,0-8,5×6,2-6,4) | ||||||
| 2004 | Ryan et al | C. suis | Cerdo | Intestino grueso | 4,6×4,2 | 69 |
| Intestino delgado | (4,4-4,9×4,3-4,0) | |||||
| 2005 | Fayer et al | C. bovis | Bovino | Intestino delgado | 5,0×4,5 | 70 |
| (4,7-5,3×4,2-4,8) | ||||||
| 2008 | Ryan et al | C. fayeri | Canguro rojo | Intestino delgado | 4,9×4,3 | 71 |
| Otros marsupiales | (4,5-5,1×3,8-5,0) | |||||
| 2008 | Jirku et al | C. fragile | Anfibio | Estómago | 6,2×5,5 | 72 |
| (5,5-7,0×5,0-6,5) | ||||||
| 2008 | Fayer et al | C. ryanae | Bovino | Desconocido | 3,73×3,16 (2,9-4,4×2,9-3,7) | 73 |
| 2008 | Power y Ryan | C. macropodum | Canguro gris | Desconocido | 4,9×5,4 (4,5-6,0×5,0-6,0) | 74 |
| 2009 | Fayer y Santin | C. xiaoi | Ovino | Desconocido | 3,9×3,4 (2,9-4,4×2,9-4) | 51 |
| 2010 | Fayer et al | C. ubiquitum | Bovino | Desconocido | 5,19×4,87 | 52 |
| Ratón | Intestino delgado | (4,9-5,6×4,4-5,4) | ||||
| Otros mamíferos | ||||||
| 2010 | Robinson et al | C. cuniculus | Conejo | Intestino delgado | 5,98×5,38 | 53,75 |
| Humano | (5,5-6,4×5,02-5,9) |
La criptosporidiosis humana ha sido clásicamente considerada una antropozoonosis que tiene como reservorios principales a bovinos y al hombre, aunque existe cierta controversia con respecto a la importancia que desempeñan los animales en la transmisión a humanos76. Se reconocen como especies más frecuentes en humanos C. parvum y C. hominis, habiéndose descrito infecciones por otras especies o genotipos minoritarios como C. felis77, C. muris78, C. meleagridis79, C. canis77, C. suis80, C. ubiquitum81, C. cuniculus82 y Cryptosporidium genotipo de mono83. C. meleagridis es la tercera especie en importancia, habiéndose descrito tanto en individuos inmunocompetentes como inmunosuprimidos, considerándose actualmente como parásito emergente49. La intensidad de las manifestaciones clínicas, la patogenicidad, el grado de excreción de ooquistes y la incidencia varía entre las distintas especies que afectan a humanos7,84. Por tanto, para conocer el riesgo para la salud pública de la criptosporidiosis es importante la correcta identificación de especies y genotipos, tanto en muestras humanas, como de otros animales o ambientales, siendo imprescindible el uso de técnicas de PCR85.
Situación epidemiológica en EspañaLa presencia de especies pertenecientes al género Cryptosporidium en España está bien documentada, habiéndose descrito tanto en humanos como en otros animales y en el medio ambiente.
El primer caso de contaminación ambiental se describió en 1994 con la detección de ooquistes de Cryptosporidium en aguas de consumo y plantas de potabilizadoras de Salamanca86. Desde entonces se han realizado varios estudios para detectar los reservorios ambientales y los posibles medios de propagación del parásito a humanos y animales. Entre ellos se han analizado: aguas de ríos y otras zonas de captación de aguas para consumo, lodos de diques, lodos y aguas procedentes de plantas de tratamiento de aguas residuales y potabilizadoras, utilizadas para riego y consumo humano respectivamente86–98; mostrando frecuencias de contaminación entre el 15,493 y el 100%86,89,95. Además, un reciente estudio ha estimado que aproximadamente el 90% de las muestas positivas para Cryptosporidium spp. presentaban ooquistes viables88.
Adicionalmente, está bien documentada la presencia de Cryptosporidium spp. en varias especies de bivalvos filtradores de consumo humano (almejas, ostras y mejillones) que acumulan grandes cantidades de ooquistes94,99–103. La posibilidad de transmisión a humanos a través del consumo estos moluscos parece evidente, al constatarse que un 53% de las muestras contaminadas contienen ooquistes viables101 y que estos no son inactivados al utilizar los habituales métodos de cocción al vapor104.
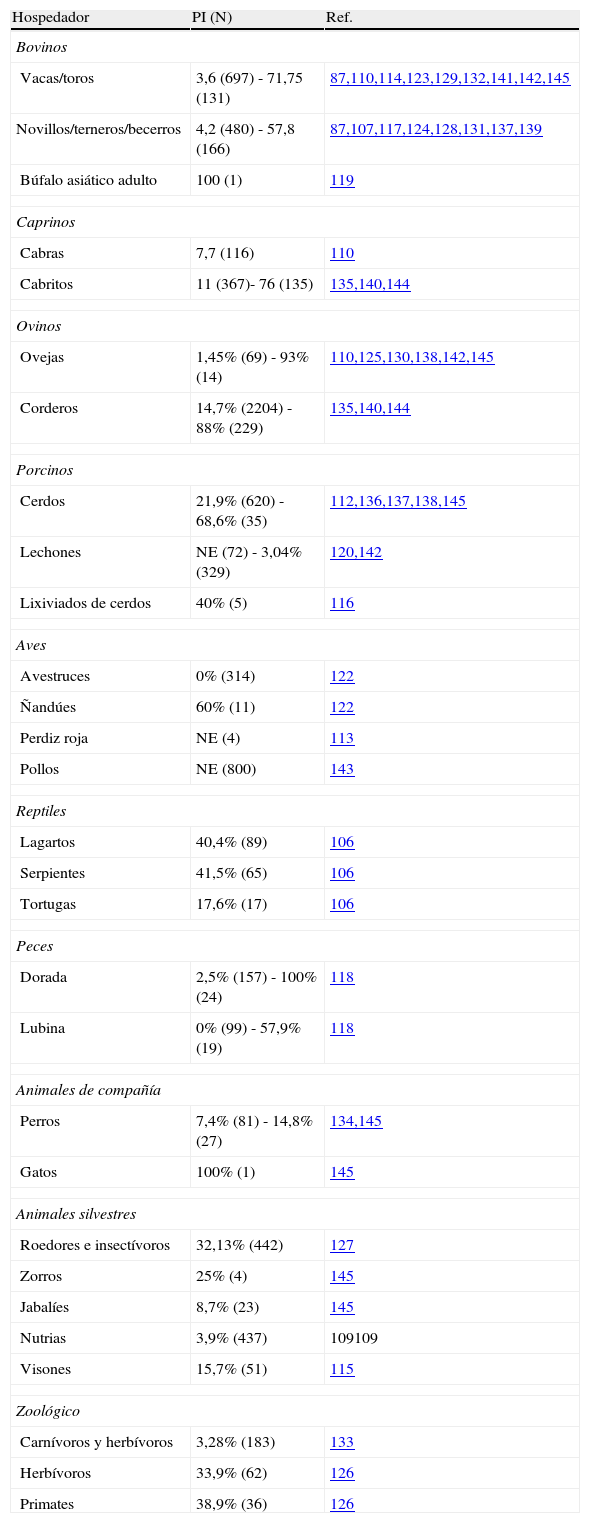
La criptosporidiosis animal se ha estudiado en España, sobre todo en animales de interés comercial y en algunos animales salvajes. Entre los animales para consumo humano, se han realizado estudios en bovinos, ovinos, porcinos, pollos, avestruces, ñandúes, peces y búfalo asiático. También se han estudiado animales de interés en peletería como el visón, animales de zoológicos (primates, carnívoros y herbívoros), animales de compañía (perros y reptiles) y fauna silvestre (jabalíes, zorros, nutrias, búhos, palomas)87,89,105–145 (tabla 2).
Detección de Cryptosporidium sp. en animales.
| Hospedador | PI (N) | Ref. |
| Bovinos | ||
| Vacas/toros | 3,6 (697) - 71,75 (131) | 87,110,114,123,129,132,141,142,145 |
| Novillos/terneros/becerros | 4,2 (480) - 57,8 (166) | 87,107,117,124,128,131,137,139 |
| Búfalo asiático adulto | 100 (1) | 119 |
| Caprinos | ||
| Cabras | 7,7 (116) | 110 |
| Cabritos | 11 (367)- 76 (135) | 135,140,144 |
| Ovinos | ||
| Ovejas | 1,45% (69) - 93% (14) | 110,125,130,138,142,145 |
| Corderos | 14,7% (2204) - 88% (229) | 135,140,144 |
| Porcinos | ||
| Cerdos | 21,9% (620) - 68,6% (35) | 112,136,137,138,145 |
| Lechones | NE (72) - 3,04% (329) | 120,142 |
| Lixiviados de cerdos | 40% (5) | 116 |
| Aves | ||
| Avestruces | 0% (314) | 122 |
| Ñandúes | 60% (11) | 122 |
| Perdiz roja | NE (4) | 113 |
| Pollos | NE (800) | 143 |
| Reptiles | ||
| Lagartos | 40,4% (89) | 106 |
| Serpientes | 41,5% (65) | 106 |
| Tortugas | 17,6% (17) | 106 |
| Peces | ||
| Dorada | 2,5% (157) - 100% (24) | 118 |
| Lubina | 0% (99) - 57,9% (19) | 118 |
| Animales de compañía | ||
| Perros | 7,4% (81) - 14,8% (27) | 134,145 |
| Gatos | 100% (1) | 145 |
| Animales silvestres | ||
| Roedores e insectívoros | 32,13% (442) | 127 |
| Zorros | 25% (4) | 145 |
| Jabalíes | 8,7% (23) | 145 |
| Nutrias | 3,9% (437) | 109109 |
| Visones | 15,7% (51) | 115 |
| Zoológico | ||
| Carnívoros y herbívoros | 3,28% (183) | 133 |
| Herbívoros | 33,9% (62) | 126 |
| Primates | 38,9% (36) | 126 |
N: número de muestras estudiadas en cada uno de los estudios mostrados; NE: no especificado; PI: porcentaje de infección (rango).
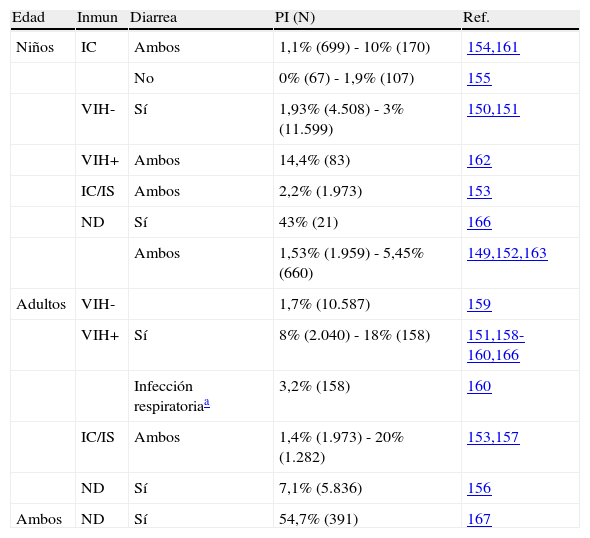
En cuanto a la infección en humanos, y debido a que hasta el año 2009 no ha sido una enfermedad que debiera ser notificada a la Red Nacional de Vigilancia Epidemiológica146, los datos obtenidos para la población general son escasos. Estos, proceden principalmente de hospitales y son remitidos de forma voluntaria, cubriendo un 25% de la población española (sólo 5 de las 19 regiones españolas participan)147. Dentro de esta Red, el Sistema de Información Microbiológica ha informado de 1.676 casos de infección entre 1995 y 2009147,148. Estos estudios han sido realizados mayoritariamente en poblaciones en riesgo que presentaba sintomatología o donde ya se sospechaba la infección. Se han publicado estudios en población adulta y en niños, tanto en inmunocompetentes como en inmunosuprimidos, sobre todo VIH-positivos en diversas regiones, como Salamanca, Zaragoza, Cádiz, Islas Canarias o Madrid145,149–165 (tabla 3). En ellos, las tasas de infección encontradas suelen ser mayores en niños e inmunosuprimidos que en adultos e inmunocompetentes. Además de estos casos esporádicos, también se han detectado varios brotes epidémicos, que deben ser informados obligatoriamente mediante un procedimiento estandarizado. El primer brote se notificó en 1997 en una escuela166. Desde ese año y hasta 2007 se notificaron 10 brotes más, involucrando hoteles, escuelas, ciudades e incluso un picnic145,147. El más importante se notificó en el año 2003, diagnosticándose 214 casos de infección en turistas británicos de un hotel de Mallorca y fue debido a la contaminación del agua de una piscina167. En los brotes no siempre ha podido determinarse la fuente de infección, sin embargo en los casos en que pudo serlo se ha atribuido siempre a aguas, tanto de la red de consumo como recreacionales145,147.
Detección de Cryptosporidium sp. en humanos.
| Edad | Inmun | Diarrea | PI (N) | Ref. |
| Niños | IC | Ambos | 1,1% (699) - 10% (170) | 154,161 |
| No | 0% (67) - 1,9% (107) | 155 | ||
| VIH- | Sí | 1,93% (4.508) - 3% (11.599) | 150,151 | |
| VIH+ | Ambos | 14,4% (83) | 162 | |
| IC/IS | Ambos | 2,2% (1.973) | 153 | |
| ND | Sí | 43% (21) | 166 | |
| Ambos | 1,53% (1.959) - 5,45% (660) | 149,152,163 | ||
| Adultos | VIH- | 1,7% (10.587) | 159 | |
| VIH+ | Sí | 8% (2.040) - 18% (158) | 151,158-160,166 | |
| Infección respiratoriaa | 3,2% (158) | 160 | ||
| IC/IS | Ambos | 1,4% (1.973) - 20% (1.282) | 153,157 | |
| ND | Sí | 7,1% (5.836) | 156 | |
| Ambos | ND | Sí | 54,7% (391) | 167 |
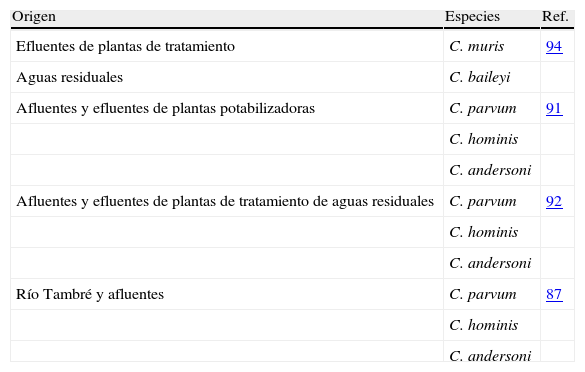
Los estudios publicados en España hasta el año 2000 sólo utilizaban métodos microscópicos tras tinción para la detección de Cryptosporidium y, por tanto, las especies involucradas no pudieron ser determinadas, aunque se asociaron generalmente a C. parvum. La disponibilidad y uso de técnicas moleculares en posteriores estudios han puesto de manifiesto la existencia en nuestro país de varias especies y genotipos de Cryptosporidium, tanto en muestras ambientales (tabla 4), como en aquellas procedentes de animales (tabla 5) y de humanos (tabla 6).
Especies/genotipos encontrados en aguas en Galicia.
| Origen | Especies | Ref. |
| Efluentes de plantas de tratamiento | C. muris | 94 |
| Aguas residuales | C. baileyi | |
| Afluentes y efluentes de plantas potabilizadoras | C. parvum | 91 |
| C. hominis | ||
| C. andersoni | ||
| Afluentes y efluentes de plantas de tratamiento de aguas residuales | C. parvum | 92 |
| C. hominis | ||
| C. andersoni | ||
| Río Tambré y afluentes | C. parvum | 87 |
| C. hominis | ||
| C. andersoni |
Especies/genotipos encontrados en animales.
| Hospedador | Procedencia geográfica | Especies | Ref. |
| Búfalo asiático | Galicia | Similar a C. parvum genotipo de cerdo | 119 |
| Bovinos | Galicia | C. parvum | 110 |
| Novillos | Galicia | C. parvum | 117 |
| Terneros | Galicia | 27C. parvum | 168 |
| Terneros | Aragón | C. parvum | 107 |
| 2C. bovis | |||
| Ovejas | Zona noroeste | 1C. bovis-like (C. xiaoi) | 111 |
| Corderos | Galicia | 14C. parvum | 168 |
| 9C. ubiquitum | |||
| Cabra | Galicia -Lugo | 2C. xiaoi | 169 |
| Ovejas y cabras | Aragón | 37C. parvum tipo Aa | 108 |
| 13C. parvum tipo Ba | |||
| Vacas, ovejas y jabalíes | Zona noroeste | 5C. parvum tipo Aa | 121 |
| 4C. parvum tipo Ba | |||
| Cerdos | Aragón | 10C. suis | 112 |
| 6 Cryptosporidium genotipo de cerdo II | |||
| Visones | Galicia | C. parvum genotipo de hurón | 115 |
| Roedores e insectívoros | Zona noroeste | 110C. parvum | 127 |
| 13C. muris | |||
| 19C. parvum/C. murisb | |||
| Perdiz roja | Cataluña | C. meleagridis | 113 |
| Búho | Cataluña | C. baileyi | 105 |
| Paloma | Islas Canarias | 2C. hominis | 89 |
| Mejillones | Galicia | C. parvum | 94 |
| Mejillones | Galicia | C. parvum | 103 |
| Mejillones, berberechos | Galicia | C. parvum | 100 |
| Mejillones, ostras yalmejas | Galicia | 1C. hominis | 102 |
| 22C. parvum | |||
| 3C. hominis/C. parvumb | |||
| Dorada | Océano Atlántico y mares Cantábrico y Mediterráneo | C. molnari | 118 |
| Lubina | |||
| Rodaballo | Zona noroeste | C. scophthalmi | 170 |
| Lagartos | Madrid | 4C. serpentis | 106 |
| 4C. varanii | |||
| 1C. parvum | |||
| 1 Cryptosporidium sp. | |||
| Serpientes | Madrid | 24C. serpentis | 106 |
| 1C. varanii | |||
| 2C. muris | |||
| 1 Cryptosporidium genotipo de ratón | |||
| 1 Cryptosporidium sp. | |||
| 1 Cryptosporidium sp. (tortuga) | |||
| Tortugas | Madrid | 2 Cryptosporidium sp. (tortuga) | 106 |
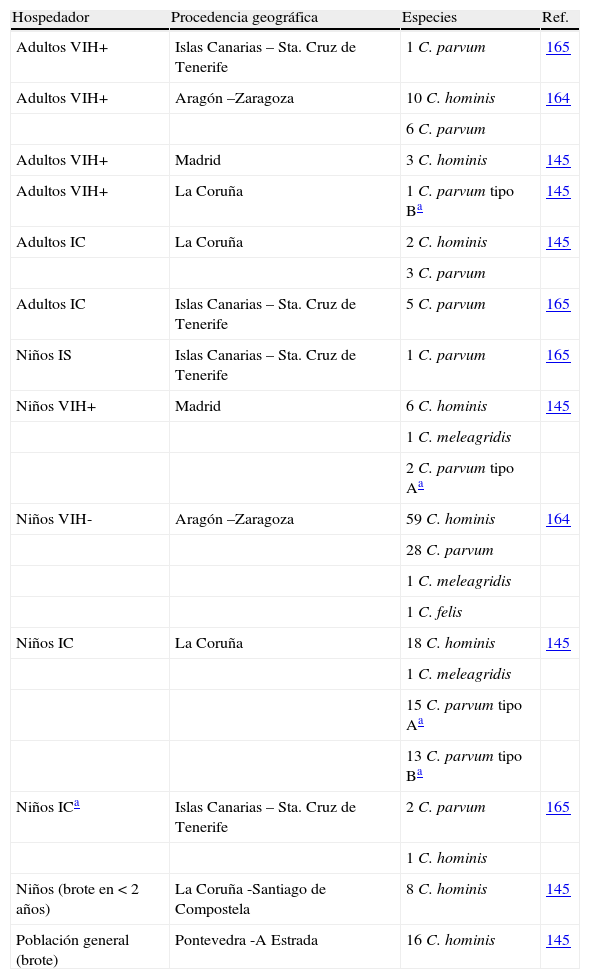
Especies/genotipos encontrados en humanos.
| Hospedador | Procedencia geográfica | Especies | Ref. |
| Adultos VIH+ | Islas Canarias – Sta. Cruz de Tenerife | 1C. parvum | 165 |
| Adultos VIH+ | Aragón –Zaragoza | 10C. hominis | 164 |
| 6C. parvum | |||
| Adultos VIH+ | Madrid | 3C. hominis | 145 |
| Adultos VIH+ | La Coruña | 1C. parvum tipo Ba | 145 |
| Adultos IC | La Coruña | 2C. hominis | 145 |
| 3C. parvum | |||
| Adultos IC | Islas Canarias – Sta. Cruz de Tenerife | 5C. parvum | 165 |
| Niños IS | Islas Canarias – Sta. Cruz de Tenerife | 1C. parvum | 165 |
| Niños VIH+ | Madrid | 6C. hominis | 145 |
| 1C. meleagridis | |||
| 2C. parvum tipo Aa | |||
| Niños VIH- | Aragón –Zaragoza | 59C. hominis | 164 |
| 28C. parvum | |||
| 1C. meleagridis | |||
| 1C. felis | |||
| Niños IC | La Coruña | 18C. hominis | 145 |
| 1C. meleagridis | |||
| 15C. parvum tipo Aa | |||
| 13C. parvum tipo Ba | |||
| Niños ICa | Islas Canarias – Sta. Cruz de Tenerife | 2C. parvum | 165 |
| 1C. hominis | |||
| Niños (brote en < 2 años) | La Coruña -Santiago de Compostela | 8C. hominis | 145 |
| Población general (brote) | Pontevedra -A Estrada | 16C. hominis | 145 |
En aguas se ha demostrado la presencia de las principales especies responsables de infección humana (C. parvum y C. hominis), favoreciendo la aparición de brotes epidémicos, como los ya descritos145,147. Además, su presencia en aguas de ríos favorece la acumulación de este parásito en animales filtradores, lo que supone un riesgo por transmisión alimenticia, al haberse hallado tanto C. hominis como C. parvum acumulados en bivalvos criados para consumo humano94,100,102,103. Otras especies detectadas en aguas, aunque no están directamente relacionadas con la criptosporidiosis humana (C. muris, C. andersoni y C. baileyii) demuestran la presencia del género en la zona y la posibilidad de circulación de otras especies con mayor repercusión en epidemiología humana.
En cuanto a la infección en animales de interés comercial, la criptosporidiosis toma importancia tanto por las pérdidas económicas que provoca la morbimortalidad asociada a la enfermedad, como por actuar como reservorios y fuente de ooquistes que podrían ser dispersados causando brotes en humanos y otros animales. De las especies halladas C. parvum es la más importante en bovinos, ovinos y caprinos (tabla 6), habiendo sido encontrada también en animales silvestres cercanos a zonas de producción ganadera121, demostrando la existencia de ciclos naturales que pueden afectar a animales de granjas o a humanos. También se han descrito otras especies minoritarias, como C. bovis107 y C. xiaoi111,169. En peces se han descrito dos especies, C. molnari66 y C. scophthalmi170, poniéndose de manifiesto el aumento de los estudios sobre este parásito a todos lo niveles en España. Otro grupo de animales que puede ejercer un papel importante en epidemiología humana son las mascotas. El único estudio molecular llevado a cabo hasta la fecha en España se centra en reptiles106. Aunque en este grupo de animales son otras las especie mayoritarias, se ha detectado la presencia de C. parvum en lagartos, pudiendo actuar como fuente de infección para sus dueños.
En infección humana son pocos los estudios publicados sobre caracterización de especies. Dos estudios llevados a cabo en Zaragoza164 y Madrid145 determinaron una mayor prevalencia de C. hominis frente a C. parvum, mientras que en otro estudio realizado en La Coruña145C. parvum fue la especie dominante. Esta observación puede ser debida a las diferencias en la distribución urbana/rural de los habitantes de estas tres regiones, siendo más común la especie zoonótica en regiones como La Coruña, con una importante distribución rural, unido a la presencia de explotaciones ganaderas. Además de estas especies también se han detectado 3 casos de infección por C. meleagridis en niños, tanto inmunocompetentes145,164 como VIH-positivos145 y una infección por C. felis en un niño VIH-negativo164. En cuanto a los brotes epidémicos hasta el momento existen datos de 4 episodios. En los años 2000 y 2003 se publicaron dos brotes asociados a aguas de piscina en turistas británicos en Mallorca, habiéndose determinado C. hominis y C. parvum como especies responsables respectivamente171. Otros dos brotes se detectaron en Santiago de Compostela en 2002 y en A Estrada en 2003, en Galicia145. En ambos casos la especie responsable fue C. hominis. En el primer caso los datos epidemiológicos apuntaban a un brote por transmisión persona a persona, mientras que en el segundo se asoció a aguas del sistema de suministro de la localidad, habiéndose detectado un vertido de una planta de reciclado de aguas residuales situada en una zona del río cercana a la de toma de las aguas de consumo, aunque en ninguno de los dos casos se pudieron corroborar estas hipótesis.
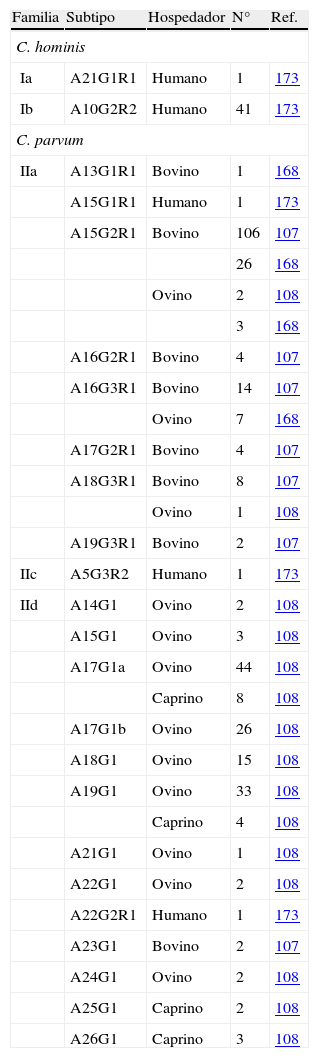
Estos estudios moleculares han demostrado la circulación, en España, de 4 especies importantes en epidemiología humana (C. parvum, C. hominis, C. meleagridis y C. felis). Especial atención merece C. parvum, de la cual se han detectado los mismos genotipos del ADNr18S infectando tanto animales como humanos108,121,145, lo que pondría de manifiesto el importante papel como reservorio que pueden desempeñar bovinos, ovinos y caprinos. Sin embargo, este marcador no es el más adecuado para la determinación a nivel subespecie. Este hecho, unido a la posibilidad de un ciclo antroponótico en C. parvum, pone de manifiesto la necesidad de utilizar marcadores moleculares hipervariables que permitan un mejor conocimiento de los subtipos y familias circulantes, facilitando la determinación de la importancia epidemiológica y el riesgo para la salud pública que este género supone172. Uno de los marcadores utilizados actualmente por su alta variabilidad y cantidad de información que aporta es el gen de la gp60. Los datos publicados en España utilizando este marcador son escasos (tabla 7). Entre los aislados de C. parvum se han detectado las mismas familias en bovinos, ovinos y caprinos que en humanos (IIa y IIb); sin embargo, no coincidieron los subgrupos de la GP60. Estos hallazgos apuntarían a la existencia de subgrupos que infectan humanos y animales independientemente; sin embargo, la escasez de datos impide confirmar esta afirmación.
Subtipos del gen de la GP60 hallados en España.
| Familia | Subtipo | Hospedador | N° | Ref. |
| C. hominis | ||||
| Ia | A21G1R1 | Humano | 1 | 173 |
| Ib | A10G2R2 | Humano | 41 | 173 |
| C. parvum | ||||
| IIa | A13G1R1 | Bovino | 1 | 168 |
| A15G1R1 | Humano | 1 | 173 | |
| A15G2R1 | Bovino | 106 | 107 | |
| 26 | 168 | |||
| Ovino | 2 | 108 | ||
| 3 | 168 | |||
| A16G2R1 | Bovino | 4 | 107 | |
| A16G3R1 | Bovino | 14 | 107 | |
| Ovino | 7 | 168 | ||
| A17G2R1 | Bovino | 4 | 107 | |
| A18G3R1 | Bovino | 8 | 107 | |
| Ovino | 1 | 108 | ||
| A19G3R1 | Bovino | 2 | 107 | |
| IIc | A5G3R2 | Humano | 1 | 173 |
| IId | A14G1 | Ovino | 2 | 108 |
| A15G1 | Ovino | 3 | 108 | |
| A17G1a | Ovino | 44 | 108 | |
| Caprino | 8 | 108 | ||
| A17G1b | Ovino | 26 | 108 | |
| A18G1 | Ovino | 15 | 108 | |
| A19G1 | Ovino | 33 | 108 | |
| Caprino | 4 | 108 | ||
| A21G1 | Ovino | 1 | 108 | |
| A22G1 | Ovino | 2 | 108 | |
| A22G2R1 | Humano | 1 | 173 | |
| A23G1 | Bovino | 2 | 107 | |
| A24G1 | Ovino | 2 | 108 | |
| A25G1 | Caprino | 2 | 108 | |
| A26G1 | Caprino | 3 | 108 | |
Por ello, y ante la reciente obligatoriedad de notificación de los casos de criptosporidiosis a la Red Nacional de Vigilancia Epidemiológica146, sería conveniente el establecimiento de un patrón de genotipado de varios loci, incluyendo genes para la determinación de especies, como el gen del ADNr18S o la HSP70, y subespecies y familias, como los microsatélites ML1, ML2, y el gen de la GP60, permitiendo la comparación entre estudios y realizando una caracterización adecuada de las poblaciones circulantes en nuestro medio.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.