Introducción
Las infecciones causadas por Staphylococcus aureus resistente a meticilina (SARM) han sido observadas desde la década de 1960 1. Aunque inicialmente este patógeno estaba relacionado casi exclusivamente con infecciones de origen nosocomial o asociadas a contactos con el sistema sanitario, en los últimos años ha habido un incremento de infecciones por SARM adquirido en la comunidad (SARM-AC) en pacientes sin factores de riesgo conocidos 2. Este incremento de SARM-AC se ha notificado en diferentes países y áreas geográficas 3-8.
Algunos estudios señalan que este incremento de infecciones por SARM-AC está siendo particularmente importante en población pediátrica 6,9,10. Un estudio poblacional realizado en Estados Unidos durante 2001 y 2002 mostraba que la incidencia anual de infecciones por SARM-AC en la población general oscilaba entre 18 y 25,7 casos por 100.000 habitantes, y esta era significativamente más alta en niños menores de 2 años 7. En ciertas áreas geográficas, como en Houston (Texas), este incremento ha sido particularmente significativo, puesto que la mayoría de las infecciones producidas por SARM en población pediátrica durante el año 2004 (76,4%) eran de adquisición comunitaria 6. Las infecciones más frecuentes asociadas al SARM-AC en población pediátrica son las relacionadas con piel y tejidos blandos 6,11,12.
Una característica adicional de las cepas de SARM-AC es que con frecuencia son portadoras de factores de virulencia, en particular de la leucocidina de Panton-Valentine (LPV) 13,14. Un estudio que examinó hasta 24 factores de virulencia y el tipo de staphylococcal cassette chromosome (SSC) mec (SSCmec) en más de 100 aislados de SARM-AC de diferentes partes del mundo, encontró como característico de estos aislamientos la presencia del tipo IV de SSCmec y de LPV 14. Otra característica es que con gran frecuencia la resistencia a oxacilina suele ser aislada, a veces asociada con resistencia sólo a la eritromicina, mientras que son generalmente sensibles a otros antibióticos como clindamicina y cotrimoxazol 15.
Aunque un estudio previo realizado en nuestro hospital durante los años 2001 y 2002 no detectó ningún caso de bacteriemia por SARM en población pediátrica 16, en los últimos años hemos observado un incremento en el número de aislamientos de SARM procedentes de pacientes pediátricos atendidos en nuestro hospital. El objetivo de este trabajo era: a) estudiar las características epidemiológicas y clínicas de los pacientes pediátricos con infecciones producidas por SARM, con especial atención a un posible origen comunitario, y b) caracterizar molecularmente este tipo de aislamientos, mediante electroforesis en campo pulsante (ECP), tipificación del elemento SCCmec y determinación de la presencia del gen LPV, para conocer si este incremento era resultado de la diseminación de algún tipo particular de clon.
Material y métodos
Pacientes
Este trabajo ha sido realizado en el Hospital 12 de Octubre de Madrid, centro de 1.300 camas que, a su vez, comprende un centro materno-infantil y un centro general para pacientes adultos. Es el hospital de referencia del Área 11 de Madrid. El período de estudio comprendió desde enero de 2002 a junio de 2005. Se incluyeron los pacientes cuya edad era inferior a los 16 años y tenían al menos un cultivo positivo de SARM. Se revisaron las historias clínicas de cada paciente mediante la recogida de los datos demográficos, clínicos y microbiológicos. Se diferenció entre adquisición intrahospitalaria, asociada o relacionada con el sistema sanitario, y comunitaria de acuerdo con los siguientes criterios:
Adquisición nosocomial: no evidencia de infección al ingreso y aislamiento de SARM después de las primeras 48 h de hospitalización.
Asociada con el sistema sanitario: aislamiento de SARM en paciente no hospitalizado con historia de ingreso en los últimos 3 meses, o sometido a diálisis continua, o portador de algún tipo de dispositivo intravascular.
Adquisición comunitaria: aislamiento de SARM en las primeras 48 h del ingreso hospitalario en un paciente sin factores de riesgo (véase párrafo anterior) o en un paciente que acude al servicio de urgencias o consultas externas y no tiene factores de riesgo.
Microbiología
La identificación y antibiograma fueron realizados por los métodos convencionales y con el uso del sistema Wider (Soria Melguizo, Madrid). Se ensayó la sensibilidad a penicilina, oxacilina, ciprofloxacino, eritromicina, clindamicina, trimetoprima/sulfametoxazol, gentamicina, tetraciclina, rifampicina, vancomicina, mupirocina y ácido fusídico mediante el método de difusión en placa de acuerdo con las normas del CLSI (Clinical and Laboratory Standards Institute) 17. Los puntos de corte utilizados para definir la resistencia a mupirocina y ácido fusídico fueron de 13 y 18 mm o menos, respectivamente 18,19. La determinación del fenotipo MLSB inducible se realizó mediante el método de difusión en placa (D-test) 17.
Tipificación molecular
La caracterización molecular de las distintas cepas se realizó mediante ECP, previa extracción y digestión con la enzima SmaI. La electroforesis se realizó en el sistema CHEF DRIII (Biorad, Richmond, EE.UU.). Los fragmentos de restricción fueron separados a 14 °C en geles de agarosa al 1%, con dos bloques de pulsos; un primer bloque durante 11,5 h con pulsos de 5 a 15 s y un segundo bloque durante otras 11,5 h con pulsos de 15 a 40 s. Como marcador molecular se utilizó lambda ladder (New England Biolabs, Beverly, EE.UU.). El análisis de los patrones moleculares se realizó con el programa informático Bionumerics (Applied Maths, Kortrijk, Bélgica). La tolerancia y la optimización para la comparación de los fragmentos de ADN eran del 1,0%. Un coeficiente de similaridad superior a 75% fue usado para definir un patrón de ECP. Mediante análisis visual de los patrones de ECP, se consideró que estos eran distintos cuando diferían en más de 3 bandas 20. Mediante la técnica de reacción en cadena de la polimerasa (PCR), se confirmó la presencia del gen mecA21, se realizó la caracterización de los distintos cassettes cromosómicos SSCmec 21, y se detectó la presencia del gen que codifica la LPV 22.
Resultados
Durante el período de estudio se obtuvieron un total de 24 aislamientos de SARM correspondientes a 17 pacientes. De éstos, tres fueron diagnosticados durante el año 2002, cinco durante 2003, siete durante 2004 y dos en enero y marzo de 2005. Del total de pacientes, 11 (64,7%) eran niños y 6 (35,3%) niñas. El rango de edad oscilaba entre los 3 días y los 16 años. Según los datos analizados, 9 casos (52,9%) fueron de adquisición intrahospitalaria, siete (41,2%) de origen comunitario y un caso (5,9%) se consideró relacionado con el sistema sanitario.
Los aislamientos de SARM de adquisición comunitaria correspondieron a 7 pacientes; seis de ellos fueron diagnosticados en el Servicio de Urgencias y uno en el Servicio de Lactantes. Cuatro pacientes eran de nacionalidad ecuatoriana. Ninguno tenía factores de riesgo para infección por SARM. Los pacientes presentaron las siguientes infecciones: cuatro eran casos de infecciones cutáneas y/o de partes blandas (un absceso en región cérvico-occipital, un absceso en región frontal, un absceso en flexura del codo, y un paciente con infección superficial en cuero cabelludo), dos otitis medias agudas, y un caso de piomiositis bacteriémica.
Los pacientes con aislamientos de SARM de adquisición intrahospitalaria fueron diagnosticados en distintos servicios: cuatro en Neonatología, tres en el Servicio de Digestivo, uno en la UCI-Pediátrica y uno en Traumatología. Todos ellos habían nacido en España y tenían algún factor de riesgo relacionado con la adquisición del microorganismo, entre los que destacaban: ingresos previos (55,5%), cirugía previa (55,5%), antibiótico previo (77,7%), catéteres (100%), otros dispositivos invasivos (66,6%), unidad de cuidados intensivos (UCI) previa (77,7%) o inmunosupresión (66,6%). Todos los casos fueron considerados como infecciones: cinco eran infecciones de la herida quirúrgica, dos eran casos de conjuntivitis, un caso era una bacteriemia asociada a catéter y otro era una infección respiratoria en una paciente con fibrosis quística. El caso asociado al sistema sanitario se consideró como una colonización nasal en un paciente en diálisis.
Se dispuso de información sobre el tratamiento antimicrobiano recibido y la evolución de la infección en 13 pacientes. En todos ellos, con excepción de un neonato que falleció (aunque no en relación con la infección por SARM), la evolución fue favorable hacia la curación. Es de reseñar que en 4 pacientes con infecciones de partes blandas por SARM que fueron vistos en urgencias, el drenaje de sus infecciones fue fundamental en la curación.
Características microbiológicas y moleculares de los aislados de SARM pediátricos
Se comprobó, mediante PCR, la presencia del gen mecA en todos los aislados clínicos de SARM. En general, las pruebas de sensibilidad a los antimicrobianos mostraron el 54% de resistencia a eritromicina, el 37% a clindamicina, el 17% a gentamicina y el 12% a ciprofloxacino. Todos los aislados de SARM fueron sensibles a cotrimoxazol, rifampicina, vancomicina, teicoplanina, mupirocina y ácido fusídico. Los aislados clínicos de origen comunitario, a excepción de uno con resistencia a ciprofloxacino, presentaron resistencia únicamente a oxacilina y eran sensibles al resto de los antibióticos ensayados. Sin embargo, de los nueve aislados de origen intrahospitalario, siete (77,7%) eran resistentes a eritromicina, cinco (55,5%) a clindamicina, incluido un aislado con MLSB inducible, cuatro (44,4%) a ciprofloxacino y tres (33,3%) a gentamicina. Todos los aislados clínicos de origen comunitario eran portadores del gen PVL; sin embargo, sólo dos (20%) relacionados con el hospital eran portadores del gen PVL.
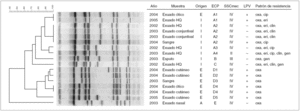
El análisis molecular mediante ECP de los 17 aislados de SARM, correspondientes a los 17 pacientes, produjo 5 clones (fig. 1). El más numeroso, genotipo A, agrupó a ocho aislados de SARM, siete de ellos de origen intrahospitalario. Un total de cuatro aislados de SARM pertenecientes al genotipo A, con patrones de ECP indistinguibles (A2) y con el tipo IV de SSCmec correspondieron a 4 pacientes ingresados en períodos de tiempo no coincidentes en el servicio de neonatología entre los años 2002 y 2004. Otros dos aislados de SARM también tuvieron patrones de ECP indistinguibles (A1), idéntico tipo de SSCmecA (tipo IV) y eran portadores del gen PVL. Éstos correspondieron a 2 pacientes, el primero de ellos aislado en 2004 y de adquisición extrahospitalaria, y el segundo en 2005 y se consideró de origen intrahospitalario. El segundo grupo clonal más frecuente fue el genotipo D, que agrupó a seis aislados de SARM, todos ellos de origen exclusivamente extrahospitalario. Todos los aislados clínicos correspondientes a este genotipo tenían el tipo IV de SSCmecA, producían la toxina LPV y sólo eran resistentes a la oxacilina (fig. 1). Los otros tres genotipos correspondieron a 3 pacientes que tuvieron cepas de SARM adquiridas o asociadas con el sistema sanitario.
Figura 1. Dendograma con los patrones de electroforesis en campo pulsante y características de los aislados clínicos de SARM de población pediátrica. Exudado HQ: exudado de herida quirúrgica; E: extrahospitalario o comunitario; I: intrahospitalario; A: asociado o relacionado con el sistema sanitario; ECP: electroforesis en campo pulsante; SSC: staphylococcal cassette chromosome; LPV: leucocidina de Panton-Valentine. La escala en la parte superior izquierda representa el porcentaje de similitud.
Discusión
Este estudio detecta un incremento de las infecciones producidas por SARM de origen extrahospitalario en población pediátrica sin factores predisponentes y sin ninguna relación con el hospital. El 42,2% de las infecciones por SARM en nuestro estudio eran de origen extrahospitalario. Es importante señalar que este incremento se produjo a expensas de la diseminación de un clon de SARM, con el mismo genotipo de ECP (genotipo D) y con el tipo IV de SSCmec (fig. 1), que fue responsable de 6 de los 7 casos producidos en nuestra área de referencia durante el período de estudio. Este clon apareció en el año 2003 y causó la mayoría de casos en 2004. Estos aislados de SARM presentaban un patrón inusual de resistencia sólo a la oxacilina, a diferencia del patrón típicamente multirresistente de los aislamientos de SARM hospitalarios 15. Por otra parte, la comparación del patrón de ECP de esta cepa de SARM-AC con la base de datos de patrones de ECP de SARM obtenidos bacteriemias por SARM en nuestro hospital (datos no mostrados) demostraba que esta cepa era genotípicamente diferente a los aislamientos de SARM hospitalarios, y que probablemente se trataba de una cepa nueva de SARM que se está diseminando en la comunidad. Estos resultados ponen de manifiesto los cambios producidos en la epidemiología de las infecciones por SARM en población pediátrica de Madrid, en la que cada vez se encuentra un mayor número de aislamientos de SARM de adquisición comunitaria 6,7,9,10.
Por otra parte, este estudio muestra que una segunda cepa de SARM, genotipo A, era la aislada con más frecuencia en los casos de niños con infecciones por SARM de adquisición hospitalaria de nuestra institución. Este clon también incluía el caso de un niño con una infección por SARM de adquisición comunitaria. Esta cepa de SARM se diseminó fundamentalmente en el Servicio de Neonatología durante el período de estudio, en el que 4 neonatos presentaron infecciones por este microorganismo, con idéntico patrón de ECP (A2), SSCmec y, salvo un caso, idéntico patrón de resistencias (fig. 1). Aunque diferentes estudios realizados en el personal sanitario de neonatología, en busca de algún portador nasal de SARM, no pudieron establecer el mecanismo de transmisión y persistencia de esta cepa en neonatología (datos no mostrados), es probable que algún trabajador sanitario que estuviera colonizado intermitentemente o alguno de los muchos trabajadores sanitarios de otros servicios que interactúan con neonatos pudieran estar en el origen y persistencia de esta cepa de SARM 23.
Un resultado destacable de este estudio es que el 100% de los aislados clínicos que causaban infecciones por SARM-AC, a diferencia de sólo el 20% de las de origen intrahospitalario, eran portadores del gen PVL. Este factor de virulencia, descubierto en 1894, es una leucocidina asociada a diferentes tipos de infecciones, en especial neumonía necrosante e infecciones de piel y partes blandas 24-27. En estos cuadros clínicos esta citotoxina desempeña un papel muy importante en la patogénesis, contribuyendo a la necrosis tisular y a la formación de abscesos. En nuestra serie, los aislados clínicos productores de esta citotoxina estuvieron asociados fundamentalmente a infecciones cutáneas y de partes blandas, y en 2 casos a bacteriemias. En general, además de estar relacionado con este tipo de cuadros clínicos, la presencia de este gen en las cepas de S. aureus se asocia con una mayor gravedad de las infecciones 24.
Un aspecto importante de las cepas de SARM-AC productoras de LPV es su facilidad en la transmisión, ya sea en familias o incluso en comunidades abiertas. Aunque en nuestro estudio no se realizó una investigación epidemiológica en los casos con SARM-AC, llama la atención que cuatro aislamientos procedían de pacientes de nacionalidad ecuatoriana, perteneciendo tres de ellos al mismo genotipo D. Se han descrito brotes en deportistas participantes en juegos de equipo, en colectivos militares y en personas que viven en prisiones 1. Por otra parte, diferentes estudios han señalado la diseminación de este tipo de cepas en la comunidad, llegando a ser alguna de ellas la más prevalente entre las que causan infecciones por S. aureus en determinadas áreas geográficas 28. Otro aspecto preocupante es la posible diseminación de estas cepas desde la comunidad hacia el hospital, que pueden causar brotes en pacientes ingresados en unidades clínicas de alto riesgo 29.
El probable incremento en la incidencia de infecciones por SARM en niños fuera del ambiente hospitalario tiene importantes implicaciones tanto para las posibles estrategias de salud pública para el control de estas infecciones, como para el diagnóstico y tratamiento de las mismas. Futuros estudios que determinen la prevalencia y factores asociados a la colonización con SARM-AC serán muy útiles para definir estrategias de prevención de la transmisión de estos clones en la comunidad.
Agradecimientos
Los autores agradecen a Mar Aguilera y Antonia Martín su excelente asistencia técnica.
Correspondencia: Dr. F. Chaves.
Servicio de Microbiología.
Hospital 12 de Octubre.
Avda. de Córdoba, s/n. 28041 Madrid. España.
Correo electrónico: fchaves.hdoc@salud.madrid.org