La identificación de levaduras se basa en el estudio de las características morfológicas, bioquímicas y nutricionales, y en la utilización de métodos moleculares. La espectrometría de masas matrix-assisted laser desorption ionization time-of-fligh (MALDI-TOF) constituye un nuevo método de identificación de microorganismos que ha demostrado gran utilidad. Nuestro objetivo ha sido evaluar este nuevo método en la identificación de levaduras.
MétodosEnsayamos un total de 600 cepas aisladas de muestras clínicas pertenecientes a 9 géneros y 43 especies. La identificación se realizó mediante secuenciación de las regiones ITS del ADN ribosómico, asimilación de compuestos de carbono (ID 32C) y espectrometría de masas en un espectrómetro Microflex (Bruker Daltonics GmbH, Alemania).
ResultadosUn total de 569 cepas (94,8%) fueron identificadas a nivel de especie por ID 32C, y 580 (96,7%) por MALDI-TOF. La concordancia entre ambos métodos comprendió un total de 553 cepas (92,2%), elevándose al 100% en las especies de interés clínico: Candida albicans, C. glabrata, C. parapsilosis, C. tropicalis, y casi del 100% en C. krusei. MALDI-TOF identificó especies que precisan métodos moleculares: Candida dubliniensis, C. nivariensis, C. orthopsilosis y C. metapsilosis. Observamos cierta irregularidad en la identificación de levaduras formadoras de artroconidias y de basidiomicetos.
ConclusiónLa espectrometría de masas MALDI-TOF es un método rápido, rentable y económico que permite la identificación de la mayoría de las levaduras aisladas en clínica, así como la diferenciación de especies estrechamente relacionadas. Sería conveniente la inclusión de más especies en su base de datos para ampliar su rentabilidad.
Identification of yeasts is based on morphological, biochemical and nutritional characteristics, and using molecular methods. Matrix-assisted laser desorption/ionization time-of-flight (MALDI-TOF) mass spectrometry, a new method for the identification of microorganisms, has demonstrated to be very useful. The aim of this study is to evaluate this new method in the identification of yeasts.
MethodsA total of 600 strains of yeasts isolated from clinical specimens belonging to 9 genera and 43 species were tested. Identification was made by sequencing of the ITS regions of ribosomal DNA, assimilation of carbon compounds (ID 32C), and mass spectrometry on a Microflex spectrometer (Bruker Daltonics GmbH, Germany).
ResultsA total of 569 strains (94.8%) were identified to species level by ID 32C, and 580 (96.7%) by MALDI-TOF. Concordance between both methods was observed for 553 strains (92.2%), with 100% in clinically relevant species: C. albicans, C. glabrata, C. parapsilosis, C. tropicalis, and almost 100% in C. krusei. MALDI-TOF identified species requiring molecular methods: Candida dubliniensis, C. nivariensis, C. metapsilosis and C. orthopsilosis. Some irregularities were observed in the identification of arthroconidia yeast and basidiomycetes.
ConclusionMALDI-TOF is a rapid, effective and economic method, which enables the identification of most clinically important yeasts and the differentiation of closely related species. It would be desirable to include more species in its database to expand its performance.
Las levaduras se asocian a cuadros clínicos en los que usualmente el huésped presenta algún factor de riesgo para adquirir la infección, entre los que se incluye la alteración de la inmunidad por inhibición de los mecanismos de defensa celular o tisular, el tratamiento con fármacos inmunosupresores, antibióticos de amplio espectro, antineoplásicos y corticoides, la diabetes mellitus, las deficiencias nutricionales, la implantación de catéteres y el empleo de procedimientos quirúrgicos o terapéuticos agresivos.
Las levaduras de interés clínico pertenecen a los géneros: Candida, Cryptococcus, Geotrichum, Kodamoea, Rhodotorula, Saccharomyces, Saprochaete y Trichosporon. Está claramente demostrado que Candida albicans es la especie que se halla presente en la mayoría de las infecciones, pero el incremento de las infecciones causadas por especies no albicans y otros géneros considerados patógenos emergentes, con sensibilidad disminuida a los antifúngicos de uso habitual, especialmente a azoles, ha supuesto un impacto significativo en la clínica y exige una identificación correcta de los aislamientos, especialmente de los procedentes de infecciones invasoras, y realizar estudios de sensibilidad para orientar un tratamiento antimicrobiano adecuado1-9.
La identificación de levaduras se basa en el estudio de las características morfológicas (morfotipo en medios cromogénicos, blastesis), bioquímicas (reducción de nitrato, producción de ureasa, perfil enzimático) y nutricionales (fermentación y asimilación de compuestos de carbono), que son lentos, exigen de 2 a 4 días o más en el caso de especies habituales, proporcionan a veces identificaciones erróneas por la inexperiencia en la interpretación y suelen tener limitaciones para la identificación de nuevas especies. La utilización de métodos moleculares es imprescindible para ciertas especies descritas recientemente, como Candida dubliniensis, C. metapsilosis, C. orthopsilosis, C. nivariensis, C. bracarensis10-14. Los métodos moleculares se basan en el análisis de los genes de las regiones genómicas del ADN ribosómico D1/D2 de la subunidad 26S, ITS1, ITS2 y 5,8S, y son muy rentables para la identificación y la diferenciación de especies pero caros y complejos para su implantación en el laboratorio clínico15-18.
La espectrometría de masas constituye un nuevo método para la identificación de microorganismos disponible desde hace más de 30 años, pero solo a partir de 2008 se ha aplicado para este fin en el laboratorio clínico por su rapidez, precisión y bajo costo. Actualmente se utiliza el método matrix-assisted laser desorption ionization time-of-fligh (MALDI-TOF) en 2 plataformas: Bruker Daltonic GmbH (Leipzig, Alemania) y Axima-SARAMIS AnagnosTec GmbH (Potsdam, Alemania)19. Los resultados obtenidos por varios autores en la identificación de levaduras parecen excelentes20-43. El objetivo de este estudio ha sido evaluar la espectrometría de masas, en comparación con los métodos convencionales, en la identificación de una amplia variedad de especies de levaduras del género Candida y otros géneros, aisladas de muestras clínicas.
Material y métodosEnsayamos un total de 600 cepas de levaduras aisladas de muestras de pacientes ambulatorios e ingresados en diversas unidades clínicas hospitalarias: 216 muestras de exudado vaginal, 184 de orina, 118 de secreciones respiratorias, 42 de sangre y 40 de otras localizaciones, incluyendo exudado cutáneo, líquido cefalorraquídeo, líquido peritoneal, catéteres y uñas. Las levaduras pertenecían a 9 géneros (Candida, Cryptococcus, Kodamaea, Geotrichum, Pichia, Rhodotorula, Saccharomyces, Saprochaete, Trichosporon) y 43 especies (tabla 1). Todas las cepas fueron identificadas por métodos convencionales, y las no pertenecientes a especies de Candida albicans y Candida tropicalis, un total de 363, fueron identificadas por métodos moleculares.
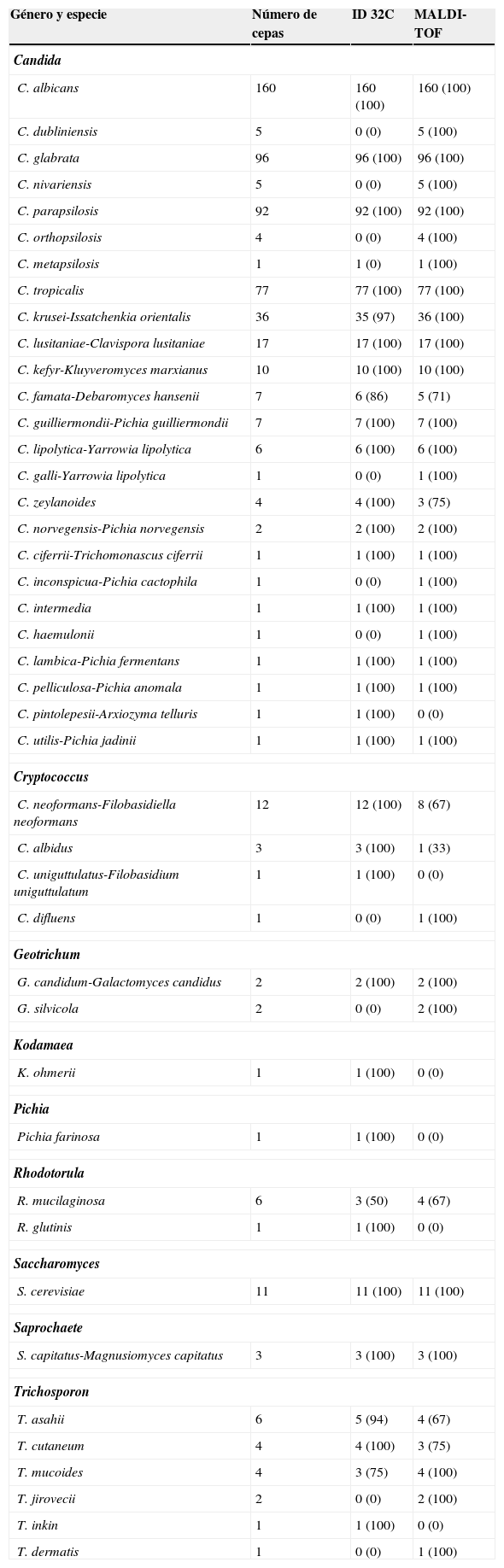
Identificación de 600 cepas de levaduras mediante asimilación de compuestos de carbono (ID 32C) y espectrometría de masas (MALDI-TOF). Porcentaje de identificación correcta entre paréntesis
| Género y especie | Número de cepas | ID 32C | MALDI-TOF |
|---|---|---|---|
| Candida | |||
| C. albicans | 160 | 160 (100) | 160 (100) |
| C. dubliniensis | 5 | 0 (0) | 5 (100) |
| C. glabrata | 96 | 96 (100) | 96 (100) |
| C. nivariensis | 5 | 0 (0) | 5 (100) |
| C. parapsilosis | 92 | 92 (100) | 92 (100) |
| C. orthopsilosis | 4 | 0 (0) | 4 (100) |
| C. metapsilosis | 1 | 1 (0) | 1 (100) |
| C. tropicalis | 77 | 77 (100) | 77 (100) |
| C. krusei-Issatchenkia orientalis | 36 | 35 (97) | 36 (100) |
| C. lusitaniae-Clavispora lusitaniae | 17 | 17 (100) | 17 (100) |
| C. kefyr-Kluyveromyces marxianus | 10 | 10 (100) | 10 (100) |
| C. famata-Debaromyces hansenii | 7 | 6 (86) | 5 (71) |
| C. guilliermondii-Pichia guilliermondii | 7 | 7 (100) | 7 (100) |
| C. lipolytica-Yarrowia lipolytica | 6 | 6 (100) | 6 (100) |
| C. galli-Yarrowia lipolytica | 1 | 0 (0) | 1 (100) |
| C. zeylanoides | 4 | 4 (100) | 3 (75) |
| C. norvegensis-Pichia norvegensis | 2 | 2 (100) | 2 (100) |
| C. ciferrii-Trichomonascus ciferrii | 1 | 1 (100) | 1 (100) |
| C. inconspicua-Pichia cactophila | 1 | 0 (0) | 1 (100) |
| C. intermedia | 1 | 1 (100) | 1 (100) |
| C. haemulonii | 1 | 0 (0) | 1 (100) |
| C. lambica-Pichia fermentans | 1 | 1 (100) | 1 (100) |
| C. pelliculosa-Pichia anomala | 1 | 1 (100) | 1 (100) |
| C. pintolepesii-Arxiozyma telluris | 1 | 1 (100) | 0 (0) |
| C. utilis-Pichia jadinii | 1 | 1 (100) | 1 (100) |
| Cryptococcus | |||
| C. neoformans-Filobasidiella neoformans | 12 | 12 (100) | 8 (67) |
| C. albidus | 3 | 3 (100) | 1 (33) |
| C. uniguttulatus-Filobasidium uniguttulatum | 1 | 1 (100) | 0 (0) |
| C. difluens | 1 | 0 (0) | 1 (100) |
| Geotrichum | |||
| G. candidum-Galactomyces candidus | 2 | 2 (100) | 2 (100) |
| G. silvicola | 2 | 0 (0) | 2 (100) |
| Kodamaea | |||
| K. ohmerii | 1 | 1 (100) | 0 (0) |
| Pichia | |||
| Pichia farinosa | 1 | 1 (100) | 0 (0) |
| Rhodotorula | |||
| R. mucilaginosa | 6 | 3 (50) | 4 (67) |
| R. glutinis | 1 | 1 (100) | 0 (0) |
| Saccharomyces | |||
| S. cerevisiae | 11 | 11 (100) | 11 (100) |
| Saprochaete | |||
| S. capitatus-Magnusiomyces capitatus | 3 | 3 (100) | 3 (100) |
| Trichosporon | |||
| T. asahii | 6 | 5 (94) | 4 (67) |
| T. cutaneum | 4 | 4 (100) | 3 (75) |
| T. mucoides | 4 | 3 (75) | 4 (100) |
| T. jirovecii | 2 | 0 (0) | 2 (100) |
| T. inkin | 1 | 1 (100) | 0 (0) |
| T. dermatis | 1 | 0 (0) | 1 (100) |
La identificación inicial de las cepas se llevó a cabo por pruebas convencionales: formación de tubos germinativos o blastesis, morfotipo de las colonias crecidas en el medio CHROMagar candida, reducción de nitratos, en el caso de Candida pelliculosa y especies de Rhodotorula, producción de ureasa en Candida lipolytica y especies de Cryptococcus, Geotrichum, Saprochaete y Trichosporon, y perfil de asimilación de compuestos de carbono utilizando el sistema comercial ID 32C (bioMérieux, Francia).
Identificación genotípicaSe realizó, tras extracción del ADN y amplificación por PCR, mediante secuenciación del espaciador transcrito interno (ITS) de las regiones ITS1-5,8S-ITS2 del ADN ribosómico, utilizando los primers ITS1 (5’-TCCGTAGGTGAACCTGCGC-3’) e ITS4 (5’-TCCTCCGCTTATTGATATGC-3’)26,44. La secuenciación se practicó en un laboratorio externo (Secugen, Madrid), a partir de 15μl del producto amplificado purificado y 1,5μl del primer ITS1 a una concentración de 5μM. Las secuencias se analizaron en el programa BLAST de GenBank (http.//blast.ncbi.nlm.nih.gov/Blast.cgi)45. Para la identificación de especies se utilizó como criterio un porcentaje de similitud ≥98% entre la secuencia desconocida y la secuencia de la base de datos.
Identificación por espectrometría de masasPara la identificación por espectrometría de masas MALDI-TOF se utilizaron cultivos puros de cada cepa en agar de Sabouraud con cloranfenicol, tras 48h de incubación a 30-37°C. La extracción de proteínas se realizó mediante 2 protocolos: uno con ácido fórmico y otro con etanol/ácido fórmico. Este último se utilizó únicamente en aquellas cepas en las que no se obtuvieron resultados fiables con la extracción mediante ácido fórmico. En cada grupo de análisis se utilizaron como controles cepas de Candida albicans ATCC 68548, C.glabrata ATCC 2001, C.parapsilosis ATCC 90018 y C.krusei ATCC 6258. El análisis de los espectros proteicos se realizó en un espectrómetro de masas MALDI-TOF Microflex (Bruker Daltonics GmbH, Leipzig, Alemania). Los espectros fueron obtenidos en el modo lineal positivo a una frecuencia de 200Hz sobre una proporción masa/carga de 2-20kD, y luego fueron comparados de manera automática a partir de algoritmos integrados en el software del sistema con la base de datos Biotyper versión 3.0 (Bruker Daltonics GmbH, Bremen, Alemania). Esta base de datos contiene los espectros de referencia de alrededor de 3.740 especies (la versión 2.0 contenía 3.476 especies) y se actualiza regularmente. Las puntuaciones de confianza en la identificación a nivel de género y especie fueron los recomendados por el fabricante: ≤1,7 identificación no fiable, 1,7-2 identificación de género, >2 identificación de género y especie.
ResultadosDel total de 600 cepas de levaduras ensayadas y de acuerdo con los resultados de la identificación genotípica, 569 cepas (94,8%) fueron identificadas correctamente a nivel de especie por asimilación de compuestos de carbono (ID 32C) y 580 (96,7%) por espectrometría de masas (MALDI-TOF), según se muestra en la tabla 1. Resultados discordantes con la identificación molecular se observaron en 47 cepas (tabla 2).
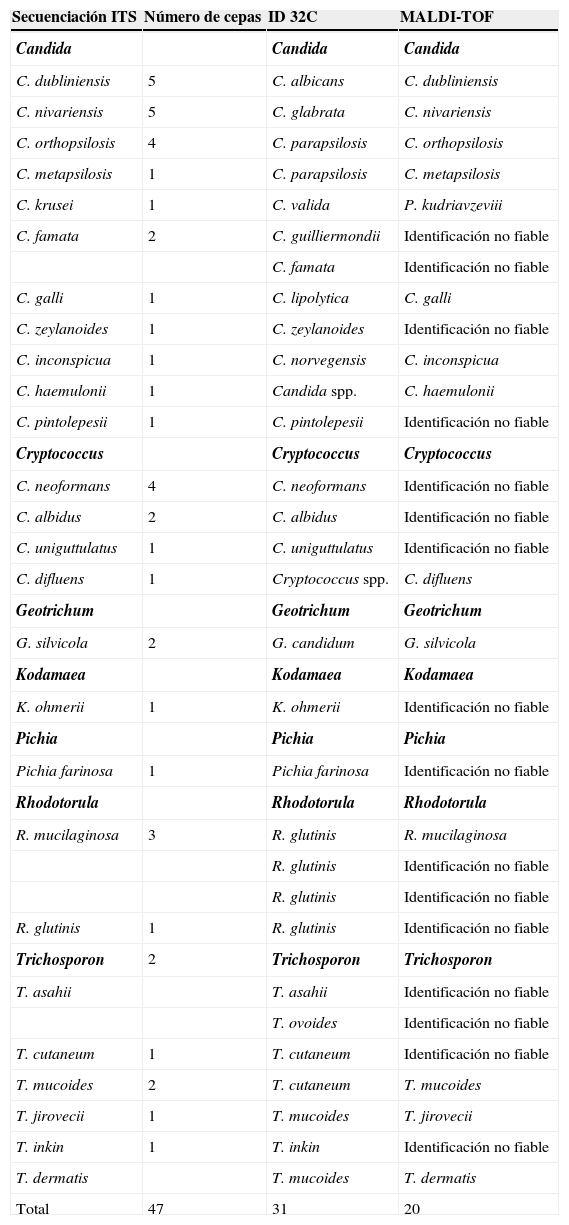
Identificación discordante con los métodos moleculares de 47 cepas de levaduras identificadas por asimilación de compuestos de carbono (ID 32C) y espectrometría de masas (MALDI-TOF)
| Secuenciación ITS | Número de cepas | ID 32C | MALDI-TOF |
|---|---|---|---|
| Candida | Candida | Candida | |
| C. dubliniensis | 5 | C. albicans | C. dubliniensis |
| C. nivariensis | 5 | C. glabrata | C. nivariensis |
| C. orthopsilosis | 4 | C. parapsilosis | C. orthopsilosis |
| C. metapsilosis | 1 | C. parapsilosis | C. metapsilosis |
| C. krusei | 1 | C. valida | P. kudriavzeviii |
| C. famata | 2 | C. guilliermondii | Identificación no fiable |
| C. famata | Identificación no fiable | ||
| C. galli | 1 | C. lipolytica | C. galli |
| C. zeylanoides | 1 | C. zeylanoides | Identificación no fiable |
| C. inconspicua | 1 | C. norvegensis | C. inconspicua |
| C. haemulonii | 1 | Candida spp. | C. haemulonii |
| C. pintolepesii | 1 | C. pintolepesii | Identificación no fiable |
| Cryptococcus | Cryptococcus | Cryptococcus | |
| C. neoformans | 4 | C. neoformans | Identificación no fiable |
| C. albidus | 2 | C. albidus | Identificación no fiable |
| C. uniguttulatus | 1 | C. uniguttulatus | Identificación no fiable |
| C. difluens | 1 | Cryptococcus spp. | C. difluens |
| Geotrichum | Geotrichum | Geotrichum | |
| G. silvicola | 2 | G. candidum | G. silvicola |
| Kodamaea | Kodamaea | Kodamaea | |
| K. ohmerii | 1 | K. ohmerii | Identificación no fiable |
| Pichia | Pichia | Pichia | |
| Pichia farinosa | 1 | Pichia farinosa | Identificación no fiable |
| Rhodotorula | Rhodotorula | Rhodotorula | |
| R. mucilaginosa | 3 | R. glutinis | R. mucilaginosa |
| R. glutinis | Identificación no fiable | ||
| R. glutinis | Identificación no fiable | ||
| R. glutinis | 1 | R. glutinis | Identificación no fiable |
| Trichosporon | 2 | Trichosporon | Trichosporon |
| T. asahii | T. asahii | Identificación no fiable | |
| T. ovoides | Identificación no fiable | ||
| T. cutaneum | 1 | T. cutaneum | Identificación no fiable |
| T. mucoides | 2 | T. cutaneum | T. mucoides |
| T. jirovecii | 1 | T. mucoides | T. jirovecii |
| T. inkin | 1 | T. inkin | Identificación no fiable |
| T. dermatis | T. mucoides | T. dermatis | |
| Total | 47 | 31 | 20 |
Con el método de asimilación de compuestos de carbono fue imposible identificar aquellas especies que precisan métodos moleculares, como Candida dubliniensis (5 cepas incorrectamente identificadas como C. albicans), C. nivariensis (5 cepas incorrectamente identificadas como C. glabrata) y las especies crípticas de C. parapsilosis (4 cepas de C. orthopsilosis y una cepa de C. metapsilosis). Tampoco fue posible identificar una cepa de C. galli, identificada como C. lipolytica, aunque ambas especies pertenecen al mimo clado Yarrowia lipolytica. Este método identificó mal otras especies de Candida no-albicans, como una cepa de C. krusei (mal identificada como C. valida por asimilar el lactato), una cepa de C. famata (mal identificada como C. guilliermondii por asimilar la melibiosa), una cepa de C. inconspicua (identificada erróneamente como C. norvegensis) y una cepa de C. haemulonii (no identificada a nivel de especie). Tampoco se pudo identificar a nivel de especie una cepa de Cryptococcus difluens, 2 cepas de Geotrichum silvicola (identificadas como G. candidum), una cepa de Trichosporon jirovecii y una cepa de T. dermitis (identificada como T. mucoides). Con este método se cometieron errores en la identificación de 3 cepas de Rhodotorula mucilaginosa (identificadas como R. glutinis), una cepa de Trichosporon asahii y una cepa de T. mucoides (identificadas como T. ovoides y T. cutaneun, respectivamente).
Sin embargo, mediante espectrometría de masas fue posible identificar las cepas que precisan métodos moleculares: Candida dubliniensis, C. nivariensis, C. orthopsilosis y C. metapsilosis, así como una cepa de C. krusei mal identificada como C. valida por ID 32C, que fue identificada como Pichia kudriavzevii, denominación antigua de su teleomorfo Issatchenkia orientalis. Este método no logró identificar un total de 20 cepas (3,3%): 2 de Candida famata, una de C. zeylanoides, una de C. pintolepesii, 4 de Cryptococcus neoformans, 2 de C. albidus, una de C. uniguttulatus, una de Kodamaea ohmeri, una de Pichia farinosa, 2 de Rhodotorula mucilaginosa, una de R. glutinis, 2 de Trichosporon asahii, una de T. cutaneum y una de T. inkin, que mostraron puntuaciones inferiores a 1,7 en la extracción con ácido fórmico, y la extracción con etanol/ácido fórmico no mejoró la puntuación, a pesar de que los espectros de masas obtenidos fueron de buena calidad.
La concordancia entre ambos métodos comprendió un total de 553 cepas (92,2%), elevándose esta concordancia a un 100% en las especies de interés clínico más frecuentes: C. albicans, C. glabrata, C. parapsilosis, C. tropicalis, C. lusitaniae, C. kefyr, y casi del 100% en C. krusei.
DiscusiónLa identificación correcta y rápida de levaduras patógenas es uno de los objetivos a alcanzar para el manejo adecuado del paciente, y especialmente para el control de la infección fúngica invasora. Conociendo la variabilidad en la susceptibilidad a antifúngicos de las diferentes especies de levaduras, como la resistencia a fluconazol en Candida glabrata, C. krusei, Rhodotorula y Trichosporon, la resistencia intrínseca a voriconazol en Rhodotorula y la resistencia intrínseca a equinocandinas en Cryptococcus, Rhodotorula y Trichosporon46-48, la correcta identificación a nivel de especie es a menudo decisiva para tomar decisiones terapéuticas. Por todo ello, es fundamental disponer de un método rápido y preciso para la identificación de levaduras en el laboratorio clínico.
Un análisis de los resultados de nuestro estudio, que compara los métodos convencionales con la espectrometría de masas, demuestra que la asimilación de compuestos de carbono es excelente para la identificación de las especies de levaduras de importancia médica más habituales (94,8% de identificaciones correctas y 100% de C. albicans, C. glabrata, C. parapsilosis y C. tropicalis). Sin embargo, este método no es válido para algunas especies de Candida no-albicans consideradas patógenas emergentes, como C. dubliniensis, C. orthopsilosis, C. metapsilosis, C. nivariensis, C. bracarensis, para las que se precisan métodos moleculares poco asequibles para el laboratorio clínico10-14,49-52. No obstante, C. dubliniensis puede diferenciarse de C. albicans por su incapacidad de crecer a 45°C y de asimilar xilosa, pruebas no siempre concluyentes. Por otra parte, C. nivariensis y C. bracarensis pueden diferenciarse de C. glabrata por presentar colonias de color blanco en el medio CROMagar candida y fermentar la trehalosa12,50. El interés de la identificación correcta de C. dubliniensis en lugar de C. albicans radica en que el desarrollo de resistencia a fluconazol es más probable en C. dubliniensis53. La identificación de las especies crípticas de C. parapsilosis es importante no solo desde el punto de vista epidemiológico, sino también por las posibles diferencias de sensibilidad a anfotericinaB y equinocandinas en estas nuevas especies54-56 y la menor patogenicidad de C. metapsilosis13,57. Igualmente, de las 2 especies crípticas de C. glabrata, se ha referido que C. nivariensis es más resistente a antifúngicos y posee la capacidad de formar biopelículas52.
La espectrometría de masas se ha propuesto como una alternativa a los métodos convencionales de identificación. Diversos estudios han demostrado que permite una rápida y precisa identificación de levaduras procedentes de cultivos20-43, e incluso directamente desde frascos de hemocultivos positivos58, además de resultar económico28. En comparación con los métodos convencionales, su precisión en la identificación de levaduras se ha estimado entre el 83 y el 100%, dependiendo del número y del tipo de levaduras y de la metodología, considerando siempre que un segundo ciclo de extracción y análisis aumenta el porcentaje de identificaciones correctas20-43. Así, cuando se analizan mayoritariamente cepas del género Candida y un número escaso de especies poco frecuentes, los resultados son mejores que cuando se analiza un número elevado de especies raras y otros géneros diferentes. La plataforma Axima-SARAMIS muestra porcentajes de identificaciones correctas ligeramente inferiores a MALDI-TOF25,27,30. La versión Biotyper 3.0 utilizada en nuestro estudio para comparar los espectros dispone de una amplia base de datos que permite identificar un mayor número de especies que la versión 2.0.
Para las especies de interés clínico, la concordancia entre ambos métodos fue excelente. La identificación mediante ID 32C se apoyó a veces en las características morfológicas y bioquímicas para corroborar algunas especies, como Candida lipolytica, C. pelliculosa, Cryptococcus, Geotrichum, Rhodotorula y Trichosporon. La identificación por MALDI-TOF dependió, en parte, de la calidad de la extracción, de los espectros presentes en la base de datos y del número de espectros de la misma. A diferencia de la asimilación de compuestos de carbono, que requirió un tiempo de incubación de 24-72h para la identificación, la espectrometría de masas pudo obtener un resultado positivo en menos de 2h y fue capaz de identificar o diferenciar especies difíciles, como C. dubliniensis, C. orthopsilosis, C. metapsilosis, C. nivariensis, C. inconspicua, C. haemulonii, Cryptococcus difluens, Geotrichum silvicola, Trichosporon jirovecii y T. dermatis59-62. En nuestra serie, sin embargo, no pudimos identificar Pichia farinosa63.
Globalmente, nuestro estudio muestra resultados similares a los descritos por otros autores, aunque con algunas diferencias. Nosotros, al igual que se refleja en otras publicaciones, hemos observado cierta irregularidad en la identificación de las levaduras formadoras de artroconidias, Geotrichum y Trichosporon, y de los basidiomicetos Cryptococcus neoformans, C. albidus, C. uniguttulatus, Rhodotorula mucilaginosa, R. glutinis y Trichosporon asahii, T. cutaneum y T. inkin. Estas levaduras están poco representadas en la literatura referente a espectrometría de masas y presentan con frecuencia fallos en la identificación, posiblemente por la particular composición de su pared celular, muy rica en azúcares, la producción de cápsula y de artroconidias, características que podrían dificultar la extracción de proteínas21,23,27-30,35-37,60,61. Sin embargo, la identificación de estas especies es importante porque, a pesar de ser poco habituales en pacientes con fungemia, se describen cada vez más en clínica1,64-78 y han demostrado in vitro altas CMI a los antifúngicos usuales, incluyendo fluconazol y caspofungina4,9, sin estar establecidos aún los puntos de corte para ellas.
Como técnica alternativa, MALDI-TOF nos ha permitido identificar y diferenciar correctamente algunas especies poco usuales en clínica pero de gran interés, como Candida nivariensis, C. orthopsilosis y C. metapsilosis, sin necesidad de recurrir a la biología molecular, y otras especies raras aisladas ocasionalmente en infecciones fúngicas: C. ciferrii79-81, C. galli (no implicada en clínica hasta el momento), C. haemulonii82-86, C. inconspicua (en nuestro caso y generalmente identificada como Pichia cactophila por MALDI-TOF87,88, C. lambica89, Cryptococcus difluens (no implicado en clínica), Geotrichum candidum90-92, G. silvicola (no implicado en clínica y filogenéticamente relacionado con G. candidum y G. bryndzae), Kodamaea ohmeri93, Saprochaete capitata94-96, Trichosporon jirovecii (no implicado en clínica) y T. dermatis70,71, ambos relacionados filogenéticamente con T. mucoides73-75.
En conclusión, la espectrometría de masas MALDI-TOF es un método rápido y rentable, además de económico, que permite la identificación rutinaria de la mayoría de las levaduras aisladas en clínica, incluidas especies poco frecuentes, así como la diferenciación de especies estrechamente relacionadas y solamente diferenciables por métodos genómicos, mucho más complejos. MALDI-TOF podría ser utilizado como una alternativa a la identificación convencional de levaduras, ya que mejoraría el diagnóstico y permitiría el reconocimiento de nuevas especies implicadas en procesos clínicos. Sería conveniente la inclusión de más especies en su base de datos, especialmente de especies raras y basidiomicetos, para ampliar su rentabilidad, aunque esta base se actualiza periódicamente y la próxima versión incluirá 8 nuevos géneros y 63 nuevas especies de levaduras.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses