Las 3 especies de bacilos gramnegativos no fermentadores más relevantes clínicamente, Pseudomonas aeruginosa, Acinetobacter baumannii y Stenotrophomonas maltophilia son, frecuentemente, multirresistentes. La resistencia de P. aeruginosa a los betalactámicos depende de la producción de betalactamasa cromosómica, betalactamasas plasmídicas, alteraciones de la permeabilidad (pérdida de la porina OprD, relacionada con la resistencia a carbapenémicos), y de bombas de expulsión activa, en especial MexAB-OprM. En las cepas resistentes a aminoglucósidos, la principal causa es la producción de enzimas inactivantes; también está implicada la bomba de expulsión MexXY-OprM. La resistencia a quinolonas en P. aeruginosa se relaciona con alteraciones de las topoisomerasas, alteraciones de las porinas y bombas de expulsión activa. La multiresistencia de A. baumannii normalmente se asocia a la adquisición de islas de resistencia que poseen genes que confieren resistencia a diversas clases de agentes antibacterianos. Si bien diversas beta-lactamasas tanto de amplio espectro como de espectro extendido han sido descritas en este microorganismo, la principal resistencia a betalactámicos se relaciona con la hiperproducción de la cefalosporinasa cromosómica (AmpC) asociada a la presencia de ISAba1 en el promotor del gen blaampC. El principal mecanismo de resistencia a carbapenémicos es la adquisición de carbapenemasas clase B (metalo-β-lactamasas) y clase D (oxacilinasas), sin embargo la perdida de una porina puede contribuir a modular la CIM final. La resistencia a aminoglucósidos se ha relacionado con enzimas modificantes y la sobreexpresión de sistemas de expulsión activa (AdeABC) y la resistencia a quinolonas con alteraciones de las dianas conjuntamente con la sobreexpresión de sistemas de expulsión activa. S. maltophilia presenta resistencia natural a carbapenémicos y otros betalactámicos por producción de dos betalactamasas (L-1 y L-2). También en esta especie se han descrito enzimas modificantes de aminoglucósidos. A diferencia de lo observado en otros muchos organismos, la resistencia de S. maltophilia a quinolonas se relaciona más con bombas de expulsión que con alteraciones de la diana.
Among non-fermenting Gram-negative rods, the most clinically important species are Pseudomonas aeruginosa, Acinetobacter baumannii, and Stenotrophomonas maltophilia, which are frequently multiresistant. P. aeruginosa resistance to beta-lactams depends on the production of chromosomal and plasmid-mediated beta-lactamases, altered permeability (loss of OprD porin is related to carbapenem-resistance) and active efflux systems, particularly MexAB-OprM. In aminoglycoside resistant strains, the main mechanism of resistance is the production of modifying enzymes; the efflux pump MexXY-OprM is also involved. Quinolone resistance in P. aeruginosa is related to changes in topoisomerases, altered permeability and efflux pumps. Multiresistance in A. baumannii may be associated with the acquisition of resistant islands carrying different resistant determining factors. Several broad- and extended-spectrum beta-lactamases have been shown in this microorganism, however the main mechanism of resistance to betalactams is the hyperproduction of the chromosomal cephalosporinase (AmpC) related to the presence of the ISAba1 in the promoter region of this gene. The main mechanism of resistance to carbapenems is the acquisition of carbapenemases type B (metallo-β-lactamases) or class D (oxacillinases), however the loss of a porin can also contribute to modulate the final MIC. The resistance to aminoglycosides has been associated with the production of modifying enzymes or the overexpression of efflux pump (AdeABC), whereas the resistance to quinolones is related to changes in the protein targets as well to the overexpression of efflux pump(s). S. maltophilia show resistance to beta-lactams including carbapenems due to the production of two beta-lactamases (L-1 and L-2). Aminoglycoside-modifying enzymes have also been described in this species. In contrast to that observed in other organisms, S. maltophilia resistance to quinolones has been mainly related to active efflux, rather than to targets alterations.
Con el término de bacilos gramnegativos no fermentadores se designa un grupo heterogéneo de microorganismos incapaces de fermentar diversos hidratos de carbono, entre ellos la glucosa. La mayoría de ellos son aerobios estrictos y abundan en reservorios naturales como el suelo y agua, formando también parte de la microbiota normal del hombre. Muchos de ellos se comportan como patógenos oportunistas y pueden causar infecciones graves en el hombre. Los más importantes desde el punto de vista clínico son: Pseudomonas aeruginosa, Acinetobacter baumannii y Stenotrophomonas maltophilia.
Muchas veces las infecciones ocasionadas por estos microorganismos se manifiestan en individuos hospitalizados, inmunodeprimidos, portadores de material protésico o ampliamente instrumentados y tratados con antibióticos. La habilidad de sobrevivir y persistir en superficies inertes probablemente asociada a la producción de biopelícula puede jugar un papel importante en la capacidad de ocasionar infecciones nosocomiales y brotes epidémicos. La existencia frecuente de multirresistencia o panresistencia en estos microorganismos, junto con la necesidad de dilucidar si se trata de una infección o una simple colonización, generan problemas y dilemas terapéuticos.
Pseudomonas aeruginosaPseudomonas aeruginosa es un microorganismo ubicuo, ampliamente distribuido en la naturaleza y responsable de diversos tipos de infecciones. El tratamiento de estas infecciones, ya sean de origen nosocomial o adquiridas en la comunidad, representa un verdadero reto terapéutico. En este sentido, el tratamiento de una infección grave como la bacteriemia es un claro ejemplo de lo que comentamos, ya que la evolución clínica favorable del proceso está influenciada por la correcta elección de la terapéutica antimicrobiana inicial1. Esta elección no suele ser fácil ya que P. aeruginosa se caracteriza por expresar una resistencia natural (intrínseca) a diversos antibióticos y por su capacidad para desarrollar resistencia a los agentes antimicrobianos durante el tratamiento mediante la adquisición de genes de resistencia situados en elementos genéticos móviles (plásmidos, integrones) o a través de mutaciones que alteran la expresión o la función de mecanismos de codificación cromosómica2,3. La tarea del laboratorio de microbiología tampoco es sencilla. Un aspecto clave es determinar correctamente la sensibilidad de P. aeruginosa a los antibióticos potencialmente activos empleando las diferentes metodologias desarrolladas para ello. En todas ellas es básico que tengamos en cuenta que existen diversos factores que pueden influir en el resultado final (inóculo correcto, medio de cultivo y su pH, concentración de iones, difusión de algunos antibióticos en el agar, temperatura y tiempo de incubación)4. El otro aspecto de enorme importancia es intentar reconocer los mecanismos de resistencia de la cepa estudiada con el empleo de antibióticos que sean buenos marcadores fenotípicos. Con frecuencia es una tarea muy complicada por diversas razones: expresión simultánea de diferentes mecanismos de resistencia que afectan, parcial o totalmente, a los mismos antibióticos; la eficacia variable de estos mecanismos en diferentes cepas y la poca capacidad de los métodos empleados habitualmente para detectar las resistencias de bajo nivel4,5. En los apartados que siguen a continuación se comentarán los mecanismos de resistencia a los antibióticos con actividad frente a P. aeruginosa y su expresión fenotípica en la lectura del antibiograma.
Resistencia naturalLa resistencia intrínseca de P. aeruginosa a múltiples antibióticos sin relación estructural se debe a varios factores: escasa permeabilidad de la membrana externa, presencia de una betalactamasa cromosómica inducible tipo AmpC y la expresión constitutiva de diversos sistemas de expulsión activa, sobre todo MexAB-OprM4,6,7. La participación conjunta de estos 3 factores condiciona resistencia natural a penicilina, aminopenicilinas, incluidas las combinadas con inhibidores de betalactamasas, cefalosporinas de primera y segunda generación, cefotaxima, ceftriaxona, cefalosporinas de tercera generación orales, cloranfenicol, nitrofurantoína, sulfonamidas, trimetoprim, tetraciclina, novobiocina y ácido nalidíxico.
En los bacilos gram negativos la membrana externa representa una barrera semipermeable a la entrada de moléculas, substratos o antibióticos. La entrada de moléculas hidrofílicas de pequeño tamaño, como los antibióticos betalactámicos, está restringida a unas estructuras concretas de la membrana externa, denominadas porinas, auténticos canales que generalmente están rellenos de agua. Esta limitación es más acusada en P. aeruginosa ya que la permeabilidad de la membrana externa es unas 90 veces inferior a la de E. coli8. La porina más abundante en la membrana externa de P. aeruginosa es OprF, pero a pesar de su elevado número es una ruta poco eficiente para el paso de los antibióticos, probablemente debido a la propia «arquitectura» de la porina8. La porina OprD permite el paso de moléculas zwiteriónicas, como los carbapenémicos (imipenem, meropenem, doripenem) y OprC y OprE son porinas inducibles en anaerobiosis aunque es probable que estén presentes en pequeñas cantidades en cepas salvajes. Mutantes triples sin oprC, oprD y oprE son sensibles a todos los antibióticos antipseudomónicos (excepto carbapenémicos, por la pérdida de OprD). Otra porina, OprB, está implicada en la entrada al interior de la célula de carbohidratos pero no hay evidencias que participe en la entrada de antibióticos8. El papel de la betalactamsa cromosómica inducible AmpC y los sistemas de expulsión activa en la resistencia a los antibióticos se comentará en los apartados correspondientes del artículo.
Resistencia a antibióticos betalactámicosEl fenotipo «salvaje» o sensible de P. aeruginosa a los antibióticos betalactámicos se caracteriza por su sensibilidad a carboxipenicilinas (carbenicilina, ticarcilina), ureidopenicilinas (azlocilina, piperacilina), ceftazidima, cefsulodina, cefepima, cefoperazona, aztreonam y carbapenémicos (imipenem, meropenem, doripenem). En P. aeruginosa, el mecanismo más importante de resistencia adquirida a los antibióticos betalactámicos se debe a la producción de β-lactamasas. En este microorganismo podemos encontrar los cuatro tipos de enzimas descritos según la clasificación molecular de Ambler: A, C, D (serina-β-lactamasas) y B (metalo-β-lactamasas).
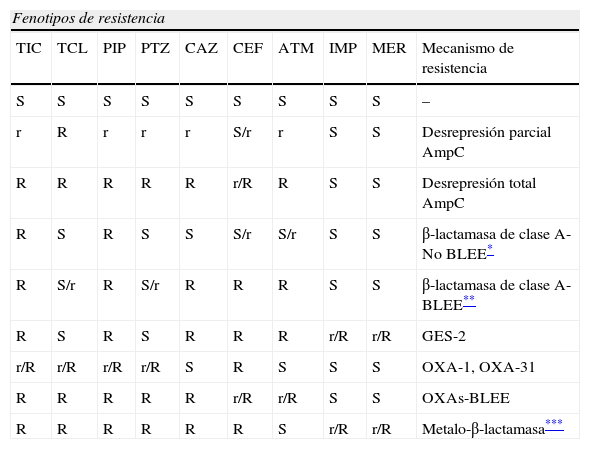
β-lactamasa AmpCP. aeruginosa produce una β-lactamasa cromosómica inducible de clase C (AmpC) codificada por el gen ampC9 similar a la de algunas enterobacterias (Enterobacter spp, Citrobacter freundii, Serratia marcescens, M. morganii). En condiciones normales esta enzima con actividad cefoslosporinasa se secreta en pequeñas cantidades (bajo nivel de expresión) y es la responsable de la resistencia a aminopenicilinas y a cefalosporinas de espectro reducido. No se inhibe con ácido clavulánico, sulbactam o tazobactam. Su producción puede incrementarse de 100 a 1.000 veces en presencia de antibióticos betalactámicos inductores, como cefoxitina o imipenem, aunque es una sobreexpresión reversible que desaparece al retirar el agente inductor. La producción de AmpC también aumenta cuando se producen mutaciones cromosómicas que afectan a las proteínas involucradas en su inducción, fenómeno que condiciona una expresión constitutiva de elevado nivel10. Sin embargo, la desrepresión de AmpC en P. aeruginosa es un fenómeno más complejo que el que tiene lugar en las especies de enterobacterias que poseen esta enzima y el fenotipo de desrepresión puede ser parcial para evolucionar posteriormente a completo o total3. Cuando la producción de AmpC está aumentada de forma significativa, P. aeruginosa expresa resistencia a todos los antibióticos betalactámicos con la excepción de los carbapenémicos. A diferencia de lo que ocurre con los miembros de la familia Enterobacteriaceae, la desrepresión de AmpC en P. aeruginosa también afecta a cefepima. Aunque se pensó que ello podría ser debido a diferencias en la actividad hidrolítica de AmpC, los datos disponibles no apoyan esta idea y probablemente la impermeabilidad de la membrana externa junto con la sobrexpresión de AmpC serían los responsables de la menor actividad de cefepima8. En la tabla 1 se presentan los diferentes fenotipos de resistencia a los antibióticos betalactámicos según la expresión de la β-lactamasa AmpC.
Fenotipos de resistencia a antibióticos betalactámicos en Pseudomonas aeruginosa. Mecanismos de resistencia enzimáticos
| Fenotipos de resistencia | |||||||||
| TIC | TCL | PIP | PTZ | CAZ | CEF | ATM | IMP | MER | Mecanismo de resistencia |
| S | S | S | S | S | S | S | S | S | – |
| r | R | r | r | r | S/r | r | S | S | Desrepresión parcial AmpC |
| R | R | R | R | R | r/R | R | S | S | Desrepresión total AmpC |
| R | S | R | S | S | S/r | S/r | S | S | β-lactamasa de clase A-No BLEE* |
| R | S/r | R | S/r | R | R | R | S | S | β-lactamasa de clase A-BLEE** |
| R | S | R | S | R | R | R | r/R | r/R | GES-2 |
| r/R | r/R | r/R | r/R | S | R | S | S | S | OXA-1, OXA-31 |
| R | R | R | R | R | r/R | r/R | S | S | OXAs-BLEE |
| R | R | R | R | R | R | S | r/R | r/R | Metalo-β-lactamasa*** |
ATM: aztreonam; CAZ: ceftazidima; CEF: cefepima; IMP: Imipenem; MER: meropenem; PIP: piperacilina; PTZ: piperacilina/tazobactam; R: resistente; S: sensible; r: sensibilidad disminuida; TCL: ticarcilina/ác. Clavulánico; TIC: ticarcilina.
Los mecanismos que regulan la expresión de ampC son similares a los estudiados en E. cloacae y en su inducción participan diversos genes3. ampG codifica una proteína de transmembrana (AmpG) que actúa como una permeasa para los muropéptidos liberados durante el proceso de formación de la pared celular. Una vez en el interior de la célula estos muropéptidos, que son considerados la molécula señal en la inducción de ampC, son modificados por otra proteína, AmpD, producto del gen ampD, dando lugar a la formación de péptidos libres y ácido murámico. Un tercer gen ampR, codifica un factor de transcripción, AmpR, perteneciente a la familia de proteínas reguladoras LysR. En la mayoría de cepas con desrepresión de ampC se detectan mutaciones en el gen ampD que conducen a una expresión disminuida o inactivación de AmpD. En estas cepas se afecta el procesamiento de los muropéptidos, lo que conlleva un incremento de su cantidad en el citoplasma y permite su unión a AmpR que cambiaría su conformación y daría lugar a un aumento significativo de la expresión de ampC3. Se ha sugerido que un cuarto gen, ampE podría participar en la regulación de AmpC pero los datos disponibles no son concluyentes11. Datos recientes demuestran que en la expresión de ampC, intervienen además de AmpD, 2 proteínas homólogas denominadas AmpDh2 y AmpDh312,13. Para que se produzca una desrepresión completa y que esta se exprese de forma constitutiva es necesario que se produzca la delección combinada de los 3 genes homólogos. Finalmente, se ha sugerido que en la compleja regulación de la expresión de ampC probablemente intervienen otros factores como por ejemplo la PBP4 codificada por el gen dacB14.
β-Lactamasas de clase ALas β-lactamasas de clase A y pertenecientes al grupo funcional 2c de Bush hidrolizan la carbenicilina, ticarcilina y piperacilina. Se inhiben con ác. clavulánico y tazobactam pero su detección no siempre es fácil ya que puede interferir la inducción de la β-lactamasa cromosómica AmpC. Se han descrito diversos enzimas pertenecientes a este grupo: TEM 1-2, PSE-1 (CARB-2), PSE-4 (CARB-1), CARB-3, CARB-415. Los enzimas PSE-1, PSE-4 y CARB-3 son similares ya que solo difieren en 1 o 2 aminoácidos pero, en cambio, solo comparten un 86,3% de homología con CARB-416. Las cepas con carbenicilinasas (CARB) pueden manifestar una sensibilidad variable a cefepima y aztreonam pero, en ausencia de otros mecanismos de resistencia, siempre son sensibles a ceftazidima y carbapenémicos (tabla 1).
Las β-lactamasas de espectro extendido (BLEE) de clase A pertenecen al grupo funcional 2b’. Su presencia es responsable de la resistencia a carboxipenicilinas, ureidopenicilinas, ceftazidima, cefepima, cefpiroma y aztreonam. También se inhiben con ácido clavulánico y tazobactam y su afinidad por los carbapenémicos es baja, por lo que se mantiene la actividad de imipenem, meropenem y doripenem (tabla 1). En P. aeruginosa, como sucede en la familia Enterobacteriaceae, se han encontrado BLEEs derivadas de enzimas tipo TEM (TEM-4, TEM-21, TEM-24, TEM-42, TEM-116) y SHV (SHV-2a, SHV-5, SHV-12), pero además se han descrito otros tipos: PER (PER-1), VEB (VEB-1, VEB-1a, VEB-1b, VEB-2), GES (GES-1, GES-2, GES-5, GES-8 (IBC-2), GES-9) y BEL (BEL-1)9,17. Los genes responsables de su síntesis están localizados en el cromosoma, plásmidos o integrones. El grado de hidrólisis de ceftazidima, cefepima y aztreonam depende del tipo de enzima que, por otra parte, se puede inhibir en mayor o menor medida con los diferentes inhibidores de β-lactamasas. Las BLEEs tipo GES tienen algunas características que debemos destacar. GES-1 se caracteriza por manifestar una actividad catalítica de bajo nivel así como una baja afinidad por la mayoría de los substratos18. A diferencia de la mayoría de las BLEEs de clase A, tiene una fuerte afinidad por la cefoxitina. GES-2, que difiere de GES-1 en una sola mutación, hidroliza a los carbapenémicos, por ello debe considerarse como una carbapenemasa de clase A19. La actividad catalítica para imipenem es 100 veces más elevada que la que expresa GES-1, pero es mucho más baja que la manifestada por las metalo-β-lactamasas de clase B.
β-lactamasas de clase D (oxacilinasas)Las oxacilinasas (enzimas tipo OXA) son enzimas de clase D que pertenecen al grupo funcional 2d. Representan un amplio grupo de enzimas con un espectro hidrolítico muy desigual que generalmente están codificadas por genes integrados en plásmidos o integrones. Una característica de estas enzimas es que no se inhiben con ácido clavulánico, sulbactam o tazobactam, con la excepción de OXA-18, lo que dificulta su detección en el laboratorio. La clasificación de las oxacilinasas es compleja. Poirel et al20 acaban de publicar una revisión en la que dividen estas enzimas según su espectro de actividad y diversidad genética.
Las enzimas OXA clásicas (OXA-1, OXA-2, OXA-10) se caracterizan por determinar resistencia a carboxipenicilinas y ureidopenicilinas, pero no a ceftazidima14. En el subgrupo OXA-1 se incluye OXA-31, que deriva de OXA-1 por la substitución de 2 aminoácidos. Ambas tienen la capacidad de hidrolizar a cefepima pero no a ceftazidima21.
Las oxacilinasas con mayor importancia clínica son las que expresan un espectro de actividad hidrolítica extendido que incluye cefotaxima, ceftazidima, cefepima, cefpiroma y aztreonam aunque el grado de actividad es variable. La mayoría de estas oxacilinasas se han encontrado en P. aeruginosa y derivan de β-lactamasas OXA con espectro reducido que han experimentado una mutación puntual. No obstante, también se han descrito enzimas con escasa similitud de aminoácidos con las clásicas OXA y que expresan el mismo espectro extendido. En general, las derivadas de oxacilinasas clásicas afectan sobre todo la actividad de ceftazidima (OXA-11, OXA-14, OXA-15, OXA-19, OXA-32) con una actividad hidrolítica variable frente a cefepima y aztreonam. OXA-15, que deriva de OXA-2 hidroliza más a ceftazidima que a cefepima y aztreonam22 y en cambio, OXA-32, que también deriva de OXA-2 hidroliza ceftazidima pero no cefotaxima y es inhibida por cefoxitina23. Las enzimas OXA-11, OXA-14 y OXA-19 afectan principalmente la actividad de ceftazidima mientras que el substrato preferido de OXA-17 es la cefotaxima. Las diferentes variantes de OXA-10 determinan un nivel de resistencia de bajo nivel a cefepima pero alto para ceftazidima. La enzima OXA-18 tiene un grado de similitud con el resto de OXAs según el análisis de los aminoácidos que la componen inferior al 50%24. Su actividad hidrolítica se parece al de una BLEE de clase A y, a diferencia del resto de oxacilinasas, se inhibe con ácido clavulánico. En el año 2003 se describió una nueva enzima parecida a OXA-18 (66% de similitud), OXA-4525. El perfil de los substratos que afecta es parecido y también se inhibe con ác. clavulánico. En la tabla 1 se incluyen algunos fenotipos de resistencia según el tipo de enzimas de este grupo.
Algunas oxacilinasas adquiridas tienen la capacidad de hidrolizar los carbapenémicos sin afectar de forma significativa a las cefalosporinas de espectro extendido. La mayoría de estos enzimas se han identificado en Acinetobacter spp. Sin embargo, recientemente se ha comunicado la detección de OXA-40 en 2 cepas de P. aeruginosa resistentes a carbapenémicos aisladas en nuestro país26.
Metalo-β-lactamasasLas carbapenemasas que necesitan la presencia de Zn2+ en su centro activo se conocen con el nombre de metalo-β-lactamasas. Son β-lactamasas de clase B que determinan resistencia a los antibióticos β-lactámicos, incluidos los carbapenémicos. Los monobactams, como el aztreonam, no se ven afectados por la actividad hidrolítica de estos enzimas (tabla 1). Las metalo-β-lactamasas no se inhiben con ácido clavulánico ni tazobactam y en cambio, sí son inhibidas por quelantes iónicos divalentes como el EDTA27. En P. aeruginosa se han identificado cuatro tipos de metalo-β-lactamasas: IMP, VIM, SPM y GIM. La primera carbapenemasa descrita en P. aeruginosa (IMP-1) se encontró en Japón durante un estudio realizado con cepas aisladas en los años 1992–199428. El gen responsable estaba localizado en un plásmido de gran tamaño (36Kb) y formaba parte de un «cassette» incluido en un integrón de clase 1 (In31). Desde entonces se han comunicado diversas variantes del enzima IMP en diferentes países: IMP-7, IMP-9, IMP-13, IMP-16, IMP-18.
La carbapenemasa VIM-1 se encontró en una cepa de P. aeuruginosa aislada en Italia en 199729. Su grado de similitud con IMP, según el análisis de los aminoácidos, es inferior al 30% aunque tiene el mismo espectro de hidrólisis26. Hasta la actualidad se han descrito diferentes variantes de la carbapenamasa VIM-1 (VIM-2, VIM-3, VIM-4, VIM-5, VIM-7, VIM-8, VIM-11, VIM-13, VIM-15 y VIM-16) con un grado de similitud muy elevado entre ellas16. Los genes responsables forman parte de un integrón localizado en un plásmido o en el cromosoma. Las descripciones más frecuentes corresponden a la VIM-2, una metalo-β-lactamasa que también se ha encontrado en nuestro país30–33. En el año 2008 se describió una nueva carbapenemasa, VIM-13, hallada en una cepa de P. aeruginosa aislada en Mallorca34. El gen responsable, blaVIM−13, se encontraba en un integrón de clase 1 que también contenía una nueva variante del gen aacA4 que confiere resistencia a gentamicina y tobramicina.
En el año 2002 se describieron dos nuevas metalo-β-lactamasas en Brasil y Alemania denominadas SPM-1 y GIM-135,36. La primera de ellas, descrita recientemente en Europa37, tiene un elevado peso molecular debido a la presencia de un bucle que contiene 23 residuos de aminoácidos y manifiesta cierta actividad frente a aztreonam. El gen responsable de la síntesis de GIM-1, blaGIM−1 se localiza en un integrón de clase 1 (In77) transportado en un plásmido de 22kb y que contiene otros genes de resistencia: aacA4, aadA1 y blaOXA−2.
Alteración de la permeabilidad (porina OprD)La porina OprD permite la entrada al interior de P. aeruginosa de aminoácidos, péptidos pequeños y carbapenémicos8. Como OprD es la principal vía de entrada de los carbapenémicos, la pérdida de esta porina comporta una disminución de la sensibilidad de P. aeruginosa a estos antibióticos7,38. En comparación con imipenem, la entrada al interior de la célula de meropenem parece verse menos afectada ya que en cepas sin OprD, la CIM de imipenem tiene un valor entre 8–32μg/ml y la de meropenem entre 2–4μg/ml (tabla 2). Por ello se sugirió que meropenem podría utilizar otras vías de entrada a través de la membrana externa de P. aeruginosa39. Estudios recientes realizados en paralelo con cepas isogénicas, empleando el fenotipo salvaje y el mutante OprD deficiente, demuestran que se produce una disminución de la sensibilidad a todos los carbapenémicos. Así, para meropenem se observa un aumento de los valores de CIM entre 4–32 veces, para imipenem entre 4–16 veces y para doripenem entre 8–32 veces38. La potencia intrínseca de meropenem y doripenem frente a P. aeruginosa es 4 veces superior a la de imipenem. Por ello, como en las cepas sin mutación los valores de CIM de imipenem ya son de 1–4μg/ml, al producirse la pérdida de la porina OprD, la CIM de este carbapenémico es más elevada que la de meropenem o doripenem.
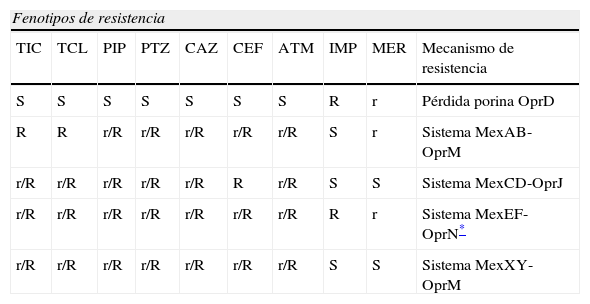
Fenotipos de resistencia a antibióticos betalactámicos en Pseudomonas aeruginosa. Deficiencia de porinas y sistemas de expulsión activa
| Fenotipos de resistencia | |||||||||
| TIC | TCL | PIP | PTZ | CAZ | CEF | ATM | IMP | MER | Mecanismo de resistencia |
| S | S | S | S | S | S | S | R | r | Pérdida porina OprD |
| R | R | r/R | r/R | r/R | r/R | r/R | S | r | Sistema MexAB-OprM |
| r/R | r/R | r/R | r/R | r/R | R | r/R | S | S | Sistema MexCD-OprJ |
| r/R | r/R | r/R | r/R | r/R | r/R | r/R | R | r | Sistema MexEF-OprN* |
| r/R | r/R | r/R | r/R | r/R | r/R | r/R | S | S | Sistema MexXY-OprM |
ATM: aztreonam; CAZ: ceftazidima; CEF: cefepima; IMP: Imipenem; MER: meropenem; PIP: piperacilina; PTZ: piperacilina/tazobactam; R: resistente; S: sensible; r: sensibilidad disminuida; TCL: ticarcilina/ác. Clavulánico; TIC: ticarcilina.
El análisis del genoma de P. aeruginosa ha revelado que este microorganismo posee diversos sistemas de expulsión activa integrados en 5 «superfamilias», aunque predominan los que pertenecen a la familia RND (resistance-nodulation-division)40. Este sistema está integrado por 3 componentes básicos: una proteína localizada en la membrana citoplasmática que actúa como transportador, un segundo componente representado por una proteína o factor de membrana externo y el sistema se completa con una tercera proteína situada en el espacio periplásmico que une las otras 23. Los sistemas de expulsión más frecuentes en P. aeruginosa son MexAB-OprM, MexCD-OprJ, MexEF-OprN y MexXY-OprM. Afectan en mayor o menor grado la actividad no solo de los antibióticos betalactámicos y carbapenémicos sino también de otros antibióticos (fluoroquinolonas, macrólidos, tetraciclinas, cloranfenicol, lincomicina, novobiocina)3,17. Los sistemas MexAB-OprM y MexXY-OprM participan en los mecanismos de resistencia natural y adquirida a varios antibióticos mientras que MexCD-OprJ, MexEF-OprN solo actúan en casos de resistencia adquirida3,41. En la tabla 2 se resumen los principales fenotipos de resistencia esperados según el sistema de expulsión activa expresado.
La sobreproducción de MexAB-OprM se produce por un incremento de la transcripción del operón mexAB-oprM debido a mutaciones en el gen de localización cromosómica mexR que codifica la proteína represora MexR. Otros genes, como nalB, nalD, nalC, también intervienen en la regulación del operón42. La sobreexpresión de MexAB-OprM condiciona una disminución de la actividad de carboxipenicilinas, ureidopenicilinas, cefotaxima, ceftazidima, cefepima y aztreonam. También se observa una disminución de la actividad de meropenem mientras que imipenem no se afecta debido a la diferente estructura molecular a nivel de la cadena lateral.
El operón mexCD-oprJ está gobernado negativamente por NfxB, producto del gen nfxB. Mutantes nfxB-conducen a una hiperexpresión de MexCD-OprJ que afecta a antibióticos betalactámicos con una preferencia sobre las cefalosporinas de 4.a generación (cefepima, cefpiroma). Se han descrito 2 tipos de mutantes, A y B, con diferente nivel de producción de MexCD-OprJ y sensibilidad a los antibióticos3.
La expresión del operón mexEF-oprN, a diferencia de lo que ocurre con los comentados previamente, no está suprimida por un regulador negativo. MexT, producto del gen mexT y miembro de la familia LysR de activadores, actúa regulando de forma positiva este operón, aunque otros genes también están implicados: mexS, mvaT y nfxC. Las cepas que sobreexpresan mexEF-oprN se caracterizan por expulsar fluoroquinolonas y por manifestar una disminución de la sensibilidad a carbapenémicos, en especial imipenem. Esta pérdida de sensibilidad se asocia a una disminución de la porina OprD en cuya expresión participa MexT3.
El operón mexXY carece de un gen que codifica para la proteína de membrana externa. Como contrapartida, MexXY puede asociarse con OprM y probablemente con otras proteínas como OpmB, OpmG, OpmH y OpmI, para formar el sistema funcional típico de 3 proteínas. El operón está regulado negativamente por el gen mexZ, cuya mutación da lugar a una sobreexpresión de mexXY que afecta a varios antibióticos. Entre los antibióticos betalactámicos el más afectado es cefepima3.
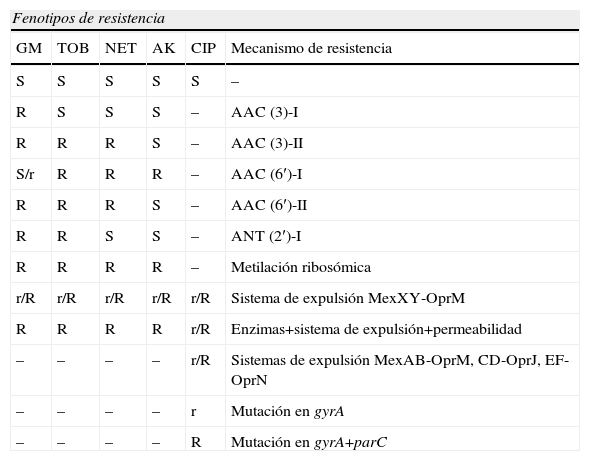
Resistencia a aminoglucósidosEl mecanismo más importante de resistencia a los aminoglucósidos en P. aeruginosa es la modificación enzimática del antibiótico, con la consiguiente disminución de la afinidad del antibiótico por la subunidad ribosómica 30S. Los enzimas responsables están codificados por genes de localización plasmídica y según el radical sobre el que actúan se denominan: fosforiltransferasas (APH), adenililtransferasas o nucleotidiltransferasas (AAD o ANT) y acetiltransferasas (AAC). En la tabla 3 se incluyen los enzimas modificantes más frecuentes en P. aeruginosa y los substratos sobre los que actúan.
Fenotipos de resistencia a aminoglucósidos y fluoroquinolonas en Pseudomonas aeruginosa
| Fenotipos de resistencia | |||||
| GM | TOB | NET | AK | CIP | Mecanismo de resistencia |
| S | S | S | S | S | – |
| R | S | S | S | – | AAC (3)-I |
| R | R | R | S | – | AAC (3)-II |
| S/r | R | R | R | – | AAC (6′)-I |
| R | R | R | S | – | AAC (6′)-II |
| R | R | S | S | – | ANT (2′)-I |
| R | R | R | R | – | Metilación ribosómica |
| r/R | r/R | r/R | r/R | r/R | Sistema de expulsión MexXY-OprM |
| R | R | R | R | r/R | Enzimas+sistema de expulsión+permeabilidad |
| – | – | – | – | r/R | Sistemas de expulsión MexAB-OprM, CD-OprJ, EF-OprN |
| – | – | – | – | r | Mutación en gyrA |
| – | – | – | – | R | Mutación en gyrA+parC |
AK: amikacina; CIP: ciprofloxacina; GM: gentamicina; NET: netilmicina; R: resistente; r: sensibilidad disminuida; S: sensible; TOB: tobramicina.
La metilación de la subunidad 16S del ARN ribosómico se debe a la acción de un enzima localizado en un transposón insertado en un plásmido43. Este nuevo mecanismo de resistencia se describió por primera vez en una cepa aislada en Japón44 y confiere resistencia de alto nivel a amicacina, tobramicina, netilmicina y gentamicina (tabla 3). En P. aeruginosa se han descrito 2 metilasas, RmtA y RmtD con un grado de similitud moderado entre ellas43.
El sistema de expulsión activa MexXY cooperando con OprM, OpmB, OpmG ó OpmI45 es un mecanismo relativamente poco frecuente de resistencia a los aminoglucósidos. Si existe un problema de permeabilidad manifiesta con dificultad para atravesar la membrana externa nos encontraremos con una cepa que expresa resistencia a todos los aminoglucósidos, una situación más habitual en aislados de pacientes con fibrosis quística46. Es muy probable que en algunas cepas también se produzca la suma de otros mecanismos no-enzimáticos, como la síntesis del lipopolisacárido (LPS), tal como sugieren los estudios de El’Garch et al47 en los que se demuestra un aumento gradual de la resistencia a aminoglucósidos.
Resistencia a fluorquinolonasLa resistencia a fluorquinolonas en P. aeruginosa se debe sobre todo a cambios estructurales en la diana (ADN girasa y topoisomerasa IV) o a sistemas de expulsión activa48. Mutaciones puntuales en el gen gyrA localizado en el motivo QRDR (quinolone-resistance-determining-region) da lugar a la síntesis de una ADN girasa o topoisomerasa II con baja afinidad por las fluoroquinolonas49. Un único cambio en un aminoácido sería responsable de un nivel de resistencia moderado mientras que mutaciones que afectan al gen gyrA y al gen parC (subunidad A de la topoisomerasa IV) condicionaría un elevado grado de resistencia50.
Las fluoroquinolonas penetran al interior de la bacteria a través de porinas y por difusión a través de la membrana citoplasmática. Si se produce una disminución en el número de las porinas disminuye la acumulación de la quinolonas, aunque esta situación no parece ser suficiente para que la bacteria manifieste resistencia51. La sobreexpresión de los 4 sistemas de expulsión activa comentados en el apartado de los antibióticos betalactámicos también afectan a las fluoroquinolonas41, aunque en general condicionan un bajo grado de resistencia. Se han encontrado otros sistemas de expulsión activa poco frecuentes que condicionan una disminución de la actividad de las fluoroquinolonas: MexVW-OprM, MexGHI-OpmD y MexPQ-OpmE3. En P. aeruginosa la resistencia de alto nivel a las fluoroquinolonas se debe a la participación conjunta de los sistemas de expulsión activa y a las mutaciones en los genes que codifican la ADN girasa y la topoisomerasa IV52. En la tabla 3 se han incluido los fenotipos de resistencia a ciprofloxacina según el mecanismo expresado.
Acinetobacter baumanniiLa taxonomía del género Acinetobacter ha sufrido diversos cambios a lo largo de la historia. Actualmente se aceptan 32 genoespecies53 definidas en base a la hibridación ADN-ADN, de estas, las de mayor interés como causa de enfermedad infecciosa en el hombre son: A. calcoaceticus (genoespecie 1); A. baumannii (genoespecie 2), Acinetobacter sp. (genoespecies 3 y 13), A. haemolyticus (genoespecie 4), A. junii (genoespecie 5), A. lwoffii (genoespecie 8). En diversos estudios se ha demostrado que algunas especies son difíciles de diferenciar por pruebas fenotípicas. El complejo A. calcoaceticus-A. baumannii consiste en 4 genoespecies (genoespecies 1, 2, 3 y 13) genotípicamente distintas pero fenotípicamente muy similares, por lo que se recomienda para la especiación utilizar una prueba genotípica como puede ser el ARDRA (ribosomal DNA restriction analysis).
Sin duda alguna, A. baumannii es la especie aislada con más frecuencia y con mayor importancia clínica, además de ser de manera significativa la especie más resistente a los antibióticos54,55, mientras que A. lwoffii, la segunda especie en frecuencia de aislamientos, es mucho más sensible a los agentes antimicrobianos. Por ello, la presente revisión se basa fundamentalmente en los fenotipos de resistencia a los antimicrobianos de A. baumannii.
La membrana externa de A. baumannii es menos permeable a los antimicrobianos que la membrana externa de E. coli. Sato y Nakae56 analizaron la permeabilidad de la membrana externa de A. calcoaceticus y encontraron que el coeficiente de permeabilidad a las cefalosporinas era de 2 a 7 veces menor que el que presenta P. aeruginosa para los mismos betalactámicos. Por todo ello, estos autores sugieren que una causa de la resistencia intrínseca que presenta A. calcoaceticus a los antibióticos puede ser atribuida a la presencia de un escaso número de porinas que además poseen un tamaño de poro pequeño. Sin embargo, recientemente se ha descrito que la expresión constitutiva del sistema de expulsión activa (AdeIJK) puede también contribuir a la resistencia intrínseca de este microorganismo a diversos agentes antibacterianos57. La resistencia de algunos antibacterianos específicos se puede también explicar por la expresión de ciertas bombas de expulsión activa, por ejemplo, la expresión de la bomba CraA que afecta al cloranfenicol58 y que parece expresarse constitutivamente y TetA que es una bomba adquirida que afecta tetraciclinas59.
Resistencia a antibióticos betalactámicosEn la actualidad, A. baumannii es resistente a la mayoría de betalactámicos, en especial penicilinas y cefalosporinas, en particular en pacientes que se encuentran en áreas de cuidados intensivos. Así pues, es infrecuente encontrar una cepa de este microorganismo con un fenotipo que presente una total sensibilidad a los betalactámicos (tabla 4). La resistencia a ampicilina, carboxipenicilinas y ureidopenicilinas se ha relacionado con la presencia de betalactamasas plasmídicas de amplio espectro como las tipo TEM-1, TEM-2, OXA-21 y la carbenicilinasa, CARB-560–63. Más recientemente, se ha descrito la penicilinasa SCO-1 en diversas especies de Acinetobacter aisladas en Argentina64.
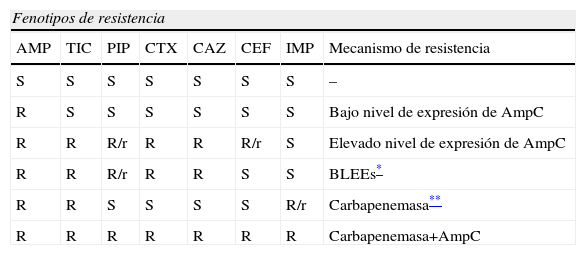
Fenotipos de resistencia a antibióticos betalactámicos en Acinetobacter baumannii
| Fenotipos de resistencia | |||||||
| AMP | TIC | PIP | CTX | CAZ | CEF | IMP | Mecanismo de resistencia |
| S | S | S | S | S | S | S | – |
| R | S | S | S | S | S | S | Bajo nivel de expresión de AmpC |
| R | R | R/r | R | R | R/r | S | Elevado nivel de expresión de AmpC |
| R | R | R/r | R | R | S | S | BLEEs* |
| R | R | S | S | S | S | R/r | Carbapenemasa** |
| R | R | R | R | R | R | R | Carbapenemasa+AmpC |
AMP: Ampicilina; CAZ: ceftazidima; CEF: Cefepima; CTX: cefotaxima; IMP: Imipenem; PIP: piperacilina; R: resistente; r: sensibilidad disminuida; S: sensible; TIC: ticarcilina.
Sin embargo, resultados recientes65–68 sugieren que la sobreexpresión de una cefalosporinasa cromosómica tipo AmpC, también denominada ADC (Acinetobacter derived cephalosporinase), es el mecanismo mas frecuente de resistencia a betalactámicos y genera un fenotipo de resistencia caracterizado por resistencia a ampicilina, cefalotina, piperacilina, cefotaxima y ceftazidima (tabla 4). Esta cefalosporinasa cromosómica se expresa a bajo nivel y en estos casos no confiere resistencia a ceftazidima. Sin embargo, la sobreexpresión del gen blaampC se asocia a la presencia de una secuencia de inserción (ISAba1) insertada en el promotor de dicho gen68–70. Esta secuencia de inserción posee un promotor que es utilizado por la RNA polimerasa para expresar dicha cefalosporinasa. La hiperproducción de AmpC confiere resistencia a ticarcilina, cefotaxima, ceftazidima, cefepima y aztreonam sin afectar cefoxitina o carbapenémicos. Diversos estudios han demostrado que de manera general aproximadamente el 50% de los aislamientos clínicos de A. baumannii presentan una hiperproducción de AmpC66,67. La secuencia de inserción ISAba1 se encuentra aproximadamente en el 69% de los aislamientos clínicos y de estas, en aproximadamente el 78%, la ISAba1 se encuentra insertada en la región del promotor del gen blaampC, confiriendo resistencia a ceftazidima. Las cepas resistentes a cefalosporinas de tercera generación, que no presentan una hiperproducción de AmpC, pueden poseer un beta-lactamasa de espectro extendido. Diversos tipos de BLEES han sido descritas en A. baumannii, entre estas encontramos las tipo PER (PER-1). Este tipo de beta-lactamasa ha sido descrito en el 31% los aislamientos clínicos de A. baumannii aislados en Turquia71. Esta beta-lactamasa ha sido también encontrada en otros países como Corea del Sur, Bélgica y Francia72–74. La BLEE PER-2 se ha descrito en Sud-América75. En España, una BLEE tipo oxacilinasa (OXA-37) se ha descrito en un 27% de las cepas de A. baumannii aisladas de diversos hospitales españoles (resultados no publicados)76. Otras BLEEs tales como CTX-M-2, TEM-92, SHV-5, SHV-12 y VEB-177–81 también se han descrito en cepas concretas de A. baumannii. Esta última BLEE fue descrita en un clon de A. baumannii causante de un brote epidémico en Francia y posteriormente se encontró en diversos hospitales de todo el país y del sur de Bélgica.
Un tipo de beta-lactamasas encontrado a menudo en aislamientos clínicos de A. baumannii son las carbapenemasas que confieren resistencia a los carbapenémicos. Dos tipos de carbapenemasas han sido descritos hasta la actualidad en A. baumannii, las oxacilinasas y las metalo-beta-lactamasas. La OXA-51/69 es otra beta-lactamasa cromosómica descrita en A. baumannii y encontrada en todas las cepas estudiadas. Hasta la actualidad se han encontrado 45 derivados de la OXA-51/6920. En condiciones normales este enzima presenta una actividad carbapenemasa débil. Sin embargo, se ha observado que puede tener lugar una sobreexpresión del gen blaOXA−51 asociada también a la inserción de ISAba1 en el promotor de dicho gen82. Además de esta oxacilinasa cromosómica con actividad carbapenemasa A. baumannii puede adquirir otras oxacilinasas con dicha actividad. Hasta la actualidad se han descrito 3 grupos: las oxacilinasas del grupo de la OXA-23 que incluye la OXA-23, OXA-27 y OXA-49; el grupo de la OXA-24/40 que incluye además la OXA-25, OXA-26 y OXA-72 y finalmente el grupo OXA-58 que incluye además las OXA-96 y OXA-97. Muy recientemente se ha descrito en Brasil la OXA-143 que presenta un 88% de identidad de aminoácidos con la OXA-4020,83. La distribución geográfica de estos diversos grupos es bastante heterogénea. En España, analizando 83 aislamientos clínicos pertenecientes a 28 clones distintos, se encontró que el 19% poseían carbapenemasa tipo OXA-58, el 42% OXA 24/40 y el 13% poseían el elemento genético ISAba1 insertado en el promotor del gen blaOXA−51, lo que favorece la sobreexpresión de dicho gen. En el mismo estudio no se detectaron carbapenemasas tipo OXA-23, VIM o IMP sugiriendo una baja prevalencia de este tipo de carbapenemasas en A. baumannii aislados en España. Por regla general, las cepas que poseen una oxacilinasa tipo OXA24/40 presentan una CIM de imipenem > 32mg/l, mientras que las que poseen una OXA-58 la CIM es de 16mg/l. Hasta la actualidad 3 grupos de metalo-beta-lactamasas han sido descritos en A. baumannii82, estos son: el grupo IMP, con IMP-1, -2, -4, -5, -6 y -11; el grupo VIM, con VIM-1 y -2 y finalmente SIM-1 solo descrita en Corea del Sur. Las variantes de IMP y VIM confieren un elevado nivel de resistencia a carbapenémicos (CIM > 32mg/l), además confieren resistencia a todos los beta-lactámicos excepto aztreonam, mientras que la presencia de SIM-1 genera niveles de resistencia a imipenem entre 8–16mg/l84.
La expresión disminuida de 3 proteínas de membrana externa se han descrito en aislamientos clínicos de A. baumannii resistentes a imipenem: una proteína con un peso molecular 33–36kDa, otra, denominada CarO de 29kDa y finalmente una proteína de 43kDa cque presenta homología con la OprD de P. aeruginosa85. Finalmente, se ha descrito que la ausencia de una PBP de 73,2kDa (PBP2a) podría relacionarse con resistencia a imipenem y/o meropenem de bajo nivel (CIM de 4mg/l), mientras que la ausencia simultánea de esta PBP y otra de 70,1kDa (PBP2b) se asocia con niveles de resistencia más elevada frente a ambos compuestos (CIM de 8–32mg/l)34.
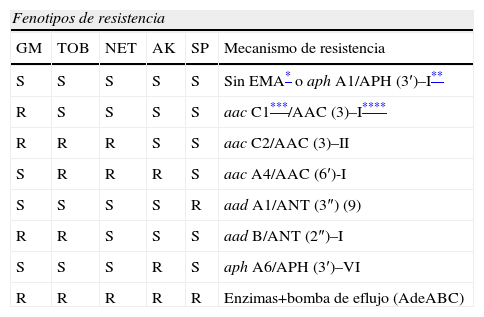
Resistencia a aminoglucósidosSe han descrito en A. baumannii diversas enzimas modificantes de los aminoglucósidos, que desempeñan un papel importante en la adquisición de resistencia a estos antibióticos en este microorganismo60,86,87. La correlación entre fenotipos de resistencia a los aminoglucósidos y enzimas modificantes se resume en la tabla 5. La frecuente presencia de 2 o más enzimas en una misma cepa determina, en muchas ocasiones, patrones de resistencia difíciles de presuponer a expensas del perfil fenotípico. Entre los fenotipos de resistencia cabe destacar por su frecuencia el de resistencia a todos los aminoglucósidos que puede ser debida a la combinación de diversas enzimas modificantes, tal vez a la disminución de la acumulación de estos antibióticos relacionada con la sobreexpresión de un sistema de expulsión activa codificado en el operón AdeABC88. Higgins y colaboradores demostraron que la sobreexpresión de AdeABC aumentaba la CIM de netilmicina a 16mg/l89, por lo que si el antibiograma presenta una CIM de netilmicina de 16mg/l puede ser un buen indicador de la sobreexpresión de esta bomba de expulsión activa, mientras que si la CIM es ≥32mg/l es probablemente debido a la expresión de un enzima modificante de aminoglucósidos.
Fenotipos de resistencia a aminoglucósidos en Acinetobacter baumannii
| Fenotipos de resistencia | |||||
| GM | TOB | NET | AK | SP | Mecanismo de resistencia |
| S | S | S | S | S | Sin EMA* o aph A1/APH (3′)–I** |
| R | S | S | S | S | aac C1***/AAC (3)–I**** |
| R | R | R | S | S | aac C2/AAC (3)–II |
| S | R | R | R | S | aac A4/AAC (6′)-I |
| S | S | S | S | R | aad A1/ANT (3″) (9) |
| R | R | S | S | S | aad B/ANT (2″)–I |
| S | S | S | R | S | aph A6/APH (3′)–VI |
| R | R | R | R | R | Enzimas+bomba de eflujo (AdeABC) |
AK: amikacina; GM: gentamicina; NET: netilmicina; R: resistente; S: sensible; SP: espectinomicina; TOB: tobramicina.
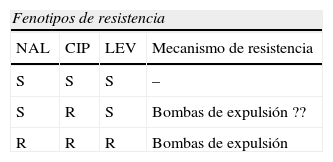
La resistencia a quinolonas en la mayoría de bacilos gramnegativos se asocia con mutaciones en los genes gyrA y parC que codifican las subunidades A de la ADN girasa y la topoisomerasa IV, respectivamente. Ambas enzimas son las proteínas diana de las quinolonas. Resultados preliminares90,91 sugieren que la sobreexpresión de una o varias bombas de expulsión también podrían desempeñar un papel fundamental en la adquisición de resistencia a estos antimicrobianos. En un estudio multicéntrico se encontró que de un total de 244 cepas de A. baumannii aisladas en diversos hospitales españoles solo el 18,6% eran sensibles a ciprofloxacino. Las cepas de A. baumannii sensibles a las quinolonas poseen un rango de CIM de ciprofloxacino (0,06–0,5mg/l) mayor al que presentan las enterobacterias (0,007–0,5mg/l), lo cual probablemente se debe a una permeabilidad disminuida a las quinolonas de este microorganismo con respecto, por ejemplo, a Escherichia coli, o bien a una expresión constitutiva de alguna bomba de expulsión activa92,93. Debido a este nivel basal de resistencia intrínseca, una mutación en gyrA ya supone una adquisición de resistencia tanto al ácido nalidíxico como al ciprofloxacino; sin embargo, las CIM de esparfloxacino y trovafloxacino se mantienen a un nivel inferior a 2mg/l, por lo que es necesario una doble mutación en los genes gyrA y parC, muchas veces acompañado de una sobreexpresión de un sistema de expulsión activa, para generar un fenotipo caracterizado por resistencia a todas las quinolonas (tabla 6).
Fenotipos de resistencia a quinolonas en Acinetobacter baumannii
| Fenotipos de resistencia | |||
| NAL | CIP | LEV | Mecanismo de resistencia |
| S | S | S | – |
| R | R | S | Mutación en gyrA ±bombas de expulsión |
| R | R | R | Mutaciones en gyrA y parC ± bombas de expulsión |
CIP: ciprofloxacino; LEV: levofloxacino; NAL: ácido nalidíxico; R: resistente; S: sensible.
En los últimos años se han descrito aislamientos clínicos de A. baumannii resistentes a todos los agentes antibacterianos incluyendo colistina. Recientemente, se ha descrito que la resistencia a colistina en A. baumannii se asocia con la expresión del gen pmrA, que codifica una proteína perteneciente a un sistema de doble componente. La expresión de este gen conduce a cambios en la expresión de ciertos genes relacionados con la modificación del lipido A. Además otros mecanismos de resistencia como cambios en la expresión de ciertas porinas como OmpW podrían contribuir a dicha resistencia (resultados no publicados).
Stenotrophomonas maltophiliaS. maltophilia es un patógeno oportunista que si bien se encuentra frecuentemente asociado con neumonías, sobre todo en pacientes con fibrosis quística, puede ocasionar una amplia variedad de infecciones nosocomiales93. Existen una serie de problemas metodológicos asociados con la determinación de la sensibilidad antibiótica de este microorganismo. El método de difusión en disco no es fiable y presenta una baja reproducibilidad94. El CLSI recomienda dilución en agar o en caldo para la determinación de la sensibilidad de S. maltophilia. Entre los factores que pueden afectar la determinación de la sensibilidad antibiótica se encuentra que la concentración de diversos cationes como Zn++, Ca++ o Mg++ pueden afectar las CIM de imipenem y carboxipenicilinas y ureidopenicilinas. Otro factor es la temperatura, pues se ha comprobado que este microorganismo presenta menor sensibilidad a aminoglucósidos cuando esta se determina a 30°C94.
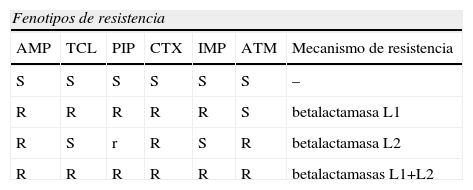
Resistencia a antibióticos betalactámicosLa permeabilidad de la membrana externa de S. maltophilia a los antibióticos betalactámicos podría explicar parte de la resistencia intrínseca basal de este microorganismo a estos antibióticos. La baja permeabilidad puede deberse al bajo número de moléculas de porinas94. Sin embargo, recientemente se han descrito diversos sistemas o bombas de expulsión activa que también podrían contribuir a esta resistencia intrínseca94–96. Dos betalactamasas tienen un papel importante en la resistencia de este microorganismo a los betalactámicos (tabla 7):
- 1.
La beta-lactamasa cromosómica L-1 es dependiente de Zn++, la presentan la mayoría de cepas y posee fundamentalmente actividad penicilinasa: aunque no hidroliza el aztreonam cabe destacar su actividad frente a imipenem y meropenem. No es inhibida por el ácido clavulánico y es sensible a la acción del EDTA.
- 2.
La beta-lactamasa cromosómica L-2 es una beta-lactamasa de serina que posee actividad cefalosporinasa y además hidroliza aztreonam. Esta enzima es sensible a inhibidores de betalactamasas. Ambas enzimas son inducibles.
Fenotipos de resistencia a betalactámicos en Stenotrophomonas maltophilia
| Fenotipos de resistencia | ||||||
| AMP | TCL | PIP | CTX | IMP | ATM | Mecanismo de resistencia |
| S | S | S | S | S | S | – |
| R | R | R | R | R | S | betalactamasa L1 |
| R | S | r | R | S | R | betalactamasa L2 |
| R | R | R | R | R | R | betalactamasas L1+L2 |
AMP: Ampicilina; ATM: aztreonam; CTX: cefotaxima; IMP: Imipenem; PIP: piperacilina; R: resistente; r: sensibilidad disminuida; S: sensible; TCL: ticarcilina+ácido clavulánico.
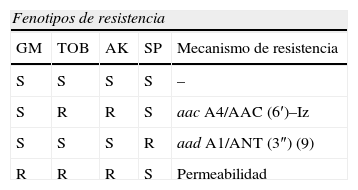
Se han descrito algunas cepas de S. maltophilia que poseen enzimas modificantes de los aminoglucósidos como acetil o O-nucleotidiltransferasas, entre ellas la AAC(6′)Iz, que inactiva tobramicina y amikacina, y se ha encontrado en un elevado número de cepas de S. maltophilia (tabla 8). Sin embargo, el principal mecanismo de resistencia que explica la baja actividad de este tipo de antibióticos frente a S. maltophilia es la disminución en la acumulación de aminoglucósidos en el interior de la bacteria. Esto puede ser debido a cambios en proteínas de membrana externa o a nivel de lipopolisacárido94.
Fenotipos de resistencia a aminoglucósidos en Stenotrophomonas maltophilia
| Fenotipos de resistencia | ||||
| GM | TOB | AK | SP | Mecanismo de resistencia |
| S | S | S | S | – |
| S | R | R | S | aac A4/AAC (6′)–Iz |
| S | S | S | R | aad A1/ANT (3″) (9) |
| R | R | R | S | Permeabilidad |
AK: amikacina; GM: gentamicina; R: resistente; S: sensible; SP: espectinomicina; TOB: tobramicina.
La resistencia a quinolonas en S. maltophilia difiere de los otros gramnegativos no fermentadores descritos en esta publicación, fundamentalmente en el hecho de que las mutaciones en los genes gyrA y parC, que codifican las subunidades A de la ADN girasa y topoisomerasa IV (proteínas diana de las quinolonas) no parecen tener un papel importante en la adquisición de resistencia a las quinolonas, probablemente debido a que en la posición equivalente a la Ser-83 de E. coli, en S. maltophilia encontramos Gln97,98. Recientemente, se ha descrito que en el genoma de este microorganismo99 se encuentra un gen similar al qnr descrito en enterobacterias y que confiere resistencia a las quinolonas mediante protección de las proteínas diana. Este gen denominado Smqnr confiere resistencia intrínseca a las quinolonas en S. maltophilia100. No es infrecuente encontrar un fenotipo de resistencia caracterizado por sensibilidad a ácido nalidíxico y resistencia a norfloxacino o ciprofloxacino (tabla 9). Aunque no se han caracterizado sistemas de expulsión activa en S. maltophilia que afecten norfloxacino y ciprofloxacino sin afectar al ácido nalidíxico, la sobreexpresión de un sistema de expulsión activo con este patrón de especificidad podría ser la causa de este fenotipo101.
Entre los agentes antimicrobianos que presentan una mayor actividad frente a este microorganismo se encuentran el cotrimoxazol, que se considera el antimicrobiano de primera elección, así como la minociclina y la doxiciclina102.
Nota: Sección acreditada por el SEAFORMEC. Consultar preguntas de cada artículo en: http://www.elsevier.es/eimc/formacion