Las variaciones del gen C originan las mutaciones precore y las core, que influyen en la producción de AgHBe. El fallo en los tratamientos antivirales se debe a la presencia de las variaciones en el gen P, que originan las mutaciones de la polimerasa viral.
MétodoHemos realizado estudio genotípico del VHB mediante secuenciación del gen P, y del gen C en pacientes con hepatitis crónica por virus B durante un periodo de cinco años.
ResultadosEl 75% presentaron alguna mutación en la región precore, core o en ambas. El 37% mostró mutaciones de resistencia al tratamiento antiviral. Los genotipos más frecuentes fueron el A y el D.
ConclusionesLa presencia de mutaciones core/precore en pacientes con hepatitis crónica hace obligado un control más estrecho de estos enfermos. La detección de resistencias debe hacerse lo más rápido posible antes de que se produzca un aumento de la carga viral.
Variations of C gene give rise to precore and basal core mutations, which influence HBeAg expression The antiviral treatment failure is due to the presence of variations in the gene P, which cause mutations in the viral polymerase.
MethodsWe performed genotyping of HBV P gene by sequencing, and gene C in patients with chronic hepatitis B over a period of five years.
ResultsA total of 75% of the patients had some mutation in precore or in basal core promoter regions, and 37% demonstrated resistance mutations to antiviral treatment. The most frequent genotypes were A and D.
ConclusionsThe presence of mutations in core/precore regions in patients with chronic hepatitis has led to tighter control of these patients. Detecting for resistance should be done as quickly as possible before there is an increase in viral load.
La infección por el virus de la hepatitis B (VHB) es una de las causas más frecuentes de enfermedad hepática crónica en el mundo1 afectando a unos 400 millones de personas2. En España la prevalencia es del 0,7%, siendo la tercera causa de enfermedad hepática crónica, por detrás de la hepatitis C y el alcohol3 Las variaciones del gen C dan lugar a las mutaciones de la región precore y del promotor basal del core (PBC), que influyen en la producción de HBeAg. La detección simultánea de mutaciones core y precore es muy frecuente, creyéndose que aparecen en primer lugar las mutaciones del core y tras la seroconversión a anti-HBe las mutaciones precore. Las mutantes precore también se han detectado junto al virus salvaje, en pacientes HBeAg positivo, formando una población viral mixta.
La existencia de estos mutantes tienen importantes implicaciones clínicas4: se ha demostrado que las mutantes precore provocan una inflamación hepática más agresiva y, en algunos casos una insuficiencia hepática aguda, además de asociarse con resistencia al tratamiento con interferón alfa, una mayor probabilidad de pérdida del injerto después de un trasplante de hígado, e incluso con el desarrollo de hepatitis fulminante. Sin embargo, también se han encontrado en portadores asintomáticos5.
En los últimos años las opciones terapéuticas de la hepatitis B crónica han aumentado y en la actualidad siete fármacos han sido aprobados para su tratamiento. El fallo en los tratamientos antivirales de la hepatitis B se debe fundamentalmente a la presencia de mutaciones en el genoma del virus, sobre todo a las variaciones en el gen P, que originan las mutaciones de la polimerasa viral, responsables de las resistencias a los tratamientos con los inhibidores de dicha enzima (análogos de nucleósidos/nucleótidos)6–8.
El objetivo de nuestro estudio ha sido estudiar la prevalencia de mutaciones core/precore en nuestra área, y su relación con el HBeAg, genotipo y carga viral, así como la selección de mutaciones en la polimerasa que implican resistencia al tratamiento antiviral.
Material y métodoHemos estudiado una cohorte de seguimiento prospectiva durante un periodo de cinco años (2004-2009), de 168 pacientes con hepatitis crónica por el virus de la hepatitis B y carga viral detectable, atendidos en nuestro hospital. Las determinaciones de los marcadores serológicos se realizaron mediante EIA con el sistema comercial de DiaSorin®. Los niveles de ADN del VHB se determinaron en suero utilizando el sistema VERSANT® HBV DNA 3.0 (bDNA) de Siemens® (factor de conversión aportado por el fabricante [1UI/mL=5,6copias/mL]).
El genotipado del VHB y las mutaciones de resistencia antiviral se determinaron en suero por secuenciación de ácidos nucleicos con el kit Trugene HBV Genotyping (Siemens®) que detecta genotipos de la A-G amplificando una secuencia definida de la polimerasa del virus. La detección de mutaciones core y precore con el kit de secuenciación abierta de Siemens ® tras doble PCR de dicha región utilizando primers específicos del VHB según la técnica descrita por Poovorawan et al.9. La extracción se llevó a cabo con el sistema automatizado MagnaPure (Roche®). La recogida y almacenamiento de muestras se hicieron siguiendo las normas del comité de ética e investigación de nuestro hospital.
Los datos recogidos de los pacientes y los obtenidos de las diferentes técnicas realizadas se incorporaron a una base de datos utilizando el paquete informático SPSS para Windows versión 15.0. Para las variables de naturaleza cualitativa se calcularon las tablas con las respectivas distribuciones de frecuencia y para las de naturaleza cuantitativa, los estadísticos descriptivos de centralización y dispersión más usuales. En todos los casos se consideraron resultados significativos cuando el nivel de significación obtenido fue inferior al 5% (p<0,05).
ResultadosDe los 168 pacientes, 114 (67,9%) eran hombres con edades comprendidas entre los 17-83años y una edad media de 47±12años. La vía de transmisión fue vertical en el 27% y desconocida en el 60%. Quince pacientes estaban coinfectados (8 VIH, 6 VHC, 1 VHD). El 30% eran portadores asintomáticos del HBsAg con niveles normales de alanino aminotransferasa. El 58,1% presentaban hepatitis crónica activa y el 11,9% tenían datos de cirrosis hepática, confirmada por la clínica, la histología y los datos ecográficos.
El 73% de los pacientes no expresaron el HBeAg, y de ellos el 87,9% seroconvirtieron a anti-HBe. 106 pacientes (63,1%) mostraron genotipo D, 39 (23,2%) genotipo A, y del resto el porcentaje fue escaso 2-3% cada uno. Noventa y nueve sujetos (58,9%) presentaron cargas virales superiores a105copias/mL.
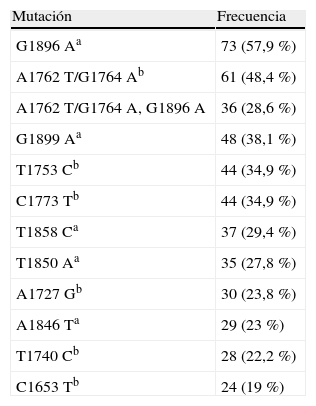
Estudiamos la presencia de las mutaciones core y precore en los 168 pacientes. De ellos, 126 (75%) presentaron alguna mutación: 17,5% en una de estas regiones (15 pacientes mutaciones precore, y 7 mutaciones core) y el 82,5% (104 pacientes) en ambas. Once pacientes (6,5%) no presentaron ninguna mutación y en 31 pacientes (18,5%) no logramos amplificar la muestra. En la tabla 1 describimos el porcentaje de las mutantes core y precore más frecuentes detectadas. Debido al escaso número de pacientes sin mutaciones, a partir de ahora solo nos referiremos a estos 126 pacientes.
Distribución de los 126 pacientes con mutaciones core y precore más frecuentes
| Mutación | Frecuencia |
| G1896Aa | 73 (57,9%) |
| A1762T/G1764Ab | 61 (48,4%) |
| A1762T/G1764A, G1896A | 36 (28,6%) |
| G1899Aa | 48 (38,1%) |
| T1753Cb | 44 (34,9%) |
| C1773Tb | 44 (34,9%) |
| T1858Ca | 37 (29,4%) |
| T1850Aa | 35 (27,8%) |
| A1727Gb | 30 (23,8%) |
| A1846Ta | 29 (23%) |
| T1740Cb | 28 (22,2%) |
| C1653Tb | 24 (19%) |
aprecore.
bcore.
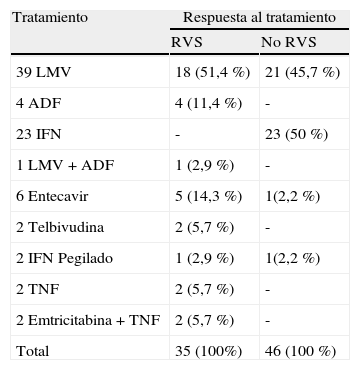
Ochenta y un pacientes (66%) con mutaciones core y precore recibieron tratamiento antiviral, y de ellos, 35 pacientes (43,2%) mantuvieron respuesta viral sostenida (RVS) 12meses tras finalizar tratamiento. El tratamiento inicial de los pacientes se resume en la tabla 2. De los pacientes que fracasaron, 18 recibieron tratamiento con adefovir, 4 con tenofovir y 3 con entecavir y en 5 se usó tratamiento antiviral combinado.
Respuesta al tratamiento entre los pacientes con mutaciones core y precore del VHB
| Tratamiento | Respuesta al tratamiento | |
| RVS | No RVS | |
| 39 LMV | 18 (51,4%) | 21 (45,7%) |
| 4 ADF | 4 (11,4%) | - |
| 23 IFN | - | 23 (50%) |
| 1 LMV+ADF | 1 (2,9%) | - |
| 6 Entecavir | 5 (14,3%) | 1(2,2%) |
| 2 Telbivudina | 2 (5,7%) | - |
| 2 IFN Pegilado | 1 (2,9%) | 1(2,2%) |
| 2 TNF | 2 (5,7%) | - |
| 2 Emtricitabina+TNF | 2 (5,7%) | - |
| Total | 35 (100%) | 46 (100%) |
En general, el 37% de los pacientes que recibieron tratamiento presentó alguna mutación del gen de la polimerasa que confería resistencia antiviral. Destacaron por su frecuencia las mutaciones seleccionadas por el tratamiento con lamivudina (LMV): 17 pacientes (21%) presentaron la mutación rtM204V acompañada en todos los casos de la mutación compensatoria rtL180M. 10 pacientes (12,3%) presentaron la mutación rtM204I, dos de ellos junto a la rtL180M.
En relación con las mutaciones de resistencia para adefovir (ADV), hemos detectado 3 pacientes (3,7%) que tenían la mutación rtA181V/T, y otros 3 (3,7%) la rtN236T en los tratamientos de rescate. Ninguno consiguió respuesta virológica. No hemos encontrado entre nuestro grupo la mutación rtI233V.
Tres (3,7%) pacientes en tratamiento con entecavir seleccionaron la mutación rt184G, y en ningún caso detectamos mutaciones relacionadas con el tenofovir.
DiscusiónLa existencia de las mutantes core/precore, llamadas también variantes mediterráneas, porque son muy frecuentes en esta área y en el sur de Europa, coincide en nuestra zona con la de regiones del noreste de nuestro país10,11, mientras que en otras series oscila entre 14 y el 54%12,13.
Si nos centramos en mutaciones concretas, hemos encontrado que el 57,9% de los pacientes tenían la mutación precore G1896A, el 48,4% la doble mutación del core A1762T/G1764A, y la combinación de esta doble mutación del core y la mutación precore G1896A, la hemos detectado en el 28,6% de los pacientes. Son resultados similares a los encontrados en la literatura, excepto en estudios como el de Choi et al.14, donde la doble mutación del core alcanza un porcentaje del 87%.
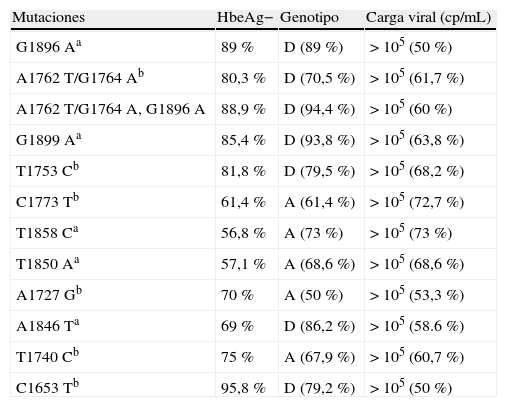
La determinación del genotipo tiene implicaciones en relación con la prevalencia de las mutaciones core y precore15. En nuestro estudio, la presencia de ciertas mutaciones en el PBC se relacionó con el genotipo A (mutaciones core C1773T, A1727G y T1740C, p<0,05), y en la región precore con el genotipo D (mutación precore G1896A, G1899A o T1846A, p<0,05) (tabla 3). En otras series se ha relacionado también la mutación G1896A con el genotipo D16,17; sin embargo, en el estudio de Jardi et al. a diferencia del nuestro, la presencia de mutaciones en PBC fueron similares en genotipos A y D11.
Distribución del HbeAg, genotipo y carga viral en los 126 pacientes con mutaciones core y precore más prevalentes de los pacientes con hepatitis B crónica
| Mutaciones | HbeAg− | Genotipo | Carga viral (cp/mL) |
| G1896Aa | 89% | D (89%) | >105 (50%) |
| A1762T/G1764Ab | 80,3% | D (70,5%) | >105 (61,7%) |
| A1762T/G1764A, G1896A | 88,9% | D (94,4%) | >105 (60%) |
| G1899Aa | 85,4% | D (93,8%) | >105 (63,8%) |
| T1753Cb | 81,8% | D (79,5%) | >105 (68,2%) |
| C1773Tb | 61,4% | A (61,4%) | >105 (72,7%) |
| T1858Ca | 56,8% | A (73%) | >105 (73%) |
| T1850Aa | 57,1% | A (68,6%) | >105 (68,6%) |
| A1727Gb | 70% | A (50%) | >105 (53,3%) |
| A1846Ta | 69% | D (86,2%) | >105 (58.6%) |
| T1740Cb | 75% | A (67,9%) | >105 (60,7%) |
| C1653Tb | 95,8% | D (79,2%) | >105 (50%) |
cp/mL: copias/mililitro; HBeAg: antígeno E.
aMutaciones precore.
bMutaciones core.
El 73% de nuestros pacientes con mutaciones core/precore, tenía el HBeAg negativo. La presencia de HBeAg en pacientes con mutaciones precore se puede explicar por la coinfección con una cepa wild type o bien con una cepa que posea una mutación en la región core18. La relación de la doble mutación del core con el HBeAg es variable según la literatura consultada. Así, mientras que en nuestro estudio más del 80% de los portadores de la doble mutación tenían el HBeAg negativo (p<0,05), en otras series19 la asociación con la doble mutación del core parece ser más fuerte en aquellos con el HBeAg positivo.
Encontramos que el 89% de pacientes con las mutaciones precore G1896A o G1899A tenían HBeAg negativo (p<0,05) al igual que un elevado porcentaje (95%) entre los pacientes con la mutación core C1653T (p<0,005), que además seroconvirtieron prácticamente en el 100% de los casos. Sin embargo, no encontramos significación estadística al comparar los pacientes con la mutación del core T1753C, ni la mutación core T1740C con el HBeAg, al contrario que Choi et al.14.
El 63,2% de pacientes con mutaciones core y precore presentaron carga viral >105copias/mL, sin que esta relación haya sido significativa para ninguna mutación en concreto. Sterneck et al.20 publicaron que la presencia de mutaciones precore asociadas con la doble del core puede aumentar de forma moderada la replicación del virus. Es conocido además que los niveles de ADN del VHB son generalmente más bajos en pacientes con hepatitis crónicas HBeAg negativo y además fluctúan21, por lo que algunos pacientes podrían ser considerados como portadores inactivos si no se les hace un seguimiento longitudinal. Entre los 92 pacientes con HBeAg negativo, el 50% presentó cargas virales >105copias/mL. Sin embargo, entre nuestros pacientes con HBeAg positivo el 91% cursó con cargas virales >105copias/mL. La presencia de las mutaciones core C1773T, A1727G o T1740C en pacientes con HBeAg positivo se asociaron en un 100% de los casos con cargas virales >105copias/mL (p<0,005).
Diversos estudios22 demuestran que una elevada carga viral de VHB, por encima de las 105copias/mL, es un factor de riesgo predictivo de cirrosis y carcinoma hepático, independientemente del estado del HBeAg y del nivel de transaminasas. En este sentido, uno de los principales estudios realizados entre más de 3.000 pacientes con hepatitis B crónica, el estudio REVEAL-HBV23, encuentra que la cuantificación de la carga viral del VHB es esencial para caracterizar el estado en que se encuentra la infección, así como para evaluar el riesgo de desarrollo de cirrosis y HCC ya que el mantenimiento de cargas virales elevadas es un fuerte factor predictor de riesgo de este tipo de cáncer24. En nuestro estudio 21 pacientes mostraron complicaciones compatibles con cirrosis o carcinoma hepático. El 81% tenía carga viral >105copias/mL.
Se ha sugerido además que las mutaciones del core pudiesen ser por sí mismas un factor de riesgo primario para el desarrollo de hepatocarcinoma18, atribuyendo el desarrollo de enfermedad hepática severa a la alta prevalencia de la doble mutación del core especialmente en los pacientes con genotipo C. En otras series13,25, la edad elevada, las transaminasas altas (>80) y la doble mutación A1762T/G1764A se asocian significativamente con cirrosis hepática en pacientes con HBeAg positivo, por lo que podría servir como un fuerte y útil marcador predictor en el desarrollo de cirrosis en estos pacientes. En nuestra serie, el 76,2% de pacientes con complicaciones tenían HBeAg negativo, el 77% presentó la doble mutación del core siendo el 81% genotipo D. En otras series se ha relacionado el genotipo D con peor progresión de la enfermedad12, pero podría deberse esta relación a la alta frecuencia de este genotipo en todas las formas clínicas en esa área geográfica.
Los objetivos del tratamiento de los pacientes con hepatitis crónica B son precisamente evitar la progresión de la enfermedad hepática, el desarrollo de cirrosis y de hepatocarcinoma. Distintos estudios han demostrado que la supresión persistente de la replicación viral se asocia a remisión de la enfermedad hepática26. El mayor inconveniente del tratamiento prolongado con análogos nucleósidos y nucleótidos es la inducción a la aparición de cepas mutantes del VHB que son resistentes a los diferentes tipos de fármacos utilizados.
En nuestro estudio, si nos centramos en las mutaciones rtM204V, rtM204I, y rt180M relacionadas con el tratamiento con lamivudina, el 90% de los pacientes que presentaban cualquiera de estas mutaciones no alcanzaron una respuesta viral sostenida. Al hacer el análisis multivariante y tomando como variable dependiente la respuesta al tratamiento, hemos observado cómo la presencia de la mutación rt204V incrementa 14 veces el riesgo de no presentar respuesta viral sostenida.
Sobre la relación entre el genotipo y la respuesta a lamivudina, algunos trabajos concluyen que hay una mayor tendencia a desarrollar mutaciones de resistencia en el genotipo A que en el D, si bien esta diferencia se aprecia tan solo durante el primer año de tratamiento, observándose así mismo diferencias en el patrón de resistencias entre los 2 genotipos: en el genotipo A el patrón más frecuente es el rtL180M+rtM204V, mientras en el genotipo D predomina la mutación rtM204I27. En cuanto al adefovir, no parecen existir diferencias entre los genotipos A y D28. En nuestra serie, el 90% de pacientes con la mutación rtM204I pertenecían al genotipo D y todos los enfermos con mutaciones de resistencia a adefovir portaban un virus con genotipo D sin que esta relación fuese significativa.
Las resistencias al tratamiento con entecavir son muy bajas: del 0% el primer y segundo año de tratamiento en pacientes sin tratamiento previo. En pacientes con tratamiento previo y con cepas del VHB resistentes a la lamivudina, las tasas de resistencias son del 7% al primer año y del 16% al segundo año de tratamiento. En nuestros resultados, de los nueve pacientes que se trataron con Entecavir, tres seleccionaron mutaciones de resistencias a este fármaco: dos portaban mutaciones rtL180M y rtM204V, y uno mutaciones rtA181V/T, y rtN236T relacionadas con adefovir, lo cual podría limitar el rescate con este fármaco en pacientes con virus que portan mutaciones por la posibilidad de seleccionar resistencias que conduzcan al fracaso del tratamiento.
Al hacer el análisis bivariante no hemos encontrado significación estadística entre las mutaciones del promotor basal del core, las mutaciones precore y la respuesta viral tras el tratamiento, a diferencias de otros autores como Chen et al.29 que encuentran una frecuencia más alta de mutaciones del promotor basal del core, C1773T, T1802C y de mutaciones precore, A1846T, la T1850A y T1858C en los pacientes que no mantenían respuesta viral sostenida.
Sin embargo, cuando hicimos en el análisis multivariante, tomando como variable dependiente la respuesta al tratamiento y como variables independientes el sexo, la vía de contagio, el HBeAg, la carga viral, las mutaciones core y precore más frecuentes en nuestro estudio y las mutaciones de resistencia antiviral, la mutación core T1740C, con un riesgo relativo menor que 1, nos dice que es un factor protector o que favorece a la respuesta viral sostenida.
Entre la limitaciones de nuestro trabajo destacan que el genotipado viral por secuenciación sin posterior clonación implica la no detección de poblaciones minoritarias (<20%) y en consecuencia no podemos descartar posibles coinfecciones con diferentes genotipos virales. Además tenemos un porcentaje de no amplificación del 18% posiblemente por condiciones inadecuadas de transporte o almacenamiento de esas muestras. Por último, nos gustaría mencionar que nuestra intención fue hacer un estudio de casos y controles, pero debido a la alta prevalencia de mutaciones core/precore en nuestra zona, tuvimos que replantearnos el estudio y acabar haciendo una cohorte de seguimiento de los pacientes con mutaciones. A pesar de estas limitaciones, nuestros resultados concuerdan con lo descrito hasta el momento en la literatura y creemos que aportan una información válida ya que la presencia de estas mutaciones en pacientes con hepatitis crónica hace obligado en cualquier caso un control más estrecho de estos enfermos, puesto que un perfil serológico caracterizado por HBeAg negativo y anti-HBe positivo no siempre es un marcador de ausencia de replicación viral y de recuperación de enfermedad hepática.
Aunque existen una serie de opciones terapéuticas para los pacientes con hepatitis crónica B, la elección del fármaco debe ser individualizada en cada caso. La detección de resistencias debe hacerse lo más rápido posible antes de que se produzca un aumento de la carga viral. A la vista de nuestros resultados, por su eficacia y su buen perfil de resistencias, el tenofovir se presenta como una de los fármacos de elección tanto en el tratamiento inicial como en el rescate, incluso en pacientes con mutaciones de resistencia a otros fármacos
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.