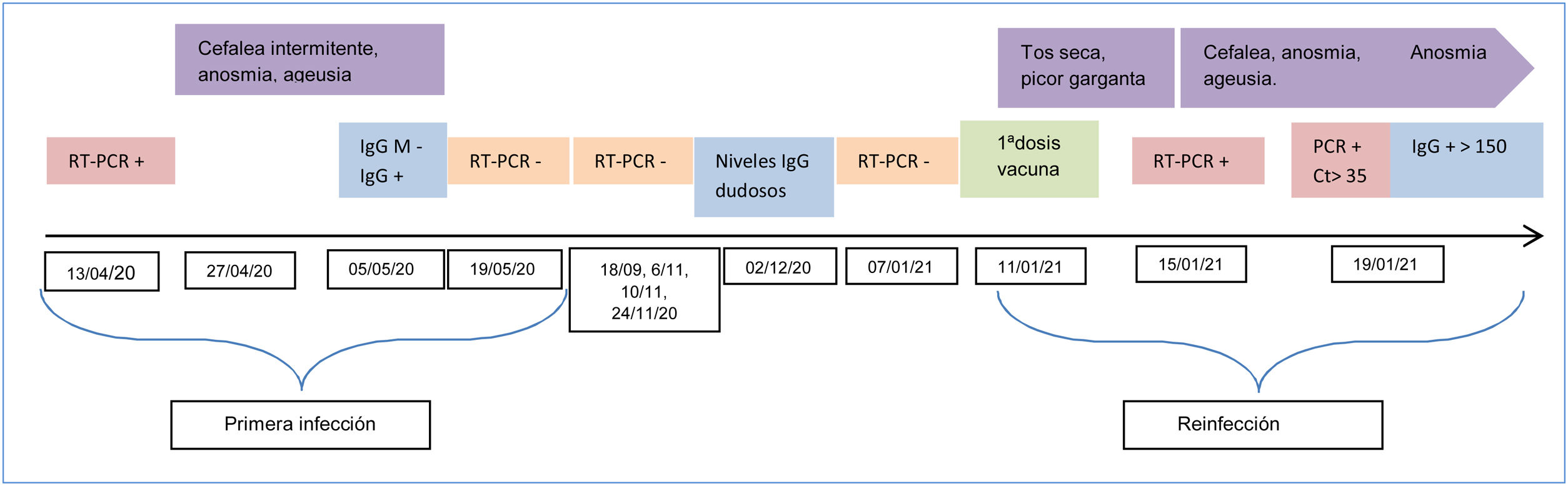
Presentamos un caso con una evidencia de reinfección sólida por SARS-COV-2. El seguimiento exhaustivo realizado a los trabajadores del hospital durante la pandemia por SARS-COV-2ha sido clave para identificar esta sospecha de reinfección confirmada por secuenciación. Cabe destacar que ambas infecciones se diagnosticaron a partir del estudio de contacto realizado entre los trabajadores al identificarse casos de COVID-19 en pacientes de hospitalización sin COVID-19. La primera infección se produjo durante el primer pico de la pandemia, en abril de 2020. La trabajadora estaba asintomática y el 13/04/2020 se le realizó una prueba de amplificación de ácido nucleico nasofaríngeo en el contexto de un estudio de contacto de origen laboral con un paciente positivo. El resultado de la RT-PCR fue positivo con Ct de 23,55 (<35). Tras la prueba positiva aparecieron los síntomas (cefalea, anosmia y ageusia), que fueron leves. A los 37 días del diagnóstico la RT-PCR ya era negativa y se identificó respuesta inmunológica (valor índice IgG 3,98; positivo> 1,6; COVID-19 VIRCLIA® IgG monotest, VIRCELL).
En los meses posteriores se le realizaron, por el programa de control de la infección intrahospitalaria, varias RT-PCR con resultado negativo (fig. 1). Ocho meses después se identificó que los niveles de IgG habían disminuido sensiblemente (IgG 1,55). La mayoría de las personas tienen respuesta inmunológica después de la infección con anticuerpos1 y, aunque suelen disminuir durante los meses posteriores, muchos estudios indican que la actividad neutralizante se mantiene hasta los 6-8 meses2,3. Esta respuesta inmunológica está asociada a la protección frente a reinfecciones, al menos, a corto plazo4.
La segunda infección también se detectó por estudio de contacto. Se diagnosticó el 15/01/2021 por RT-PCR. La trabajadora presentaba síntomas leves (tos y picor de garganta), que no había notificado. Creemos que aunque el programa de detección precoz de la infección por SARS-COV-2 entre los trabajadores era conocido y estaba correctamente implantado, el haber pasado la infección y ser conocedora de su respuesta inmunológica no le generó una sospecha de infección por SARS-COV-2 y, por ello, no lo notificó a salud laboral. Además, los días previos (11/01/21) había recibido la primera dosis de la vacuna de Pfizer. En este caso, podemos identificar que la reinfección se produjo después del 07/01/21, ya que ese día obtuvimos una RT-PCR con resultado negativo. Creemos que la trabajadora ya debía estar infectada el día de la vacunación, puesto que el 15/01/21 ya tenía síntomas y el período de incubación medio es de 5-6 días. Además, a los 4 días del diagnóstico se le practicó una RT-PCR y determinación de IgG de la subunidad S1 de SARS-CoV-2 (anti-S), en la que se detectó carga viral baja (Ct > 35) y niveles muy altos de respuesta inmunológica (>150). La dinámica de la respuesta inmune en humanos está descrita. Es posible detectar anticuerpos totales, IgM e IgG, con una sensibilidad creciente en el curso de la infección, que es superior al 90% en la segunda semana tras el inicio de los síntomas5,6. En nuestro caso, el hecho de detectar niveles de IgG anti-S tan altos a los pocos días del inicio de la sintomatología nos hace pensar en un posible efecto booster, principalmente asociado a la vacuna, aunque no disponemos de las IgG anti-N. Se ha evidenciado que la inoculación de una sola dosis de las vacunas mRNA en un individuo que haya tenido la infección da lugar a un incremento exponencial de la respuesta inmunológica7. En otra publicación8, describen que una sola dosis de vacuna mRNA da lugar a una respuesta inmune inmediata en personas seropositivas, con títulos de anticuerpos posvacunación similares o superiores a los seronegativos con 2dosis de la vacuna.
La secuenciación es necesaria para confirmar la reinfección y diferenciarla de la RT-PCR persistente. Hay descritos casos esporádicos de reinfección confirmada por secuenciación en el mundo9,10. En nuestro caso, la posibilidad de secuenciar las 2muestras confirmó la reinfección, al mostrar linajes diferentes. La tecnología de secuenciación utilizada fue Illumina (plataforma MiSeq) GISAID para la primera infección EPI_ISL_1595836 y GISAID para la reinfección EPI_ISL_1595838. En caso de muestras con cargas virales bajas, el laboratorio aumenta la cantidad de extracto de RNA para disponer de material suficiente para la secuenciación. La primera infección corresponde a un miembro de Clade 20B (linaje B.1.1) y la segunda corresponde a Clade 20E (linaje B.1.177). El linaje de la segunda muestra comenzó a circular en España a partir del verano de 2020.
La implementación del programa de diagnóstico y seguimiento exhaustivo de los trabajadores durante la pandemia de COVID-19 en nuestro centro permitió detectar este caso. Consideramos que esto es importante para el buen control de la infección intrahospitalaria.