La virulencia bacteriana ha experimentado un largo proceso evolutivo dependiente de la relación huésped/patógeno, mientras que la resistencia antimicrobiana ha tenido una evolución muy diferente, más corta y cambiante debido a la presión biológica provocada por la introducción de los antimicrobianos en medicina por parte del hombre. Esta fuerte presión ha obligado a los microorganismos a adaptarse a esas condiciones cambiantes, adquiriendo o desarrollando nuevos mecanismos de resistencia de manera continuada, provocando cambios importantes en las funciones celulares, influyendo finalmente sobre la virulencia y el fitness bacterianos. Múltiples son los factores que pueden mediar en la relación entre virulencia y resistencia, y a menudo genes implicados en ambos fenómenos presentan el mismo medio de transporte y dispersión, como los plásmidos. Islas, integrones, transposones y otros elementos genéticos podrían también facilitar la coselección de genes implicados en virulencia y resistencia. La ganancia de resistencia puede afectar de manera muy variable a la virulencia, dependiendo principalmente de la especie bacteriana, del ambiente y del propio mecanismo de resistencia. En la revisión se exponen diferentes fenómenos en los cuales el mecanismo genético que proporciona una ventaja frente a los antimicrobianos afecta directamente a la virulencia y al fitness, como son cambios en la estructura de la pared celular, bombas de expulsión, porinas o sistemas reguladores de dos componentes. La coselección de factores de virulencia y resistencia antimicrobiana, así como la relativa facilidad de las bacterias para desarrollar mutaciones compensatorias, puede favorecer, especialmente en ambientes con una alta presión antibiótica, la aparición de clones prevalentes, virulentos y además con escasas opciones terapéuticas, lo que podría ser un importante problema de salud en un futuro cercano.
While bacterial virulence has experienced a long host/pathogen-dependent evolutionary process, antimicrobial resistance has had a very different, shorting and changing evolution due to the biological pressure caused by the introduction of the antimicrobials in human medicine. This strong pressure has forced the microorganisms to adapt to these changing conditions, continuously acquiring or developing new resistance mechanisms, causing major changes in cellular functions and finally influencing the virulence and bacterial fitness. Multiple factors may mediate in the relationship between virulence and resistance. The genes often involved in both phenomena have the same transport and dispersion mediums. Islands, integrons, transposons and other genetic elements could also facilitate the combined selection of virulence and resistance genes. The increase in resistance can affect virulence in different ways, mainly depending on the bacterial species, the environment, and the mechanism of resistance. This review presents the different phenomena in which the genetic mechanism that provides an advantage over the antimicrobials directly affects the virulence and fitness, such as changes in the structure of the cellular wall, efflux pumps, porins or two-component regulatory systems. The co-selection of virulence and antimicrobial resistance factors and the relative ease of bacteria to develop compensatory mutations can favour, particularly in environments with high antibiotic pressure, the emergence of prevalent clones. These can be virulent and with few treatment options, and could be a major health problem in the near future.
Los mecanismos bacterianos implicados tanto en la resistencia antimicrobiana como en la virulencia son en la actualidad objeto de numerosos estudios en el ámbito de la microbiología infecciosa. Es sin embargo más difícil localizar trabajos en los que se estudien conjuntamente ambos aspectos, que son esenciales para entender el comportamiento de los patógenos bacterianos y diseñar adecuadas estrategias terapéuticas para combatirlos de una manera eficaz.
Para poder entender con mayor facilidad esta revisión sobre mecanismos de resistencia y virulencia, es necesario introducir una serie de conceptos básicos sobre la patogénesis bacteriana. Varios son los mecanismos implicados en la «virulencia bacteriana», como la colonización, la capacidad de sobrevivir en superficies ambientales y la infección1. Un factor común a los tres procesos es el «fitness bacteriano», definido como la capacidad de la bacteria de sobrevivir o replicarse2. Además del fitness, otros factores que contribuyen al desarrollo de la colonización son la capacidad de adherencia a las células epiteliales hospedadoras, la resistencia a los agentes inhibitorios, la formación de biofilms y el quorum sensing (QS). Respecto a la supervivencia en superficies ambientales durante largos períodos de tiempo, destacan la resistencia a desinfectantes y antimicrobianos, la utilización de diversas vías metabólicas para su crecimiento, la formación de biofilms en superficies y aparatos invasivos, así como la respuesta QS entre bacterias. Finalmente, entre los mecanismos implicados en el desarrollo del proceso infeccioso destacan la obtención de una respuesta inflamatoria, los sistemas de adquisición del hierro, la resistencia a la actividad bactericida del suero humano y la citotoxicidad1.
Aunque la adquisición de resistencia a los antimicrobianos no es específicamente un factor de virulencia, ambos aspectos guardan entre sí una estrecha relación. Así, por ejemplo, los factores de resistencia ofrecen al microorganismo la capacidad de continuar replicándose en un individuo tratado con antimicrobianos, desplazando al resto de microorganismos sensibles y expresando así su carga genética de patogenicidad. Patógenos multirresistentes como Pseudomonas aeruginosa o Acinetobacter baumannii, resistentes en muchos casos a la práctica totalidad de los antibióticos, son capaces de colonizar y multiplicarse en ambientes donde el resto de especies bacterianas son incapaces, lo que les confiere una ventaja evolutiva y la capacidad de causar brotes infecciosos epidémicos, lo que en cierta manera puede considerarse un factor de virulencia. Es en especial en el ambiente hospitalario, donde un patógeno debe ser no solo virulento sino también resistente a los antimicrobianos para poder causar una enfermedad infecciosa3,4. Las infecciones nosocomiales son producidas en gran parte por patógenos oportunistas y no por patógenos primarios, y una gran parte de ellos son especies medioambientales capaces de sobrevivir y desarrollarse en el ambiente hospitalario con una mayor facilidad que los patógenos tradicionales gracias a su plasticidad y a su capacidad para adaptarse adquiriendo mecanismos de resistencia o persistencia en el ambiente.
Los fenómenos de virulencia y resistencia presentan bastantes características en común, y entre las más importantes se encuentran los mecanismos de transmisión horizontal que pueden existir entre diferentes especies. Los plásmidos son posiblemente los principales vehículos de transmisión de los elementos genéticos que codifican los factores de virulencia y resistencia, los cuales se describen en detalle en esta revisión. Tanto la virulencia como la resistencia son mecanismos adaptativos similares que cumplen su función bajo condiciones de estrés, tales como el proceso de infección o colonización y la presión antibiótica, respectivamente. Estas condiciones de infección y de presión antibiótica dan lugar a un cuello de botella en la población bacteriana que limita enormemente la capacidad de crecimiento y disminuye a la vez la diversidad microbiana4. A menudo la resistencia antimicrobiana se encuentra también asociada a la infección y por tanto también vinculada a virulencia, como en el caso de microorganismos productores de biofilm o en infecciones intracelulares5,6. Otras características que presentan en común virulencia y resistencia son la implicación directa de bombas de expulsión7,8, porinas9,10, alteraciones de la pared celular o los sistemas de dos componentes, sensor-receptor, que en función de las condiciones del ambiente pueden activar o reprimir la expresión de diferentes genes, entre ellos genes implicados en resistencia y virulencia11.
Por otro lado, la práctica médica por parte del hombre ha limitado la evolución o la diseminación de los microorganismos infecciosos, pero a costa de un aumento de la resistencia antibiótica de manera global. Los determinantes de virulencia y resistencia han tenido una evolución muy diferenciada a lo largo del tiempo. Los diferentes mecanismos de virulencia existentes se encuentran integrados en el cromosoma bacteriano (islas de patogenicidad, integrones) o bien en elementos genéticos móviles (plásmidos, transposones…). La adquisición de un fenotipo de patogenicidad en genomas bacterianos adaptados al hospedador mediante la integración de diferentes factores de virulencia ha tenido lugar posiblemente durante largos periodos de coevolución patógeno/huésped. Por el contrario, la selección y diseminación de los factores de resistencia es un fenómeno mucho más reciente que ha tenido lugar mayoritariamente en los últimos 50 años, desde el inicio de la utilización de los antibióticos por parte del hombre. Sin embargo, pese a estas diferencias en su evolución, a menudo ambos tipos de genes presentan similares mecanismos de dispersión, llegando a compartirlos en muchos casos, como veremos más adelante12,13.
Aunque hemos centrado esta revisión en el estudio de la relación virulencia/resistencia, en algunos casos hemos decidido incluir el papel que la ganancia de resistencia tiene sobre el fitness, ya que en buena medida virulencia y fitness se encuentran asociados directamente frente a la ganancia de resistencia (esta ganancia modifica directamente las tasas de virulencia y fitness a la vez) o indirectamente (la ganancia de resistencia tiene un coste de fitness y este afecta a la virulencia). Cuando se han analizado el coste de fitness y la virulencia en un mismo estudio frente a la ganancia de resistencia, generalmente se ha visto un descenso de ambos en los mutantes resistentes2,14-16. Sin embargo en los últimos años, debido a la enorme presión antibiótica a que se ven sometidas las bacterias, especialmente en los ambientes nosocomiales, cada vez con más frecuencia se publican trabajos en los que la ganancia de resistencia presenta un efecto positivo sobre la virulencia; por ejemplo, diseminándose los mecanismos de resistencia y virulencia de manera conjunta, o bien porque el mecanismo de resistencia está también directamente implicado en la virulencia de la bacteria.
En esta revisión nos centraremos en intentar dar respuesta a las siguientes cuestiones:
- •
¿Cuál es el efecto que tiene la adquisición de resistencia sobre la virulencia en una especie patógena?
- •
Resistencia antibiótica y virulencia: ¿mecanismos íntimamente ligados en el desarrollo del proceso infeccioso?
- •
En el futuro, los microorganismos que provoquen patología infecciosa ¿serán cada vez más resistentes a los antimicrobianos y a su vez más virulentos?
La adquisición horizontal de mecanismos de resistencia está comúnmente asociada a un coste biológico y una pérdida de fitness2. Funciones o estructuras importantes para las células se ven implicadas en los procesos de resistencia a los antimicrobianos, como son el reciclaje de la pared celular, la modificación de proteínas diana esenciales, la disminución de porinas (canales de entrada de diferentes moléculas, como solutos o agua), el aumento de sistemas de expulsión activa, etc. Se entiende así que generalmente la ganancia de resistencia conlleve una disminución del fitness bacteriano, asociado a menudo a una disminución de la virulencia2,14,17. Existen múltiples estudios, revisados por Andersson et al.2, que demuestran in vitro e in vivo el coste biológico de la adquisición de mecanismos de resistencia a múltiples familias de antimicrobianos. Sin embargo, y especialmente en ambientes con una fuerte presión antimicrobiana, es relativamente fácil para las bacterias desarrollar mutaciones cromosómicas compensatorias que aumenten el fitness, manteniendo e incluso incrementando la resistencia a los antibióticos previamente adquirida2. Por tanto, el coste de la adquisición de la resistencia a los antimicrobianos será muy variable en función de las condiciones ambientales, la especie o el propio mecanismo de resistencia adquirido. Un ejemplo donde la ganancia de resistencia provoca una ganancia de la virulencia se da en Escherichia coli. En esta especie son frecuentes las mutaciones en los genes parC y gyrA (implicados en la replicación del ADN y en la transcripción del ARN) como mecanismo de resistencia a las fluorquinolonas. A menudo estas mutaciones puntuales conllevan una bajada del fitness de estos patógenos. Por el contrario, se han obtenido mutantes de laboratorio en los que la adición de una sola mutación en parC sobre una cepa resistente a fluorquinolonas por mutaciones en gyrA y marR y con un fitness disminuido aumenta significativamente tanto la resistencia a estos antibióticos como su fitness, lo que podría aumentar las posibilidades de dispersión de cepas resistentes aun en ausencia de antibióticos18. Es importante también el ambiente en el que se encuentran las bacterias a la hora de analizar el coste sobre el fitness. Así, en el caso de bacterias capaces de sobrevivir en diferentes condiciones ambientales, podrían adquirir o desarrollar mecanismos de resistencia en un ambiente determinado, disminuyendo así su fitness, y posteriormente experimentar una serie de mutaciones seleccionadas para compensar esa pérdida de fitness; pero estas mutaciones, beneficiosas en ese ambiente, pueden ser perjudiciales y suponer una pérdida de fitness todavía mayor en otro ambiente diferente19.
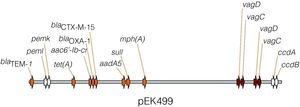
Diseminación horizontal, fenómenos de coseleccciónPlásmidosProbablemente el mecanismo más importante de diseminación de genes de virulencia y de resistencia antibiótica es la transmisión mediada por plásmidos. Estos son elementos extracromosómicos no esenciales para la bacteria que a menudo portan genes que confieren ciertas características a las bacterias como son resistencia, virulencia, persistencia en condiciones extremas, etc. A menudo presentan una gran facilidad de conjugación entre especies, lo que les adjudica un importante papel en la evolución de las bacterias por su fácil transmisión tanto horizontal como vertical20. Un ejemplo de ello son los plásmidos IncF, ampliamente distribuidos en enterobacterias, que portan genes de resistencia a múltiples familias de antibióticos, así como factores de virulencia como hemolisinas, invasinas o toxinas. Un ejemplo claro del aumento de virulencia y resistencia de manera conjunta tiene lugar cuando ese aumento de virulencia es dependiente de la adquisición y distribución de plásmidos portadores de genes de resistencia. Destaca el ampliamente distribuido clon ST131 de E.coli como un claro representante de este fenómeno. Su distribución a nivel mundial fue descrita en 200821, y retrospectivamente se ha visto que ha pasado de ser un clon puntualmente aislado a principios de los 2000, a ser posiblemente el clon resistente a antibióticos más ampliamente distribuido, actualmente con una alta prevalencia a nivel mundial (el 30-60% de los aislamientos resistentes a fluorquinolonas pertenecen a este clon). Aunque la explicación de su éxito no está clara, parece que los plásmidos del grupo IncFII, como el pEK499 (fig. 1), portadores a menudo de β-lactamasas CTX-M, están directamente relacionados. Entre los mecanismos de resistencia que se encuentran codificados en estos plásmidos se encuentran además otras β-lactamasas, genes de resistencia a aminoglucósidos, a tetraciclina, a trimetoprim, a fluorquinolonas, etc.21,22. Entre los genes de virulencia codificados en estos plásmidos se encuentran por ejemplo los sistemas toxina-antitoxina pemI-pemK o ccdA-ccdB, o los genes vagC-vagD relacionados con virulencia, posiblemente implicados en la división celular y necesarios para mantener la virulencia en cepas de Klebsiella pneumoniae portadoras de plásmidos con estos genes13,20. El hecho de portar sistemas de toxina-antitoxina favorece por sí mismo el mantenimiento del plásmido, incluso en ausencia de presión antibiótica. Aunque existe una gran diversidad en la estructura de estos plásmidos y en la evolución de los mecanismos de resistencia que ha experimentado el clon ST131, es básico el papel de estos plásmidos portadores de genes de resistencia en la rápida expansión de estos virulentos clones de E.coli en la actualidad13,23,24.
Representación parcial del plásmido pEK499, formado por la fusión de dos plásmidos de los grupos FII y FIA. Porta hasta 10 genes que confieren resistencia a 8 clases de antibióticos, entre ellos, las β-lactamasas blaTEM-1, blaCTX-M-15 y blaOXA-1; genes de resistencia aminoglucósidos, aac6-Ib-cr; macrólidos, mph(A); cloranfenicol, catB4; tetraciclina, tet(A); estreptomicina, aadA5 y sulfonamida, sulI. Posee dos copias del sistema vagD-vagC, asociado a virulencia, y los sistemas toxina-antitoxina, pemI-pemK y ccdA-ccdB, implicados en el mantenimiento del plásmido, mediante el proceso de muerte post-segregación13.
No solo la presión antibiótica fomenta la dispersión de plásmidos con marcadores de resistencia y a menudo también de virulencia, sino que también determinados ambientes bacterianos fomentan la aparición y distribución de estos mecanismos por localizarse en ellos una enorme diversidad biológica, con su correspondiente carga genética. El plásmido pRSB107 de 120 megabases fue localizado en bacterias de los lodos de una planta de tratamiento de aguas residuales; en cepas transformantes de E.coli expresaba resistencia a al menos 10 antimicrobianos diferentes, y al mismo tiempo codificaba cuatro factores de virulencia, dos sistemas de captación de hierro, un sistema de transporte de glicerol-fosfato y los genes vagCD asociados a virulencia20, siendo así el plásmido pRSB107 una especie de quimera formada a partir de diferentes plásmidos provenientes de múltiples ambientes adquiridos mediante transmisión horizontal.
El hecho de portar plásmidos que codifiquen a la vez para determinantes de resistencia y virulencia capacita a las bacterias en condiciones de presión antibiótica a seleccionar las virulentas, y en condiciones de infección, a seleccionar las resistentes a los antimicrobianos4. La distribución de este tipo de plásmidos entre los patógenos humanos habituales puede ser en un futuro cercano un importante problema de salud.
Otro elemento que asocia virulencia y resistencia en los plásmidos son los genes codificantes de la lipoproteína de membrana externa TraT, implicada en la conjugación de los plásmidos, y en diversos mecanismos de virulencia como son la resistencia al suero, fagocitosis o formación de biofilms. Los plásmidos conjugativos juegan de esta manera un doble papel importante, por extender fácilmente diferentes mecanismos de resistencia y por la propia implicación directa de los genes traT sobre la virulencia de la bacteria4,25.
Elementos genéticos movilizables y captadores de genes: transposones, integrones, islas…Los elementos transponibles son moléculas sencillas de ADN que se encuentran siempre localizadas en otra molécula de ADN mayor con capacidad replicativa, como pueden ser plásmidos o cromosomas. Son elementos genéticos con capacidad para escindirse e integrarse en diferentes regiones del ADN celular, y aunque el ADN de algunos fagos también tiene la capacidad de moverse sobre el ADN de la célula hospedadora, la mayoría de los episodios relacionados con la movilización en el genoma de factores de virulencia o resistencia son debidos a los transposones o integrones. Estos elementos son capaces de moverse gracias al proceso de transposición, mediante la acción de una transposasa o una integrasa, respectivamente. Suelen además portar genes extra, lo que les confiere una gran importancia desde un punto de vista evolutivo.
Los integrones son sistemas muy importantes a la hora de captar genes de resistencia. Se encuentran a menudo formados por casetes de genes colocados en tándem, y la expresión de todos ellos se regula por un potente promotor26. Existe poca bibliografía hasta el momento que recoja información de genes de virulencia asociados a integrones, pero teóricamente son un sistema que podría también captar este tipo de genes y dispersarlos de manera conjunta con los genes de resistencia antibiótica. En un ejemplo descrito recientemente se ha visto que la expresión de una pequeña molécula de ARN (small RNA), la sRNA-Xcc1, se encuentra positivamente regulada por HrpG y HrpX, reguladores de virulencia, y esta a su vez se encuentra posiblemente implicada en la virulencia de Xanthomonas campestris mediante la regulación del QS. Este small RNA se encuentra codificado dentro de un integrón por el que fue capturado y posteriormente distribuido horizontalmente entre diferentes especies mediante transposones y plásmidos27.
Otras estructuras genéticas que pueden ayudar a co-seleccionar mecanismos de virulencia y resistencia son las islas genéticas, regiones hot spot, con una alta tasa de genes con funciones similares, como pueden ser islas de patogenicidad o islas de resistencia. Se ha visto muy recientemente en Streptococcus suis, un agente causante de zoonosis en cerdos jóvenes, un elemento integrativo conjugativo (ICE), que se trata de una isla de patogenicidad de89 kb sin capacidad de movilización pero sí de captación de genes, que además de portar diversos mecanismos de resistencia a antibióticos, como aminoglucósidos o tetraciclina, portan una proteína de membrana LPXTG, un factor de virulencia que facilita la unión de la bacteria a la célula eucariota, localizado también en Streptoccocus agalactiae12,28. En A.baumannii se ha visto, mediante secuenciación del genoma entero de la cepa ATCC 17978, la existencia de hasta 28 posibles islas alien (islas formadas por genes obtenidos de otras especies), en 7 de las cuales se expresan, basándose en sus secuencias, genes de resistencia y genes con posible implicación en la virulencia de esta especie29,30.
En la isla de patogenicidad 2 de Salmonella se encuentra codificado el operón OmpR-EnvZ, implicado en la regulación de resistencia y virulencia a la vez. Este sistema responde a las condiciones ambientales modificando la expresión de OmpC y OmpF, porinas implicadas en la resistencia a antibióticos en esta especie. Por otro lado, el sistema OmpR-EnvZ regula también otro sistema codificado en la misma isla de patogenicidad, el operón SsrA-SsrB, que a su vez regula el sistema de secreción tipo III, necesario para la replicación de la bacteria en el interior de los macrófagos y para la infección en ratones. Mutantes de OmpR son incapaces de replicar en el interior celular de los macrófagos, disminuyendo su virulencia en ausencia de la función de este operón31,32.
Mecanismos comunes implicados en virulencia bacteriana y resistencia a los antimicrobianosEn este apartado analizaremos los sistemas básicos para el desarrollo de un fenotipo resistente que pueden estar implicados a su vez en el desarrollo o en la pérdida de un fenotipo virulento.
BiofilmsEn algunos casos la formación de biofilms bacterianos presenta un efecto beneficioso para el hospedador, ya que ciertos biofilms tienen un papel protector dentro de nuestro organismo. Por ejemplo, las comunidades de lactobacilos presentes en la vagina fermentan el glucógeno producido por las células epiteliales (al ser inducidas por los estrógenos), produciendo ácidos que disminuyen el pH vaginal y previenen de esa manera la colonización por microorganismos patógenos. La desaparición de este biofilm, con la consiguiente neutralización del pH, favorece el desarrollo de infecciones por Gardnerella vaginalis y otros microorganismos anaerobios.
Sin embargo, las bacterias con capacidad de formar biofilms están asociadas en muchos casos a infecciones crónicas, así como a infecciones en catéteres y prótesis. Diversos estudios han analizado que los biofilms bacterianos son más resistentes a los antimicrobianos que las bacterias planctónicas33. Varios son los factores que determinan dicha resistencia:
- •
La barrera de difusión física y química a la penetración de los antimicrobianos que constituye la matriz extracelular del biofilm.
- •
El crecimiento ralentizado de las bacterias del biofilm, debido a la limitación de nutrientes y de oxígeno, lleva a un cambio de la actividad metabólica que puede hacer ineficaz la acción del antimicrobiano.
- •
Las concentraciones subinhibitorias de los aminoglucósidos inducen la formación de biofilms en P.aeruginosa y E.coli.
Se ha confirmado que la expresión de un gen denominado regulador que responde a aminoglucósidos (arr) es fundamental para la inducción de resistencia a esta clase de antibióticos y, además, para el desarrollo de biofilms, lo que indica que este proceso puede representar un mecanismo de reacción defensiva a la presencia de agentes antimicrobianos33. En general, se puede decir que los mecanismos asociados a la resistencia a antimicrobianos en biofilms se deben a procesos multifactoriales y que varían según el microorganismo6. Un problema adicional de la práctica clínica relacionado con la resistencia de los biofilms a los antimicrobianos es la ausencia de métodos estandarizados de uso rutinario para determinar la sensibilidad de las bacterias de un biofilm. Por todo ello, se han desarrollado varios sistemas para comparar la resistencia a los antimicrobianos de las bacterias que crecen formando biofilms, aunque en la actualidad no se ha adoptado ningún protocolo estándar para este fin34,35.
Bombas de expulsiónLos sistemas de bombeo múltiple de drogas o «bombas de expulsión» (MDR, multidrug resistance) se caracterizaron inicialmente como mecanismos de resistencia antimicrobiana, aunque comienzan a ser relacionados con otras funciones en la bacteria, entre ellas la resistencia a los biocidas36 y su implicación en la virulencia, favoreciendo el desarrollo de la infección. Usualmente, estos sistemas suelen estar reprimidos o con bajos niveles de expresión, pero en respuesta a determinadas condiciones del medio ambiente se produce una desrepresión génica de estos mecanismos.
Los sistemas MDR presentan un importante papel en la señalización intercelular y en el proceso de infección y colonización mediante la expulsión de compuestos tales como las sales biliares, los ácidos grasos y los péptidos antimicrobianos catiónicos. En relación con la señalización intercelular, destacamos los estudios realizados en P.aeruginosa. Esta bacteria presenta más de 10 sistemas MDR, alguno de ellos como MexGHI-OpmD, necesarios para la síntesis de moléculas inductoras del QS como acil-homoserina-lactonas (acil-HSL) o 2-heptil-3-hidroxi-4(1H)-quinolonas (PSQ), responsables del inicio de la transcripción de un elevado número de genes implicados en virulencia37; mutantes de MexI u OpmD presentan una inhibición de los genes responsables de la síntesis de PSQ y una inhibición de la virulencia, la resistencia y la comunicación célula a célula. En una situación normal, las bacterias disparan la producción de factores de virulencia cuando se sobrepasa una concentración umbral de señales QS. Sin embargo, existen trabajos en los que se analiza que la hiperexpresión de sistemas MDR como MexAB-OprM y MexCD-OprJ en P.aeruginosa provoca la expulsión no regulada de señales activadoras de QS entre otros metabolitos hacia el exterior de la célula, impidiendo así la acumulación intracelular de estas señales y, por tanto, que se alcance la concentración umbral necesaria para activar el QS. De esta manera, la hiperexpresión de los sistemas de bombeo puede tener también una función inhibitoria de la virulencia o el fitness38,39.
Otro sistema MDR implicado en virulencia, resistencia a antimicrobianos y respuesta al estrés oxidativo es el operón Mar (multiple antibiotic resistance) en cepas de E.coli7. Por otro lado, hay que destacar dos sistemas MDR: el AcrAB, relacionado con la resistencia a sales biliares en enterobacterias y, por tanto, en la virulencia de la bacteria8, y el sistema MDR CmeABC, que determina la capacidad infectiva de Campylobacter jejuni. Finalmente cabe destacar los sistemas MDR MtrCDE y FarAB, que expulsan ácidos grasos y péptidos antimicrobianos y son necesarios para la colonización de mucosas por parte de Nesseria gonorrhoeae40.
PorinasLa resistencia a los antibióticos carbapenémicos en E.coli se relaciona con la disminución de la expresión de las porinas OmpF y OmpC en asociación con la expresión de β-lactamasas41. Recientemente, en un análisis proteómico, se ha estudiado la implicación de las proteínas de membrana externa TolC y OmpC con la resistencia al ácido nalidíxico42. En relación con virulencia, en el E.coli LF82 la OmpC se ha relacionado con la adhesión, la invasión celular y la colonización intestinal en pacientes con enfermedad de Crohn43. Por otro lado, son diversos los trabajos que confirman la ausencia de participación de la ompF en la adhesión a las células Hep-244.
En otros microorganismos como Shigella flexneri, la resistencia a β-lactámicos se asoció con alteraciones en las porinas ompC y ompF9. Además, Berdardini et al. investigaron en 1993 el papel de OmpC en la virulencia de esta especie. En dicho estudio trabajaron con mutantes del gen ompC que presentaron una disminución de la virulencia, en relación con la cepa salvaje. Asimismo, mutantes de ompF no mostraron efecto alguno en la capacidad de invasión de la bacteria tanto in vivo como in vitro al compararla con la cepa silvestre45. Finalmente, la introducción de un plásmido multicopia conteniendo el gen ompC clonado de E.coli bastó para que se recobrara la virulencia, lo que indica que esta proteína es suficiente y necesaria para producir virulencia en un mutante de S.flexneri45.
En Salmonella enterica se ha estudiado la interacción de proteínas periplásmicas (Pp) con porinas. Conviene destacar tres ejemplos; en primer lugar, la Pp Ydel/OmdA, que actúa sobre la porina OmpD, incrementando la difusión al exterior de péptidos antimicrobianos como la catelicidina46. En segundo lugar, la Pp YgiW, que al actuar sobre la OmpF se asocia con resistencia a la polimixina B46. Y finalmente la proteína Pp YdeI, que ha sido implicada en la persistencia y la supervivencia del patógeno S.enterica en el tracto gastrointestinal del ratón47. Vemos, por tanto, que dichas proteínas periplásmicas son un claro ejemplo de mecanismo de conexión entre virulencia y resistencia.
Otro ejemplo de enterobacterias en las que las porinas juegan un importante papel tanto en resistencia como en virulencia es K.pneumoniae. La deleción conjunta de las proteínas ompK35 y ompK36 provocó la resistencia a cefalosporinas y carbapenemes, así como un descenso en la virulencia de dicho patógeno en un modelo de ratón10.
En último lugar, hay que destacar el patógeno nosocomial A.baumannii. Dos son las principales porinas implicadas en la resistencia a carbapenemes: la Omp33-36 y la CarO48,49. La Omp33-36 ha sido asociada con muerte celular mediante apoptosis a través de experimentos en líneas celulares (48th ICCAC and 46th IDSA: Interscience Conference on Antimicrobial Agents and Chemotherapy, C1-1054). Sería este un ejemplo con efectos opuestos en el binomio resistencia-virulencia: la ausencia de la porina conllevaría aumento de la resistencia a los carbapenémicos concomitante a la reducción en la virulencia. Recientemente, Islam et al.50 han determinado que dicha porina presenta una respuesta antigénica específica para A.baumannii y es un buen marcador para el desarrollo de un test diagnóstico de infecciones por este patógeno.
Alteraciones en la pared celularEn las modificaciones en la permeabilidad de la membrana externa, tanto porinas como sistemas de bombeo múltiple modulan diferentes procesos relacionados con la colonización, la virulencia y la capacidad bacteriana de adaptación al huésped. En un estudio sobre la implicación de la permeabilidad en la resistencia y virulencia en Enterobacter aerogenes se observan cambios en ella tras el tratamiento antibiótico. En un primer estadio el aislamiento presenta resistencia a β-lactámicos y a otras familias de antibióticos, principalmente a fluorquinolonas, debido a la hiperexpresión de sistemas de bombeo múltiple (MDR) y a mutaciones en la topoisomerasa. En un segundo estadio la cepa de E.aerogenes presenta menor susceptibilidad al imipenem, con un importante descenso en la expresión de porinas junto con la hiperexpresión de sistema MDR. En un tercer y último estadio, el aislamiento presenta alta resistencia al imipenem y a la polimixina en respuesta a la acción conjunta de la disminución en la expresión de las dos principales porinas, la hiperexpresión de MDR y la alteración del lipopolisacárido (LPS). Estas importantes modificaciones en la permeabilidad de la bacteria provocan una disminución de la supervivencia y del fitness bacteriano51.
Modificaciones del LPS de la membrana externa están también relacionadas con el aumento de resistencia e implicadas también en un aumento de virulencia que se produce en A.baumannii. Una de las principales características de este patógeno es la plasticidad que presenta a la hora de modificar su background genético, mediante mutaciones o adquisición horizontal, para adaptarse al ambiente que le rodea. Aunque todavía son esporádicas, desde la reintroducción de la colistina como tratamiento en este tipo de infecciones en la última década han surgido cepas resistentes a este antibiótico, tanto in vitro52,53 como en infecciones clínicas54,55; incluso se han descrito fenómenos de selección de clones resistentes in vivo durante el tratamiento con colistina56,57. Hasta el momento se han descrito dos mecanismos de resistencia a la colistina en A.baumannii. El primero, la pérdida total de LPS debida a un déficit en la biosíntesis de lípido A por inactivación de uno de los tres genes implicados, lpxA, lpxC o lpxD58. En el segundo mecanismo conocido hasta la fecha, estudiado por nuestro grupo, la modificación del lípido A mediada por la adición de fosfoetanolamina da lugar a la pérdida de la afinidad por la colistina y la subsiguiente resistencia; estas modificaciones están reguladas en este caso por el sistema de dos componentes pmrAB52. El LPS y el lípido A de A.baumannii se encuentran implicados en diversas actividades biológicas como son toxicidad, pirogenicidad, mitogenicidad y activación de la respuesta proinflamatoria del sistema inmune. Por esto es de suponer que cambios importantes en el LPS o en sus componentes tengan una relación directa sobre la virulencia de la bacteria.
Recientemente, en mutantes de laboratorio resistentes a la colistina debido a mutaciones en pmrB se ha demostrado la pérdida de parte de su fitness y de su virulencia, tanto in vitro como in vivo53,55, lo que sugiere que la modificación o pérdida de LPS pueden aumentar la resistencia a la colistina, pero a costa de disminuir la virulencia en esta especie. Aunque se ha observado la pérdida de capacidad infectiva en una cepa con elevada resistencia a la colistina seleccionada durante un tratamiento con este antimicrobiano57, sigue siendo esta una cuestión sujeta a debate. También se ha encontrado un clon en España resistente a la colistina seleccionado durante el tratamiento y con su capacidad infectiva intacta, aunque con un perfil de resistencias antimicrobianas alterado, volviéndose sensible a antimicrobianos como la cefepima o el sulbactam (de manera inversa al clon original, colistina-sensible y cefepima/sulbactam-resistente). Similares resultados se han observado también en otros estudios58. Parece haber por tanto una relación inversa entre la ganancia de resistencia a la colistina en A.baumannii mediada por cambios en el LPS y la virulencia de la especie, o al menos en la pérdida de resistencia frente a otros antimicrobianos, lo cual puede considerarse en cierto modo, por ejemplo en un ambiente con alta presión antibiótica, como una pérdida de virulencia.
P.aeruginosa, caracterizada por presentar también altas tasas de multirresistencia, porta al igual que muchos de los patógenos gramnegativos una β-lactamasa AmpC. En condiciones normales la expresión de esta β-lactamasa se encuentra a niveles basales, pero es fácil el aumento de expresión en presencia de antibióticos inductores como la cefoxitina o el imipenem. La expresión de AmpC se encuentra regulada por diversos genes implicados en el reciclaje de peptidoglucano de la pared bacteriana, entre los que se encuentra AmpD. La inactivación mediante mutación del gen ampD da lugar a una desrepresión de la β-lactamasa AmpC, y este es el mecanismo de resistencia a β-lactámicos habitualmente presente en esta especie59,60. Sin embargo, la desrepresión de AmpC debido a inactivaciones en ampD es una desrepresión parcial, y esto se debe a la existencia de dos genes homólogos a ampD: ampDh2 y ampDh3. Cuando estos 3 genes son inactivados en mutantes de laboratorio se observa una activación de la expresión mucho mayor (1.000 veces superior a la cepa salvaje). El coste de fitness en términos de virulencia es mínimo cuando se inactiva el gen ampD y se hiperexpresa la β-lactamasa AmpC, pero se produce una pérdida total de la virulencia cuando los tres genes son inactivados, pérdida debida a la incapacidad de la bacteria para reciclar el peptidoglucano61. Se presenta de esta manera un modelo en P.aeruginosa donde resistencia y virulencia se encuentran cromosómicamente regulados, donde la ganancia efectiva de resistencia no supone una pérdida de patogenicidad, debido a la presencia de varios genes homólogos implicados en la regulación de la β-lactamasa cromosómica y el mantenimiento de la pared celular.
Sistemas de dos componentesLos sistemas reguladores de dos componentes, sensor-receptor (SDC, two component systems), son mecanismos reguladores fundamentales, capaces de controlar múltiples funciones celulares entre las que se encuentran la expresión de factores de virulencia y la sensibilidad antibiótica. Los sistemas reguladores de dos componentes están compuestos por una proteína histidina quinasa, anclada en la membrana citoplasmática, cuya función es captar los estímulos ambientales y activar el segundo componente, una proteína reguladora de respuesta citoplasmática, que controla la expresión génica por unión a las regiones promotoras62.
En P.aeruginosa recientemente se ha descrito que el sistema de dos componentes CbrA-CbrB, implicado en principio en la utilización metabólica del carbono y nitrógeno, se encuentra asimismo implicado en diversos mecanismos de resistencia y virulencia. Se mostró que este sistema tiene la capacidad de regular la expresión de otros sistemas de dos componentes, como PmrA-PmrB o PhoP-PhoQ (también implicados en la regulación de la resistencia y la virulencia). La inactivación del sensor CbrA provocó, entre otros fenotipos, un aumento de la citotoxicidad de la cepa mutante y una mayor resistencia a diferentes familias de antibióticos, como la colistina, el ciprofloxacino y la tobramicina11. En este mismo patógeno, el sistema PhoP-PhoQ incrementa la resistencia a la colistina mediante la inducción de cambios del LPS63, pero además se ha visto recientemente que se encuentra también directamente implicado en la regulación de la virulencia, observándose en un mutante de PhoP una disminución de la formación de biofilm, de la citotoxicidad y de la virulencia en dos modelos in vivo, así como su importancia en la resistencia a péptidos antimicrobianos64. El sistema PmrA-PmrB en A.baumannii, como ya hemos comentado anteriormente, es otro ejemplo de cómo estos sistemas se encuentran implicados, regulando, entre otros, la virulencia y la resistencia a la colistina.
DiscusiónTras los ejemplos aquí descritos deberían tenerse en cuenta una serie de reflexiones:
Resistencia antibiótica y virulencia: ¿mecanismos íntimamente ligados en el desarrollo del proceso infeccioso?Es necesario desarrollar más estudios que permitan conocer mejor la implicación de ambos mecanismos. Tras hacer esta revisión, podríamos establecer que a medida que se investigan estos procesos de manera conjunta, se detecta mayor vinculación. Dicha asociación es en determinadas ocasiones positiva o de potenciación para uno o ambos mecanismos, o por el contrario, en otros casos, es una vinculación con efecto negativo o de disminución de uno sobre otro4.
¿Efecto de la adquisición de resistencia sobre la virulencia en una especie patógena?Parece claro que el efecto positivo o negativo de la asociación entre la resistencia a los antimicrobianos y la virulencia bacteriana depende de múltiples factores como el ambiente, la especie bacteriana y el mecanismo implicado. La presencia o ausencia de antibióticos en un nicho determinado generará una enorme presión, dirigiendo la selección hacia el éxito de los patógenos más resistentes; por el contrario, durante la colonización y la infección serán los patógenos más virulentos los que tengan mayores posibilidades de éxito y, por tanto, de subsistir. Así, en un ambiente nosocomial es fácil pensar que las poblaciones más resistentes y a la vez más virulentas sean las que tengan un mayor porcentaje de éxito; sin embargo, en ambientes sin una presión selectiva directa posiblemente la ganancia de virulencia o de resistencia tenga un efecto negativo en esta asociación. Por otro lado, la especie bacteriana y el propio mecanismo implicado son imprescindibles a la hora de valorar el efecto de la ganancia de resistencia sobre la virulencia. La variabilidad de los mecanismos de resistencia que es capaz de desarrollar una especie y el efecto positivo o negativo que pueda ejercer sobre la virulencia hacen que apenas se pueda generalizar dentro del mundo microbiano y que cada especie/mecanismo implicado deba ser estudiado de una manera muy específica.
Perspectivas de la asociación entre virulencia y resistencia en el futuroMecanismos comunes capaces de seleccionar resistencia y virulenciaLa utilización de antibióticos ha dado lugar a una selección de las cepas resistentes; así, cuando estos mecanismos de resistencia son codificados desde elementos genéticos como plásmidos, transposones, islas, etc., capaces de diseminar también genes de virulencia, de manera colateral se seleccionarán asimismo cepas virulentas. Además, a menudo portan resistencias a diferentes familias de antibióticos, lo cual supone que la no utilización de una familia de antibióticos no provocaría la disminución o la desaparición de las cepas resistentes a esa familia. El hecho de portar genes de virulencia permite además tener cierta ventaja evolutiva durante los procesos de colonización e infección, seleccionando así las cepas resistentes. Esta plasticidad permite a los patógenos disponer de nuevas estrategias para explorar nuevos ambientes en los cuales la flora normal estaría en desventaja. La coevolución de la virulencia y la resistencia en este tipo de clusters podría dar lugar en un futuro cercano a una explosión de patógenos altamente virulentos y resistentes. Un claro ejemplo sucede con la virulenta cepa de E.coli ST13123, cuyo éxito en su amplia y rápida distribución está relacionado directamente con plásmidos IncFII o IncFI, entre otros; al ser en muchos casos resistente a la gran mayoría de antibióticos, es en la actualidad un importante problema de salud pública.
Mutaciones compensatorias del fitness en cepas resistentes y que, por tanto, permanecerán en el tiempoEs relativamente fácil para las bacterias restaurar eficazmente el coste de una bajada de fitness debida a la adquisición de un nuevo mecanismo de resistencia; mediante mutaciones compensatorias los patógenos resistentes son además capaces de mantener esa resistencia tras recuperar o incluso aumentar su fitness. Por tanto, la evolución compensatoria podría estabilizar las poblaciones resistentes, incluso en ausencia de antibióticos, al competir con el resto de la flora comensal en igualdad de condiciones. Esta serie de compensaciones suelen seguir una de estas tres estrategias: a) utilizar una vía alternativa para realizar la misma función; b) reducir la necesidad de llevar a cabo esa función, y c) restaurar la eficacia de esa función. Todavía no está claro el papel que estas compensaciones juegan en la estabilidad de las poblaciones bacterianas resistentes fuera del laboratorio, pero existen estudios que han señalado este hecho en el ambiente clínico, lo cual puede suponer un problema creciente en los próximos años con los actuales tratamientos utilizados2.
En conclusión, la interrelación entre la resistencia a los antibióticos y la virulencia en una población bacteriana concreta va a depender de una compleja red de interacciones, siendo el impacto final dependiente principalmente de la especie bacteriana concreta, del tipo de mecanismo específico, de la resistencia al antimicrobiano y del medio ambiente en el que se encuentre.
FinanciaciónEste trabajo ha sido financiado por el Ministerio de Sanidad y Consumo, Instituto de Salud Carlos III, a través de la Red de Investigación en Patología Infecciosa (REIPI RD 06/0008/0025) y el Fondo de Investigaciones Sanitarias (PI08/1368, PS09/00687 y PI10/00056), SERGAS (PS07/90) y Xunta de Galicia (07CSA050916PR).
Conflicto de interesesA. Beceiro posee un contrato Ángeles Alvariño de la Consellería de Economía e Industria, Xunta de Galicia. M. del Mar Tomás posee un contrato Miguel Servet del Instituto de Salud Carlos III, Ministerio de Sanidad y Consumo. Los autores declaran que no existe ningún conflicto de intereses.