se considera éxito terapéutico cuando un paciente con el virus de la inmunodeficiencia humana (VIH) sometido a tratamiento antirretroviral de gran actividad (TARGA) consigue una carga viral plasmática<50copias/ml. Sin embargo, es frecuente encontrar a pacientes cuya carga viral se sitúa en 50–1.000copias/ml, en estos casos las guías de tratamiento no recomiendan realizar un test genotípico de resistencias aduciendo una baja rentabilidad. El objetivo principal de este estudio fue examinar la utilidad de una técnica de concentración para la secuenciación de VIH-1 desde muestras que contienen menos de 1.000copias/ml, y a partir de aquí determinar las consecuencias virológicas de los cambios guiados por el test de resistencia genotípica del TARGA de estos pacientes.
Métodosse estudió a 51 pacientes con cargas virales de 50–1.000copias/ml, de los que 27 presentaron estos valores de carga viral durante al menos 12 meses consecutivos. Antes de la extracción del ARN, se concentró una muestra de 3ml de plasma y, a continuación, se genotipó siguiendo el procedimiento estándar.
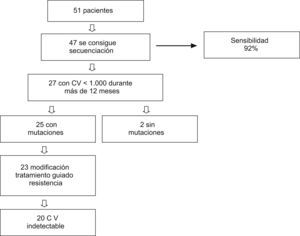
Resultadosse pudo secuenciar 47 muestras de las 51, con una sensibilidad del 92%. De estos 47 pacientes, 27 mantienen carga viral de 50–1.000copias/ml durante 12 meses, de ellos, 20 consiguen carga viral indetectable al cambiar el TARGA guiado por el genotipo (análisis por intención de tratar; no consta=fracaso; 20 de 27 [74,1%]) (análisis en tratamiento; 20 de 23 [86,9%]).
Conclusionesmostramos una modificación sencilla de la secuenciación genotípica en pacientes con grados de viremia persistentemente bajos, que resulta en una modificación en el régimen del TARGA que consigue una viremia plasmática indetectable.
Highly active antiretroviral therapy (HAART) in HIV patients is considered successful when plasma viral load (VL) reaches<50copies/ml. However, many patients have a persistent VL of 50 to 1000copies/ml, and treatment guidelines do not recommend genotypic resistance testing at these levels because of poor performance. The aim of this study was to evaluate the usefulness of a concentration technique for HIV-1 sequencing in samples with<1000copies/ml, and determine the virological consequences of HAART treatment changes guided by resistance testing in this scenario.
MethodsObservational study performed in 51 patients with plasma VL between 50 and 1000copies/m; 27 patients had these levels for at least 12 consecutive months. Prior to RNA extraction, virions were concentrated from 3-ml plasma samples and then genotyped following standard procedures.
ResultsForty-seven of the 51 samples were successfully sequenced, resulting in a sensitivity of 92%. Among these 47 patients, 27 showed a persistent viral load of 50–1000copies/ml for 12 months, and 20 patients achieved undetectable viral load following the genotype-guided HAART change (intention-to-treat analysis: NC=F; 20 of 27 [74.1%]; on-treatment analysis: 20 of 23 [86.9%]).
ConclusionsWe report a simple method for genotype sequencing in patients with persistent low-level viremia that allowed a modification of the HAART regimen leading to undetectable plasma viremia.
De acuerdo con las guías de tratamiento antirretroviral, se considera éxito terapéutico en pacientes con el virus de la inmunodeficiencia humana (VIH) a la supresión de la carga viral por debajo de las 50copias/ml. Sin embargo, la completa erradicación viral no es posible debido a la existencia de reservorios de larga vida, como los linfocitos T CD4+ memoria infectados, que poseen el genoma del VIH integrado.
Una de las principales causas del fracaso del tratamiento antirretroviral (TAR) es la aparición de resistencias a fármacos1. Varios estudios han demostrado que los test de resistencia genotípica mejoran la respuesta virológica en pacientes cuyo TAR ha de ser modificado tras el fracaso2,3. La evidencia obtenida de estos estudios apoya la recomendación actual del uso del test de resistencia para todos los pacientes que que no responden al tratamiento, para los que se plantea un cambio de terapia4,5.
Aunque la resistencia a los fármacos antirretrovirales puede desarrollarse con valores bajos de carga viral, las técnicas para su detección no están recomendadas cuando ésta es<1000copias/ml por la alta frecuencia de fallo en la secuenciación6. Esto complica el correcto mantenimiento clínico de los pacientes con carga viral detectable, pero por debajo de este umbral. Sin embargo, se ha publicado recientemente que es posible detectar mutaciones relacionadas con resistencia a fármacos antirretrovirales de pacientes con carga viral baja7–10, aunque la relevancia clínica de estos hallazgos aún no ha sido establecida con claridad.
El objetivo principal de este estudio fue examinar la utilidad de una técnica de concentración para la secuenciación de VIH-1 desde muestras de plasma que contienen menos de 1.000copias/ml, y a partir de aquí determinar las consecuencias virológicas de los cambios guiados por el test de resistencia genotípica del tratamiento antirretroviral de gran actividad (TARGA) de estos pacientes.
Pacientes y métodosSe realizó un estudio observacional con 51 pacientes con VIH-1, del subtipo B, con una carga viral baja, de dos centros que atienden a pacientes con VIH en Granada (Hospital Universitario San Cecilio y Hospital Universitario Virgen de las Nieves), con los siguientes criterios de inclusión: a) infección crónica por VIH-1 documentada; b) TARGA en régimen estable durante al menos 12 meses, y c) dos cargas virales detectables consecutivas pero por debajo de 1.000copias/ml al mismo tiempo que reciben TARGA. El período de estudio comprendió los años 2002–2005. Para evaluar las consecuencias virológicas de los cambios guiados por el test genotípico, seleccionamos a pacientes con carga viral<1.000copias/ml, pero detectable durante un período de, al menos, 12 meses. De este modo, descartamos los casos que desarrollaron un repunte transitorio de carga viral. De los 51 pacientes, sólo 27 cumplían dicho criterio. En estos pacientes, después de ese período, se realizó un cambio de tratamiento y se estudió la repercusión del cambio en la evolución virológica e inmunológica (cambios en carga viral y CD4 a los 3 y 6 meses).
Los análisis para la detección del ARN del VIH-1 se realizaron con el estudio Ultrasensitive de Roche Amplicor Monitor System v1.5 (Roche Diagnostics, Barcelona), cuyo límite inferior de detección es de 50copias/ml. La técnica de genotipificación se realizó con el sistema TruGene HIV-1 Genotyping Kit (Siemens, Barcelona). Antes de la extracción de ARN, se procedió a la concentración de las muestras de plasma de acuerdo con el siguiente protocolo: las muestras fueron sometidas a tres ciclos de ultracentrifugación en Biofuge Stratos (Heraeus, Barcelona). Una alícuota inicial de 1ml de plasma se ultracentrifuga a 23.600g durante 1h a 4°C; se desecha el sobrenadante y otro ml de plasma se añade al pellet en el mismo tubo y se repite la centrifugación; después se desecha el sobrenadante, se añade otro ml, y se somete a una tercera centrifugación; finalmente, se desechan 860μl del sobrenadante y el pellet se resuspende intensamente con el remanente de 140μl, que se somete a la extracción con QUIAMP Viral RNA mini-kit (Qiagen, Barcelona). El eluido de ARN se empleó directamente para el test con Trugene HIV-1 genotyping Kit (Siemens, Barcelona), con las precauciones estándar para evitar contaminaciones cruzadas por PCR. Además, para excluir la contaminación cruzada, todas las secuencias se analizaron con la herramienta genetic fingerprinting del software de Trugene (Siemens, Barcelona) para explorar las coincidencias entre secuencias de diferentes pacientes. Esta herramienta enfrenta la secuencia de estudio (con una longitud de unos 900 pares de bases) a todas las ya existentes en la base de datos, de modo que diferencias entre secuencias por debajo de 20 pares de bases permiten establecer una elevada homología entre ellas.
Las secuencias se analizan con TruGene HIV-1 Report Generator, con Guidelines 7.0 (Bayer, Barcelona) y Retrogram v 1.6 (www.retrogram.com), para conocer las mutaciones de resistencia y su interpretación.
La eficacia del cambio de TARGA guiado por genotipo, definido como la proporción de pacientes que consiguen carga viral indetectable, se midió con un análisis por intención de tratar (ITT; NC=F [no consta=fracaso]) y un análisis en tratamiento (OT).
ResultadosPara valorar la sensibilidad de nuestro método de concentración, se seleccionó a 51 pacientes con carga viral<1.000copias/ml. Se pudo secuenciar 47 de las 51 muestras, con una sensibilidad del 92%. Las cuatro muestras que no se pudo secuenciar tenían una carga viral<200copias/ml. De los 51 pacientes incluidos inicialmente, 27 (53%) mantuvieron una carga viral de 50-1.000copias/ml durante 12 meses, y se seleccionaron para análisis posteriores (fig. 1).
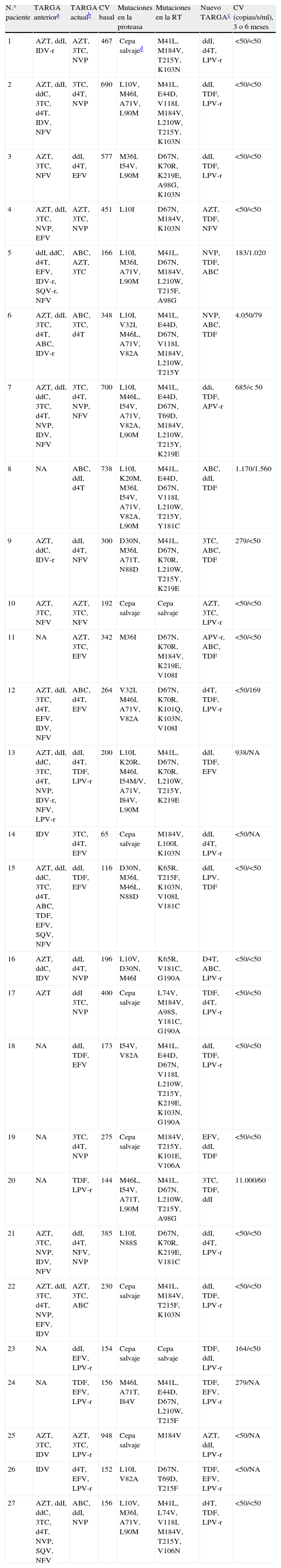
De estos 27 pacientes, la mayoría eran varones (66,7%) con una media de edad de 42 años. La mediana de carga viral basal fue 264 (intervalo, 65–948)copias/ml, y la mediana de recuento de linfocitos CD4 fue 327 (intervalo, 72–844) células/μl. El 48,1% de estos pacientes recibían, al comienzo del estudio, tratamiento con un régimen con análogos de nucleósidos/nucleótidos y no análogos; el 18,5%, con análogos e inhibidores de la proteasa; otro 18,5% recibe tratamiento con los tres grupos antirretrovirales, y el 14,8% restante, sólo con análogos. Las mutaciones de resistencia en el gen de la transcriptasa inversa (RT) y/o proteasa se detectaron en 25 (93%) pacientes. Las mutaciones detectadas en la RT fueron: M41L (48,1%), E44D (22,2%), K65R (7,4%), D67N (55,5%), T69D (7,4%), K70R (22,2%), L74V (7,4%), V118I (18,5%), M184V (48,1%), L210W (37%), T215Y/F (59,2%), K219Q/E (22,2%), A98G (11,1%), L100I (3,7%), K101E/Q (11,1%), K103N (40,7%), V106A (7,4%), V108I (11,1%), Y181C (22,2%), G190A (11,1%), y en la proteasa: L10I/V (44,4%), K20M/R (7,4%), D30N (7,4%), M36I (25,9%), M46I/L (33,3%), I54M/V (25,9%), A71V/T (40,7%), V82A (22,2%), I84V (7,4%), N88D/S (11,1%), L90M (29,6%). La genotipificación mostró sensibilidad a algunos de los fármacos antirretrovirales incluidos en el régimen terapéutico que recibían los pacientes en 10 (37%) de los 27 casos, actividad disminuida en 7 (26%) y resistencia a todos los fármacos del régimen en 8 (30%). El patrón de resistencias se muestra en la tabla 1, junto con los regímenes previos, los cambios de tratamiento y la evolución virológica e inmunológica.
TARGA, mutaciones de resistencia, interpretación de la resistencia y modificaciones al TARGA de la cohorte estudiada
| N.° paciente | TARGA anteriora | TARGA actualb | CV basal | Mutaciones en la proteasa | Mutaciones en la RT | Nuevo TARGAc | CV (copias/s/ml), 3 o 6 meses |
| 1 | AZT, ddI, IDV-r | AZT, 3TC, NVP | 467 | Cepa salvajed | M41L, M184V, T215Y, K103N | ddI, d4T, LPV-r | <50/<50 |
| 2 | AZT, ddI, ddC, 3TC, d4T, IDV, NFV | 3TC, d4T, NVP | 690 | L10V, M46I, A71V, L90M | M41L, E44D, V118I, M184V, L210W, T215Y, K103N | ddI, TDF, LPV-r | <50/<50 |
| 3 | AZT, 3TC, NFV | ddI, d4T, EFV | 577 | M36I, I54V, L90M | D67N, K70R, K219E, A98G, K103N | ddI, TDF, LPV-r | <50/<50 |
| 4 | AZT, ddI, 3TC, NVP, EFV | AZT, 3TC, NVP | 451 | L10I | D67N, M184V, K103N | AZT, TDF, NFV | <50/<50 |
| 5 | ddI, ddC, d4T, EFV, IDV-r, SQV-r, NFV | ABC, AZT, 3TC | 166 | L10I, M36I, A71V, L90M | M41L, D67N, M184V, L210W, T215F, A98G | NVP, TDF, ABC | 183/1.020 |
| 6 | AZT, ddI, 3TC, d4T, ABC, IDV-r | ABC, 3TC, d4T | 348 | L10I, V32I, M46L, A71V, V82A | M41L, E44D, D67N, V118I, M184V, L210W, T215Y | NVP, ABC, TDF | 4.050/79 |
| 7 | AZT, ddI, ddC, 3TC, d4T, NVP, IDV, NFV | 3TC, d4T, NVP, NFV | 700 | L10I, M46L, I54V, A71V, V82A, L90M | M41L, E44D, D67N, T69D, M184V, L210W, T215Y, K219E | ddi, TDF, APV-r | 685/< 50 |
| 8 | NA | ABC, ddI, d4T | 738 | L10I, K20M, M36I, I54V, A71V, V82A, L90M | M41L, E44D, D67N, V118I, L210W, T215Y, Y181C | ABC, ddI, TDF | 1.170/1.560 |
| 9 | AZT, ddC, IDV-r | ddI, d4T, NFV | 300 | D30N, M36I, A71T, N88D | M41L, D67N, K70R, L210W, T215Y, K219E | 3TC, ABC, TDF | 279/<50 |
| 10 | AZT, 3TC, NFV | AZT, 3TC, NFV | 192 | Cepa salvaje | Cepa salvaje | AZT, 3TC, LPV-r | <50/<50 |
| 11 | NA | AZT, 3TC, EFV | 342 | M36I | D67N, K70R, M184V, K219E, V108I | APV-r, ABC, TDF | <50/<50 |
| 12 | AZT, ddI, 3TC, d4T, EFV, IDV, NFV | ABC, d4T, EFV | 264 | V32I, M46I, A71V, V82A | D67N, K70R, K101Q, K103N, V108I | d4T, TDF, LPV-r | <50/169 |
| 13 | AZT, ddI, ddC, 3TC, d4T, NVP, IDV-r, NFV, LPV-r | ddI, d4T, TDF, LPV-r | 200 | L10I, K20R, M46I, I54M/V, A71V, I84V, L90M | M41L, D67N, K70R, L210W, T215Y, K219E | ddI, TDF, EFV | 938/NA |
| 14 | IDV | 3TC, d4T, EFV | 65 | Cepa salvaje | M184V, L100I, K103N | ddI, d4T, LPV-r | <50/NA |
| 15 | AZT, ddI, ddC, 3TC, d4T, ABC, TDF, EFV, SQV, NFV | ddI, TDF, EFV | 116 | D30N, M36I, M46L, N88D | K65R, T215F, K103N, V108I, V181C | ddI, LPV, TDF | <50/<50 |
| 16 | AZT, ddC, IDV | ddI, d4T, NVP | 196 | L10V, D30N, M46I | K65R, V181C, G190A | D4T, ABC, LPV-r | <50/<50 |
| 17 | AZT | ddI 3TC, NVP | 400 | Cepa salvaje | L74V, M184V, A98S, Y181C, G190A | TDF, d4T, LPV-r | <50/<50 |
| 18 | NA | ddI, TDF, EFV | 173 | I54V, V82A | M41L, E44D, D67N, V118I, L210W, T215Y, K219E, K103N, G190A | ddI, TDF, LPV-r | <50/<50 |
| 19 | NA | 3TC, d4T, NVP | 275 | Cepa salvaje | M184V, T215Y, K101E, V106A | EFV, ddI, TDF | <50/<50 |
| 20 | NA | TDF, LPV-r | 144 | M46L, I54V, A71T, L90M | M41L, D67N, L210W, T215Y, A98G | 3TC, TDF, ddI | 11.000/60 |
| 21 | AZT, 3TC, NVP, IDV, NFV | ddI, d4T, NFV, NVP | 385 | L10I, N88S | D67N, K70R, K219E, V181C | ddI, d4T, LPV-r | <50/<50 |
| 22 | AZT, ddI, 3TC, d4T, NVP, EFV, IDV | AZT, 3TC, ABC | 230 | Cepa salvaje | M41L, M184V, T215F, K103N | ddI, TDF, LPV-r | <50/<50 |
| 23 | NA | ddI, EFV, LPV-r | 154 | Cepa salvaje | Cepa salvaje | TDF, ddI, LPV-r | 164/<50 |
| 24 | NA | TDF, EFV, LPV-r | 156 | M46I, A71T, I84V | M41L, E44D, D67N, L210W, T215F | TDF, EFV, LPV-r | 279/NA |
| 25 | AZT, 3TC, IDV | AZT, 3TC, LPV-r | 948 | Cepa salvaje | M184V | AZT, ddI, LPV-r | <50/NA |
| 26 | IDV | d4T, EFV, LPV-r | 152 | L10I, V82A | D67N, T69D, T215F | TDF, EFV, LPV-r | <50/NA |
| 27 | AZT, ddI, ddC, 3TC, d4T, NVP, SQV, NFV | ABC, ddI, NVP | 156 | L10V, M36I, A71V, L90M | M41L, L74V, V118I, M184V, T215Y, V106N | d4T, TDF, LPV-r | <50/<50 |
3TC: lamivudina; ABC: abacavir; AZT: zidovudina; CV: carga viral; d4T: estavudina; ddC: zalcitabina; ddI: didanosina; EFV: efavirenz; IDV: indinavir; NA: no disponible; NFV: nelfinavir; NVP: nevirapina; RT: transcriptasa inversa; SQV: saquinavir; TARGA: tratamiento antirretroviral de gran actividad; TDF: tenofovir.
Se realizó una modificación del TARGA en 23 de los 27 pacientes. En 6 el cambio en el tratamiento fue sólo de un fármaco; en 8, de 2 fármacos; en 8, de tres y en un paciente se modificó un régimen con cuatro fármacos por otro con tres distintos. Hubo un paciente que no aceptó la modificación del tratamiento por lo que continuó con el mismo. Para 3 pacientes no había fármacos activos disponibles, por lo que se realizó el cambio de terapia empíricamente, en ninguno de ellos se obtuvo respuesta terapéutica.
En 23 pacientes se realizó la modificación guiada por el genotipo. Después de que el TARGA fuese modificado, los valores de carga viral a los 3 meses fueron indetectables en 19 de 27 (70,4%) pacientes en el análisis ITT, y en 19 de 23 (82,6%) en el análisis OT. A los 6 meses, había 5 pacientes de los que carecíamos del valor de carga viral, por lo tanto, los resultados fueron: 17 de 27 (63%), ITT; 17 de 18 (94,4%), OT. Los resultados que obtenemos, independientemente de si la carga viral indetectable se obtuvo a los 3 o a los 6 meses, cuando analizamos el porcentaje de pacientes que logra la indetectabilidad, fueron 20 de 27 (74,1%) pacientes en el análisis ITT, y 20 de 23 (86,9%) en el análisis OT. Los hallazgos inmunológicos de los pacientes en que fue posible la modificación guiada por el genotipo fueron como sigue: mediana de recuento de linfocitos CD4 basal de 327 (intervalo, 72-823) células/μl; en el tercer mes, 356 (intervalo, 62-882), y a los 6 meses, 386 (intervalo, 120-797). Estos cambios no fueron significativos.
DiscusiónLos pacientes con cargas virales plasmáticas de 51–1.000copias/ml durante al menos 3 meses se consideran con viremias persistentemente bajas. Diferente es el concepto de blips, que define los casos con repuntes de carga viral de 50–1.000copias/ml, precedida y seguida por otra medida<50copias/s/ml. Los repuntes transitorios de carga viral durante el TARGA pueden observarse en hasta el 25–53% de los pacientes11,12. Se ha descrito la relación entre los pacientes con carga viral persistentemente baja y una mayor probabilidad de fracaso virológico. Los virus plasmáticos que causan estos episodios durante el TARGA parecen originarse desde dos procesos distintos: a) producción de partículas virales a partir de células de larga vida infectadas de meses a años antes del inicio del TARGA, y b) a partir de una escasa replicación con selección de nuevas mutantes13. Tobin et al13 demuestran que las secuencias de los virus plasmáticos y las secuencias procedentes de las células memoria (polimorfonucleares) son las mismas. Por lo tanto, estos episodios transitorios de viremia se relacionan con un incremento de la replicación de virus competente en reservorios celulares, este pool de virus que se replica potencia la falta de respuesta del fármaco mediante mutaciones que permiten incrementar el fitness y la resistencia al fármaco. Si se han seleccionado virus resistentes con una terapia no supresiva anterior, habrá producción continua de estos virus resistentes, aunque el paciente haya modificado su tratamiento. Identificar estas mutaciones puede ser útil en la elección de fármacos antirretrovirales14–16. Sin embargo, también se ha descrito que la producción de escasos virus puede ocurrir durante varios años sin seleccionar mutaciones de resistencia a ninguno de los fármacos del régimen16.
Por razones técnicas, los sistemas genotípicos comerciales no se recomiendan para pacientes que tengan resistencia terapéutica, pero que tienen una carga viral<1.000copias/s/ml6. Estas recomendaciones se basan en el hecho de que el éxito de las técnicas con muestras con carga viral baja es pequeño y sobre la creencia de que la amplificación de muestras con carga viral baja podría permitir seleccionar cepas no representativas motivando cambios prematuros en el tratamiento antirretroviral. Esta última razón, sin embargo, nunca ha sido apoyada por datos científicos17. Por contraste, las actuales guías de tratamiento indican que el TARGA debería cambiarse después de dos cargas virales consecutivas de VIH>400copias/s/ml; la razón para este cambio es intentar reducir la posibilidad de desarrollo de resistencias farmacológicas al régimen que fracasa. Una publicación de la cohorte de EuroSIDA de Cozzi-Lepri18 ha demostrado que en el 77% de los pacientes con carga viral baja y con un régimen estable que no responde aparece al menos una nueva mutación de resistencia en un segundo genotipo. Estos datos son similares a los de la cohorte Scope19 y, por lo tanto, el retraso hasta conseguir una carga viral suficientemente alta puede resultar en una evolución adicional del patrón de resistencias7, especialmente en los pacientes que han recibido más de dos o tres líneas de tratamiento20. Gunthard et al15 demuestran que las resistencias pueden desarrollarse cuando la viremia está en el intervalo de 20-400. Si bien, Napravnik et al21 afirman que hay una mayor probabilidad de desarrollar resistencia con carga viral entre 3 y 4 logaritmos. La importancia de la detección de resistencias a un fármaco en pacientes con viremia baja es crucial, ya que un cambio terapéutico oportuno podría evitar continuar con un régimen que no responde cuando la resistencia es todavía limitada y un cambio puede ayudar a preservar futuras opciones de tratamiento y evitar resistencias cruzadas, además de prevenir el desarrollo de toxicidades. Por lo tanto, es obvia la necesidad de genotipificación con carga viral baja. Algunos autores7,8 han publicado que es técnicamente factible realizar la secuención de muestras con carga viral baja, pero la relevancia clínica de estos hallazgos todavía es controvertida. Recientemente, Gale et al9 han presentado datos de cómo diluyendo 20 muestras para conseguir cargas virales<1.000copias/s/ml (100 a 500copias/s/ml) logran genotipificarlas con éxito usando TruGene Bayer Assay y con un paso previo de centrifugación. Waters et al10, mediante el secuenciador ABI 3730XL, obtienen una amplificación que varía del 70 al 92%, según el valor de la carga viral de partida, que varía de 50 a 1.000copias/s/ml.
Nuestro trabajo aporta un método fácil para concentrar viriones de muestras de plasma, con una sensibilidad excelente para pacientes con carga viral<1.000copias/s/ml. La única modificación a las recomendaciones del protocolo del fabricante es una ultracentrifugación de las muestras y el empleo de un mayor volumen de muestra. En nuestro caso, lo hacemos en tres pasos, pero si se dispone de una ultracentífuga que permita el empleo de volúmenes mayores, entonces se podrá realizar en un único paso. En contraste con otros estudios8, no clonamos el producto de la PCR, ya que la población secuenciada podría reflejar mejor el trabajo de rutina que la clonación. La población que estudiamos mostraba una gran prevalencia de mutaciones de resistencia (93%), probablemente porque estaba compuesta principalmente por pacientes con amplia experiencia antirretroviral, el 70% de los pacientes no había respondido a dos o más regímenes antirretrovirales.
La carga viral persistentemente baja puede ser una condición peculiar en la cual su supresión se obtiene más fácilmente, incluso con la adición secuencial de un único fármaco, como han demostrado algunos estudios de intensificación22. Sin embargo, actualmente no está claro que la sustitución puntual del fármaco, o fármacos, para los cuales hay resistencia pueda impedir una nueva evolución del VIH-1, que permita el desarrollo de resistencias cruzadas, e interfiera en futuras opciones terapéuticas. En este contexto, como en nuestros pacientes, incluso con carga viral baja, los ensayos de resistencia son útiles para conseguir carga viral indetectable después de un cambio de TARGA guiado por el genotipo (el 78%, carga viral<50copias/s/ml a los 3 y 6 meses en el análisis ITT). Además, consideramos que, aunque es posible, no es probable que encontremos un resultado falso negativo (ausencia de mutaciones) después de genotipificar la muestra con carga viral baja, ya que, con la modificación que describimos aquí, incrementamos la sensibilidad de los métodos de genotipificación; de hecho, se pudo genotipificar 47/51 muestras con carga viral<1 000copias/s/ml y sólo 2/27 pacientes con bajo grado de viremia persistente tienen virus wild-type. En 14 de los 27 pacientes disponíamos de estudios genotípicos previos o posteriores cuando su carga viral fue >1.000copias/s/ml, en éstos la herramienta genetic fingerprinting da coincidencias entre las distintas cepas del mismo paciente. Esta herramienta enfrenta la secuencia de estudio (con una longitud de unos 900 pares de bases) a todas las ya existentes en la base de datos, de modo que diferencias entre secuencias por debajo de 20 pares de bases permiten establecer una elevada homología entre ellas. En nuestro estudio, en los 14 casos en los que disponíamos de secuencias previas o posteriores, las diferencias entre secuencias nunca superaron el valor de 20 para las que pertenecían a un mismo paciente, lo que se puede interpretar como garantía de que las secuencias obtenidas con ese valor de baja carga viral son correctas. Además, distintos autores ya han demostrado que no hay diferencias entre las mutaciones detectadas cuando se amplifican y secuencian virus de muestras de un mismo paciente diluidas por debajo de 1.000copias/s/ml7,23.
En resumen, un grado de viremia bajo pero detectable mientras se recibe TARGA es suficiente para generar virus con mutaciones de resistencia, lo que permite que la replicación hasta cargas virales que alcancen más de 1.000copias/s/ml puede resultar en una evolución adicional del patrón de resistencias. La realización del test de resistencia con cargas virales bajas es posible empleando un método de ultracentrifugación para concentrar el virus y un mayor volumen de muestra de partida, y en nuestro estudio la posterior modificación del TARGA guiada por el genotipo conllevó una buena respuesta virológica después de 3 o 6 meses. Nuestros resultados, junto con otros7,8, sientan las bases para investigaciones futuras para establecer la recomendación de realizar estudios de resistencia a fármacos en pacientes con carga viral persistentemente baja.
FinanciaciónConsejería de Innovación, Ciencia y Empresa, Junta de Andalucia (CTS213; PO6-CTS-01498), Consejería de Salud, Junta de Andalucía (377/05; PI-0129; PI/0408/08) y Fondo de Investigación Sanitaria (PI051513, ISCIII-RETIC RD06/00006/0016).