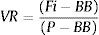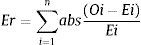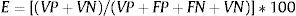La glucoproteína gp36 del VIH2 es muy utilizada en los ELISA. Pretendimos evaluar los índices de diagnóstico de mezclas de antígenos con el péptido sintético del VIH2 gp36 (5).
MétodosSe prepararon 5 mezclas con gp36 (5) y proteínas recombinantes del VIH1/2. Se evaluaron 1.306 muestras con el UMELISA HIV1+2 RECOMBINANT como referencia.
ResultadosLa mezcla (V-1) mostró muy buena concordancia respecto a la referencia.
ConclusiónLa variante V-1 demostró elevada eficacia en el inmunodiagnóstico del VIH1/2.
The HIV-2 glycoprotein 36 (gp36) is often used in ELISA. An evaluation of the diagnostic indexes of antigen mixtures with a synthetic peptide of HIV2 gp36 (5) is performed in this study.
MethodsFive mixtures of gp36 (5) and the recombinant proteins of HIV1/2 were prepared. A total of 1306 samples were evaluated with UMELISA HIV1+2 RECOMBINANT used as reference.
ResultsThe variant (V-1) showed very good agreement as regards the reference method.
ConclusionThe V-1 variant was shown to be highly effective in the immunodiagnosis of HIV 1/2.
Según datos de la Organización Mundial de la Salud, en el mundo existen más de 37 millones de personas infectadas por el virus de la inmunodeficiencia humana tipo1 y 2 (VIH1/2), agentes causales del síndrome de inmunodeficiencia humana adquirida (sida)1.
El rápido progreso de la infección por este virus ha impuesto el desarrollo de pruebas diagnósticas más simples, de bajo costo y de alta sensibilidad y especificidad para la detección de anticuerpos anti-VIH1/2 en muestras de sangre. Entre las más empleadas se encuentran técnicas inmunoenzimáticas de tercera y cuarta generación, como la quimioluminiscencia (ChLIA) y los ELISA2. Los antígenos usados en el recubrimiento de los ELISA pueden ser producto de lisados virales, proteínas recombinantes y péptidos sintéticos, siendo estos últimos ampliamente utilizados en la actualidad, en busca de mayor sensibilidad y especificidad3.
En el caso específico del VIH2, la glucoproteína de transmembrana gp36 es la más utilizada en el serodiagnóstico, y está descrito que péptidos sintéticos de esta glucoproteína muestran una elevada antigenicidad y especificidad en la detección de anticuerpos en sueros de pacientes infectados con VIH24.
Es por ello que en este trabajo nos propusimos como objetivo evaluar los índices diagnósticos: sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo de diferentes mezclas de antígenos, que usan el péptido sintético monomérico del VIH2 gp36 (5).
MétodosSíntesis química, purificación y caracterización del péptido gp36 (5) del VIH-2El péptido gp36 (5) se obtuvo en el Centro de Inmunoensayo (CIE, Cuba) mediante síntesis química en fase sólida, empleando la estrategia Boc en bolsas de polipropileno4.
Proteínas recombinantes de los VIH1/2Las proteínas recombinantes gp120, gp41 y p24 del VIH1, y gp36 del VIH2, fueron suministradas por el Centro de Ingeniería Genética y Biotecnología de Sancti Spiritus (CIGB SS), Cuba.
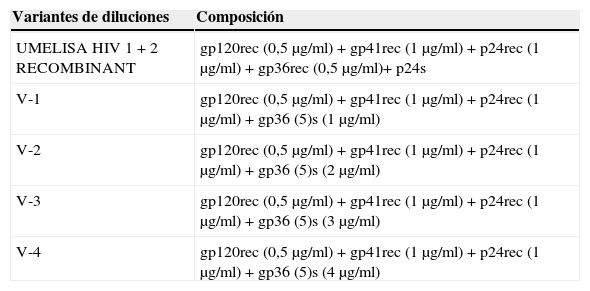
Recubrimiento de las placas y ensayo UltramicroElisa (UMELISA)Con el péptido sintético gp36 (5) y las proteínas recombinantes se prepararon 5 mezclas diferentes a las concentraciones deseadas (tabla 1). Se recubrieron entonces placas de poliestireno de 96 pocillos (placas UMELISA, Greiner Bio-One, Alemania). El ensayo se realizó según instrucciones del fabricante para el estuche UMELISA HIV1+2 RECOMBINANT (TecnoSUMA Internacional S.A.).
Composición de las variantes de mezclas de antígenos empleadas
| Variantes de diluciones | Composición |
|---|---|
| UMELISA HIV1+2 RECOMBINANT | gp120rec (0,5μg/ml)+gp41rec (1μg/ml)+p24rec (1μg/ml)+gp36rec (0,5μg/ml)+ p24s |
| V-1 | gp120rec (0,5μg/ml)+gp41rec (1μg/ml)+p24rec (1μg/ml)+gp36 (5)s (1μg/ml) |
| V-2 | gp120rec (0,5μg/ml)+gp41rec (1μg/ml)+p24rec (1μg/ml)+gp36 (5)s (2μg/ml) |
| V-3 | gp120rec (0,5μg/ml)+gp41rec (1μg/ml)+p24rec (1μg/ml)+gp36 (5)s (3μg/ml) |
| V-4 | gp120rec (0,5μg/ml)+gp41rec (1μg/ml)+p24rec (1μg/ml)+gp36 (5)s (4μg/ml) |
gp: glucoproteína; p: proteína; rec: recombinante; s: sintético.
Para la validación de los resultados se utilizó un programa de computación para trabajo con la tecnología SUMA (Strips Reader Software SRS V. 9.0.). El nivel de corte (NC) utilizado en el procesamiento de los datos fue de 0,27, valor de corte del estuche UMELISA HIV1+2 RECOMBINANT (TecnoSUMA Internacional S.A.) usado como referencia en el estudio.
El valor de relación (VR) se definió como:
donde Fi: fluorescencia de la muestra; BB: valor promedio del blanco; P: menor valor de fluorescencia de los duplicados del control positivo.
Aquellas muestras cuya media de los duplicados tuvieron un VR/NC≥1 fueron consideradas reactivas, y las clasificadas como repetir fueron revaluadas hasta alcanzar un resultado preciso.
Muestras de sueroSe emplearon 10 paneles de sueros.
Los paneles 1-7 incluyeron todas las muestras positivas al VIH1 y/o 2 (174) y 9 negativas. Del panel 8-10 se encontraron las restantes muestras negativas (1.123), incluyendo algunas reactivas al virus de la hepatitisB (VHB [n=30]), virus de la hepatitis C (VHC [n=32]), virus linfotrópico de célulasT humanas (HTLV [n=10]), virus dengue (n=10), Mycobacterium leprae (n=3) y Trypanosoma cruzi (n=5), además de 10 muestras hemolíticas.
Análisis estadístico de los resultadosCon el apoyo del programa EPIDAT 3.1 se calcularon los índices diagnósticos de sensibilidad, especificidad y valores predictivos positivos y negativos. Con el programa estadístico Tonystat se analizó el ajuste a la distribución normal de los resultados de las muestras no reactivas de los paneles 1-7. Se seleccionó la mejor de las variantes de antígenos con el apoyo del error relativo, calculado como:
donde O: valor observado; E: valor esperado.
Además, se calculó la eficacia:
donde VP: verdaderos positivos; VN: verdaderos negativos; FP: falsos positivos; FN: falsos negativos.
Mediante el índice kappa (k) se analizó la concordancia entre UMELISA HIV1+2 RECOMBINANT y la más idónea de las variantes de antígenos.
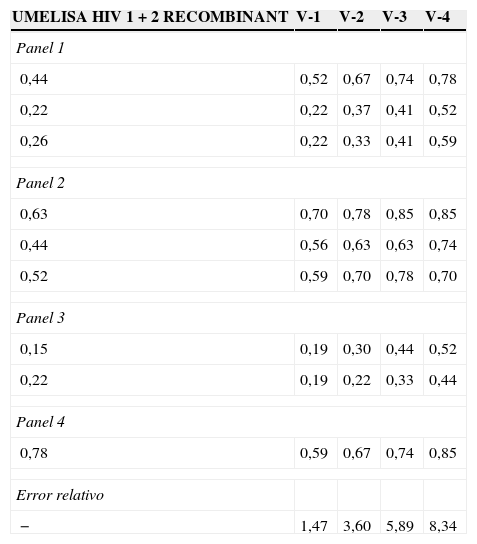
ResultadosAl analizar los paneles 1-7, todas las variantes de mezclas de antígenos detectaron la totalidad de las muestras reactivas, para un 100% de sensibilidad. Así también, obtuvieron valores de VR/NC<1 ante las muestras negativas incluidas en dichos paneles (tabla 2). Sin embargo, la variante1 tuvo mayor similitud con UMELISA HIV1+2 RECOMBINANT, al presentar el menor valor de error relativo (tabla 2). Por tal motivo se eligió solo esta mezcla para continuar el estudio.
Resultados y error relativo de las variantes de antígenos frente a las muestras negativas para VIH (paneles 1-7)
| UMELISA HIV 1+2 RECOMBINANT | V-1 | V-2 | V-3 | V-4 |
|---|---|---|---|---|
| Panel 1 | ||||
| 0,44 | 0,52 | 0,67 | 0,74 | 0,78 |
| 0,22 | 0,22 | 0,37 | 0,41 | 0,52 |
| 0,26 | 0,22 | 0,33 | 0,41 | 0,59 |
| Panel 2 | ||||
| 0,63 | 0,70 | 0,78 | 0,85 | 0,85 |
| 0,44 | 0,56 | 0,63 | 0,63 | 0,74 |
| 0,52 | 0,59 | 0,70 | 0,78 | 0,70 |
| Panel 3 | ||||
| 0,15 | 0,19 | 0,30 | 0,44 | 0,52 |
| 0,22 | 0,19 | 0,22 | 0,33 | 0,44 |
| Panel 4 | ||||
| 0,78 | 0,59 | 0,67 | 0,74 | 0,85 |
| Error relativo | ||||
| − | 1,47 | 3,60 | 5,89 | 8,34 |
Los valores de las muestras se expresan como (VR/NC), y son consideradas positivas cuando VR/NC≥1.
Con el objetivo de evaluar la especificidad de la variante de antígenos V-1, se emplearon las muestras descritas en los paneles 8-10. Para todas ellas, la mezcla de antígenos V-1 mostró un 100% de especificidad.
Después se realizó el cálculo de los valores predictivos negativos y positivos, y de la eficiencia, teniendo en cuenta los resultados de todos los paneles. Tanto para V-1 como para la referencia se obtuvo un 100% en todos los parámetros. Finalmente el índice kappa evidenció una concordancia muy buena (igual a 1) entre V-1 y UMELISA HIV1+2 RECOMBINANT.
DiscusiónEl uso de péptidos sintéticos en la fase sólida de los ELISA ha posibilitado aumentar su sensibilidad y su especificidad5. Por tanto, el empleo del antígeno sintético gp36 (5) del VIH2 con proteínas recombinantes del VIH1 podría ser eficaz en el diagnóstico del VIH1/2.
La elevada sensibilidad de las variantes de antígenos reflejó su valía para detectar anticuerpos en sueros positivos al VIH. Sobre todo el péptido sintético gp36 (5), que permitió el reconocimiento de las 26 muestras positivas al VIH2 empleadas, coincidiendo con otros autores, que informaron alta sensibilidad y especificidad para este péptido de forma independiente4,6.
La variante V-1 evidenció, además, la alta especificidad de los antígenos utilizados en el ensayo, sobre todo porque otros autores han descrito homología de secuencias entre el VIH y el HTLV7 y discrepancia en la interferencia de muestras hemolíticas en los inmunoensayos8. Resultados similares han sido descritos ante sueros positivos a los agentes estudiados en este trabajo9,10. No obstante, es necesario ampliar la población de este tipo de muestras que esclarezcan la interferencia de las mismas en nuestro ensayo.
Además, la reducción del tiempo de obtención, la disminución del peligro de contaminación durante el proceso de síntesis, la alta reproducibilidad y la elevada pureza de los péptidos sintéticos con respecto a las proteínas recombinantes9 validan las ventajas que tendría el uso del péptido gp36 (5) en el ensayo. También dotaría al Centro de Inmunoensayo de independencia productiva, ya que la proteína recombinante gp36 del UMELISA HIV1+2 RECOMBINANT es adquirida de manera comercial.
De forma general se evaluaron diferentes variantes de mezclas de antígenos que incluyeron un péptido sintético de la proteína de transmembrana del VIH2 [gp36 (5)] las cuales posibilitaron la correcta identificación de todas las muestras positivas analizadas. De las variantes probadas, V-1 presentó el menor error relativo, asemejándose más al ensayo de referencia UMELISA HIV1+2 RECOMBINANT. Las altas sensibilidad y especificidad de la variante V-1 demostró su eficacia en el inmunodiagnóstico de los sueros reactivos al VIH1 y 2 utilizados.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Queremos hacer notar la labor decisiva para la culminación de este trabajo de la MSc. Idialis Hernández Spengler, la Lic. Dunia Clara Bequer Ariza y la MSc. Aurora Delahanty Fernández, todas del Centro de Inmunoensayo. Gracias por sus aportes.