El objetivo del presente trabajo fue demostrar la utilidad de un algoritmo de identificación de micobacterias no tuberculosas (MNT) que integra diferentes técnicas de biología molecular y características fenotípicas básicas. Además se ha realizado una actualización del algoritmo de interpretación del análisis del patrón de restrición de hsp65 (PRA hsp65).
MétodosLa manera elegida de trabajar consistió en la identificación mediante hibridación con sondas de ADN seguido de PRA hsp65 en aquellos aislados que no pudieron ser identificados mediante hibridación con sondas de ADN. En caso necesario se realizó secuenciación del 16S rDNA y hsp65.
ResultadosSe aislaron 236 MNT. De ellos, 102 (43,2%) aislados fueron identificados mediante hibridación con sondas de ADN y 76 (32,2%) mediante PRA hsp65. En los 58 (24,5%) aislados restantes se secuenció 16S rDNA, lo cual permitió la identificación de 53 (22,4%). Para 5 (2,1%) aislados se secuenció hsp65 y permitió la identificación de un aislado más. Cuatro (1,7%) aislados no pudieron ser identificados. Tres nuevos patrones de PRA hsp65 fueron encontrados. Siete aislamientos hibridaron con la sonda AccuProbe Mycobacterium avium complex Identification pero no lo hicieron con las sondas específicas de especie incluidas en el MAC. Cinco y 2 aislados fueron identificados como M. intracellulare y Mycobacterium colombiense, respectivamente.
ConclusiónEste esquema de trabajo nos permitió la identificación de casi todas las MNT encontradas en este estudio, incluyendo especies recientemente descritas.
The aim of the present work was to demonstrate the utility of a non-tuberculous mycobacteria (NTM) identification algorithm, which integrates different PCR-based techniques and basic phenotypic features. Moreover, the algorithm for pattern restriction analysis of hsp65 (hsp65 PRA) interpretation has been updated.
MethodsThe workflow chosen consisted of the identification by a DNA hybridization probe method, followed by PCR-restriction enzyme analysis of hsp65 (hsp65 PRA) in those isolates that cannot be identified by hybridization probes. If necessary, 16S rRNA gene and hsp65 gene sequencing were used for speciation.
ResultsA total of 236 NTM were collected, in which 102 (43.2%) isolates were identified by DNA specific probes and 76 (32.2%) isolates were identified with hsp65 PRA. Partial sequencing of the 16S rRNA gene was used for species identification of the remaining 58 (24.5%) isolates. Fifty-three (22.4%) were identified using this method. Five isolates (2.1%) were submitted for partial sequencing of hsp65 gene and one isolate was identified with this method. Four strains (1.7%) could not be identified at species level. Three new PRA patterns were found. Seven isolates tested positive with the AccuProbe Mycobacterium avium complex identification test but did not test positive with the M. avium or Mycobacterium intracellulare specific probes. Five and two of these isolates were identified as M. intracellulare and Mycobacterium colombiense, respectively.
ConclusionThis approach allowed us to identify almost all NTM isolates found in this study, including some recently described species.
Las micobacterias no tuberculosas (MNT) cada vez son más reconocidas como patógenas en seres humanos1. Los métodos fenotípicos para su identificación están bien establecidos y son económicos, pero tienen el inconveniente de que son tediosos y requieren personal especializado2. La introducción, en las 2 últimas décadas, de distintas herramientas moleculares ha revolucionado el proceso de identificación al reducirse el tiempo necesario para llevarlo a cabo, además de ser imprescindibles para la caracterización de nuevas especies3. Los tests basados en la hibridación con sondas de ADN (AccuProbe, Gen-Probe) son rápidos, sensibles y fáciles de realizar pero cubren solo pocas especies. En 1989, Hance et al. propusieron un test basado en la amplificación de un fragmento del gen que codifica para la proteína del shock térmico de 65 KDa (hsp65) para detectar e identificar micobacterias a partir de muestras clínicas4. Después de éste, Telenti et al. desarrollaron un método basado en este gen para diferenciar micobacterias mediante el análisis del patrón de restricción (PRA)5. Desde entonces, este método ha sido utilizado por distintos autores6,7, conduciendo a un algoritmo de identificación para 54 especies2. Recientemente han sido descritos patrones adicionales8,9. Los esquemas de trabajo basados en la secuenciación para detectar polimorfismos en el gen que codifica para el ARN ribosomal 16S (16S rDNA) han sido útiles para la identificación de MNT. De cualquier forma, hay poca variabilidad en el 16S rDNA en Mycobacterium spp., lo que hace a esta diana un pobre discriminador para especies muy relacionadas genéticamente como Mycobacterium abcessus y Mycobacterium chelonae10. El hsp65 es más polimórfico que el 16S rDNA. Kapur et al. fueron los primeros en secuenciar hsp65 para identificar micobacterias11. Otros autores han demostrado la utilidad de la secuenciación parcial de hsp65 para identificar MNT12.
El esquema de trabajo elegido en nuestro laboratorio consiste en la identificación de Mycobacterium sp. mediante hibridación con sondas de ADN (AccuProbe), seguido de PRA hsp65 en aquellos aislados que no pueden ser identificados mediante hibridación. Si el patrón obtenido no es concluyente utilizamos la secuenciación parcial de 16S rDNA y hsp65 para llegar a una identificación definitiva.
El motivo del presente trabajo es describir los resultados obtenidos utilizando este algoritmo de identificación durante un periodo de 5 años. También hemos actualizado el algoritmo de identificación mediante hsp65 PRA basado en su secuencia nucleotídica.
MétodosAislados de micobacterias no tuberculosasEn el Laboratorio de Microbiología Clínica del Hospital de la Santa Creu i Sant Pau de Barcelona fueron recuperados 236 aislados clínicos de MNT durante el periodo 2002-2006. Solo se consideró un aislamiento por paciente. Los aislados fueron examinados al microscopio y subcultivados en medio de Löwenstein-Jensen para realizar los tests de velocidad de crecimiento, producción de pigmento y capacidad para crecer a diferentes temperaturas. La identificación mediante hibridación con sondas AccuProbe (Gen-Probe, San Diego, CA, EE. UU.) de Mycobacterium avium complex (MAC), M. avium, Mycobacterium intracellulare, Mycobacterium kansasii y Mycobacterium gordonae se realizó de acuerdo con las instrucciones del fabricante. La sonda AccuProbe (MAC, M. kansasii y M. gordonae) fue elegida en función de las características fenotípicas. En caso de positividad para la sonda específica de MAC se procedió a la hibridación con las sondas específicas de M. avium y M. intracellulare. Este estudio no requirió aprobación por el comité ético del Hospital de la Santa Creu i Sant Pau.
Extracción de ADNEl ADN genómico fue extraído mediante Instagene matrix siguiendo las instrucciones del fabricante (Bio-Rad, Glattbrugg, Suiza).
Análisis del patrón de restricción de hsp65 (hsp65 PRA)Un fragmento de hsp65 fue amplificado y posteriormente digerido con BstEII y HaeIII (Roche Diagnostics GmbH, Mannheim, Alemania) como describió Telenti et al.5 con las siguientes modificaciones: 15μl de la reacción de amplificación fue añadida a una mezcla que contenía 1μl (10 U/μl) de enzima, 3μl de tampón de restricción específico (10X) y 11μl de agua destilada. Las mezclas fueron incubadas durante 120 min a 60°C para la digestión con BstEII y a 37°C para la digestión con HaeIII. Los fragmentos obtenidos en la restricción fueron sometidos a electroforesis y los pesos de las bandas obtenidas fueron analizados con Bio Image System (Millipore, EE. UU.), con Marker VIII (Roche Diagnostics GmbH, Mannheim, Alemania) y un marcador de 25 pares de bases (Invitrogen, Karlsruhe, Alemania) como estándares de peso molecular. En la digestión de HaeIII no se tuvieron en cuenta los fragmentos menores de 50 pb. Los patrones de PRA obtenidos fueron interpretados según el algoritmo publicado por Brunello et al.2. Hemos utilizado este algoritmo debido a que los patrones de PRA obtenidos fueron validados mediante secuenciación.
Amplificación de 16S rDNALa amplificación de 16S rDNA fue realizada utilizando iniciadores universales.
Secuenciación parcial de 16S rDNA (primeros 500 pares de bases) y hsp65Los productos de PCR amplificados fueron purificados con High Pure PCR Product Purification Kit® (Roche Diagnostics GmbH, Mannheim, Alemania) según las instrucciones del fabricante. La secuenciación fue llevada a cabo en ambos sentidos con el método dideóxido con iniciadores fluorescentes (fmol DNA Cycle Sequencing System, Promega, Madison, EE. UU.) utilizando para ello un secuenciador de ADN (ALF, Pharmacia, Upsala, Suecia).
Análisis de las secuenciasLas secuencias nucleotídicas obtenidas fueron analizadas mediante el programa BLAST (www.ncbi.nlm.nhi.gov). Además, las secuencias nucleotídicas de 16S rDNA fueron analizadas con la base de datos de Ribosomal Differentiation of Medical Microorganisms (RIDOM) (http://www.ridom.de). Para los análisis de 16S rDNA y hsp65, la identificación de especie fue confirmada si la secuencia nucleotídica obtenida presentaba una homología ≥ 99,5%13 y ≥ 97%12, respectivamente, con una secuencia depositada en las bases de datos públicas y publicada. El nuevo algoritmo basado en la secuenciación para interpretación del PRA hsp65 fue realizado de la siguiente manera. Los patrones de PRA que no estaban incluidos en el algoritmo publicado por Brunello et al.2 fueron calculados in silico a partir de secuencias de hsp65 depositadas en GenBank con el programa LaserGene (DNASTAR 4.0, Madison, WI, EE. UU.). Los patrones de PRA estimados, basados en datos de la electroforesis, han sido incluidos cuando ninguna secuencia que correspondiera a ese patrón estaba depositada en GenBank. Algunos errores menores y cambios taxonómicos detectados en el algoritmo de Brunello et al.2 fueron corregidos.
Números de acceso de las secuencias depositadas en GenBankLas secuencias de hsp65 únicas determinadas en el presente trabajo han sido depositadas en GenBank con los números de acceso EU732652, EU732653 y EU732654.
ResultadosDurante el periodo de estudio se aislaron 236 MNT. Fenotípicamente, 134 (56,7%) aislados correspondieron a crecedores lentos pigmentados (PSG), 54 (22,8%) a crecedores lentos no pigmentados y 48 (20,3%) a crecedores rápidos. Dentro del grupo de PSG, 100 (74,6%) aislados fueron escotocromógenos y 34 (25,4%) fueron fotocromógenos.
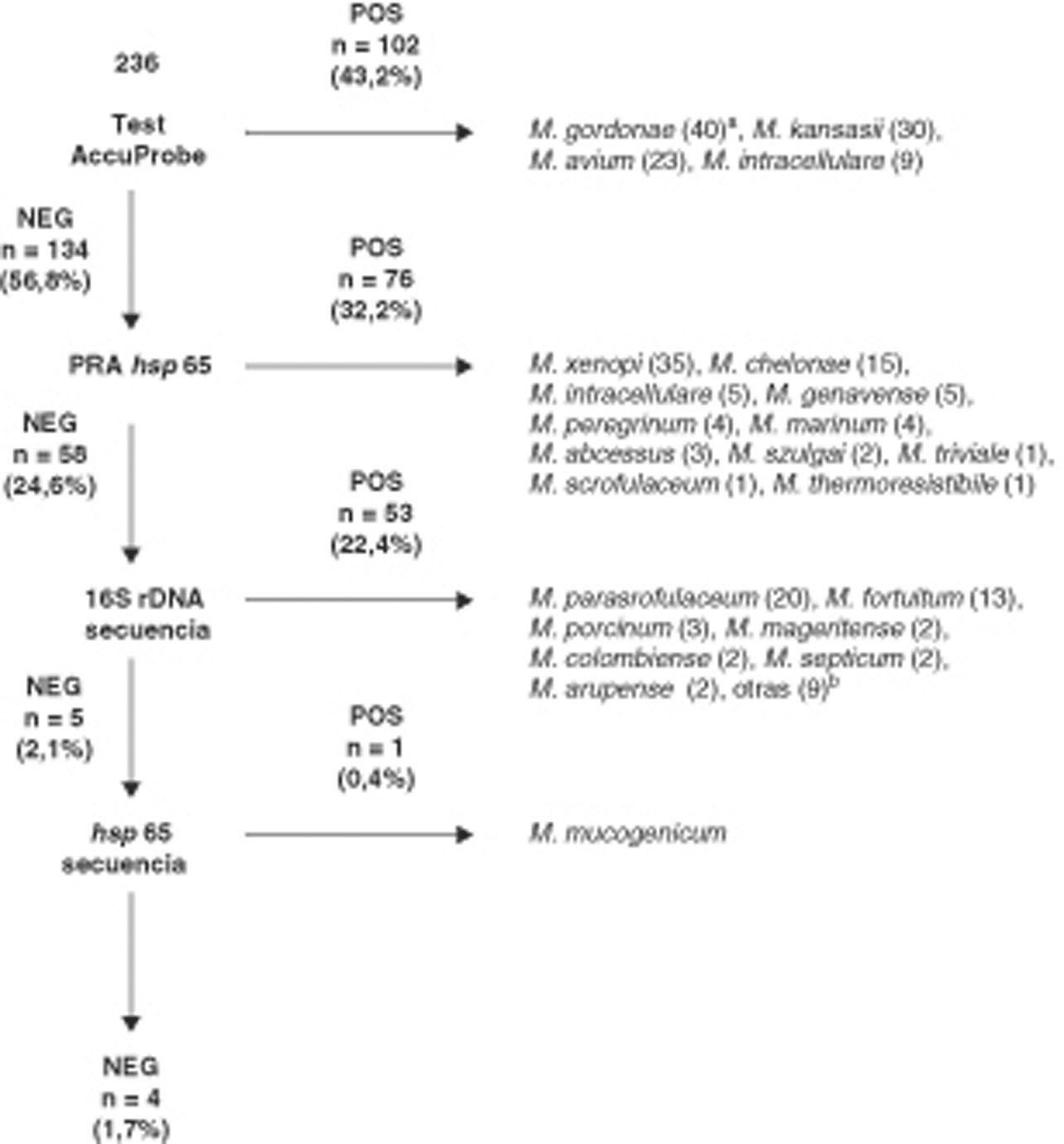
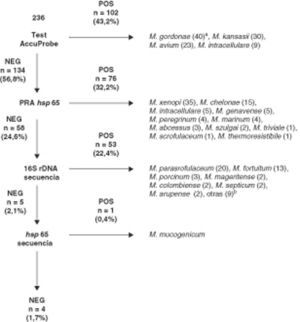
Los resultados obtenidos con nuestro algoritmo de identificación se muestran en la figura 1. Un total de 102 (43,2%) aislados fueron identificados mediante hibridación con sondas de ADN (AccuProbe). En 134 aislados (56.8%) se realizó PRA hsp65. Fueron identificados mediante esta técnica 76 (32,2%) aislados. La secuenciación de 16S rDNA se realizó para la identificación de especie de los 58 (24,5%) aislados restantes. Un total de 53 (22,4%) fueron identificados con este método. En los 5 aislados no identificados mediante secuenciación de 16S rDNA, se realizó secuenciación de hsp65, identificándose un aislado como M. mucogenicum. Finalmente, 4 aislados no pudieron ser identificados y, en función de sus secuencias únicas de 16S rDNA y hsp65, probablemente correspondan a taxones no descritos. No se realizaron pruebas bioquímicas o de HPLC para estos 4 aislados no identificados.
Resultados obtenidos con nuestro algoritmo de identificación de micobacterias no tuberculosas.
aNúmero de aislados.
bM. wolinskyi (1), M. alvei (1), M. lentiflavum (1), M. terrae (1), M. peregrinum (1), M. nonchromogenicum (1), M. celatum (1), M. kumamotonense (1) y M. sherrisii (1).
Un total de 7 aislados presentaban un resultado positivo para AccuProbe M. avium complex Identification Test pero no presentaban un resultado positivo con las sondas AccuProbe específicas de M. avium y M. intracellulare. Cinco de estos aislados fueron identificados mediante PRA como M. intracellulare pero los otros 2 aislados no presentaban un patrón de PRA incluido en el algoritmo de Brunello et al.2. Estos 2 aislados fueron identificados mediante secuenciación de 16S rDNA como Mycobacterium colombiense.
Entre los 58 aislados que no pudieron ser identificados mediante hsp65 PRA, 48 presentaban patrones que no estaban incluidos en el algoritmo propuesto por Brunello et al.2. Estos aislados fueron identificados mediante secuenciación de 16S rDNA como M. parascrofulaceum (n = 20), M. fortuitum (n = 13), M. mageritense (n = 2), M. colombiense (n = 2), M. arupense (n = 2), M. wolinskyi (n = 1), M. alvei (n = 1), M. lentiflavum (n = 1), M. terrae (n = 1), M. peregrinum (n = 1), M. nonchromogenicum (n = 1), M. celatum (n = 1), M. kumamotonense (n = 1) y M. sherrisii (n = 1). Cinco aislados presentaron un patrón de PRA específico de M. porcinum y M. septicum. Esta última especie no está incluida en el algoritmo de Brunello et al. pero es conocido que comparte el patrón de hsp65 PRA con M. porcinum14,15. Tres de estos 5 aislados fueron identificados con secuenciación de 16S rDNA como M. porcinum y 2 aislados como M. septicum.
Los 5 aislados restantes no pudieron ser identificados mediante secuenciación del 16S rDNA. Cuatro aislados presentaban menos de un 99,5% de homología con alguna secuencia depositada en las bases de datos públicas y un aislado presentó un 100% de homología con la secuencias correspondientes a M. mucogenicum (GenBank accession number AF480586) y M. phocaicum (GenBank accession number AY859682).
Desarrollo del nuevo algoritmo para interpretación de PRA hsp65 basado en la secuenciaTres nuevos patrones de PRA hsp65 se encontraron en este estudio y fueron confirmados mediante secuenciación de hsp65. Las secuencias obtenidas fueron alineadas con la secuencia de hsp65 de M. tuberculosis (GenBank accession number M15467).
Una secuencia correspondió a un nuevo patrón (tipo II) de M. alvei (GenBank accession number EU732653), diferente del tipo I (CIP103464) debido a cambios en los lugares de restricción de HaeIII (T618→C y G798→C). La secuenciacíon de hsp65 demostró que M. alvei tipo II difería de M. alvei tipo I en 13 de 441 nucleótidos (96,9% de homología).
La segunda secuencia correspondió a un nuevo patrón (tipo VIII) de M. terrae (GenBank accesion number EU732654), diferente del tipo I (ATCC15755) por cambios en los lugares de restricción de BstEII (T631→A) y HaeIII (G522→C, C593→G, C594→T y G741→C). La secuenciación del amplicón demostró que M. terrae tipo VIII difería de M. terrae tipo I en 25 de 441 nucleótidos (94,3% de homología), de M. terrae tipo II en 32 nucleótidos (92,7% de homología), de M. terrae tipo III en 18 nucleótidos (95,9% de homología) y de M. terrae tipo IV en 24 nucleótidos (94,5% en homología).
Finalmente, la tercera secuencia correspondió a un nuevo patrón (tipo III) de M. mageritense (GenBank accesion number EU732652) que difería de M. mageritense tipo I (CIP104973) debido a cambios en los sitios de restricción de HaeIII (T537→C y C588→T). La secuenciación de hsp65 mostró una diferencia de 11 de 441 nucleótidos (97,5% de homología) con M. mageritense tipo I y de 12 de 441 nucleótidos (97,2% de homología) con M. mageritense tipo II.
Todos los resultados obtenidos en este estudio junto con una actualización de observaciones previamente publicadas y el algoritmo publicado por Brunello et al.2 han sido integradas en el algoritmo ilustrado en la tabla 1.
Algoritmo de patrones de hsp65 PRA basados en el análisis in silico del fragmento de Telenti de hsp65
| Longitud de los fragmentos digeridos con BstEII (pb) | Longitud de los fragmentos digeridos con HaeIII (pb)a,b | Especieb | Referencia |
| No digestión | 197, 87, 58 | M. confluentis | AJ310228 |
| No digestión | 184, 107 | M. gilvum | AJ310236 |
| No digestión | 175, 87, 59 | M. gadium CIP105388 | AJ310233 |
| No digestión | 171, 87 | M. tusciae | AJ307660 |
| No digestión | 168, 127 | M. triviale DSM44153 | AJ307659 |
| No digestión | 161, 87, 59, 58 | M. vaccae CIP105934 | AF547889 |
| No digestión | (160), (85), (55) | M. flavescens Type III | 22 |
| No digestión | (145), (130) | M. simiae Type V | 22 |
| No digestión | 145, 127 | M. florentinum DSM44852 | DQ350162 |
| No digestión | 145, 127 | M. lentiflavum Type I CIP105465 | AF547851 |
| No digestión | 145, 87, 59, 58, 52 | M. komossense CIP105293 | AY438649 |
| No digestión | 145, 87, 59, 58 | M. parafortuitum CIP106802 | AF547864 |
| No digestión | 145, 69, 58, 54 | M. brumae | AJ310224 |
| No digestión | 139, 98, 58, 51 | M. holsaticum DSM44478 | AJ310469 |
| No digestión | 139, 87, 58 | M. flavescens Type II | DQ986499 |
| No digestión | 139, 58, 51 | M. flavescens CIP104533 | AF547831 |
| No digestión | 139, 58, 51 | M. novocastrense CIP105546 | AF547862 |
| No digestión | 134, 127, 65 | M. duvalii CIP104539 | AJ310229 |
| No digestión | 127, 114, 70, 58 | M. aurum Type II CIP 104465 | AY438081 |
| No digestión | 127, 103, 69 | M. szulgai CIP104532 | AF547878 |
| No digestión | 127, 103, 59 | M. nebraskense ATCCBAA-837 | AY368457 |
| 325, 116 | 196, 69, 58, 54 | M. aichiense | AJ310218 |
| 325, 116 | 185, 145 | M. fallax CIP81.39 | AF547829 |
| 325, 116 | 181, 127 | M. terrae Type I CIP104321 | AF547879 |
| 325, 116 | 161, 123, 58 | M. rhodesiae CIP106806 | AF547870 |
| 325, 116 | (145), (130) | M. lentiflavum Type II | 22 |
| 325, 116 | (145), (130) | M. simiae Type IV | 22 |
| 325, 116 | 145, 127, 59 | M. montefiorense ATCCBAA-256 | AY027785 |
| 325, 116 | 145, 127, 51 | M. triplex CIP106108 | AF547882 |
| 325, 116 | (145), (115), (70) | M. terrae Type V | 8 |
| 325, 116 | 145, 65, 59, 58 | M. mucogenicum Type IV | 23 |
| 325, 116 | 145, 65, 59, 58 | M. phocaicum Type III | 23 |
| 325, 116 | 145, 58 | M. arupense Type I ATCCBAA-1242 | DQ168662 |
| 325, 116 | 145 | M. cookii CIP105396 | AF547824 |
| 325, 116 | (140), (85), (60) | M. terrae Type VII | 24 |
| 325, 116 | 139, 94, 58 | M. nonchromogenicum Type II | AJ307639 |
| 325, 116 | 139, 87, 58 | M. chitae | AJ310227 |
| 325, 116 | 139, 65, 58 | M. mucogenicum Type V | 24 |
| 325, 116 | 139, 58 | M. terrae Type III | AY550212 |
| 325, 116 | (130), (110), (70), (60) | M. gordonae Type VIII | 22 |
| 325, 116 | (130), (95), (75), (60) | M. kansasii Type V | 22 |
| 325, 116 | 127, 112, 70, 59 | M. gordonae Type IV | AJ310239 |
| 325, 116 | 127, 112, 69 | M. kumamotonense | AB239920 |
| 325, 116 | 127, 103 | M. genavense | AJ310235 |
| 310, 131 | 197, 60, 58, 54 | M. chelonae | AJ310226 |
| 310, 131 | 197, 60, 58, 54 | M. salmoniphilum ATCC13758 | DQ866777 |
| 310, 131 | 175, 87, 58 | M. sphagni DSM44076 | AJ307665 |
| 310, 131 | 161, 123, 58 | M. madagascariense CIP104538 | AF547852 |
| 310, 131 | 160, 112, 111 | M. haemophilum | AJ307630 |
| 310, 131 | 145, 69, 58, 54 | M. immunogenum CIP106684 | AY458081 |
| 310, 131 | 139, 134, 51 | M. elephantis Type I CIP106831 | AF547828 |
| 310, 131 | 139, 134, 51 | M. pulveris | AF547869 |
| 310, 131 | (140), (100), (60) | M. elephantis Type II | 25 |
| 310, 131 | 139, 94, 78 | M. canariasense CIP 107998 | AY255477 |
| 310, 131 | 139, 94, 78 | M. cosmeticum ATCC BAA-878 | AY449731 |
| 310, 131 | 139, 65, 58 | M. mucogenicum TypeI ATCC49650 | 14 |
| 310, 131 | 127, 114, 58 | M. botniense DSM44537 | AF547812 |
| 310, 131 | 127, 94, 78 | M. lacus DSM44577 | AY438090 |
| 310, 116 | 244, 139 | M. fluoranthenivorans DSM44556 | DQ350157 |
| 310, 116 | 181, 161 | M. fredericksbergense DSM44346 | AF547834 |
| 310, 116 | 181, 139 | M. terrae Type II | AY550211 |
| 310, 116 | 172, 139 | M. neoaurum CIP105387 | AF547860 |
| 310, 116 | 146, 139, 58 | M. diernhoferi CIP105384 | AF547825 |
| 310, 116 | 145, 78, 58 | M. augbanense CIP 108543 | AY859677 |
| 310, 116 | 145, 65, 58 | M. phocaicum CIP108542 Type I | AY859676 |
| 310, 116 | 145, 65, 58 | M. mucogenicum TypeII CIP105223 | 24 |
| 310, 116 | 139, 87, 58 | M. mucogenicum Type III | 23 |
| 310, 116 | 139, 87, 58 | M. phocaicum Type II | 23 |
| 310, 116 | 139, 65, 58 | M. phocaicum Type IV | 23 |
| 231, 210 | 223, 106 | M. shottsii ATCC700981 | AY550225 |
| 231, 210 | 197, 87, 58 | M. moriokaense CIP105393 | AF547857 |
| 231, 210 | 197, 69, 58 | M. bolletii CIP 108541 | AY859675 |
| 231, 210 | 197, 69, 58 | M. massiliense CCUG 48898 | AY596465 |
| 231, 210 | (190), (105), (80) | M. ulcerans Type II | 7 |
| 231, 210 | 188, 126 | M. simiae Type I | AJ307652 |
| 231, 210 | (185), (130) | M. genavense Type II ATCC51233 | 22 |
| 231, 210 | 178, 134, 72, 51 | M. thermoresistibile CIP105390 | AF547880 |
| 231, 210 | 178, 98, 51 | M. hassiacum CIP105218 | AF547842 |
| 231, 210 | 162, 94, 51 | M. poriferae | AJ307645 |
| 231, 210 | 161, 58, 51 | M. austroafricanum CIP 105395 | AF547807 |
| 231, 210 | 161, 58, 51 | M. vanbaalenii DSM7251 | AY438091 |
| 231, 210 | (155), (140) | M. simiae Type II | 7 |
| 231, 210 | 146, 139, 98, 52 | M. peregrinum Type I CIP105382 | AF547865 |
| 231, 210 | 145, 139, 75 | M. parascrofulaceum Type V | AY337278 |
| 231, 210 | (145), (130), (60) | M. parmense CIP107385 | 22 |
| 231, 210 | (145), (130) | M. seoulense DSM44998 | 22 |
| 231, 210 | (145), (130) | M. intracellulare Type III | 22 |
| 231, 210 | 145, 129, 127 | M. intermedium CIP104542 | AF547847 |
| 231, 210 | 145, 127, 95 | M. scrofulaceum Type I | AJ307648 |
| 231, 210 | 145, 127 | M. saskatchewanense ATCCBAA-544 | AY208859 |
| 231, 210 | 145, 127 | M. simiae Type VI | AY550224 |
| 231, 210 | 145, 127 | M. avium Type III | AF126033 |
| 231, 210 | 145, 127 | M. interjectum Type I | AJ307632 |
| 231, 210 | 145, 106, 78 | M. ulcerans CIP105425 | AF547888 |
| 231, 210 | 145, 106, 78 | M. pseudoshottsii ATCCBAA-883 | AY550226 |
| 231, 210 | 145, 106, 78 | M. marinum | AJ307636 |
| 231, 210 | 145, 106 | M. bohemicum CIP 105811 | AF547811 |
| 231, 210 | (145), (105), (80) | M. malmoense Type II | 22 |
| 231, 210 | 145, 69, 58, 52 | M. abscessus | AJ310215 |
| 231, 210 | (140), (125), (60), (50) | M. senegalense Type III ATCC35796 | 22 |
| 231, 210 | (140), (105), (80) | M. intracellulare Type II | 22 |
| 231, 210 | 140, 81, 58, 51 | M. phlei CIP105389 | AF547866 |
| 231, 210 | 139, 123, 98, 52 | M. peregrinum Type II | AY379072 |
| 231, 210 | 139, 123, 98, 52 | M. septicum ATCC700731 | 15 |
| 231, 210 | 139, 123, 98, 52 | M. porcinum ATCC33776 | 15 |
| 231, 210 | 139, 123, 58, 52 | M. boenickei CIP107829 | AY943175 |
| 231, 210 | 139, 112, 69 | M. terrae Type IV | 19 |
| 231, 210 | 139, 103, 58 | M. kubicae CIP106428 | AF547850 |
| 231, 210 | 139, 103 | M. conspicuum CIP105165 | AF547823 |
| 231, 210 | 139, 87, 59, 58 | M. chubense CIP106810 | AF547821 |
| 231, 210 | 139, 87, 58 | M. chlorophenolicum CIP104189 | AF547820 |
| 231, 210 | 139, 87, 58 | M. obuense | AJ307640 |
| 231, 210 | 127, 112, 95 | M. intracellulare Type V | 26 |
| 231, 210 | 127, 112 | M. gordonae Type V | 27 |
| 231, 210 | 127, 112 | M. interjectum Type II | AY550236 |
| 231, 210 | 127, 112 | M. heidelbergense CIP105424 | AF547844 |
| 231, 210 | 127, 103, 78 | M. kansasii Type I CIP104589 | AF547849 |
| 231, 210 | 127, 103, 78 | M. branderi CIP104592 | AJ310223 |
| 231, 210 | 127, 103, 59 | M. avium Type II | AF126032 |
| 231, 210 | 127, 103 | M. avium Type I ATCC25291 | DQ284768 |
| 231, 210 | 127, 103 | M. colombiense CIP108962 | AM062765 |
| 231, 210 | 127, 94, 81, 78 | M. parascrofulaceum Type III | AY337276 |
| 231, 210 | 127, 94 | M. palustre DSM44572 | AY943200 |
| 231, 210 | 127, 78, 58 | M. celatum Type I CIP106109 | AF547817 |
| 231, 210 | (120), (115), (110) | M. intracellulare Type IV | 22 |
| 231, 210 | 112, 106 | M. asiaticum DSM44297 | AF547806 |
| 231, 131, 79 | 181, 161 | M. doricum DSM44852 | AF547826 |
| 231, 131, 79 | (175), (80) | M. aurum Type I | 7 |
| 231, 131, 79 | 161, 145, 59 | M. agri | AJ310217 |
| 231, 131, 79 | 161, 87, 58 | M. monacense | DQ381730 |
| 231. 131, 79 | 146, 145, 98, 52 | M. alvei CIP103464 | AF547805 |
| 231, 131, 79 | 146, 139, 98, 58 | M. peregrinum Type III | DQ986500 |
| 231, 131, 79 | 145, 127 | M. simiae Type III | AF247569 |
| 231, 131, 79 | 145, 123, 58, 52 | M. mageritense Type I CIP104973 | AF547853 |
| 231, 131, 79 | 145, 123, 58, 52 | M. alvei Type II | Our study |
| 231, 131, 79 | 145, 123, 58 | M. smegmatis CIP104444 | AF547876 |
| 231, 131, 79 | 145, 123, 58 | M. goodii ATCC700504 | AY458071 |
| 231, 131, 79 | 145, 123, 58 | M. mageritense Type II | DQ184958 |
| 231, 131, 79 | 145, 81, 58 | M. murale CIP105980 | AF547859 |
| 231, 131, 79 | 139, 81, 58 | M. tokaiense CIP106807 | AF547881 |
| 231, 131, 79 | (140), (85) | M. wolinskyi Type II | 28 |
| 231, 131, 79 | (140), (120), (95) | M. gordonae Type VI | 7 |
| 231, 131, 79 | 139, 103, 69 | M. shimoidei DSM44152 | AJ307650 |
| 231, 131, 79 | 139, 123, 98, 52 | M. senegalense Type II | AY684049 |
| 231, 131, 79 | 139, 123, 59 | M. fortuitum Type II ATCC19542 | AJ310232 |
| 231, 131, 79 | 139, 123, 58 | M. wolinskyi Type I ATCC700010 | AY458064 |
| 231, 131, 79 | 139, 87, 58 | M. mageritense Type III | Our study |
| 231, 131, 79 | 139, 81, 58 | M. hodleri CIP104909 | AF547845 |
| 231, 131, 79 | (130), (105), (80) | M. celatum Type II ATCC51130 | 22 |
| 231, 131, 79 | (130), (95), (70) | M. kansasii Type III | 22 |
| 231, 131, 79 | 127, 103, 101 | M. kansasii Type II | AY438086 |
| 231, 131, 79 | 127, 103, 69 | M. gastri | AJ310234 |
| 231, 131, 79 | 127, 103, 69 | M. kansasii Type VI | AY550233 |
| 231, 131, 79 | 127, 94 | M. parascrofulaceum Type IV | AY337277 |
| 231, 116, 94 | 161, 112, 59 | M. gordonae Type IX | AM398452 |
| 231, 116, 94 | 161, 103, 59 | M. heckeshornense DSM44428 | AF547843 |
| 231, 116, 94 | (155), (110) | M. gordonae Type VII | 6 |
| 231, 116, 94 | (145), (130) | M. lentiflavum Type III | 24 |
| 231, 116, 94 | 145, 127, 59 | M. chimaera CIP107892 | AY943198 |
| 231, 116, 94 | 145, 127, 57 | M. intracellulare Type I | AJ307633 |
| 231, 116, 94 | 145, 127 | M. scrofulaceum Type III | 9 |
| 231, 116, 94 | 145, 105, 78 | M. malmoense | AJ307635 |
| 231, 116, 94 | (145), (70), (60) | M. terrae Type VI | 8 |
| 231, 116, 94 | (140), (125), (100) | M. brisbanense CIP107830 | 29 |
| 231, 116, 94 | 139, 123, 58 | M. terrae Type VIII | Our study |
| 231, 116, 94 | 139, 58 | M. hiberniae | AJ307631 |
| 231, 116, 94 | 127, 112, 95 | M. gordonae Type X | 19 |
| 231, 116, 94 | 127, 112 | M. gordonae tipo III | AJ310238 |
| 231, 116, 94 | 127, 94, 59, 51 | M. intracellulare Type VI | AY536637 |
| 231, 116, 79 | 210, 115 | M. gordonae Type II | 2 |
| 231, 116, 79 | 181, 139, 52 | M. senegalense Type IV ATCC35796 | AY684045 |
| 231, 116, 79 | 161, 112, 57 | M. gordonae Type I | AJ310237 |
| 231, 116, 79 | 161, 104, 59 | M. xenopi | AJ307661 |
| 231, 116, 79 | 152, 127, 69 | M. tuberculosis complex | 2 |
| 231, 116, 79 | 145, 123, 58, 52 | M. fortuitum Type I ATCC6841 | AY458072 |
| 231, 116, 79 | 145, 127 | M. sherrisii ATCCBAA-832 | DQ523524 |
| 231, 116, 79 | 145, 58, 54 | M. nonchromogenicum Type I DSM44164 | 24 |
| 231, 116, 79 | 141, 123, 58, 52 | M. farcinogenes | AJ310231 |
| 231, 116, 79 | 139, 123, 58, 52 | M. conceptionense CIP108544 | AY859678 |
| 231, 116, 79 | 139, 123, 58, 52 | M. houstonense ATCC49403 | AY458077 |
| 231, 116, 79 | 139, 123, 58, 52 | M. neworleansense ATCC49404 | AY496143 |
| 231, 116, 79 | 139, 123, 58, 52 | M. senegalense Type I CIP104971 | AF547872 |
| 231, 116, 79 | 139, 94 | M. parascrofulaceum Type I | AY337275 |
| 231, 116, 79 | (135), (90), (85) | M. fortuitm type III | 6 |
| 231, 116, 79 | (130), (115), (75), (60) | M. kansasii Type IV | 30 |
| 231, 116, 79 | (130), (95) | M. lentiflavum Type IV | 22 |
| 231, 116, 79 | 127, 94 | M. parascrofulaceum Type II ATCCBAA-614 | AY337274 |
| 231, 116, 79 | (125), (95) | M. scrofulaceum Type II | 8 |
Clásicamente, Mycobacterium spp. han sido identificadas con métodos fenotípicos. En las últimas 2 décadas, el desarrollo de herramientas basadas en la biología molecular ha llevado a profundas modificaciones en los métodos de identificación y en la clasificación taxonómica de este grupo. La introducción del sistema AccuProbe (Gen-Probe, San Diego, CA) ha acelerado enormemente la identificación de algunas especies de Mycobacterium. Desarrollado para la identificación de M. tuberculosis complex, MAC, M. avium, M. intracellulare, M. kansasii y M. gordonae, es un ensayo simple y rápido. Consiste en una sonda única cuya elección puede ser orientada en función de la morfología al microscopio o las características de la colonia. En nuestro estudio, AccuProbe fue capaz de identificar 102 de 236 (43,2%) aislados de MNT, resultados similares a los publicados por otros autores12.
Siete aislados presentaron un resultado positivo con AccuProbe MAC Identification Test pero negativa para las sondas específicas de M. avium y M. intracellulare. Estudios recientes han llamado la atención acerca de la amplia diversidad de los aislados que pueden ser detectados dentro del MAC16.
La especie M. colombiense fue descrita en 2006 como un crecedor lento incluido en el MAC17 y ha sido asociaao a enfermedad humana. En nuestro estudio encontramos 2 aislados de esta especie. Otras especies como M. palustre y M. saskatchewanense pueden ser confundidas con MAC debido a su positividad con AccuProbe MAC Identification Test aunque genéticamente están distantes del MAC16.
La identificación de micobacterias mediante PRA hsp65 fue desarrollada por Telenti et al.5 y ha sido una práctica establecida desde 1993. Fueron identificados originalmente por Telenti et al. 33 patrones de PRA; después de esto, otros autores introdujeron patrones adicionales en el algoritmo publicado por estos autores7,18. En 2001, Brunello et al. publicaron un algoritmo para la identificación mediante PRA hsp65, el cual incluyó 50 especies de micobacterias2. Dado que se han descrito más de 130 especies y subespecies de micobacterias (J.P. Euzéby, List of Prokaryotic Names with Standing in Nomenclature [http://www.bacterio.citc.fr]) hemos considerado necesaria una actualización de este procedimiento. Chimara et al.19 recientemente han publicado un nuevo algoritmo de identificación mediante PRA hsp65.
En nuestro estudio, nosotros identificamos 76 de 134 (56,7%) aislados con esta técnica. En varios estudios6,9,19 PRA hsp65 identificaba cerca del 90% de las cepas evaluadas, aunque en ellos se incluían especies que pueden ser identificadas mediante sondas AccuProbe. Si se tiene como referencia el algoritmo publicado por Chimara et al.19 el número de aislados no identificados mediante PRA hsp65 sería de 32 aislados, en vez de los 58 aislados para los que fue necesario realizar la secuenciación del 16S rDNA. Un total de 26 aislados sí presentaron un PRA hsp65 incluido en el algoritmo publicado por Chimara et al.19. Estos aislados son M. parascrofulaceum (n = 7), M. fortuitum (n = 13), M. wolinskyi (n = 1), M. nonchromogenicum (n = 1), M. celatum (n = 1), M. sherrisii (n = 1), M. kumamotonense (n = 1) y M. mucogenicum (n = 1). No obstante, en el caso de M. wolinskyi (n = 1) según el algoritmo presentado en nuestro estudio comparte PRA hsp65 con M. fortuitum ATCC195422. Una diferencia importante de nuestro algoritmo de hsp65 PRA con el algoritmo propuesto por Chimara et al.19 es que el nuestro incluye los fragmentos 325/116 y 310/116, obtenidos tras digestión con BstEII, que separamos por ser claramente diferentes (15 nt).
Distintos factores críticos deben ser considerados cuando se usa PRA hsp65 como método de identificación de MNT. Uno es la pérdida de estandarización debido a las diferentes condiciones de electroforesis que son utilizadas. Esto causa diferencias en los patrones comunicados en la literatura científica. Además, esta metodología está afectada por la interpretación subjetiva si no se utiliza software para reconocer el patrón. Para evitar estas dificultades, nosotros utilizamos estrictas condiciones de electroforesis y software apropiado. Reconociendo las limitaciones del PRA hsp65, Kapur et al.11 fueron los primeros en secuenciar el amplicón de hsp65 generado con los iniciadores de Telenti como método de identificación de MNT. Desde entonces, otros autores han demostrado la utilidad de esta aproximación diagnóstica12. La secuenciación de 441 pares de bases del hsp65 permite la construcción de algoritmos basados en la secuencia. De cualquier forma, los patrones de bandas deducidos de la secuencia no reflejan exactamente los patrones de bandas observados en los geles de electroforesis. Además, algunas especies de Mycobacterium pueden compartir patrón de PRA o tienen diferencias de menos de 5 pb (tabla 1). Finalmente, nuevos patrones para nuevas especies o especies conocidas son descritos con frecuencia. Aunque tales patrones nuevos, si son fácilmente reconocidos2, no deberían ser un inconveniente para esta metodología, sí que es verdad que requiere una actualización constante. En nuestro trabajo, 3 aislados presentaban patrones de PRA hsp65 que no coincidían con alguno previamente descrito. Esta circunstancia también ha pasado en otros estudios6,8,9.
El algoritmo de patrones de cepas de referencia calculados in silico a partir de las secuencias depositadas en las bases de datos creemos que es útil porque refleja la realidad mejor que los algoritmos comunicados con patrones estimados, aunque el PRA hsp65 es una estimación, no una secuenciación.
El gen más ampliamente utilizado en identificación bacteriana es el 16S rDNA y es considerado el gold standard10. Su utilización ha contribuido a la descripción de nuevas especies de MNT en los últimos años10. Sin embargo, como existe poca variabilidad dentro del 16S rDNA del género Mycobacterium, no permite diferenciar especies cercanamente relacionadas como M. chelonae o M. abcessus10.
La secuenciación parcial de 16S rDNA nos ha permitido identificar un total de 53 (22,4%) aislados, incluyendo algunas especies recientemente descritas como M. sherrisii, M. kumamotonense y M. arupense. Destaca la elevada frecuencia con la que aislamos M. parascrofulaceum. Esta especie fue descrita en 200420 como un crecedor lento escotocromógeno relacionado con M. simiae. Trece aislados fueron identificados como M. fortuitum mediante secuenciación del 16S rDNA. Todos presentaron un patrón de PRA hsp65 que corresponde a M. fortuitum tipo I. Este patrón no está incluido en el algoritmo publicado por Brunello et al.2.
Nosotros encontramos 5 de 58 aislados (8,6%) que no pudieron ser identificados mediante secuenciación de 16S rDNA. Uno de ellos fue identificado como M. mucogenicum mediante secuenciación de hsp65. Finalmente, 4 aislados (1,7%) no pudieron ser identificados y podrían corresponder a nuevos taxones, en función de sus secuencias únicas de 16S rDNA y hsp65. Otros autores21 han comunicado resultados similares.
Actualmente ninguna técnica de biología molecular es capaz de identificar el 100% de los aislados de Mycobacterium sp. La técnica de HPLC se encuentra en laboratorios de referencia y requiere personal muy especializado. La secuenciación parcial del 16S rDNA se ha convertido en la actual prueba de referencia «práctica» para la identificación de especie (la prueba de referencia real para la identificación de especie es el estudio de reasociación de ADN o hibridación ADN-ADN). El problema de este gen para Mycobacterium sp. es que presenta poca variabilidad para este género (el punto de corte para el diagnóstico de especie está fijado en 99,5% de homología, frente al 99% general para otras especies bacterianas), de ahí que se utilicen otros genes como hsp65 o rpoB, los cuales presentan más variabilidad que el 16S rDNA. Las inmunocromatografías de membrana, como GenoType® CM/AS (Hain Lifescience GmbH, Nehren, Alemania), si bien son fáciles y rápidas de realizar, sólo identifican 30 especies (o complejos, como M. tuberculosis complex). Además, en ocasiones, son necesarias 2 hibridaciones, ya que GenoType CM identifica 12 especies, un complejo (M. tuberculosis complex) y M. marinum/ulcerans; GenoType AS permite la identificación de 16 especies. En nuestro estudio un total de 42 cepas no hubieran podido ser identificadas mediante esta técnica y necesitarían otra técnica de identificación. Estas especies serían M. triviale (n = 1), M. thermoresistibile (n = 1), M. parasrofulaceum (n = 20), M. porcinum (n = 3), M. mageritense (n = 2), M. colombiense (n = 2), M. septicum (n = 2), M. arupense (n = 2), M. alvei (n = 1), M. terrae (n = 1), M. nonchromogenicum (n = 1), M. kumamotonense (n = 1), M. sherrisii (n = 1) y Mycobacterium sp. (probables taxones no descritos) (n = 4).
La identificación correcta de MNT clínicamente relevantes es esencial para establecer un sistema taxonómico detallado y su rápida identificación está llegando a ser cada vez más importante para el buen cuidado del paciente. Nosotros integramos diferentes técnicas de biología molecular como AccuProbe, PRA hsp65 y secuenciación de 16S rDNA y hsp65, junto a características fenotípicas básicas en un algoritmo de identificación de MNT. Este algoritmo nos permitió identificar casi todas las MNT aisladas en este estudio, incluyendo algunas especies recientemente descritas. En el presente trabajo hemos actualizado el algoritmo para interpretación del PRA hsp65.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este estudio ha sido parcialmente financiado por el Ministerio de Sanidad y Consumo, el Instituto de Salud Carlos III-Feder y la Red Española de Investigación en Patología Infecciosa (REIPI C03/14 y RD06/0008/0013).