Describir el perfil bacteriano del biofilm supragingival de niños con dentición temporal (NDT) y dentición mixta temprana (NDMT), con la técnica de secuenciación de próxima generación HOMINGS.
MétodoSe realizó un estudio descriptivo comparativo con 30 niños de 5 a 7 años de edad sistémicamente sanos de escuelas públicas de Cartagena (Colombia). Todos los participantes estaban libres de caries, según los criterios del Sistema Internacional de Detección y Evaluación de Caries (ICDAS II) y sin experiencia de caries según el índice de dientes cariados, perdidos y obturados (DCPO). Se recolectaron muestras de biofilm supragingival. Se extrajo el ADN bacteriano y se usó para su análisis mediante HOMINGS (identificación de microorganismos orales humanos utilizando secuenciación de próxima generación) basado en la secuenciación de la región V3-V4 del gen 16S rRNA con la plataforma Illumina MiSeq.
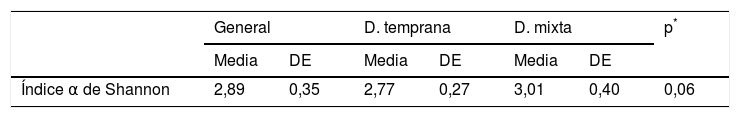
ResultadosSe identificaron 360 especies específicas y 65 géneros específicos de las sondas: Streptococcus, Actinomyces, Veillonella y Fusobacterium (29,2% del total de ADN bacteriano presente), mientras que en el grupo de dentición mixta temprana se encontraban Streptococcus, Leptotrichia, TM7 y Porphyromonas (24,5% del ADN bacteriano presente). Las especies bacterianas con mayor abundancia relativa en el microbioma oral de biofilm de NDT fueron Streptococcus sanguinis, Rothia aeria, Gemella haemolysans, mientras que en NDMT fueron S. sanguinis, Leptotrichia sp. HOT-417, Leptotrichia sp. HOT-498. El índice de diversidad de Shannon fue 2,77 (DE=0,26) para NDT y 3,01 (DE=0,39) para NDMT (p=0,06).
ConclusionesEl análisis del perfil bacteriano del biofilm dental supragingival en niños con NDMT mediante HOMINGS mostró baja diversidad microbiológica tanto en presencia como en abundancia relativa a nivel de género y de especies bacterianas.
Tdescribe the bacterial profile of the supragingival biofilm of children with temporary dentition (CTD) and early mixed dentition (CEMD), with the next-generation sequencing (HOMINGS) technique.
MethodA comparative descriptive study was carried out with 30 systemically healthy children aged between 5 and 7 years old from public schools in Cartagena-Colombia. All participants were caries-free applying the criteria of the International Caries Detection and Assessment System (ICDAS II) and had no caries experience according to the Decayed, Missing and Filled Teeth (DMFT) index. Supragingival biofilm samples were collected. Bacterial DNA was extracted and used for analysis using HOMINGS (Human Oral Microbe Identification using Next-Generation Sequencing) based on the sequencing of the V3-V4 region of the 16S rRNA gene using the Illumina MiSeq platform (V3-V4 primers).
ResultsA total of 360 species-specific and 65 genus-specific probes were identified. The bacterial genus most predominant in CTD were Streptococcus, Actinomyces, Veillonella and Fusobacterium (29.2% of all bacterial DNA present), while in CEMD the most predominant were Streptococcus, Leptotrichia, TM7 and Porphyromonas (24.5% of all bacterial DNA present). The bacterial species with the highest relative abundance in the oral biofilm microbiome from CTD were Streptococcus sanguinis, Rothia aeria, Gemella haemolysans, while in CEMD they were S. sanguinis, Leptotrichia spp. HOT-417 and Leptotrichia spp. HOT-498. The Shannon diversity index was 2.77 (SD=0.26) for CTD and 3.01 (SD=0.39) for CEMD (P=0.06).
ConclusionsThe analysis of the bacterial profile of the supragingival dental biofilm in children with DMFT, by means of HOMINGS showed low microbiological diversity both in presence and in relative abundance in terms of genus as well as bacterial species.
El microbioma humano juega un papel importante en el bienestar y el estado de salud. Se ha hecho en los últimos años un gran esfuerzo en la caracterización de las diferentes comunidades microbianas que colonizan el cuerpo humano; entre otros sitios, la cavidad oral, que representa una de las más diversas comunidades microbianas asociadas con cualquiera de los sitios humanos1,2.
Con el advenimiento de técnicas moleculares de la identificación de bacterias orales basadas en el ADN, se ha demostrado que la microbiota oral es mucho más compleja y diversa de lo que originalmente se había previsto3. La cavidad oral está colonizada por más de 700 especies de microorganismos, el 35% de los cuales son aún no cultivables. Dicha comunidad microbiana puede diferir entre la infancia y la edad adulta4.
Los biofilm son acumulación heterogénea de una comunidad microbiana, rodeada por una matriz intercelular de polímeros. Estos microorganismos pueden adherirse o depositarse sobre las paredes de los dientes, en la mucosa oral y en la saliva, entre otros5. Algunos colonizadores de dientes y favorecedores en la formación del biofilm oral son Streptococcus sanguis, S. mitis y S. oralis, Actinomyces naeslundii, S. mutans, S. salivarius, S. gordonii S. parasanguis, Neisseria spp., Prevotella loescheii, P. intermedia, Capnocytophaga spp., Fusobacterium nucleatum, Porphyromonas gingivalis y otros6,7.
La técnica de secuenciación de próxima generación HOMINGS (Human Oral Microbe Identification using Next Generation Sequencing) fue desarrollada recientemente y permite la identificación simultánea a nivel de especie de casi 600 taxones bacterianos orales. Entre sus ventajas: es computacionalmente eficiente, rápida, reproducible y puede identificar la mayoría del microbioma oral a nivel de especie8. HOMINGS se ha utilizado en varias investigaciones recientes, Richards et al., en el 2017, investigaron el microbioma de la placa dental en niños con diferentes estados de caries9 y Belstrom et al. en el 2016 estudiaron la estabilidad temporal del microbiomas salivales10.
El propósito del presente estudio fue identificar el perfil bacteriano del biofilm dental en niños con dentición temporal y mixta temprana, aplicando la técnica de secuenciación de próxima generación HOMINGS, ya que la tecnología proporcionaría información más completa sobre el bacterioma supragingival.
MétodosModelo de estudio descriptivo de corte transversal en el que se describió el perfil bacteriano del biofilm oral en niños con dentición temporal y con dentición mixta temprana. La población de estudio fueron niños entre los 5 y 7 años de edad, de 3instituciones de educación primaria en la ciudad de Cartagena de Indias (Colombia). El tamaño de la muestra se determinó mediante el software estadístico Epidat v. 4.0. Para esto se tuvo en cuenta una prevalencia esperada de caries en la región caribe del 90%, según se reporta en ENSAB IV11, con un nivel de confianza del 95% y error del 5%. La muestra estuvo constituida por 30 niños.
Se excluyeron niños con enfermedad sistémica, hematológica, autoinmune, que estén bajo tratamiento farmacológico al menos tres meses anteriores al inicio del estudio, que presenten malformaciones congénitas involucrado la cavidad oral, que la última visita al odontólogo fuese menos de seis meses y haber recibido profilaxis dental, sellantes y/o topicaciones de flúor en gel o barniz durante este tiempo, ya que estos procedimientos puede modificar el microambiente oral.
Este proyecto se desarrolló en el marco del proyecto «Caracterización del microbioma oral en saliva y biopelícula bacteriana de niños con lesiones cariosas. Estudio de casos y controles prospectivo», código N.°141965741160, el cual fue financiado por Colciencias (contrato 673-2014) y ejecutado por la Corporación Universitaria Rafael Núñez. Las actividades aquí descritas, la muestra de estudio y la financiación son completamente derivadas del proyecto antes mencionado.
Técnicas y procedimientos para recolectar la informaciónSe recolectaron muestras de biofilm supragingival en niños sanos, en el primer molar permanente, con dentición mixta temprana y en el segundo molar caduco en niños con dentición temporal. En aquellos casos en los que el diente no presentaba biofilm supragingival, se recolectó placa de las superficies cercanas. El biofilm supragingival se recolectó con un aplicador y se colocó en tubos de 1,5mL con 150 μL de buffer TE y se almacenaron a −20°C hasta su posterior análisis.
Extracción de ADN microbianoPara la extracción de ADN bacteriano se suspendieron las muestras (biofilm) en 150μL de buffer TE (10mM Tris-HCl, 1M EDTA, pH 8,0) y 1μL de Ready-Lyse Lysozyme (Epicentre Cat. No. R1802M, EE. UU.), que se dejó incubar a 37°C toda la noche. Posteriormente se agregaron 150μL de solución de lisis TyC 2X y 1μL de proteinasa K. Se dejó incubar por 30 min a 65°C bajo agitación continua.
Se colocaron las muestras en hielo durante 3-5 min. Se agregó 175μL del reactivo de precipitación de proteína MPC a 300μL de las muestras lisadas; se agitó vigorosamente por 10 s. Se centrifugaron las muestras por 10 min a 4°C a 10.000xg. Se transfirió el sobrenadante a tubos de 1,5mL estériles. Se agregaron 500μL de isopropanol al sobrenadante recuperado y se mezclaron de 30 a 40 veces. Se incubó por 10 min sobre hielo. Se centrifugó a 10.000xg a 4°C; se descartó el isopropanol evitando la pérdida del precipitado. Se enjuagó el precipitado con 500μL de etanol al 75%, se mezcló, se centrifugó brevemente y se descartó el etanol; se repitió este paso. Se resuspendió el ADN con 25μL de buffer TE. A cada muestra se le analizó la concentración y calidad del ADN mediante un espectrofotómetro Multiskan Go® (ThermoFisher Scientific) bajo densidades ópticas de 260/280. Las muestras se almacenaron a −20°C hasta su posterior análisis.
Identificación bacterianaEl perfil microbiológico obtenido del ADN purificado de las muestras de biofilm se realizó mediante la tecnología HOMINGS (http://homings.forsyth.org). HOMINGS es una técnica molecular basada en secuenciación de nueva generación que emplea la plataforma MiSeq System (Ilumina, Inc., San Diego, CA, EE. UU.), que secuencia la región 16S rRNA V3-V4 usando cebadores descritos12. Esta técnica fue realizada en el instituto Forsyth (Boston, MA, EE. UU.) que, en combinación con el programa ProbeSeq for HOMINGS, Programa BLAST personalizado (Forsyth Institute, Cambridge, MA, EE. UU.) es capaz de identificar simultáneamente alrededor de 600 taxones orales Belstrom12. La base de datos de ProbeSeq contiene secuencias de sondas (17-40 bases) recogidas en la base de datos de HOMD (Human Oral Microbiome Database), en concreto, 598 sondas especie-específicas y 94 sondas género-específicas (sondas dirigidas a 2o más taxones estrechamente relacionados dentro del mismo género).
Análisis estadísticoLos datos sociodemográficos como género, grado escolar, abundancia relativa y presencia del organismo son reportados mediante tablas de frecuencia. La variable edad se describe en valores de media y desviación estándar (DE). El contraste entre variables demográficas y tipo de dentición se analizó mediante la prueba de Chi cuadrado de Pearson. A partir del número de lecturas obtenidas por muestra, se estimó el índice de diversidad microbiológica α de Shannon. El contraste de este índice entre los grupos de estudio se realizó mediante la prueba t de Student. Para estos análisis, un valor de p <0,05 fue considerado significativo.
La abundancia relativa y la presencia de las especies y géneros bacterianos no mostraron distribución normal conforme la prueba de normalidad de Shapiro-Wilk. El contraste de estas variables entre los grupos según el tipo de dentición se realizó mediante la prueba U de Mann-Whitney con corrección de Benjamini-Hochberg para comparaciones múltiples. Esta fue usada para controlar el descubrimiento de falsos positivos del 10% (66). Para estos análisis, un valor de p ajustado <0,001 fue considerado estadísticamente significativo. Los análisis estadísticos se realizaron con los programas estadísticos SPSS v20 (IBM, Nueva York, EE. UU.) y MeV 4 8 1 Multi Experiment Viewer. www.tm4.org13,14.
Consideraciones éticasAntes de iniciar cualquier procedimiento los padres o tutores legales aprobaron la participación del menor mediante un consentimiento informado. De igual forma, se indagó en el niño su voluntad de participación en el estudio mediante un asentimiento. Según la resolución 008430 de 1993, el presente estudio representa un riesgo mayor que el mínimo para los participantes: los posibles riesgos fueron plasmados con claridad en el consentimiento informado.
ResultadosFormaron parte del estudio 30 niños: 15 con dentición temporal y 15 con dentición mixta temprana. Se observó un promedio de edad de 6,23±0,8 años de edad. El 46,7% (n=14) eran de género masculino y el 53,3% (n=16) de género femenino. Estaban matriculados en 3instituciones de educación básica primaria ubicadas en la Localidad 1 Histórica y del Caribe Norte en la ciudad de Cartagena (Bolívar).
El análisis HOMINGS permitió identificar el perfil bacteriano en muestras de biofilm oral procedentes de superficies supragingivales y se pudo observar la diversidad bacteriana de algunas especies bacterianas identificadas y su contribución (proporción o abundancia relativa) en el microbioma de cada individuo. En total, 65 sondas género-específicas amplificaron en las muestras de estudio; estas recopilaron información de 44 géneros bacterianos presentes en el biofilm oral analizado. Entre estos, los géneros de Streptococcus, Actinomyces, Veillonella, TM7, Porphyromonas, Fusobacterium, Neisseria, Rothia, Selenomonas y Centipeda, Leptotrichia, Gemella, Actinomyces y Corynebacterium estuvieron presentes en el 100% de las muestras. Por su parte, la abundancia relativa (proporción relativa) de cada uno de los géneros denota la contribución de estos sobre el perfil bacteriano del biofilm oral. Por consiguiente, Streptococcus sonda 4 (20,67%±12,1), Streptococcus sonda 1 (2,68%±2,67), Actinomyces sonda 3 (1,07%±1,3), Leptotrichia sonda 3 (0,96%±1,7), Veillonella sonda 2 (0,91%±1,4), TM7 (0,90%±2,0) y Porphyromonas sonda 2 (0,82%±1,3) son los 6géneros bacterianos más abundantes en el biofilm oral, que representan el 28,02% de este.
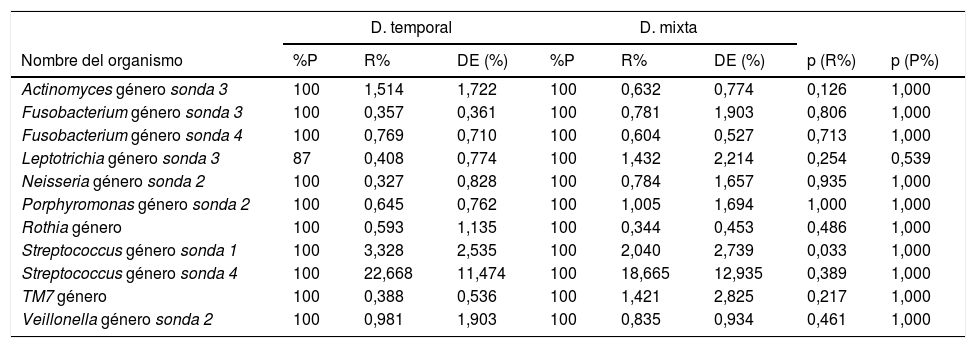
Perfil bacteriano de biofilm oral según el tipo de denticiónEl perfil bacteriano del biofilm oral varía tanto en presencia como en abundancia relativa en el género y en las especies bacterianas entre los 2tipos de dentición. En los géneros bacterianos se observó que los que mayor abundancia relativa tiene en las muestras de biofim procedentes de niños con dentición temporal fueron Streptococcus sonda 4, Streptococcus sonda 1, Actinomyces género sonda 3, Veillonella sonda 2, Fusobacterium sonda 4 y Porphyromonas género sonda 2. Mientras que en los niños con dentición mixta temprana se observan Streptococcus sonda 4, Streptococcus sonda 1, Leptotrichia sonda 3, TM7 género, Porphyromonas género sonda 2, Veillonella género sonda 2 y Neisseria género sonda 2 (tabla 1).
Géneros bacterianos según tipo de dentición
| D. temporal | D. mixta | |||||||
|---|---|---|---|---|---|---|---|---|
| Nombre del organismo | %P | R% | DE (%) | %P | R% | DE (%) | p (R%) | p (P%) |
| Actinomyces género sonda 3 | 100 | 1,514 | 1,722 | 100 | 0,632 | 0,774 | 0,126 | 1,000 |
| Fusobacterium género sonda 3 | 100 | 0,357 | 0,361 | 100 | 0,781 | 1,903 | 0,806 | 1,000 |
| Fusobacterium género sonda 4 | 100 | 0,769 | 0,710 | 100 | 0,604 | 0,527 | 0,713 | 1,000 |
| Leptotrichia género sonda 3 | 87 | 0,408 | 0,774 | 100 | 1,432 | 2,214 | 0,254 | 0,539 |
| Neisseria género sonda 2 | 100 | 0,327 | 0,828 | 100 | 0,784 | 1,657 | 0,935 | 1,000 |
| Porphyromonas género sonda 2 | 100 | 0,645 | 0,762 | 100 | 1,005 | 1,694 | 1,000 | 1,000 |
| Rothia género | 100 | 0,593 | 1,135 | 100 | 0,344 | 0,453 | 0,486 | 1,000 |
| Streptococcus género sonda 1 | 100 | 3,328 | 2,535 | 100 | 2,040 | 2,739 | 0,033 | 1,000 |
| Streptococcus género sonda 4 | 100 | 22,668 | 11,474 | 100 | 18,665 | 12,935 | 0,389 | 1,000 |
| TM7 género | 100 | 0,388 | 0,536 | 100 | 1,421 | 2,825 | 0,217 | 1,000 |
| Veillonella género sonda 2 | 100 | 0,981 | 1,903 | 100 | 0,835 | 0,934 | 0,461 | 1,000 |
p<0,001. Prueba U de Mann-Whitney con corrección de Benjamini-Hochberg (tasa de descubrimientos falsos: 10%) de las comparaciones múltiples. Se muestran aquellos géneros bacterianos con una proporción relativa promedio ≥0,3% en el microbioma de los individuos.
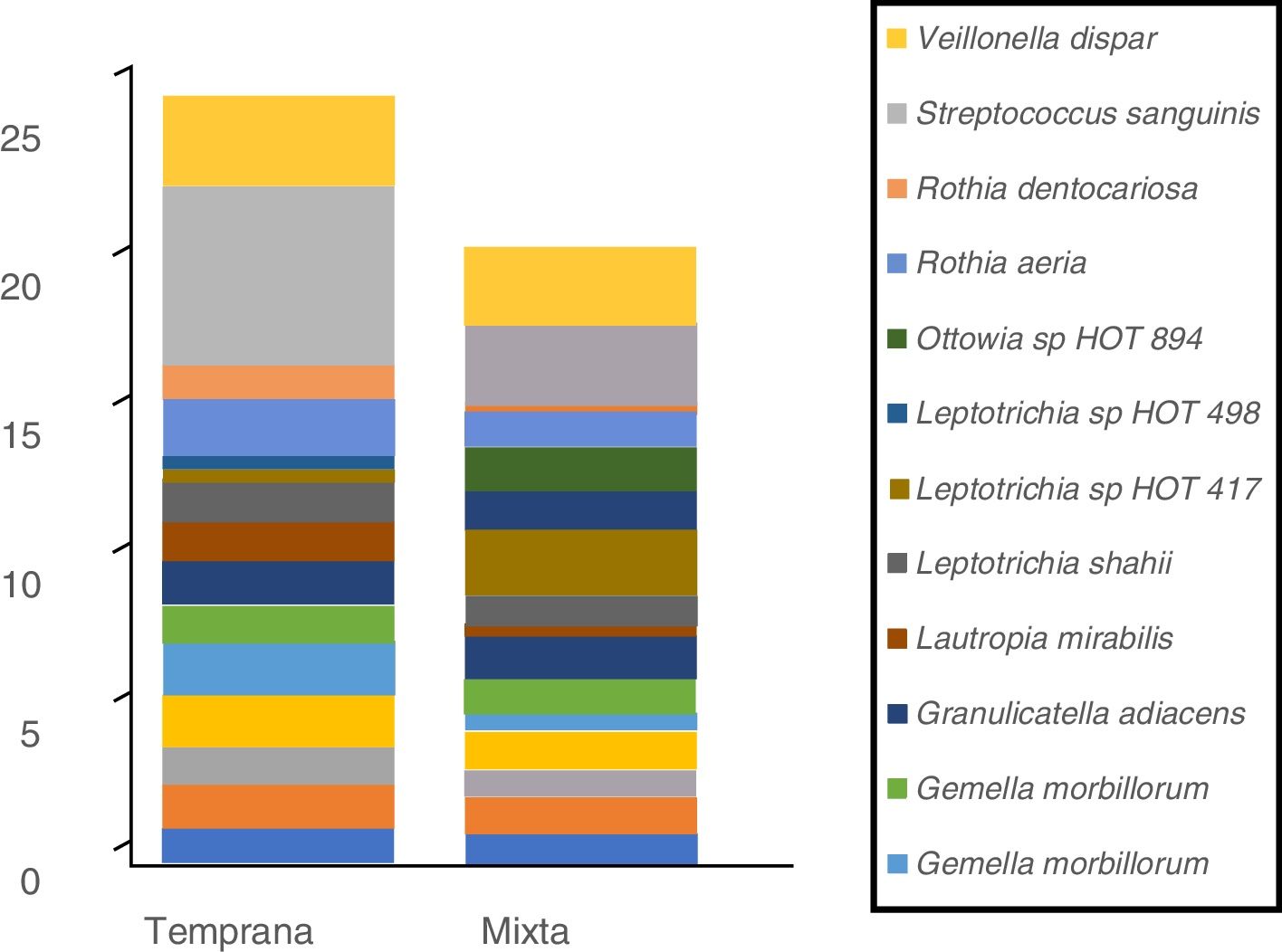
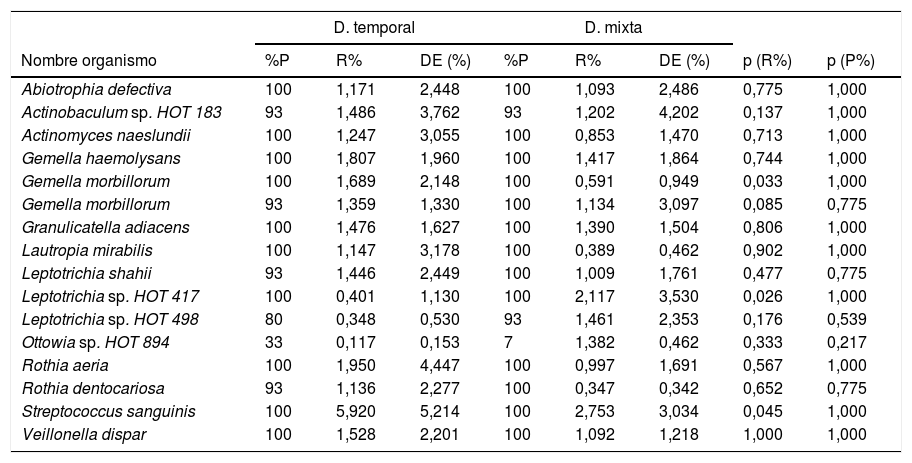
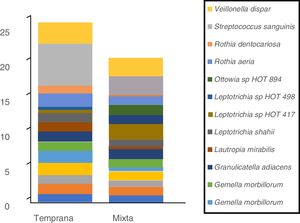
En cuanto a las especies bacterianas, se observó que las 6especies con mayor abundancia relativa en las muestras de biofilm procedentes de niños con dentición temporal fueron: Streptococcus sanguinis, Rothia aeria, Gemella haemolysans, Gemella morbillorum, Veillonella dispar y Actinobaculum sp. HOT 183. Por su parte, en los niños con dentición mixta temprana se observó: Streptococcus sanguinis, Leptotrichia sp. HOT 417, Leptotrichia sp. HOT 498, Gemella haemolysans, Granulicatella adiacens y Ottowia sp. HOT 894 (tabla 2). En la figura 1 se muestran las especies bacterianas con mayor abundancia relativa.
Especies de microorganismos según dentición
| D. temporal | D. mixta | |||||||
|---|---|---|---|---|---|---|---|---|
| Nombre organismo | %P | R% | DE (%) | %P | R% | DE (%) | p (R%) | p (P%) |
| Abiotrophia defectiva | 100 | 1,171 | 2,448 | 100 | 1,093 | 2,486 | 0,775 | 1,000 |
| Actinobaculum sp. HOT 183 | 93 | 1,486 | 3,762 | 93 | 1,202 | 4,202 | 0,137 | 1,000 |
| Actinomyces naeslundii | 100 | 1,247 | 3,055 | 100 | 0,853 | 1,470 | 0,713 | 1,000 |
| Gemella haemolysans | 100 | 1,807 | 1,960 | 100 | 1,417 | 1,864 | 0,744 | 1,000 |
| Gemella morbillorum | 100 | 1,689 | 2,148 | 100 | 0,591 | 0,949 | 0,033 | 1,000 |
| Gemella morbillorum | 93 | 1,359 | 1,330 | 100 | 1,134 | 3,097 | 0,085 | 0,775 |
| Granulicatella adiacens | 100 | 1,476 | 1,627 | 100 | 1,390 | 1,504 | 0,806 | 1,000 |
| Lautropia mirabilis | 100 | 1,147 | 3,178 | 100 | 0,389 | 0,462 | 0,902 | 1,000 |
| Leptotrichia shahii | 93 | 1,446 | 2,449 | 100 | 1,009 | 1,761 | 0,477 | 0,775 |
| Leptotrichia sp. HOT 417 | 100 | 0,401 | 1,130 | 100 | 2,117 | 3,530 | 0,026 | 1,000 |
| Leptotrichia sp. HOT 498 | 80 | 0,348 | 0,530 | 93 | 1,461 | 2,353 | 0,176 | 0,539 |
| Ottowia sp. HOT 894 | 33 | 0,117 | 0,153 | 7 | 1,382 | 0,462 | 0,333 | 0,217 |
| Rothia aeria | 100 | 1,950 | 4,447 | 100 | 0,997 | 1,691 | 0,567 | 1,000 |
| Rothia dentocariosa | 93 | 1,136 | 2,277 | 100 | 0,347 | 0,342 | 0,652 | 0,775 |
| Streptococcus sanguinis | 100 | 5,920 | 5,214 | 100 | 2,753 | 3,034 | 0,045 | 1,000 |
| Veillonella dispar | 100 | 1,528 | 2,201 | 100 | 1,092 | 1,218 | 1,000 | 1,000 |
p<0,001. Prueba U de Mann-Whitney con corrección de Benjamini-Hochberg (tasa de descubrimientos falsos: 10%) de las comparaciones múltiples. Se muestran aquellas especies bacterianas con una proporción relativa promedio ≥1% en el microbioma de ambos grupos.
Perfil de especies bacterianas de biofilm oral según tipo de dentición.
Se muestran las especies bacterianas con mayor abundancia relativa. El área de cada color corresponde a la abundancia relativa promedio (proporción relativa promedio) de cada especie bacteriana con relación al bacterioma del biofilm oral.
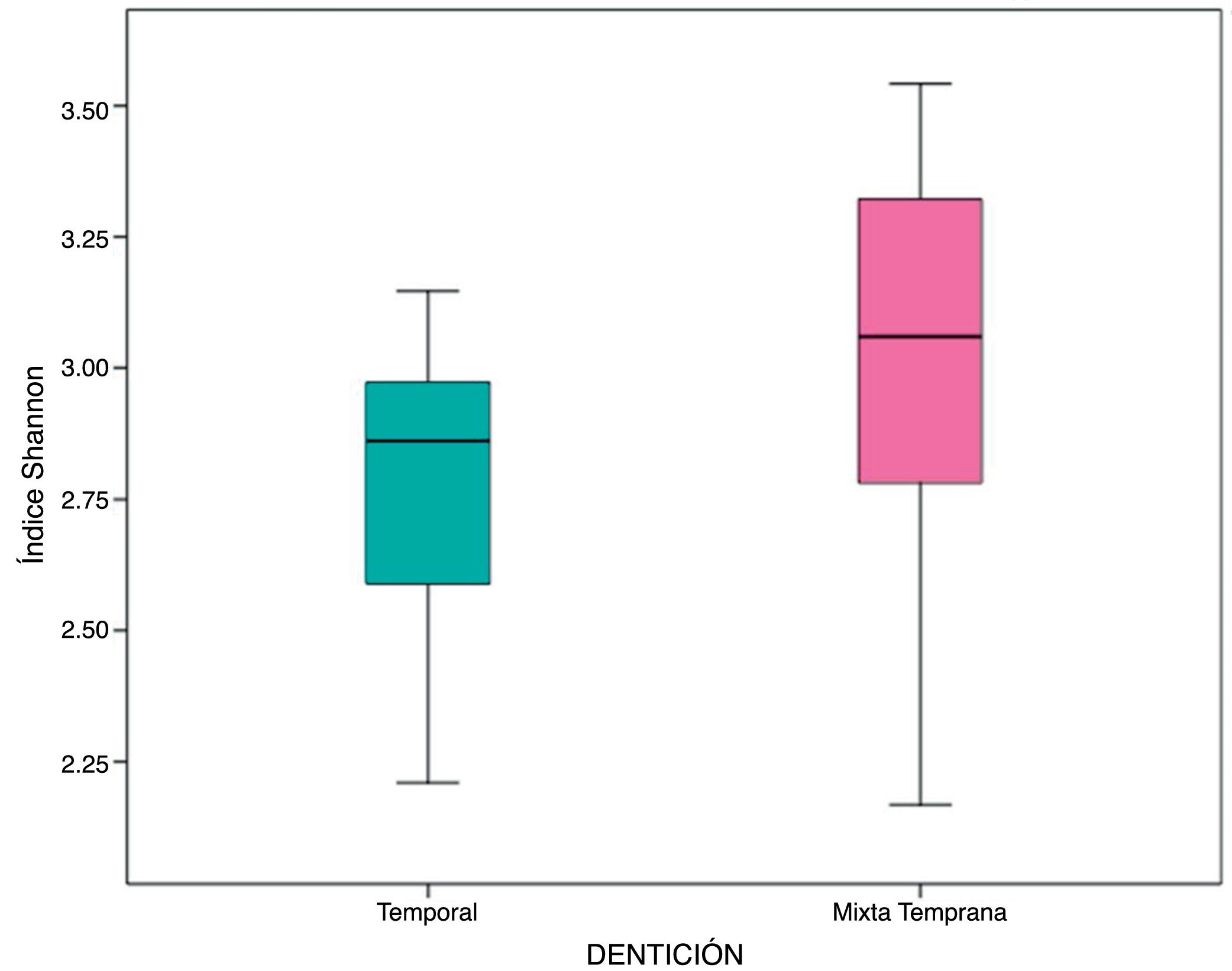
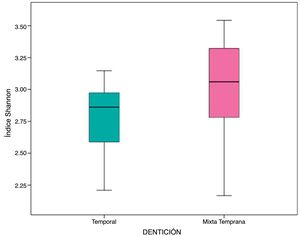
En las muestras de biofilm oral analizadas se observó riqueza de especies bacterianas representada por el índice α de Shannon 2,89 (DE: 0,35). Esta riqueza microbiológica se mantuvo entre los grupos de estudio, sin mostrar diferencia estadísticamente significativa (p=0,06) (tabla 3, fig. 2).
Índice α de Shannon de diversidad microbiológica
| General | D. temprana | D. mixta | p* | ||||
|---|---|---|---|---|---|---|---|
| Media | DE | Media | DE | Media | DE | ||
| Índice α de Shannon | 2,89 | 0,35 | 2,77 | 0,27 | 3,01 | 0,40 | 0,06 |
En los dientes encontramos una gran cantidad de bacterias que forman la biopelícula bacteriana. Algunas han sido implicadas en enfermedades bucales. Son importantes las investigaciones relacionadas con el conocimiento de la composición del bacterioma del biofilm supragingival: Kamma et al. investigaron la composición de la microbiota de la placa subgingival de los diferentes grupos de dientes en los niños con dentición mixta. En los resultados aseveraron que se identificaron grupos de microrganismos diferentes, tanto en dientes temporales como permanentes, y 45 especies microbianas diferentes fueron aisladas15. Xin et al. identificaron la diversidad microbiana oral de los niños sanos chinos. Tomaron muestras de la placa dental de 10niños y en los resultados reportaron un total de 41 géneros y 112 especies bacterianas16. Shi et al. realizaron un estudio en el que investigaron los perfiles microbianos de la placa supragingival de dientes temporales y permanentes en niños sanos con dentición mixta y detectaron un total de 21 clases, 38 órdenes, 66 familias y 74 géneros17, lo que confirma la variedad microbiana entre los dientes temporales y permanentes. Esto coincide con el presente estudio, en el que se describió el perfil bacteriano del biofilm dental en niños con dentición temporal y mixta temprana: se encontraron 44 géneros y 360 especies bacterianas presentes en el biofilm oral analizado, lo que demuestra las diferencias considerables en la diversidad microbiana entre la dentición mixta y temporal. Dichas diferencias podrían relacionares con factores físicos, químicos y ambientales.
Con relación al factor tiempo, Facal et al. estudiaron la topografía de las caries en los molares temporales y su relación con la edad cronológica. Afirmaron que la forma física de la corona de un diente termporal varía de la de un diente permanente: el temporal presenta una corona pequeña, con fosas y surcos poco profundos; el permanente, una corona de mayor tamaño, con fosas y surcos con más profundidad, lo que genera zonas de retención18. En el actual estudio, los dientes temporales tuvieron mayor oportunidad para el desarrollo y la maduración del biofilm supragingival, ya que el tiempo de exposición al ambiente en la cavidad bucal es más extenso (6,5 años) que el de los dientes permanentes (5 años). El factor nutricional es importante para el ambiente de la cavidad oral: la dentición temporal ha pasado por sustancias químicas involucradas en la nutrición como la leche artificial y también con alimentos de consistencia semisólida y sólida; mientras que la dentición mixta no se le relaciona con leche artificial, pero si con alimentos sólidos. Todos estos factores alimenticios podrían estar relacionados con la diversidad microbiológica encontrada.
Con respecto a los géneros bacterianos, Shi et al. estudiaron los perfiles microbianos del biofilm supragingival de dientes temporales y permanentes, y afirmaron que existen diferencias en la diversidad microbiana: de las muestras obtenidas, se encontraron diferencias en los dientes permanentes en el 25,2%; al compararlos con los dientes temporales, en un 20,9% y la diferencia en la comunidad microbiana a nivel de género se debió principalmente a Actinomyces para dentición permanente y a Treponema en dentición temporal17, lo que concuerda con el actual reporte, en el que se presentó diversidad microbiana en las muestras de estudio de dentición mixta temprana y temporal. Sin embargo, se discrepa respecto a la diferencia en la comunidad bacteriana a nivel de género, ya que los géneros bacterianos con mayor abundancia relativa que difieren de la comunidad microbiana en la dentición mixta temprana fueron Leptotrichia sonda 3, TM7 género y Neisseria género sonda 2, mientras que en dentición temporal fueron Actinomyces género sonda 3 y Fusobacterium sonda 4.
Xu et al. estudiaron el microbioma del biofilm bacteriano en niños con dentición temporal con caries y sin ella. Los géneros de mayor frecuencia presentes en el grupo de pacientes sin caries fueron Leptotrichia, Streptococcus, Actinomyces, Prevotella, Porphyromonas Neisseria y Veillonella19; lo que coincide con el presente estudio en algunos géneros como Streptococcus sonda 4, Streptococcus sonda 1, Actinomyces sonda 3, Veillonella sonda 2 y Porphyromonas sonda 2 y discrepa con el género Fusobacterium sonda 4.
ConclusiónLos géneros con mayor abundancia relativa en las muestras de biofilm procedentes de niños con dentición temporal fueron Streptococcus sonda 4, Streptococcus sonda 1, Actinomyces sonda 3, Veillonella sonda 2, Fusobacterium sonda 4 y Porphyromonas sonda 2; mientras que en los niños con dentición mixta temprana se observan Streptococcus sonda 4, Streptococcus sonda 1, Leptotrichia sonda 3, TM7 género, Porphyromonas sonda 2, Veillonella sonda 2 y Neisseria género. Respecto a las especies bacterianas, las de mayor abundancia relativa en las muestras de niños con dentición temporal fueron Streptococcus sanguinis, Rothia aeria, Gemella haemolysans, Gemella morbillorum, Veillonella dispar y Actinobaculum sp. HOT 183. Por su parte, en los niños con dentición mixta temprana se encontraron Streptococcus sanguinis, Leptotrichia sp. HOT 417, Leptotrichia sp. HOT 498, Gemella haemolysans, Granulicatella adiacens y Ottowia sp. HOT 894.
El perfil bacteriano del biofilm dental supragingival en niños con dentición temporal y mixta temprana muestra poca diversidad microbiológica tanto en presencia como en abundancia relativa y tanto a nivel de género como de especies bacterianas, sin mostrar resultados estadísticamente significativos.
RecomendacionesSe propone como recomendaciones para futuros estudios aumentar el tamaño de la muestra.
FinanciaciónEl presente trabajo ha sido financiado por Colciencias (contrato 673-2014), código N.°141965741160, y ha sido ejecutado por la Corporación Universitaria Rafael Núñez.
Conflicto de interesesLos autores declaran no tener conflicto de intereses.
Los autores agradecen a Colciencias por la financiación de este proyecto con el contrato N.° 673 de 2014 y a los participantes por su incorporación al estudio.