El diagnóstico asistido por computador (DAC) constituye una herramienta con gran potencial para ayudar a los endoscopistas en las tareas de detección y clasificación histológica de los pólipos colorrectales. En los últimos años se han descrito diferentes tecnologías y ha aumentado la evidencia sobre su potencial utilidad, lo que ha generado grandes expectativas en las sociedades científicas. Sin embargo, la mayoría de estos trabajos son retrospectivos y utilizan imágenes de diferente calidad y características que son analizadas off-line. En esta revisión se pretende familiarizar a los gastroenterólogos con los métodos computacionales y las particularidades de la imagen endoscópica con impacto en el análisis del procesamiento de imágenes. Finalmente, se exponen las bases de datos de imágenes disponibles de forma pública que son necesarias para poder comparar y confirmar los resultados obtenidos con diferentes métodos.
Computer-aided diagnosis (CAD) is a tool with great potential to help endoscopists in the tasks of detecting and histologically classifying colorectal polyps. In recent years, different technologies have been described and their potential utility has been increasingly evidenced, which has generated great expectations among scientific societies. However, most of these works are retrospective and use images of different quality and characteristics which are analysed off line. This review aims to familiarise gastroenterologists with computational methods and the particularities of endoscopic imaging, which have an impact on image processing analysis. Finally, the publicly available image databases, needed to compare and confirm the results obtained with different methods, are presented.
A pesar de las mejoras significativas producidas en los endoscopios, hasta un 21% de los pólipos no son visualizados durante la colonoscopia1. Es bien conocido que la tasa de detección de adenomas tiene una correlación inversa con la incidencia del cáncer colorrectal de intervalo y con la mortalidad asociada al cáncer colorrectal2. Por ello, es fundamental realizar una colonoscopia de buena calidad y disminuir el número de pólipos no detectados.
Por otro lado, para mejorar la eficacia de la colonoscopia, varios autores han propuesto la estrategia de «resecar y descartar»3 o «dejar in situ»4 basándose en una predicción de histología in vivo. De acuerdo con esto, un pólipo diminuto (≤5mm) localizado en el recto o colon sigmoide, con un diagnóstico óptico de alta confianza de pólipo hiperplásico y sin un síndrome de poliposis serrada asociado, podría dejarse in situ dado que su potencial maligno es nulo y tampoco precisa vigilancia5. Sin embargo, para poder aplicar esta estrategia se requiere un valor predictivo negativo (VPN) para el diagnóstico de adenoma ≥90% (cuando es utilizado con alta confianza), siguiendo las recomendaciones PIVI (Preservation and Incorporation of Valuable Endoscopic Innovations) de la Sociedad Americana de Endoscopia Digestiva (ASGE)6. Por otro lado, un diagnóstico óptico de alta confianza de un pólipo adenomatoso de menos de 10mm permitiría hacer una resección, sin necesidad de remitirlo a Anatomía Patológica para su diagnóstico histopatológico. Estas estrategias únicamente se podrían llevar a cabo por parte de endoscopistas que cumplan estas recomendaciones y, por tanto, muy expertos.
Para superar estas limitaciones, el diagnóstico asistido por computador (DAC) es una alternativa atractiva ya que podría ayudar a mejorar la tasa de detección y evitar los errores en la caracterización de los pólipos. En consecuencia, se podrían tomar decisiones de diagnóstico in vivo y esto optimizaría el manejo de los pacientes (tiempo, recursos y evitar complicaciones asociadas a polipectomías innecesarias).
Para poder desarrollar sistemas de DAC eficientes, el contenido de los vídeos e imágenes de colonoscopia debe ser analizado minuciosamente por expertos en procesamiento de imagen y aprendizaje computacional, que definirán modelos que permitan buscar aquellas lesiones o indicadores definidos por los médicos. En este contexto, la mayoría de los trabajos existentes se han centrado en el desarrollo de métodos para caracterizar con precisión los diferentes elementos de la escena endoluminal, prestando especial atención a los pólipos. Asimismo, la generación y adquisición de imágenes en alta calidad también es crucial para que estos métodos computacionales funcionen según lo previsto, ya que la presencia de artefactos de imagen tiene un impacto negativo en el rendimiento de los métodos de localización de pólipos7.
Particularidades de la imagen endoscópica con impacto en el análisis del procesamiento de imágenesLos desafíos más importantes que debe afrontar un determinado método computacional para brindar un soporte eficiente son aquellos relacionados con la adquisición y formación de la imagen y los vinculados a la caracterización de las estructuras anatómicas que aparecen en la escena endoluminal (fig. 1)8.
En cuanto a la adquisición y formación de imagen, algunas de las características que pueden afectar al rendimiento de un método de visión por computador son:
- -
La aparición de reflejos especulares y la ausencia de iluminación uniforme
- -
Los artefactos de color
- -
La baja resolución de la imagen
- -
El entrelazado
- -
La superposición de información (por ejemplo, información del paciente)
- -
El marco negro que rodea la imagen
- -
La compresión de la imagen
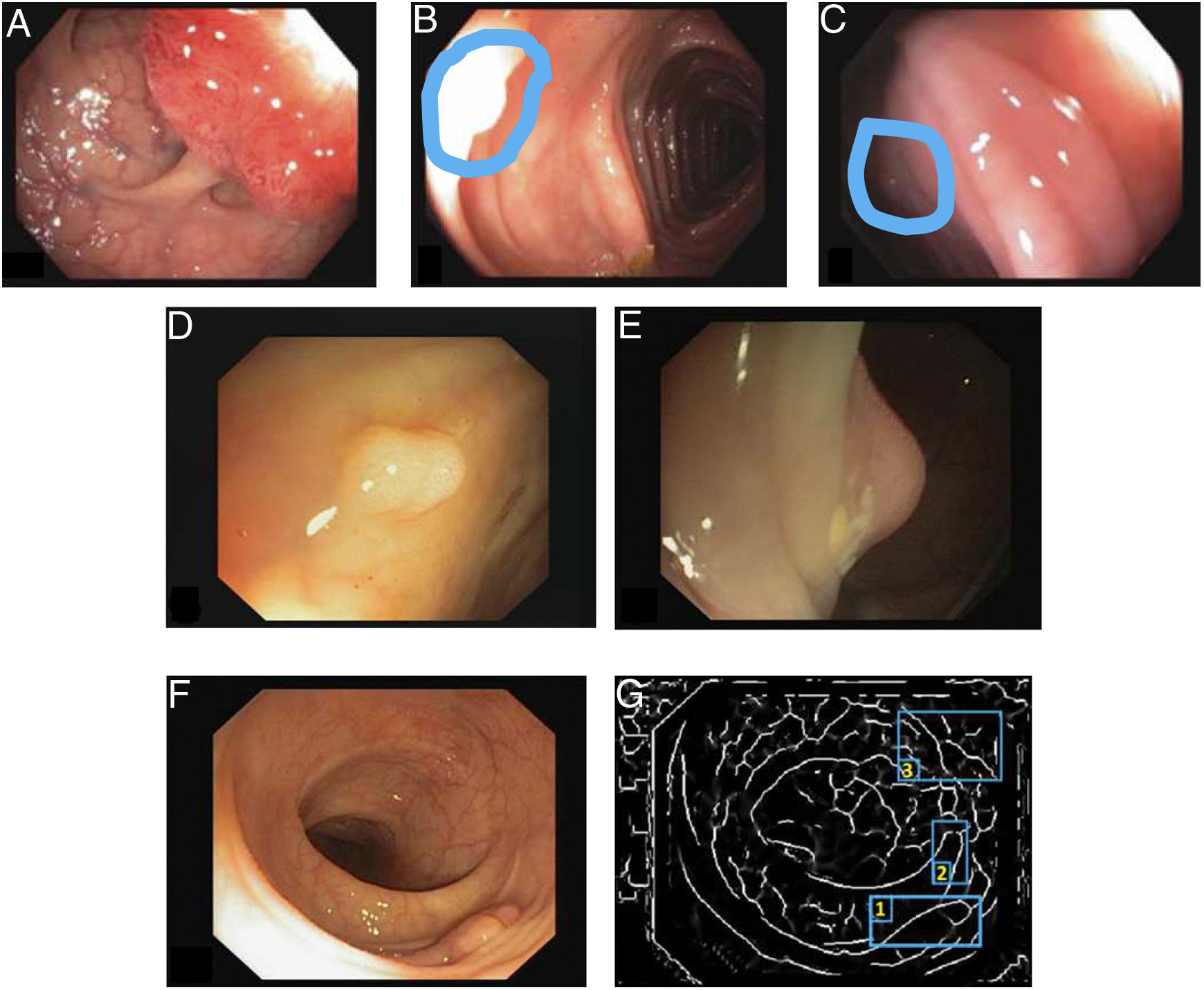
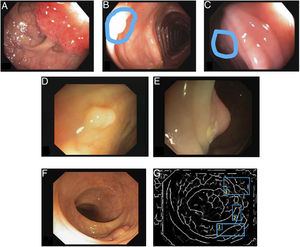
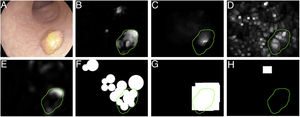
Los reflejos especulares son muy frecuentes cuando la iluminación es perpendicular a la superficie del pólipo y dificultan el procesamiento de la imagen7, ya que aparecen como estructuras muy prominentes que además ocultan la información de color y textura sobre las superficies en las que aparecen. Asimismo, la iluminación axial no es uniforme, de modo que las estructuras más cercanas al endoscopio se verán más brillantes que otras más lejanas (fig. 2A-C).
Particularidades de la imagen endoscópica con impacto en el análisis del procesamiento de imágenes. Ejemplos de efectos de iluminación: (A) reflejos especulares; (B) pólipo sobreexpuesto, y (C) pólipo subexpuesto. Los pólipos en las imágenes B y C se delimitan con una máscara azul para facilitar la visualización. Variabilidad en la apariencia del pólipo: (D) vista frontal; (E) vista lateral. Ejemplo de similitud de respuesta de diferentes estructuras (F y G). El número 1 representa un pólipo, el número 2 un pliegue y el número 3 varios vasos sanguíneos.
Con respecto a la caracterización de las estructuras anatómicas, la descripción de los diferentes elementos que aparecen en la escena endoluminal supone varios desafíos para los sistemas basados en visión por computador:
- -
Ausencia de apariencia uniforme de la estructura. Los pólipos no tienen una apariencia única y uniforme, sino que depende en gran medida del punto de vista en el que se observa (visión frontal o lateral) (fig. 2D y E). Por tanto, la mayoría de los métodos de procesamiento de imagen se centran en buscar características intrínsecas de cada estructura, de modo que puedan ser observadas en la mayoría de los casos, independientemente de cómo haya sido tomada la imagen.
- -
Impacto de otros elementos de la escena en la caracterización de un elemento particular. Siguiendo con el ejemplo de pólipo, la mayoría de los trabajos se basan en la identificación de los contornos de los pólipos que, en términos de procesamiento de imágenes, no son muy diferentes de los vasos sanguíneos y los pliegues, como se puede observar en la figura 2F y G. Teniendo esto en cuenta, un método computacional debe considerar el impacto de todas las estructuras presentes y encontrar elementos adicionales para diferenciarlas entre ellas.
Los métodos de detección tienen como objetivo decidir si hay un pólipo o no en la imagen, y habitualmente están diseñados para alertar a los endoscopistas con un sonido o marcador cuando se sospecha la presencia de un pólipo durante la realización de una colonoscopia.
Los métodos de localización de pólipos pueden entenderse como una extensión de los métodos de detección de pólipos. En este caso, además de revelar la presencia del pólipo, resaltan el área de la imagen donde está localizado.
Los métodos de detección y localización se suelen agrupar en 3 grupos según la metodología en la que se basan: (a) hand-crafted («a medida»), (b) machinelearning, y (c) híbridos (que combinan características de ambos grupos)9.
Métodos hand-craftedLos métodos de este grupo suelen basarse en una definición previa de un modelo de apariencia de la estructura anatómica a modelar y se enfocan en definir filtros y operaciones matemáticas específicas para cada problema a resolver, no garantizando su aplicación en otros dominios en los cuales fueron definidos. Debido a la naturaleza de estos, los métodos pertenecientes a este grupo no requieren de gran cantidad de datos para ser desarrollados, aunque su capacidad de adaptación a las apariencias reales de las estructuras depende de la complejidad del modelo que se haya usado como base.
En el caso de los pólipos de colon, la mayoría de las metodologías existentes se basan en el uso de herramientas de procesado de imagen de bajo nivel (tal como la extracción de contornos) para obtener aquellas partes de la imagen que son candidatas a ser parte de un contorno de pólipo. Para tal efecto, Iwahori et al.10 usan filtros de la matriz Hessiana, Bernal et al.7,11 usan los valles de intensidad, y Silva et al.12 usan la transformada de Hough. Esta información es usada para luego definir restricciones que sean solo aplicables a los contornos de los pólipos: Zhu et al.13 usan la curvatura de dichos contornos como característica clave, mientras que Kang y Doraiswami14 se centran en discriminar aquellos contornos con forma elíptica, típicamente asociada a los pólipos.
Métodos machine learningLos sistemas de aprendizaje computacional son aquellos que, una vez han sido entrenados para realizar una tarea determinada a partir de ejemplos, son capaces de replicarla en un conjunto de datos nuevos. Se suelen definir 3 etapas diferentes en el diseño de estos sistemas: entrenamiento, validación y prueba. Las 2 primeras se hacen sobre un mismo conjunto de datos que se conoce completamente (tanto datos de entrada como salida esperada), mientras que los datos del conjunto de prueba son desconocidos en la fase de desarrollo del sistema.
Diversos grupos de investigación han utilizado métodos de machine learning para la detección de pólipos, definiendo el conjunto de características a extraer de la imagen (de forma, color o textura) y el método de aprendizaje a emplear (desde árboles de decisión a support vector machines).
Respecto al tipo de características empleadas, Karkanis et al.15 propusieron el empleo de wavelets de color mientras que Ameling et al.16 exploraron el uso de características de textura a través de las matrices de coocurrencia. El trabajo de Gross et al.17 también se enfocó en el uso de características de textura mediante local binary patterns. En cuanto al método de aprendizaje, Angermann et al.18 propusieron el empleo de técnicas de aprendizaje activo mediante el cual el sistema mejora a partir del aprendizaje de ejemplos positivos y negativos.
Debido al incremento de la disponibilidad de herramientas de computación de alta capacidad, como las GPU (unidades de procesamiento gráfico), el uso de técnicas basadas en redes neuronales convolucionales (deep learning) también se ha explorado para tareas de detección de pólipos. La ventaja del uso de técnicas basadas en deep learning es el hecho de que pueden aprender a partir de grandes volúmenes de datos, sin tener que especificar qué tipo de característica han de buscar en ello. Dentro de este grupo de métodos, las diferencias principales radican en la arquitectura de red específica empleada y en las bases de datos utilizadas para el entrenamiento y validación. Liu et al.19 emplean la arquitectura ResNet para la detección de pólipos, obteniendo un sistema que funciona en tiempo real (procesa más de 25 imágenes por segundo) con una tasa de detección cercana al 80% una vez testado en la base de datos CVC-VideoClinicDB. Zheng et al.20 tratan el problema de la detección de pólipos en 2 etapas: en la primera intentan delimitar todas las estructuras de la imagen empleando la arquitectura U-Net y luego aplican técnicas de seguimiento (tracking) para obtener la localización final del pólipo. Su metodología alcanza valores de sensibilidad superiores al 95%, testado también en la base de datos CVC-VideoClinicDB21. Un último ejemplo interesante lo encontramos en el trabajo de Qadir et al.22, que combina una primera arquitectura Faster RCNN para seleccionar regiones candidatas a contener pólipo y una segunda etapa enfocada en la eliminación de falsos positivos mediante la inclusión del análisis de imágenes anteriores. Este sistema alcanza valores de precisión superiores al 90% en la base de datos CVC-VideoClinicDB.
Validación de metodologíasComo se puede extrapolar, es complicado realizar una comparativa entre los diferentes métodos debido a la falta de bases de datos públicas donde realizar una validación de estos. Con el ánimo de solventar este problema, y animados por el uso creciente y exitoso de técnicas de aprendizaje computacional en otros ámbitos, se ha llevado a cabo un esfuerzo en la recolección, anotación y publicación de bases de datos que contienen imágenes y vídeos de colonoscopias. A partir de ello, se han llevado a cabo diversos challenges internacionales donde concurren diferentes equipos poniendo a prueba sus diferentes propuestas para la detección automática de pólipos. A fecha de hoy se han llevado a cabo 4 ediciones diferentes: 1) Automatic polyp detection challenge en la conferencia IEEE International Symposium on Biomedical Imaging (ISBI), celebrada en Nueva York en 2015 y organizada conjuntamente por Arizona State University (ASU) y el Centro de Visión por Computador (CVC), 2) Automatic polyp detection in colonoscopy videos, dentro del marco de la conferencia International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI), celebrada en Múnich en 2015 y organizada conjuntamente por ASU, CVC y el laboratorio ETIS de ENSEA. A partir del año 2017 se amplió el alcance del challenge incluyendo tareas adicionales como la segmentación de pólipos y la detección de lesiones en imagen de cápsula endoscópica, dentro del marco del challenge GIANA (Gastrointestinal Image Analysis). Se han celebrado 2 ediciones de GIANA dentro de la conferencia MICCAI: en 2017 en Quebec y en 2018 en Granada, ambas seguidas con un gran interés por parte de la comunidad científica, participando más de 20 equipos en la última edición.
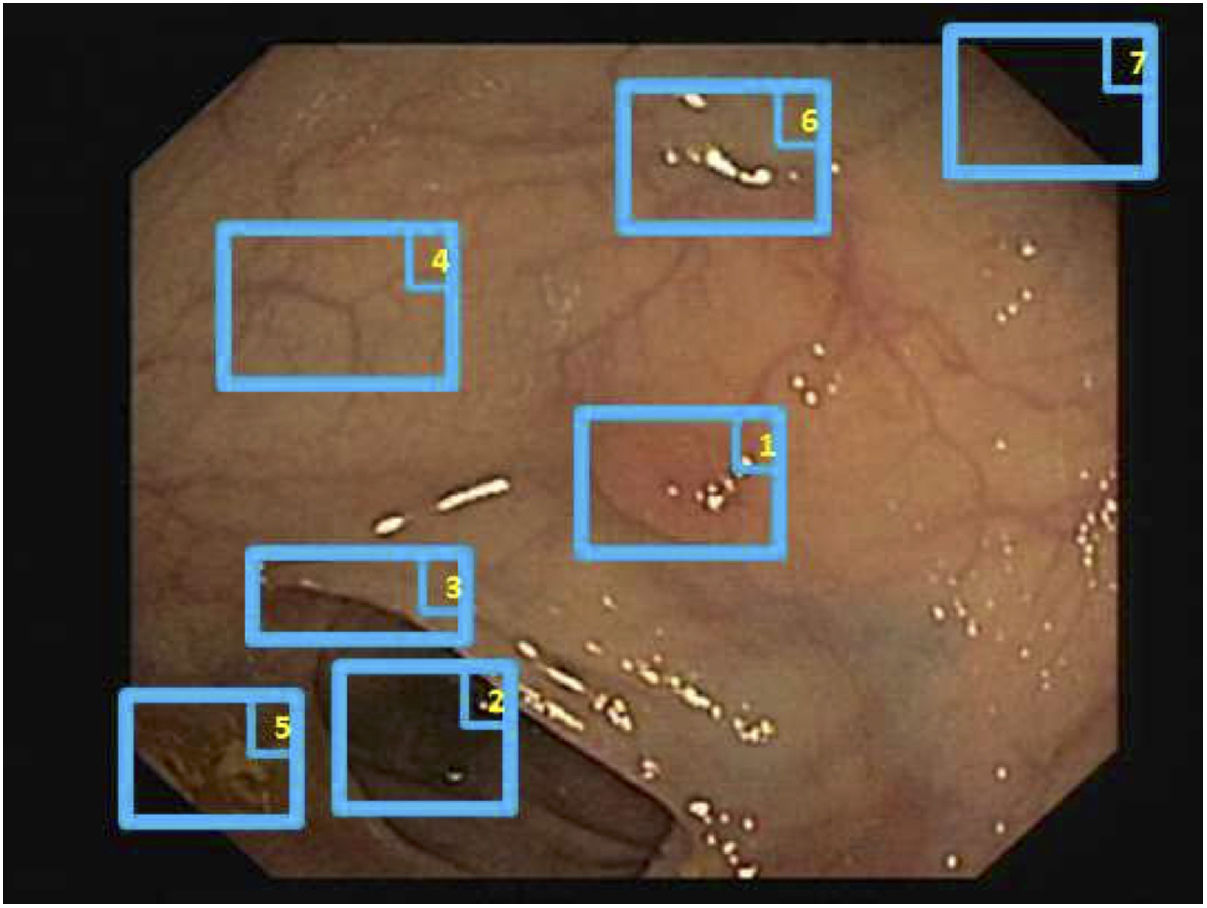
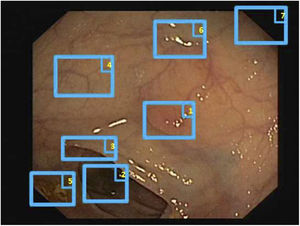
El primer challenge de detección de pólipos celebrado dentro del marco de la conferencia MICCAI en 2015 sirvió asimismo para realizar un estudio comparativo entre 8 metodologías de detección automática de pólipos usando la misma base de datos, definiendo asimismo un marco común de validación que puede servir para comparar metodologías en un futuro9 (fig. 3), mostrando un rendimiento superior en los que utilizaron metodología de machine learning. Dicha tendencia se ha ido manteniendo en los challenges posteriores, donde se ha visto también la generalización del uso de técnicas de deep learning debido a su rendimiento y, sobre todo, a la rapidez de cálculo asociado al uso de tarjetas gráficas de alta capacidad.
Comparación de los mapas de energía proporcionados por cada método participante en el challenge de detección de pólipos celebrado en el congreso MICCAI de 2015: (A) Imagen original; (B) CUMED; (C) CVC-CLINIC; (D) PLS; (E) UNS-UCLAN; (F) ETIS-LARIB; (G) OUS, y (H) SNU. La línea verde de cada imagen representa la máscara del pólipo de referencia.
Varios grupos han desarrollado sistemas de DAC para la caracterización de los pólipos colorrectales23,24. La mayoría de estos métodos requieren equipamiento de diagnóstico óptico avanzado, como NBI (narrow band imaging), endocitoscopia u otros y algunos de ellos alcanzan el umbral requerido del 90% de VPN en pólipos diminutos de recto-sigma, descrito en el documento PIVI de la ASGE6. El análisis de las imágenes también utiliza metodología muy diferente que va desde el análisis automático de algunas características de la textura de superficie del pólipo o el color hasta los métodos de deep learning. Sin embargo, es común a todos ellos la identificación semiautomática de la zona correspondiente al pólipo ya que el endoscopista debe indicar la región de interés centrando el pólipo en la imagen25 o poniéndolo en contacto con el endoscopio26,27. Los resultados de los estudios publicados se encuentran resumidos en la tabla 1.
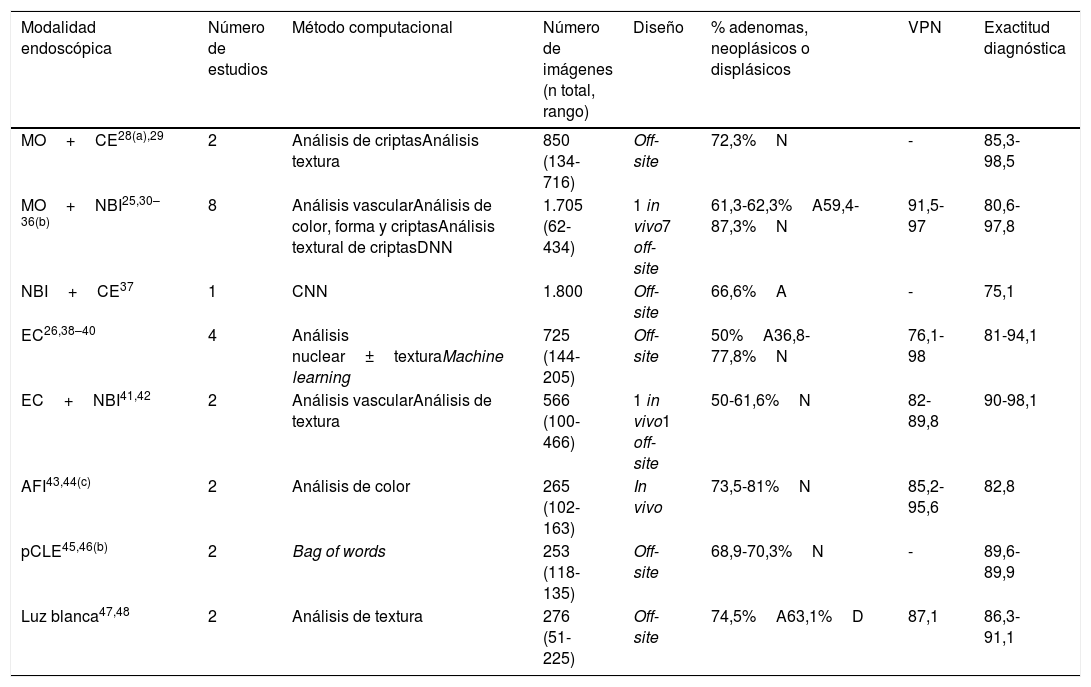
Estudios de predicción histológica computacional de pólipos colorrectales basados en análisis de imagen
| Modalidad endoscópica | Número de estudios | Método computacional | Número de imágenes (n total, rango) | Diseño | % adenomas, neoplásicos o displásicos | VPN | Exactitud diagnóstica |
|---|---|---|---|---|---|---|---|
| MO+CE28(a),29 | 2 | Análisis de criptasAnálisis textura | 850 (134-716) | Off-site | 72,3%N | - | 85,3-98,5 |
| MO+NBI25,30–36(b) | 8 | Análisis vascularAnálisis de color, forma y criptasAnálisis textural de criptasDNN | 1.705 (62-434) | 1 in vivo7 off-site | 61,3-62,3%A59,4-87,3%N | 91,5-97 | 80,6-97,8 |
| NBI+CE37 | 1 | CNN | 1.800 | Off-site | 66,6%A | - | 75,1 |
| EC26,38–40 | 4 | Análisis nuclear±texturaMachine learning | 725 (144-205) | Off-site | 50%A36,8-77,8%N | 76,1-98 | 81-94,1 |
| EC+NBI41,42 | 2 | Análisis vascularAnálisis de textura | 566 (100-466) | 1 in vivo1 off-site | 50-61,6%N | 82-89,8 | 90-98,1 |
| AFI43,44(c) | 2 | Análisis de color | 265 (102-163) | In vivo | 73,5-81%N | 85,2-95,6 | 82,8 |
| pCLE45,46(b) | 2 | Bag of words | 253 (118-135) | Off-site | 68,9-70,3%N | - | 89,6-89,9 |
| Luz blanca47,48 | 2 | Análisis de textura | 276 (51-225) | Off-site | 74,5%A63,1%D | 87,1 | 86,3-91,1 |
A: adenoma; AFI: autofluorescence imaging; CE: cromoendoscopia; CNN: convolutional neural network; D: displásico; DNN: deep neuronal network; EC: endocitoscopia; MO: magnificación óptica; N: neoplásico; NBI: narrow band imaging; pCLE: probe-based confocal laser endomicroscopy; VPN: valor predictivo negativo.
Los estudios de Takemura et al.28 y Hafner et al.29 utilizaron cromoendoscopia y magnificación óptica. El primero analizó computacionalmente el patrón de criptas de modo cuantitativo en 134 imágenes y el segundo examinó diversas características de textura en 716 imágenes (72,3% pólipos neoplásicos). Takemura obtuvo una extraordinaria exactitud diagnóstica (98,5%), pero el patrón oro fue la clasificación de Kudo. En cambio, el estudio de Hafner obtuvo una exactitud diagnóstica del 85,3%; la sensibilidad y especificidad fue del 88,6 y 76,6%, respectivamente.
Magnificación óptica y NBIVarnavas et al.30 describieron en 2009 un primer estudio clínico con 62 imágenes (61,3% de adenomas) empleando un método de DAC basado en el cálculo de características vasculares en imágenes con NBI y magnificación, con una exactitud diagnóstica, sensibilidad y especificidad del 80,6, 81,6 y 79,2%, respectivamente, en la diferenciación de adenomas colorrectales. Posteriormente, Tischendorf et al.31 publicaron en 2010 un estudio clínico prospectivo con la misma modalidad endoscópica cuya metodología computacional propuesta incluye las siguientes etapas: 1) preprocesado de imágenes, 2) segmentación de estructuras vasculares, 3) extracción de características de las estructuras vasculares (longitud, brillo, perímetro y otras) y 4) clasificación final de la imagen. Estos autores utilizaron 209 imágenes (76,6% pólipos neoplásicos) y obtuvieron una sensibilidad, especificidad y exactitud diagnóstica del 90, 70,2 y 85,3%, respectivamente, en la diferenciación entre pólipo neoplásico y no neoplásico. Gross et al.32 en 2011 alcanzaron un rendimiento diagnóstico del 93,1% también con una metodología basada en NBI y magnificación y análisis del mapa vascular en 434 imágenes (59,4% pólipos neoplásicos); la sensibilidad, especificidad y valores predictivos también superaron el 90%. Takemura et al.33 en 2012 también utilizaron un número elevado de imágenes (n=371, 87,3% de pólipos neoplásicos), analizaron el color, la forma y el tamaño de las criptas y obtuvieron todavía mejores resultados: exactitud diagnóstica 97,8%, con similar sensibilidad y especificidad. También con la misma modalidad endoscópica el estudio de Tamai et al.34 analizó computacionalmente características de la vascularización del pólipo (121 imágenes, 82,6% de pólipos neoplásicos) y obtuvo los siguientes resultados: sensibilidad 83,9%, especificidad 82,6%, valor predictivo positivo (VPP) 53,1%, VPN 95,6% y exactitud diagnóstica 82,8%. El estudio de Chen et al.35 recientemente publicado utilizó la metodología computacional deep neural network en 284 imágenes (66,2% de pólipos neoplásicos) y obtuvo una sensibilidad del 96,3%, especificidad 78,1%, VPP 89,6%, VPN 91,5% y exactitud diagnóstica 90,1%.
Por otra parte, Byrne et al.36 también aplicaron la metodología computacional deep neural network para el reconocimiento en tiempo real (pero no in vivo) de pólipos adenomatosos/hiperplásicos, con un 94% de eficacia diagnóstica, una sensibilidad del 98%, especificidad 83%, VPP 90% y VPN 97%, analizando vídeos (no imágenes aisladas) de colonoscopia con NBI y near focus.
Sin embargo, estos estudios se basaban en imágenes analizadas posteriormente y no durante la exploración. En cambio, Kominami et al.25 describieron un algoritmo más robusto y adecuado para su uso clínico en tiempo real, que ofrecía clasificación in vivo de pólipos durante la endoscopia. El estudio incluyó 118 imágenes (62% de pólipos neoplásicos), analizó características texturales de las criptas, y obtuvo un rendimiento del 93,2% para la diferenciación de lesión neoplásica/no neoplásica; la sensibilidad, especificidad y valores predictivos también superaron el 90%.
NBI y cromoendoscopiaKomeda et al.37 utilizaron las redes neuronales convolucionales (convolutional neural network) en 1.800 imágenes (66,6% adenomas) extraídas de vídeos de colonoscopia con luz blanca, NBI y cromoendoscopia, alcanzando una exactitud diagnóstica para la diferenciación de adenomas del 75,1%.
EndocitoscopiaLa endocitoscopia también ha sido utilizada para la clasificación automática de pólipos, ya que proporciona imágenes enfocadas y de tamaño fijo que permiten un análisis de imagen más fácil y robusto mediante DAC. Los primeros modelos publicados por Mori et al.26,38,39 se basaron en la extracción automática de características magnificadas 380× del núcleo y de los elementos con vascularización. Estas características se integran en un sistema clasificador de machine learning y permiten alcanzar una exactitud diagnóstica en torno al 90% para la identificación de adenomas/lesiones neoplásicas, y con un retardo al proporcionar el resultado de solo 0,2-0,3 segundos, una vez la imagen ha sido capturada. De este modo, el tipo de pólipo se predice en tiempo real y se da una probabilidad. Si esta es >90%, el sistema indica «alta confianza» de tratarse por ejemplo de un adenoma26. Posteriormente, Takeda et al.40 utilizaron también endocitoscopia con una magnificación de aproximadamente 400×. Entrenaron el método con 5.543 imágenes de 238 lesiones y lo aplicaron a 200 imágenes. La sensibilidad, especificidad, VPP, VPN y exactitud diagnósticas obtenidas en esta serie fueron: 89,4, 98,9, 98,8, 90,1 y 94,1%. En 2018, Mori et al.39 publicaron otra serie con 144 imágenes (36,8% pólipos neoplásicos) y obtuvieron los siguientes resultados: sensibilidad 98%, especificidad 71%, VPP 67%, VPN 98% y exactitud diagnóstica 81%.
Endocitoscopia y NBITambién se han publicado estudios con endocitoscopia y NBI. Misawa et al.41 utilizaron 100 imágenes magnificadas 380× (50% pólipos neoplásicos) y analizaron la vascularización. Obtuvieron un 90% de exactitud diagnóstica, sensibilidad 84,5%, especificidad 97,6%, VPP 98% y VPN 82%. Recientemente Mori et al.42 han mejorado sus resultados utilizando endocitoscopia con magnificación 520× y NBI en 466 imágenes (61,6% de pólipos neoplásicos) en tiempo real e in vivo, analizando características de textura y obteniendo una exactitud diagnóstica en la valoración de pólipos diminutos neoplásicos/no neoplásicos del 98,1% (457/466); la sensibilidad, especificidad y valores predictivos fueron en torno al 90%.
AutofluorescenciaAFI (autofluorescence imaging) es otra modalidad endoscópica que ha sido utilizada para la predicción histológica endoscópica de los pólipos colorrectales mediante el análisis computacional numérico del color. Se ha descrito un rendimiento diagnóstico del 82,8% con esta técnica43,44. Se trata de estudios in vivo que utilizaron 102 y 163 imágenes (73,5 y 81% de pólipos neoplásicos, respectivamente) y obtuvieron una sensibilidad, especificidad, VPP y VPN del 94,2, 88,9, 95,6 y 85,2% en el estudio de Aihara et al.43 y del 83,9, 82,6, 53,1 y 95,6% en el estudio de Inomata et al.44.
Endomicroscopia láser confocalTambién se ha desarrollado un software que usa la técnica de extracción de características y machine learning, conocida como Bag of words, mediante la cual se detectan y caracterizan regiones de interés en imágenes de endomicroscopia láser confocal (CLE), alcanzando un rendimiento equivalente a endoscopistas expertos (exactitud diagnóstica próxima al 90%). Andre et al.45 utilizaron 135 imágenes (68,9% de pólipos neoplásicos) y obtuvieron una sensibilidad, especificidad y exactitud diagnóstica del 92,5, 83,3 y 89,6%, respectivamente. Tafreshi et al.46 utilizaron 118 vídeos (70,3% de pólipos neoplásicos) y sus resultados fueron, respectivamente: 90,4, 88,6 y 89,9%.
Luz blancaFinalmente, nuestro grupo (Sánchez et al.47) realizó un primer análisis utilizando patrones de superficie (textones) de los pólipos con imágenes de luz blanca de alta definición. Tras un preprocesado de la imagen que mitigaba los reflejos especulares, se analizó la tubularidad de los textones. En total se incluyeron 38 adenomas y 13 pólipos no adenomatosos. Mediante una curva ROC se obtuvo el punto de corte de tubularidad de 13,14 y se utilizó este valor para discriminar entre adenoma y no adenoma. Con este punto de corte se obtuvo una exactitud diagnóstica global del 86,3% (44/51); para los adenomas fue del 94,7% (36/38), pero para los no adenomas descendió al 61,5% (8/13). Posteriormente se perfeccionó el método, analizándose también la ramificación y el contraste de los patrones texturales en superficie y se amplió el número de imágenes (n=225, 63,1% displásicos), mejorando la exactitud diagnóstica global al 91,1%48; en cambio, el VPN global alcanzado fue del 87,1% y por tanto no cumplía el criterio PIVI de la ASGE6. No obstante, el VPN ascendía al 96,7% en el subgrupo de pólipos diminutos de recto y sigma (n=54), con una exactitud diagnóstica del 90,6%.
Combinación de métodos de detección y predicción histológicaUn sistema ideal debería combinar la detección automática de pólipos seguida de su caracterización a tiempo real e in vivo. En este sentido, Mori et al. han descrito un modelo preliminar (sin análisis estadístico) basado en un algoritmo de deep learning para detectar pólipos en imágenes de luz blanca y otro algoritmo que predice su histología mediante endocitoscopia49. Hasta el momento no existen otros modelos que combinen ambas metodologías de detección y predicción histológica, no obstante, los estudios in vivo de clasificación histológica descritos previamente son los que de entrada podrían combinarse con métodos de detección automática25,42–44.
Bases de datosLas bases de datos de imágenes apropiadamente anotadas son necesarias para validar los métodos computacionales, así como para facilitar la comparativa entre ellos y garantizar que estos representen adecuadamente el problema. Por tanto, los casos incluidos en una base de datos deben representar toda la variabilidad clínica que el médico puede encontrar durante las intervenciones. Además, cuanto más diferentes sean los ejemplos, más robusto será el método; y si además logramos que un método determinado ofrezca un buen rendimiento en una base de datos, será fácil extrapolarlo para una posible aplicación clínica.
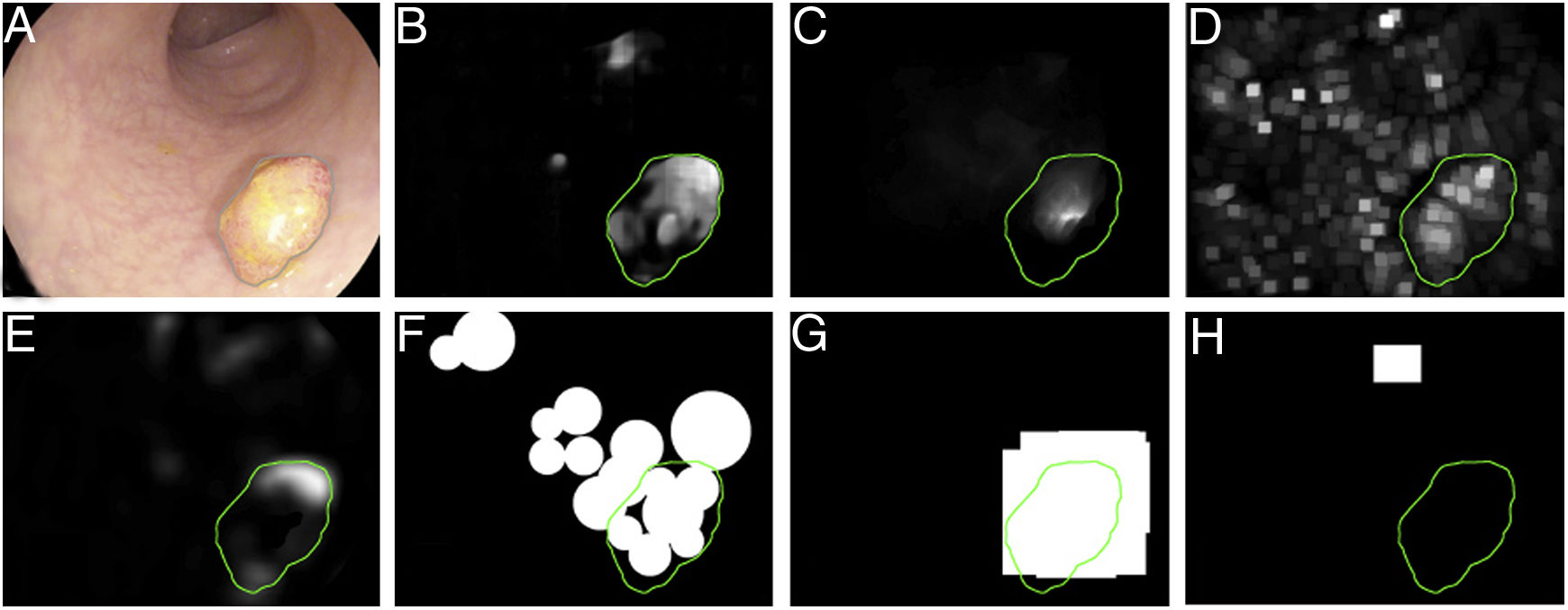
Una vez que se ha definido la base de datos, debe crearse un ground truth para evaluar/validar el rendimiento del método que será totalmente dependiente del tipo de aplicación. Por ejemplo, en un método de detección de pólipos, el ground truth consistirá en un archivo Excel que indica si hay o no un pólipo en cada imagen y únicamente se requerirá el etiquetado de la imagen como verdadero/falso. En cambio, un método de localización implica definir el área de la imagen que ocupa el pólipo y hacer máscaras ajustadas al pólipo en la imagen, obteniendo una imagen binaria que represente la estructura8. Finalmente, la clasificación de pólipos requiere asignar una etiqueta a la imagen según la categoría histológica.
Existe la necesidad de colaboración de los médicos para realizar estas bases de datos por los siguientes motivos: (a) un ground truth realizado por personal clínico es siempre más fiable que el realizado por personal técnico, (b) el médico conoce la naturaleza clínica del problema y puede decidir con mejor criterio qué casos incluir y cuáles descartar y (c) involucrar al personal facultativo especialista en la validación de los métodos es clave para asegurar un posible uso clínico de las herramientas, al estar ya sometidos a estándares médicos.
Las diferentes bases de imágenes de colonoscopia existentes y sus aplicaciones se detallan en la tabla 27,9,21,50.
Principales características y aplicaciones de las bases de datos disponibles para el análisis de imágenes de colonoscopia
| Base de datos | Número de imágenes/vídeos | Resolución imágenes (píxeles) | Ground truth realizada por médicos | Pública | Contenido del ground truth | Aplicaciones | |||
|---|---|---|---|---|---|---|---|---|---|
| Detección | Localización | Segmentación | Clasificación | ||||||
| CVC-ColonDB50 | 380 imágenes de 15 secuencias diferentes con un pólipo (definición estándar) | 500×574 | No | Sí | Para cada imagen con un pólipo se proporcionan las siguientes imágenes: 1) imagen original; 2) máscara de pólipo; 3) máscara de regiones no informativas y 4) contorno de pólipo | No | Sí | Sí | No |
| CVC-ClinicDB7 | 612 imágenes de 24 secuencias diferentes con un pólipo (definición estándar) | 384×288 | Sí | Sí | Para cada imagen, se proporcionan tanto la original como una con máscara que cubre el pólipo. Para cada pólipo, se proporcionan metadatos clínicos asociados (tamaño, clasificación de París, histología del pólipo) | Sí | Sí | Sí | No |
| ASU-Mayo9 | Entrenamiento: 20 vídeos (10 con un pólipo y 10 sin pólipos)Validación: 18 vídeos (definición estándar) | 712×480 | No | No | Para cada imagen del vídeo, se proporciona una imagen binaria. La ausencia de pólipo en la imagen puede identificarse al tener una imagen completamente negra asociada. En caso de presencia de pólipos, se proporciona una aproximación de la región del pólipo | Sí | Sí | No | No |
| ETIS-Larib9 | 196 imágenes con 44 pólipos de 34 secuencias (alta definición) | 1.225×966 | No | Sí | Para cada imagen con un pólipo se proporciona la imagen original y otra con las máscaras del pólipo | No | Sí | Sí | No |
| CVC-VideoClinicDB21 | Entrenamiento: 18 vídeosValidación: 18 vídeos (definición estándar) | 384×288 | Sí | Sí | Para cada imagen del vídeo, se proporciona una imagen binaria. La ausencia de pólipo en la imagen puede identificarse al tener una imagen completamente negra asociada. En caso de presencia de pólipos, se proporciona una elipse que incluye la región del pólipo | Sí | Sí | No | No |
A pesar del tremendo potencial de los métodos de DAC, ninguno de los métodos de detección y clasificación de pólipos colorrectales descritos hasta la actualidad ha sido implementado ampliamente en la práctica habitual por las siguientes razones9: (a) necesidad de rapidez para su funcionamiento en tiempo real; (b) algunos métodos de detección se construyen a partir de un modelo teórico de la apariencia del pólipo7 que puede estar limitado a ciertas morfologías; (c) no consideran la presencia de otros elementos tales como pliegues, lesiones subepiteliales, compresiones extrínsecas, vasos sanguíneos o la luz7; (d) algunos métodos solo se han probado en imágenes (no vídeos) seleccionadas de buena calidad que se adaptan bien a sus requisitos técnicos (buena limpieza, pólipo centrado, pólipo de un cierto tamaño y forma, …); (e) la existencia de desafíos específicos de la imagen de colonoscopia, como los artefactos de imagen generados, debido a la iluminación (reflejos especulares, regiones sobreexpuestas) o a la configuración específica del videoprocesador adjunto al colonoscopio, que podría superponer información sobre la escena a visualizar, y (f) validación frecuente en bases de datos pequeñas y restringidas, con un dispositivo específico (marca de endoscopio, resolución y método auxiliar —NBI, AFI, endocitoscopia, luz blanca, etc.), e incluso dedicándose solo a un tipo específico de pólipo o morfología.
ConclusiónLa colonoscopia tiene un papel primordial en el diagnóstico y prevención del cáncer colorrectal, pero no es infalible y depende de factores como la calidad de la exploración y la experiencia del endoscopista. El DAC (o inteligencia artificial) ha emergido en los últimos años como un complemento a la colonoscopia y es una alternativa muy atractiva para estandarizar la práctica de esta exploración y homogeneizar los resultados. Sin embargo, a pesar del tremendo potencial de los métodos de DAC, los resultados de que disponemos son aún insuficientes debido a la limitada calidad de las publicaciones y que ninguno de los métodos descritos hasta la actualidad ha sido implementado ampliamente en la práctica habitual. Por último, para poder corroborar los resultados obtenidos y realizar estudios comparativos es necesario disponer de bases de imágenes con datos clínicos asociados que sean compatibles con todos los métodos. Por tanto, es necesario seguir investigando para encontrar un método de DAC útil en tiempo real, que sea compatible con diferentes marcas de endoscopios y que no se base en ninguna técnica compleja, sea poco disponible y que requiera además de un elevado nivel de experiencia.
Conflicto de interesesG. Fernández-Esparrach ha recibido honorarios por organizar cursos de Norgine Iberia y Olympus España en los últimos dos años y ha sido consultora de CDx Diagnostics.
Este trabajo fue apoyado por el Gobierno de España a través de los proyectos financiados iVENDIS (DPI2015-65286-R) e HISINVIA (PI17/00894), por la Fundación de la Sociedad Española de Endoscopia Digestiva (FSEED), por la Secretaria d’Universitats i Recerca de la Generalitat de Catalunya (2014-SGR-1470, 2014-SGR-135, SGR-2017-1669 y SGR-2017-653), y por el programa CERCA de la Generalitat de Catalunya.