Existen dos métodos de recuperación de información de documentos propios de la ciencia genómica y de medicina en general, a saber: uno está basado en el uso combinado de las relaciones determinadas por el Medical Subject Headings, mientras que el otro emplea las terminologías particulares, como pueden ser folksonomías, nombres alternativos de los términos médico-genómicos de uso en el lenguaje más general o los acrónimos o apocópes comunes en áreas como la genómica. Numerosos teóricos e indizadores consideran que la combinación de dos métodos puede funcionar mejor y es capaz de ofrecer mejoras significativas. Pese a que son pocos los autores que pugnan por no modificar la estructura de los vocabularios controlados, construidos a través de años de interpretación de contenidos, la multiplicidad de propuestas se reúnen bajo la tendencia de expandir el horizonte de búsqueda de los tesauros, ya sea con la catalogación social, el análisis de dominio realizado con algoritmos que contrastan indicadores o la web semántica, a través de la propuesta de marcado de unidades lexicales significativas en los textos digitalizados.
There are two methods of retrieving information from documents in the field of genomic science and medicine in general, namely: 1) through the combined use of associations determined by the Medical Subject Headings, and 2) by employing specific terminologies, such as in folksonomies, alternative medical-genomic terms in use in the general language, or acronyms or apocopes from the genomics field. To some extent, many thinkers and indexers hold that the combination of two methods may be the best approach. While few authors advocate for keeping the structure of controlled vocabularies, built up over many years of content interpretation, unchanged, there are numerous proposals for expanding the search horizons of thesauri, whether through social cataloging, algorithmic domain analyses that contrast indicators or the semantic web using markers of meaningful semantic lexicons contained in digitized text.
Los encabezamientos de temas médicos o MeSH, por sus siglas en inglés (Medical Subject Headings), son un corpus de términos conformados por la National Library of Medicine (NLM) de Estados Unidos de América. Estas unidades léxicas son utilizadas para indizar y recuperar documentos sobre biomedicina y ciencias genómicas —además de áreas tangenciales— en la base de datos de citas y resúmenes MEDLINE, a través de PubMed, que es un motor de búsqueda de libre acceso. Ambos recursos también son ofrecidos por la Biblioteca Nacional de Medicina de Estados Unidos. PubMed cuenta con una amplia variedad de herramientas automatizadas (PubMed Tools) entre las cuales destacan por su eficacia BioSample, Assembly o Genome, entre otras; por ejemplo, este último recurso organiza la información genómica e incluye secuencias, mapas, representación gráfica de cromosomas y anotaciones mediante tres procedimientos principales. Cada una de las herramientas utiliza diferentes métodos para recuperar información. En el caso de Genome se ha corroborado que el desarrollo de las estructuras tesaurales es la forma más idónea y eficaz de representación y recuperación de información (Chute, 2005). No obstante, desde hace algunos años, se han planteado otros métodos y enfoques (algunos novedosos) que buscan expandir las posibilidades de indización y consulta de los documentos, sin que esto implique abandonar el uso de los MeSH (Bodenreider, Rindflesch y Burgun, 2002: 54).
Algunas de estas propuestas funcionan a partir del hecho de que la mayoría de los términos, por ejemplo, utilizan varias denominaciones, ya sean sinónimos (fitomenadiona=vitamina K), siglas (dihidroxiacetona-fosfato=DHAP) o apócopes (coccidioidomicosis → coccidiosis), e incluso falta consenso en la escritura (Zweigenbaum y Grabar, 2004), y a que por lo general sólo se utiliza un término para la consulta de un documento, es decir, este método aprovecha que un término tiene varias derivaciones y nombres alternativos y los utiliza para enriquecer la búsqueda. En este caso la variedad de nombres no implica que uno deba fungir como el término correcto y el resto sean no autorizados, ya que, en el caso de la información genómica, cada uno es empleado en diferentes situaciones. No es igual un texto recuperado de una revista de divulgación científica sobre salud pública que la publicación de los resultados sobre el mapeo del genoma del cáncer en un fascículo de alta especialización. Ambos son documentos de carácter científico, pero con una mirada disímil; van dirigidos a diferentes comunidades y sobre todo, satisfacen diferentes necesidades. No es lo mismo disertar en un artículo académico sobre el ácido ascórbico (C6H8O6) que hablar en una cápsula informativa acerca de la vitamina C, se trata de la misma sustancia pero el nombre denota una diferente intención para informar.
ENFOQUES DE RECUPERACIÓN A TRAVÉS DEL USO DE LA ESTRUCTURA DE UN TESAUROEl nivel de especificidad o la comunidad no son los únicos criterios para discernir entre la utilización de un término o de otro. La BIREME (Centro Latinoamericano y del Caribe de Información en Ciencias de la Salud), que surge como la Biblioteca Regional de Medicina en 1967 apoyada por la Organización Mundial de la Salud (OMS) y la Organización Panamericana de la Salud (OPS), ha conformado los Descriptores en Ciencia de la Salud (DeCS), basados en los Mesh, donde incorpora términos en español, portugués y agrega algunas áreas de medicina como homeopatía o vigilancia sanitaria, también en inglés. Este vocabulario apoya a la Biblioteca Virtual de Salud (BVS) y a lilacs, que es el índice más importante de la salud en América Latina y el Caribe. Hay comunidades, como la francesa, que no siempre optan por el uso de los encabezamientos médicos y a veces se inclinan por emplear nombres que divergen de los autorizados por la nlm. Esto se debe en parte a los juegos lingüísticos implícitos en la vida cotidiana o por la adopción léxica de su comunidad, y quizá para este escenario estaríamos hablando de folksonomías (Zweigenbaum et al., 2003). Por ejemplo, el sistema médico CISMeF (Catalogue et Index des Sites Médicaux de Langue Française) releva el uso de los Mesh o de otros vocabularios con los metadatos, es decir, que alternan el rigor de un vocabulario controlado con nombres alternativos emanados de los estudios de comunidad para adoptar la catalogación social como otra opción (Deacon, Smith y Tow, 2001). Algunos autores como Mary Rajathei David y Selvaraj Samuel han propuesto un método diseñado para PubMed, denominado FNeTD (Frequent Nearer Terms of the Domain), basado en el uso de los términos más utilizados en una determinada comunidad médica para recuperar información de una manera más eficiente. Estos términos pueden ser o no los autorizados por la nlm, o incluso ser derivaciones, el criterio primordial es que sean los utilizados verdaderamente en el acontecer diario de la práctica médica (Rajathei David y Samuel, 2012: 20).
El CISMeF busca establecer como su línea editorial las descripciones precisas de los documentos, basadas la mayoría de los casos en los mesh, pero modificadas y mejoradas. Busca de manera constante nuevos enfoques de descripción y recuperación de información médica (Kerdelhué, 2007). Algunos colaboradores del CISMeF, adscritos al Hospital Universitario de Rouen, encabezados por Magaly Douyère, han buscado adaptar la terminología médica más amplia y general usada en Internet, en lugar de recurrir, en primera instancia, a los artículos científicos de la base de datos bibliográfica MEDLINE. Como ya se mencionó, CISMeF utiliza dos herramientas estándar para la organización de la información: los mesh y varios conjuntos de elementos de metadatos en formato Dublin Core. Sin embargo, el carácter heterogéneo de los recursos de información sobre salud en Internet llevó al equipo CISMeF a emprender la búsqueda para mejorar los mesh; primero, con el diseño de un algoritmo aleatorio que se basó en otorgar valores determinados a los enlaces semánticos (Névéol, 2004), cuestión que resultó sumamente exhaustiva e insuficiente; segundo, con la introducción de dos nuevos conceptos: los tipos de recursos y los meta-términos. Un tipo de recurso describe la naturaleza del documento y no sólo su materia, como sucede con las palabras clave y los calificadores basados en los mesh. Un meta-término suele ser un término amplio (como el nombre de una disciplina o tratamiento médico) que ofrece conexiones semánticas entre los mesh y los tipos de recursos. CISMeF permite dos opciones de búsqueda: la simple y la avanzada. La búsqueda simple requiere que el usuario dé entrada a un solo término o expresión, esto se complementa con una búsqueda de texto completo. En la búsqueda avanzada, las búsquedas complejas se realizan con la combinación de posibles operadores booleanos con meta-términos, palabras clave, nombres alternativos y tipos de recursos. En este tipo de búsqueda se opta por combinar dos herramientas de búsqueda: el mesh y el formato de metadatos Dublin Core. Por tanto, los documentos son descritos con las dos herramientas en conjunto: el título, el autor o creador, el tema y las palabras clave, la descripción, los editores, fecha, tipo de recurso, formato, identificador y el lenguaje (Darmoni, 2001: 167).
Bundschus y sus colaboradores de la Universidad de Múnich y la compañía Siemens han optado por el término meta-información, el cual utilizan para complementar información, a diferencia de la distinción explícita entre tipos de recursos y meta-términos realizada en Francia. La cuestión es que al enriquecer los sistemas de información médica con la inclusión de nuevos documentos, y cuando se indizan con los términos del mesh, pueden complementarse las descripciones de éstos con información adicional: “Esta meta-información proporciona una rica fuente de conocimiento, que puede ser explotada para el descubrimiento de conocimiento biomédico y las tareas de minería de datos” (Bundschus et al., 2008: 11). Además se agrega que: El modelo término/concepto descubre información novedosa de un conjunto de textos sobre biomedicina, incluyendo la extracción de la estructura del concepto de un tema oculto, utilizando todos los términos mesh que concurren en ese conjunto […]. En contraste con los modelos temáticos estándar, en donde los temas son representados exclusivamente por las palabras más probables, la estructura tema-concepto puede ser interpretada como una representación temática más rica, sobre todo por la vinculación con los conceptos del mesh. Por lo tanto, esta representación temática enriquecida proporciona una importante información adicional, como una ontología terminológica (Bundschus et al., 2008: 16. Las cursivas son nuestras).
El equipo de Bundschus, especialista en cienciometría, ha explorado paralelamente aplicaciones como la extracción de relaciones estadísticas entre los temas genéricos y los términos mesh, encaminada a la extracción automatizada de información (Leydesdorff, Rotolo y Rafols, 2012).
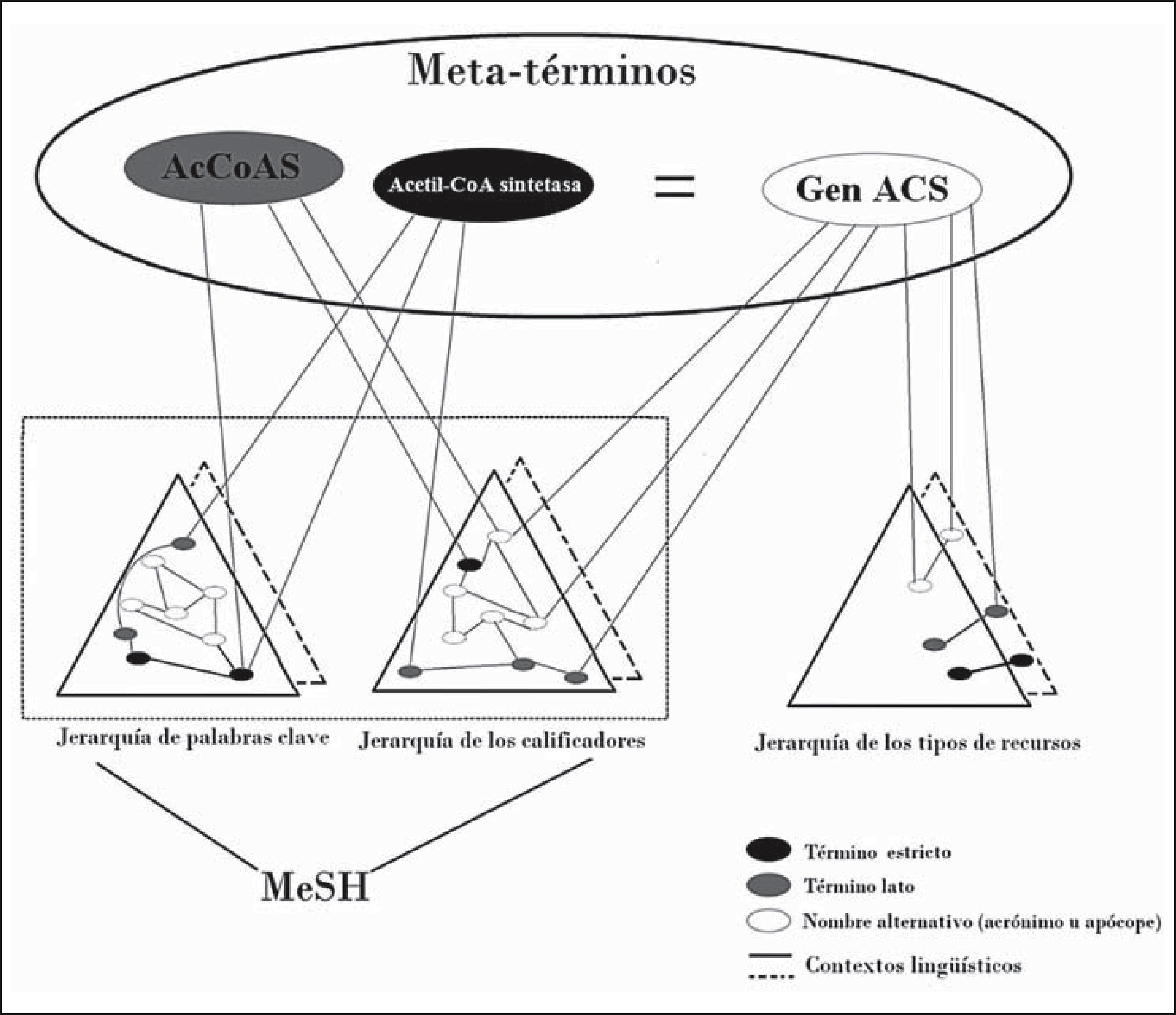
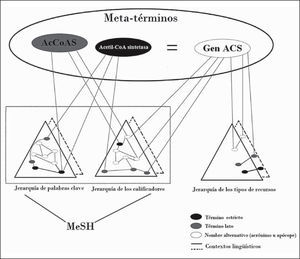
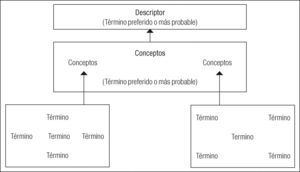
De acuerdo con las propuestas que no circunscriben su búsqueda sólo a un término autorizado, se encuentra la investigación de dos españoles en la cual se expone que el gen accoas tiene nueve nombres alternativos dispersos en la literatura biomédica, todos registrados en el principal catálogo sobre genes humanos, el Online Mendelian Inheritance in Man (OMIM): CG9390, acetato-coenzima-A-ligasa, acetil-CoA sintetasa, acetil coa sintasa, Acetil CoA sintasa, acs, Acetil-CoA synthasa, Acetil CoA sintetasa, best:gh2840 (Galveza y Moya-Anegón, 2006: 345). Con base en este ejemplo, es posible mostrar el funcionamiento de la propuesta del CISMeF (Figura 1).
Ejemplificación del funcionamiento de la propuesta de CISMeF.
Fuente: Douyère et al., 2004: 255.
Pero el esfuerzo del CISMeF por renovar el mesh no es nuevo. En los albores del siglo XXI, la Red de Información sobre la Salud de Austria (Gesundheitsinformationsnetz Österreich o GIN, por sus siglas en alemán) ofrecía información a los pacientes (denominados por ellos como consumidores de conocimiento) no sólo de tipo preventiva, sino conocimiento médico fiable sobre las enfermedades, el bienestar y la gestión de la enfermedad de una manera fácil de comprender que les permitiera el acceso rápido a la información acerca de sus padecimientos para entender la diagnosis, además de ofrecer datos específicos sobre el sistema de salud y las organizaciones sanitarias austriacas. Pese a que las descripciones ofrecidas por la GIN estaban controladas inicialmente por el tesauro mesh, se percataron que los usuarios a menudo no utilizan los términos y expresiones científicas para entender su diagnóstico, por ello buscaron adecuar la terminología coloquial con la más rigurosa a partir de un método vectorial, de tal forma que los usuarios pudieran hacer uso del sistema de información (Göbel, 2001: 242-244). Al igual que el CISMeF, utilizaron un algoritmo informático para realizar las búsquedas automatizadas, en este caso el algoritmo de Floyd-Warshall.
Por otro lado, Radu Serban y Annette ten Teije (2009), holandeses, ponen en alta estima al vocabulario controlado como herramienta de representación y recuperación de información, y sopesan que su estructura no debe ser modificada, ni mucho menos alternar el vocabulario especializado de sentido más estricto con uno más lato. Por el contrario, Edgar Meij y su grupo de archivistas de la Universidad de Ámsterdam establecen que el mejor método para recuperar información debe basarse en las relaciones complejas de un vocabulario controlado, pero consideran que cada registro descriptivo de un término mesh debe ser equivalente a un documento sobre ese término (y no que un término equivalga o recupere varios documentos). Con esta idea sería inadmisible descartar la inmensa variedad de nombres alternativos que tiene un término, mismos que son útiles para la expansión de los métodos de recuperación de información e, incluso, pueden representar mejor a un documento que un propio MeSH (Meij, 2005; Nelson, Johnson y Humphreys, 2001: 177).
En otro caso, en una comunidad epistémica en Suiza, se emplea una terminología particular para un grupo de investigadores, de manera específica en el Swiss-Prot Group (cuestión que acaece con frecuencia en este tipo de comunidades). Este grupo de investigación alterna el uso de los mesh con su terminología particular para darle mayor fidelidad a la búsqueda (Mottaz, 2006: 18). Dieuwke Brand-de Heer aducía hace más de diez años que: […] ciertamente medline no cubre “la totalidad” de la literatura médica. Otras bases de datos contienen información adicional a medline, por ejemplo Excerpta Medica, otra base de datos que cubre temas médicos, y que en algunos campos se comporta mejor que medline, por ejemplo en farmacología. También está biosis previas que contiene información adicional relevante para médicos, misma que no contempla medline. (2001: 112)
Tomaz Bartol, especialista esloveno sobre literatura médica, considera que el uso de términos más amplios es útil, ya que mejora la recuperación de los documentos pertinentes. Él realizó un estudio muy reciente sobre la información en medicina herbal y arguyó que: En nuestro estudio, hemos puesto especial importancia a la cuestión de la co-ocurrencia de términos diferentes, especialmente descriptores, en el mismo documento. Este tipo de investigación implica generalmente descriptores basados en diccionario de sinónimos, por ejemplo mesh. Los términos y nombres alternativos de los términos obtienen sentido sólo en los “contextos de su uso”. Los sistemas de clasificación tradicionales, sin embargo, con frecuencia son resistentes al contexto. Los términos de indización en los tesauros se basan generalmente en vastas estructuras rígidas y en jerarquías predefinidas por lo que no siempre se pueden servir con eficacia a los efectos de un tema específico y a su consiguiente recuperación. (Bartol, 2012: 286)
En clara sintonía con Meij, la sentencia de Bartol expone la necesidad de expandir los horizontes de búsqueda con vocabularios controlados y enriquecer su propia estructura. Bartol pone como ejemplo el caso del término origanum, que en algunas bases de datos se sustituye con su equivalente salvia. Si consideramos la visión de Meij y su equipo, no se podrían recuperar los mismos documentos cuando se usa origanum que cuando se usa salvia, pese a que ambos refieren a la misma planta, ya que sus denominativos denotan contextos diferentes, por ello deben describir a documentos diferentes (cada término para cada documento). Bartol señala que es un tanto erróneo vincular ambos términos en la descripción con la finalidad de que en una base de datos se pueda recuperar el mismo documento a través de ambos puntos de acceso. Cada uno debe representar una necesidad de información diferente.
LA UNIFICACIÓN DEL LENGUAJE MÉDICOEl uso de los nombres alternativos poco a poco ha influenciado a la propia estructura de los mesh, y más ahora que se incluyen registros que contienen descripciones de los encabezamientos médicos, pero también de sus nombres alternativos. Estas descripciones incluyen notas de alcance sobre las nomenclaturas, siglas y referencias a los nombres utilizados con anterioridad.
La National Library of Medicine desde hace unos años ha agregado otra herramienta lingüística además de los encabezamientos de temas médicos, el Metatesauro del Sistema de Lenguaje Médico Unificado (umls por sus siglas en inglés), en el que también colaboran otros sistemas de información médica. A medida que avanzan los apoyos tecnológicos son aprovechados para crear nuevas formas de indizar con la finalidad de recuperar información. El Macrotesauro pretende en lo inmediato erigirse como una ontología que integre el conocimiento de diversos tesauros y de otras fuentes, no para expandir la búsqueda, sino para especificar la recuperación de información (Humphreys y Schuyler, 1993).
En este sentido, Hassan, Htroy y Palombi (2010) proponen dos enfoques principales para representar el conocimiento médico:
- •
Enfoque basado en imágenes: atlas clásicos, atlas informáticos y atlas probabilísticos. Estos atlas proporcionan un modelado para algunos órganos y el etiquetado de estos órganos suele ser manual.
- •
Enfoque basado en ontologías. Una ontología es, por definición, una representación formal de un conjunto de conceptos dentro de un dominio, además de las relaciones entre estos conceptos.
“Una ontología es una especificación formal y explícita de una conceptualización compartida”, esta definición se acuñó en 1998 por Studer, Benjamin y Fensel. Pastor Sánchez la retoma y explica de la siguiente manera: Con el término conceptualización se refieren a un modelo abstracto de una realidad concreta que se obtiene tras haber identificado los conceptos relevantes de la misma. Por explícita se alude a que el tipo de concepto usado y a las restricciones para su uso son explícitamente definidas. Formal se refiere al hecho de que la ontología debería ser legible por ordenador, y compartida refleja la noción de que una ontología captura conocimiento que no es objeto de un solo individuo, sino aceptado por un grupo de forma consensuada. (2011: 29. Las cursivas son nuestras)
El Metatesauro es sólo una de las herramientas del umls, el cual también está integrado por otras herramientas como el Semantic Network y specialistLexicon. El umls ha sido un proyecto trabajado lentamente, que pretende combinar sus tres herramientas para conseguir una eficaz recuperación de la información. El Metatesauro se encarga de los conceptos, Semantic Network de las categorías y relaciones y specialist Lexicon de los recursos y herramientas (Kostoff, 2004: 518). El Metatesauro inició en 1988 y está constituido a partir de las versiones automatizadas de diversos tesauros y listas de encabezamientos en diferentes idiomas además del inglés (español, francés, holandés, italiano, japonés y portugués), códigos y listas de términos controlados utilizados en la atención al paciente —como la mencionada GIN—, las estadísticas de salud pública y la indización de la literatura biomédica.
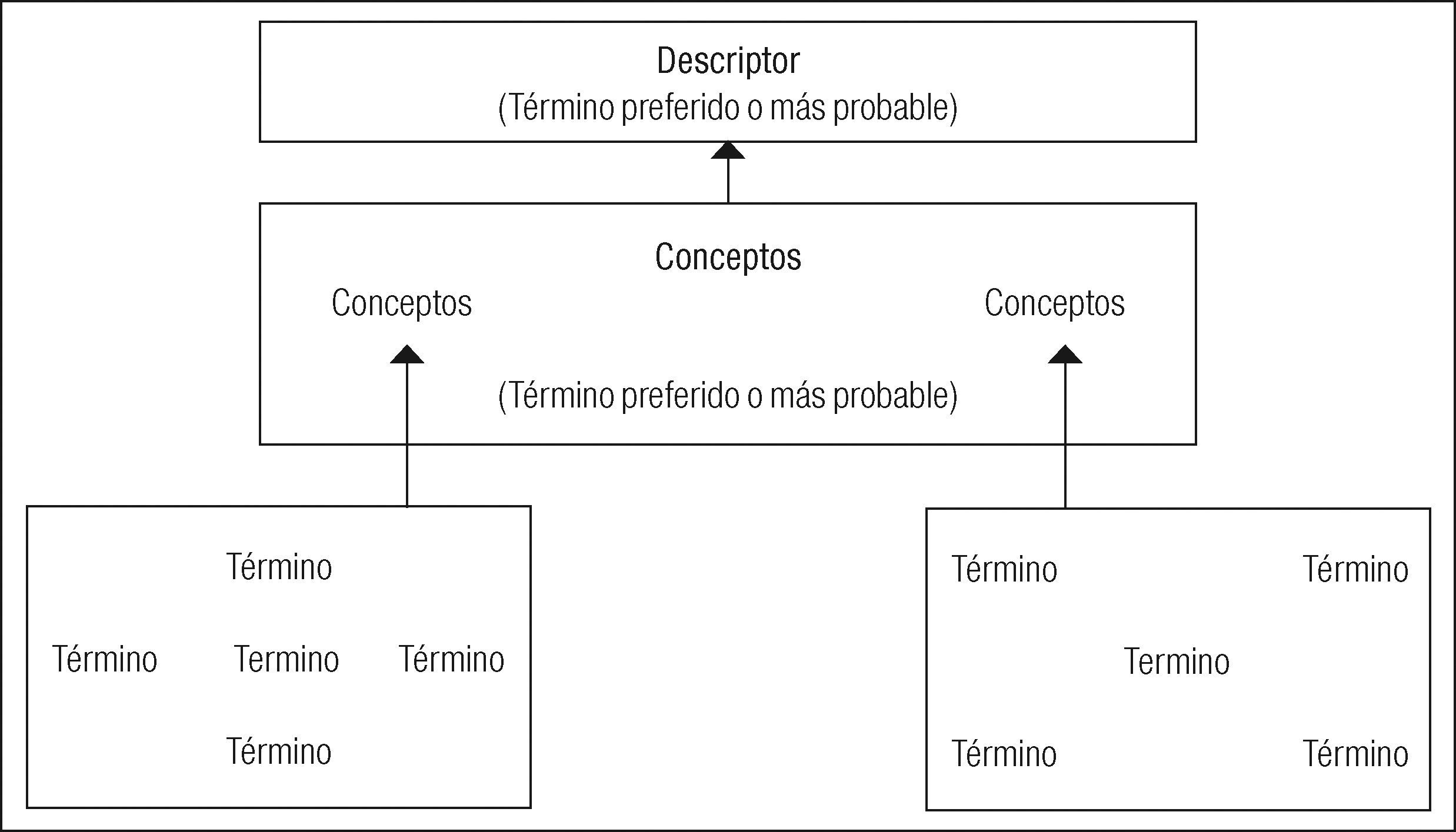
Los términos del Metatesauro se organizan por sentido y se les asigna un identificador único de concepto (con varios identificadores léxicos vinculados), se organizan todos los datos originales del vocabulario fuente, las definiciones o variantes de escritura. Los MeSH han tenido la limitante del retraso en la adopción de una nueva terminología. Incluso el Metatesauro no siempre puede incorporar lo más novedoso en temas de manera oportuna. Por esta razón, el uso de los metadatos por parte del Metatesauro a partir de 2004 produjo un severo cambio en la forma en que se gestionaban los documentos y formatos de la nml (Figura 2).
Para dar cabida a la descripción compleja que incluye a las siglas y a las abreviaturas indizadas en el Systematized Nomenclature of Medicine, umls desarrolló su propio formato denominado Rich Release Format (RRF) (Chute, 2005: 176). Para una recuperación integral de información, tanto en términos mesh como con nombres alternativos se deben incorporar mejores estrategias de búsqueda. No obstante, la salvedad que tiene el Metatesauro es que se vale de metadatos para dar mayor alcance a la búsqueda, que con los encabezamientos resulta insuficiente. Cuenta con cerca de cuarenta etiquetas, de ellas las más representativas son las siguientes (Tabla 1):
Principales eitquetas del formato Rich Release Format (RRF)
| Etiquetas | Características |
|---|---|
| MRCONSO.RRF | Nombres, sinónimos, términos, tipos de términos |
| MRREL.RRF | Relaciones semánticas |
| MRFILES.RRF | Todos los archivos de un subconjunto |
| MRHIER.RRF | Jerarquías |
| MRSAT.RRF | Atributos |
| MRDEF.RRF | Definiciones |
| MRMAP.RRF | Asignaciones |
| MRSMAP.RRF | Asignaciones simplificadas |
| MRSTY.RRF | Tipos semánticos (organismos, estructuras anatómicas, funciones biológicas, conceptos e ideas) |
Con estas funciones la búsqueda con el Metatesauro se enriquece y tan sólo se trata del funcionamiento de una de las tres herramientas del UMLS (Figura 3).
Funcionamiento de la búsqueda del Metatesauro umls a partir de los ficheros de metadatos y las relaciones establecidas entre términos, conceptos y descriptores.
Fuente: Mottaz, 2006: 8.
Christopher Chute agrega, desde una perspectiva más bien informática: “Anteriormente, el proceso de formateo de UMLS daba lugar a una transferencia de información ‘con pérdidas’. La visión moderna del umls es convertirse en la fuente definitiva y el formato para la publicación de los principales terminologías biomédicas, lo que significa mucho avance” (2005: 176-177). Esto indica que la pretensión de la unificación del lenguaje por parte de la nml no es sólo en cuanto a los términos, sino el mejoramiento de los sistemas computacionales. UMLS ha pretendido establecer un formato de intercambio de información para el área médica que, poco a poco, contribuye a su cometido. No obstante, el proyecto más adelantado sigue siendo el del CISMeF: En CISMeF los recursos se describen utilizando un conjunto de metadatos sobre la base de una terminología estructurada que “encapsula” el tesauro MeSH en su versión francesa. Ahora, el objetivo es migrar la terminología CISMeF, y por lo tanto el MeSH, a una ontología formal, a fin de obtener una más potente herramienta de búsqueda (Soualmia, Golbreich y Darmoni, 2004: 1).
En la actualidad, la terminología particular de CISMeF se ha “formalizado” en Web Ontology Language (OWL), en su versión DL, a diferencia de los tesauros ontologizados que están en la versión owl-Full.
CONCLUSIONESLos diversos casos en casi todo el plano internacional apuntan a que es necesario expandir el horizonte de representación y de recuperación de información. La estructura de los vocabularios controlados requiere ser “complementada” con otro tipo de métodos. Pese a la creencia generalizada acerca de que el uso de un lenguaje lato arroja una lista con una ingente cantidad de resultados, en realidad en el ámbito de la salud la terminología amplia puede ser muy específica a la hora de describir un documento y es probable recuperar términos en sentido estricto.
Finalmente, tarde o temprano, los tesauros tendrán que integrarse de lleno a la web semántica, quizá como ontotesauros (si se le quiere ver a los tesauros como ontologías). La combinación, en ciencias genómicas y de la salud, de las relaciones semánticas de un tesauro, una terminología alternativa y los metadatos y motores de búsqueda crearían una herramienta con una potencialidad inimaginable, y esta es una oportunidad en la que la bibliotecología tiene mucho que aportar.