Las alteraciones genómicas desequilibradas (duplicaciones o deleciones) causantes de trastornos del neurodesarrollo (TND) son en su mayoría episodios de novo. Sin embargo, también pueden surgir como consecuencia de reordenamientos equilibrados no detectados en uno de los progenitores, cambiando radicalmente el riesgo de recurrencia y el consejo genético de estos casos. La técnica de fluorescence in situ hybridization (FISH, «hibridación in situ fluorescente») permite la identificación y localización de reordenamientos cromosómicos tanto equilibrados como desequilibrados, identificando la ubicación de los segmentos duplicados. En este trabajo se pretende localizar en el genoma los segmentos duplicados detectados en pacientes con TND, e identificar aquellos casos debidos a reordenamientos heredados.
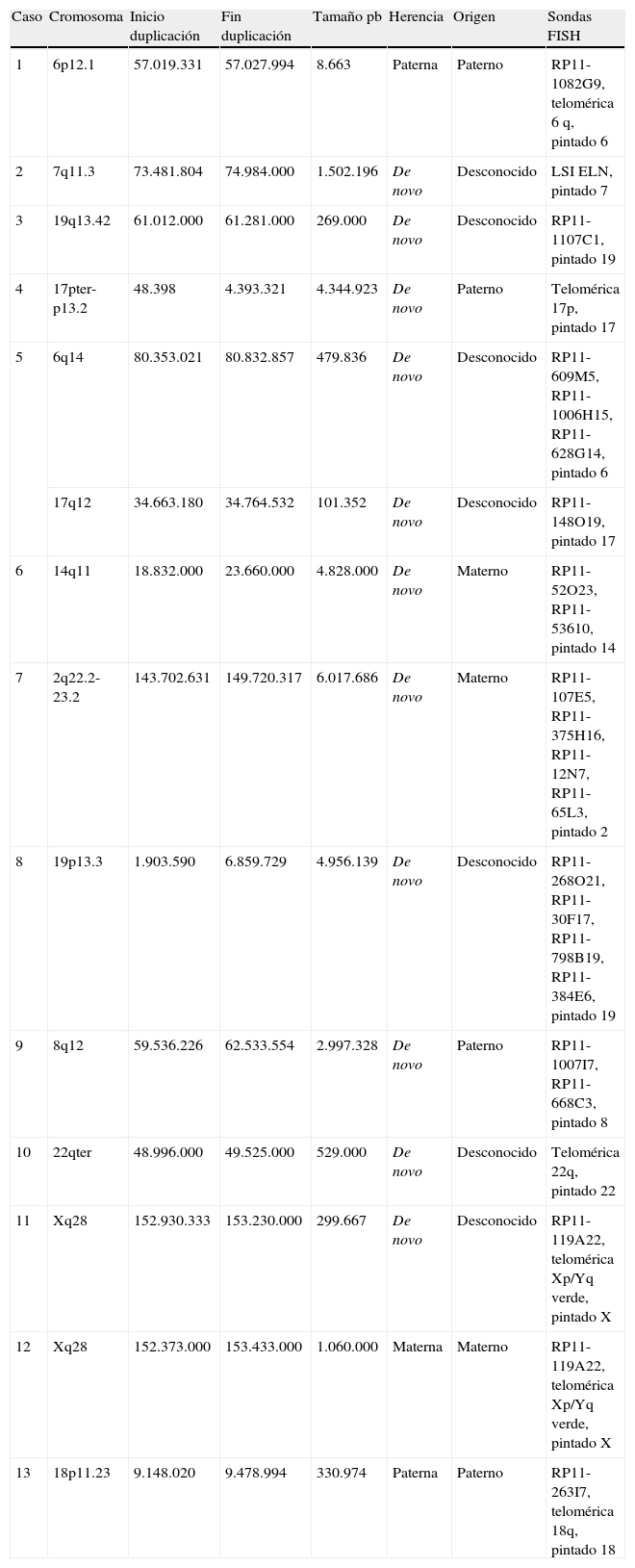
Pacientes y métodoEl estudio se llevó a cabo en 13 pacientes con TND y portadores de duplicaciones génicas detectadas por compared genomic hybridization-array (CGH-array, «hibridación genómica comparada sobre arrays»). Se utilizaron 2 aproximaciones de la técnica FISH: hibridación con sondas de pintado cromosómico y con sondas específicas de cada duplicación.
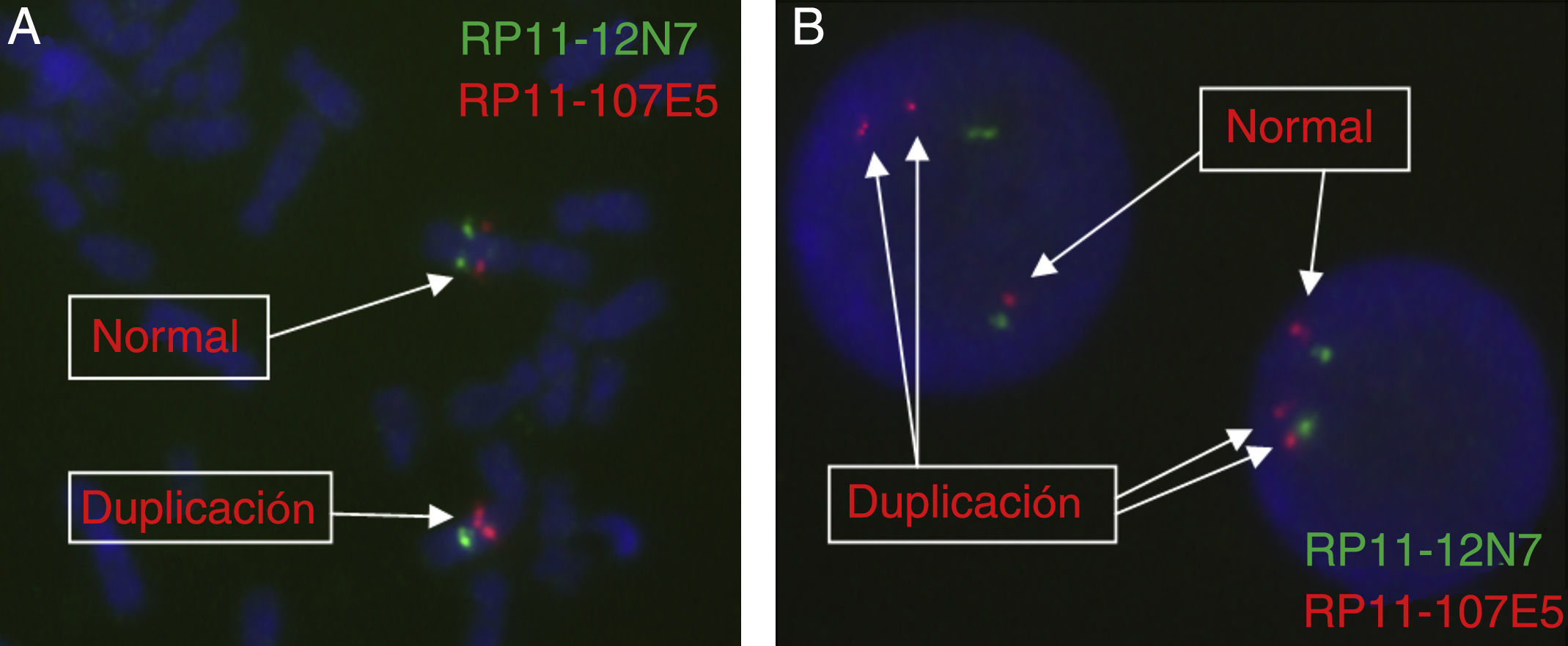
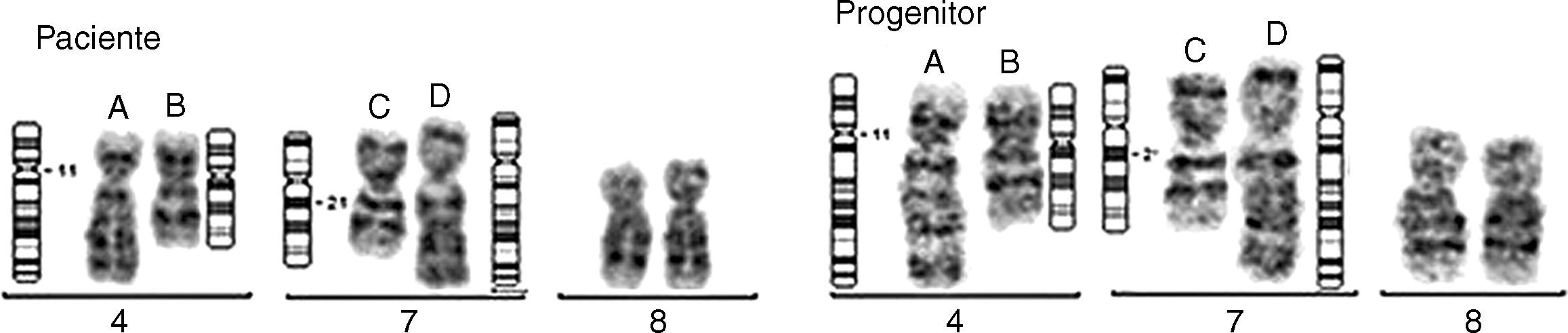
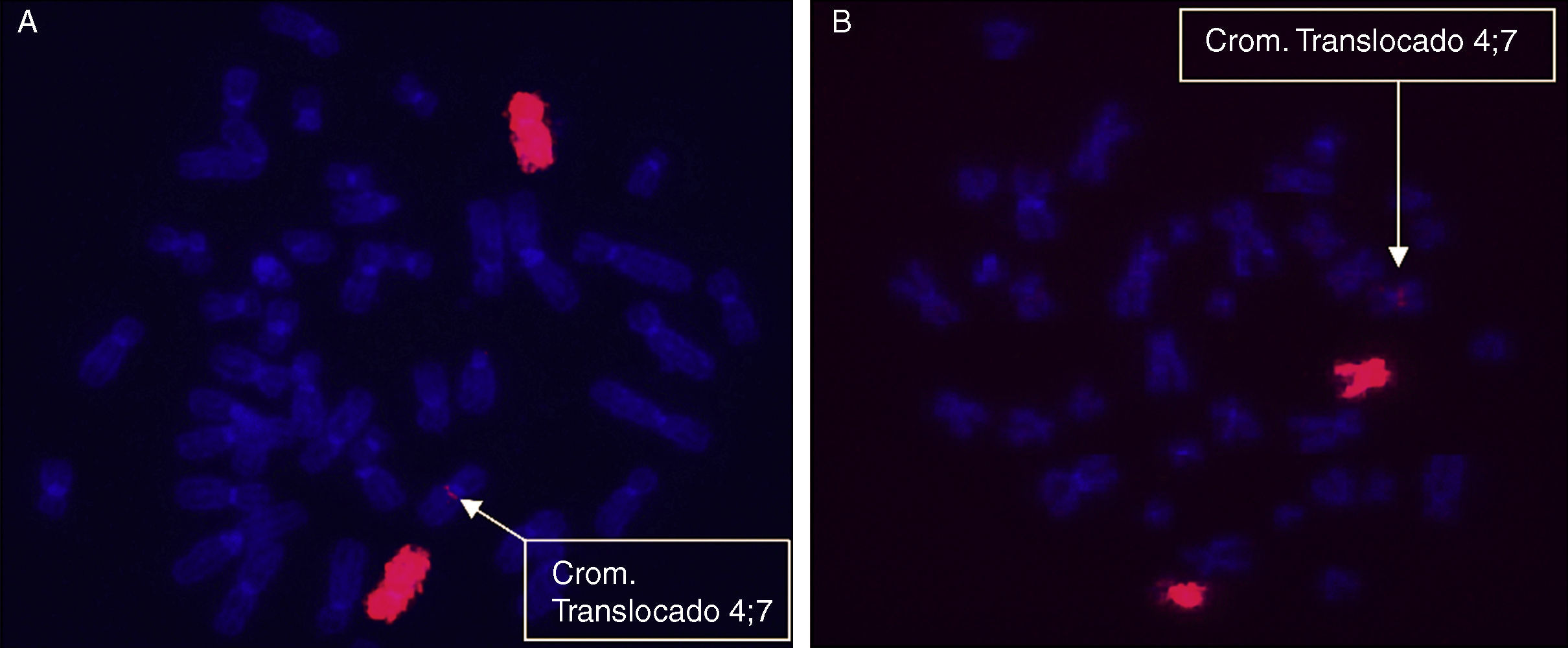
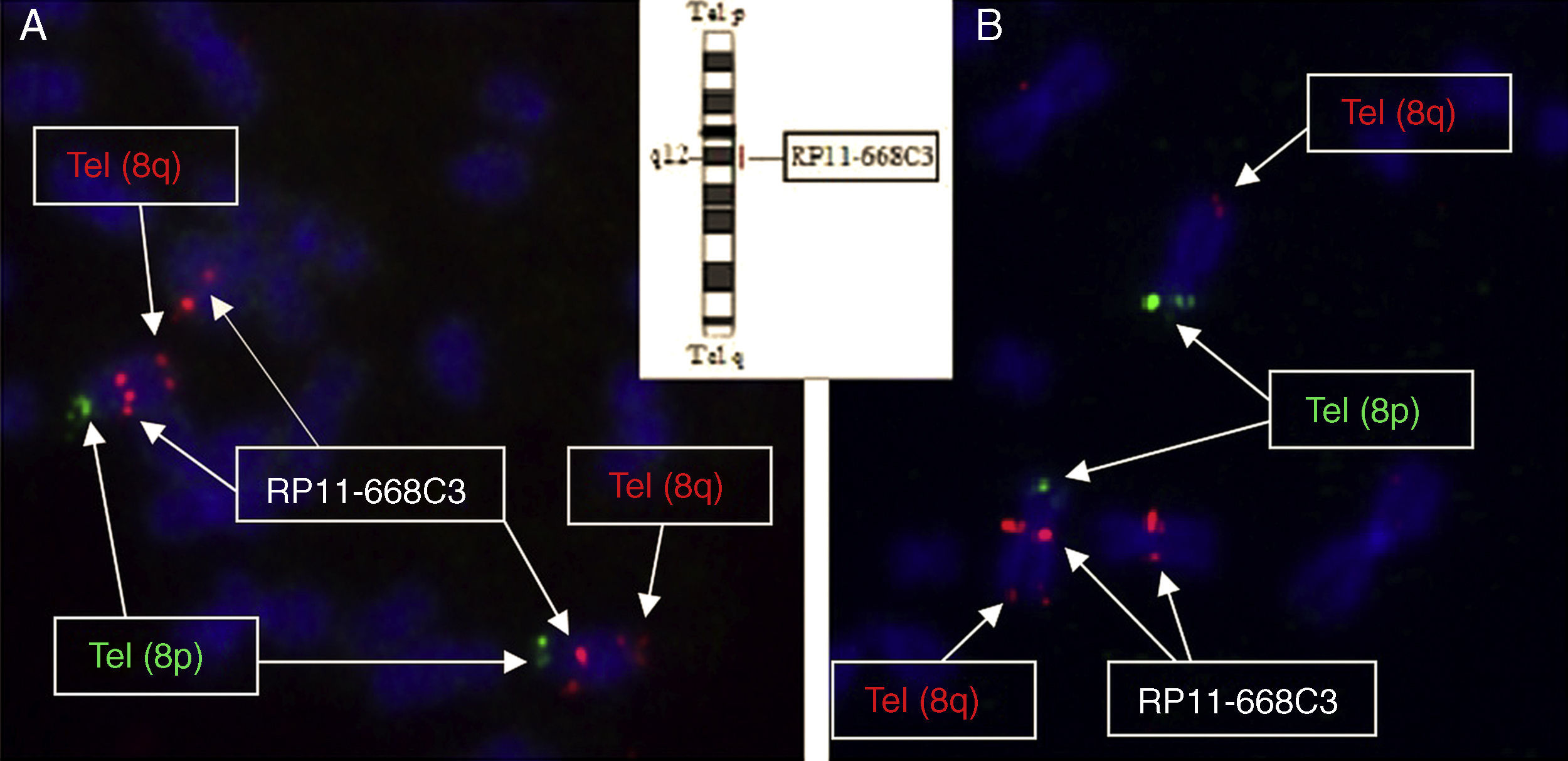
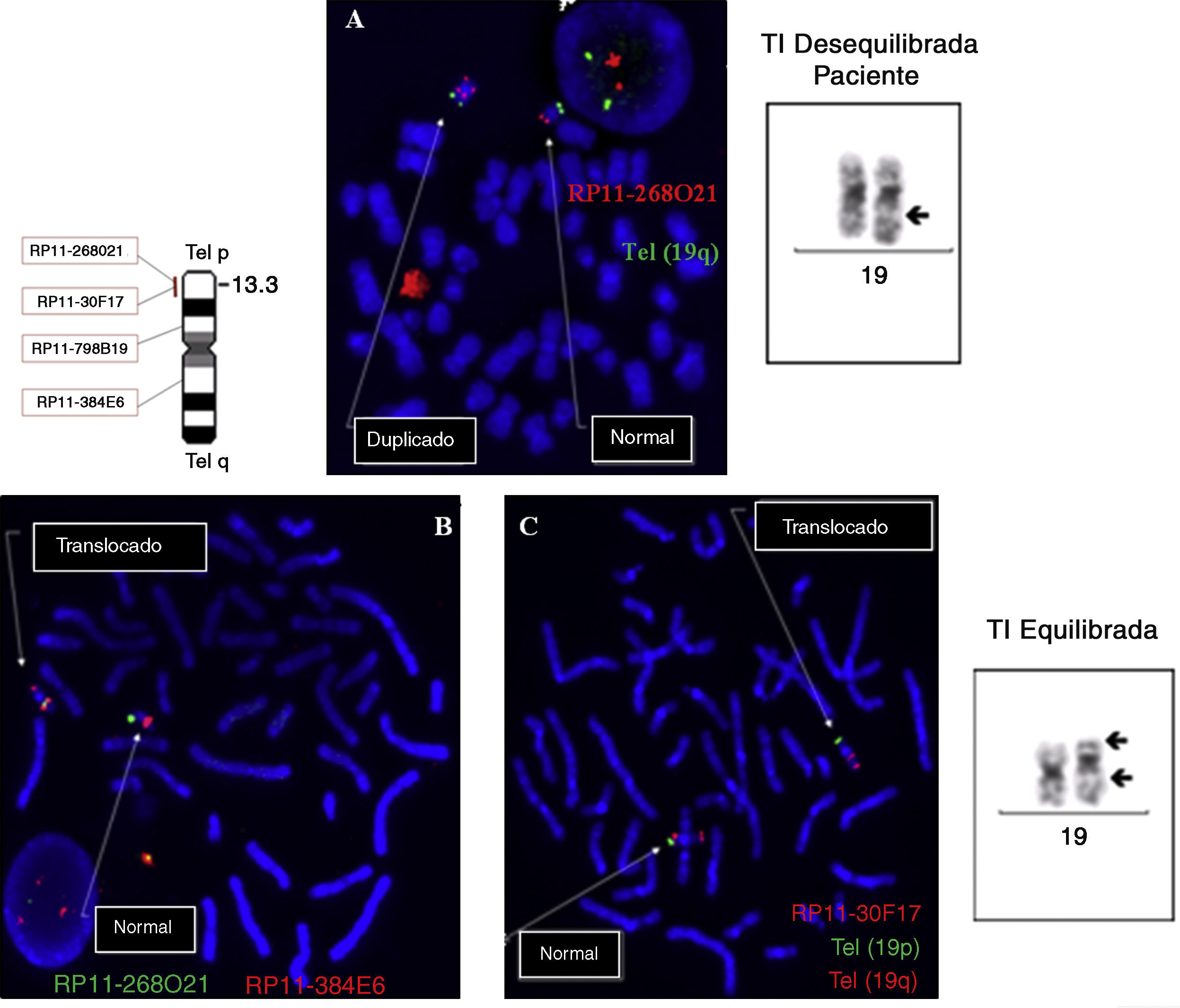
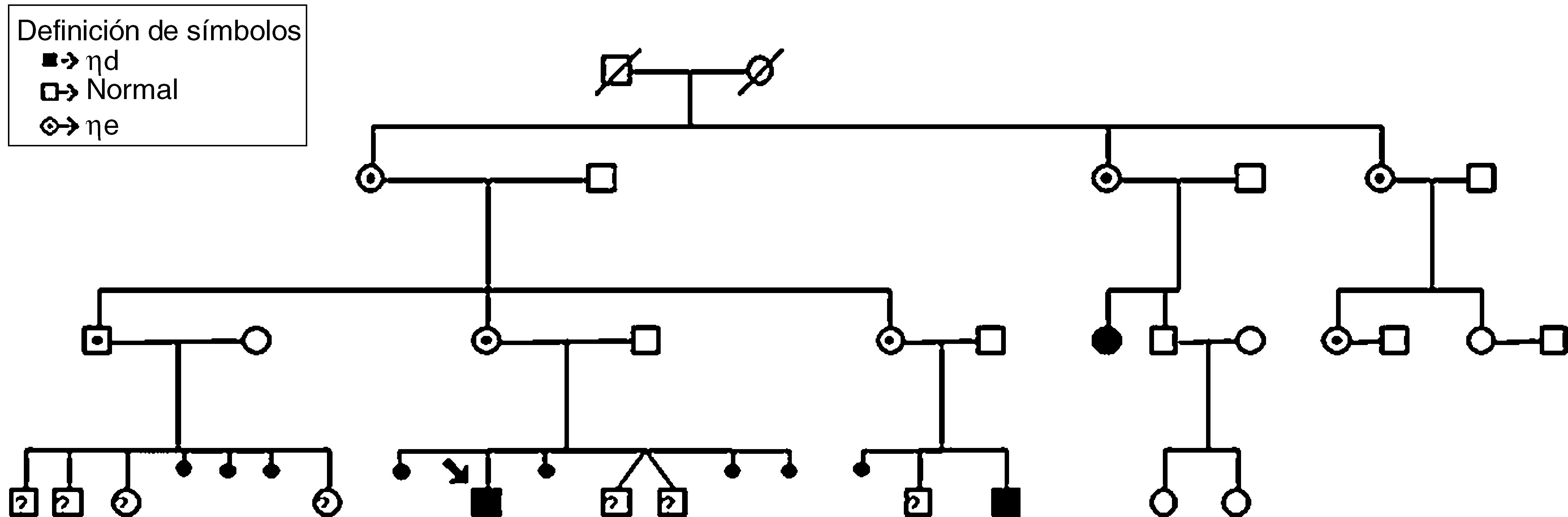
ResultadosEn la serie de 13 pacientes con duplicación estudiados, se han encontrado 11 con duplicaciones en tándem, un caso con una traslocación insercional intracromosómica, y otro con una traslocación insercional intercromosómica. Por tanto, 2 de las duplicaciones que se habían considerado de novo habían sido, en realidad, heredadas de forma desequilibrada de un progenitor que era portador equilibrado del reordenamiento.
ConclusiónLos resultados ponen de manifiesto la necesidad de caracterizar, mediante la técnica de FISH, los reordenamientos que se detectan por CGH-array, para identificar los casos con un elevado riesgo de recurrencia y realizar un correcto asesoramiento genético.
An important proportion of neurodevelopmental disorders (NDDs) results from unbalanced genomic alterations (duplication or deletion). These chromosomal rearrangements may be considered as de novo, despite they arise as a result of a balanced rearrangement not detected in a phenotypically normal parent. Therefore, if the rearrangements are inherited, the recurrence risk and the genetic counseling of these cases change radically. Fluorescence in situ hybridization (FISH) is a technique that allows detecting both balanced and unbalanced rearrangements, identifying also the location of duplicated segments. We tried to locate in the genome the duplicated segments detected in patients with NDDs in order to identify those cases due to inherited rearrangements.
Patients and methodThe study was conducted in 13 patients with NDDs and genomic duplications detected by compared genomic hybridization-array (CGH-array). Two approaches of FISH technique were taken: hybridization with painting chromosome probes and with specific probes for each duplication.
ResultsIn the studied series of 13 patients with duplication, 11 patients were found to carry tandem duplications, one with an intrachromosomal insertional translocation, and another with an interchromosomal insertional translocation. Therefore, 2 of the duplications considered de novo were actually an unbalanced rearrangement inherited from a parent who is a balanced carrier.
ConclusionThe results illustrate the need to characterize by FISH technique the rearrangements that are detected by CGH-array to identify those cases with a high risk of recurrence.
Artículo
Comprando el artículo el PDF del mismo podrá ser descargado
Precio 19,34 €
Comprar ahora