La tuberculosis humana es una enfermedad producida por bacterias del género Mycobacterium, y dentro de éste por tres especies que pertenecen al complejo M.
tuberculosis: M. tuberculosis es la especie aislada con más frecuencia, M. bovis produce una enfermedad indistinguible clínicamente de la producida por M. tuberculosis y M. africanum causa enfermedad en África tropical, pudiendo ser considerada como una forma intermedia entre M. tuberculosis y M. bovis1,2.
Características bacteriológicas del género Mycobacterium
Son bacilos delgados de forma recta o ligeramente curvada, de tamaño de 1-10 µ de largo por 0,2-0,6 µ de ancho. Ocasionalmente forman ramificaciones verdaderas observadas en cultivos enriquecidos y en frotis de ganglios linfáticos. Son bacilos aerobios estrictos, inmóviles, sin cápsula, que no forman esporas y se tiñen con dificultad con la tinción de Gram (irregularmente grampositivos) (fig. 1).
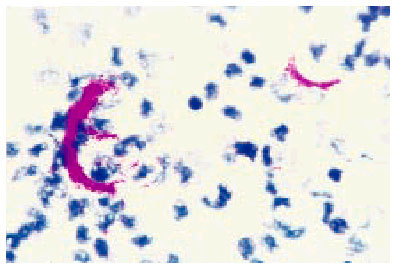
Fig. 1. Tinción de Ziehl-Neelsen positiva (*100). Cord-factor de M. tuberculosis en tuberculosis renal.
La estructura de su gruesa pared celular, responsable de su característica coloración ácido-alcohol resistente, presenta en su lado interno una capa de polímeros de arabinosa y galactosa, seguida de otra formada por ácidos micólicos (ácidos grasos de gran importancia en la taxonomía del género Mycobacterium, y otra superficial formada por lípidos, como los sulfolípidos y micósidos, lo que hace que presenten un alto contenido en lípidos. Es probable que los ácidos grasos y los lípidos sean los responsables de su gran resistencia a la desecación y a la acción de los descontaminantes poderosos, tanto ácidos como alcalinos. Son sensibles al calor húmedo y se destruyen a temperatura de pasteurización.
El crecimiento bacteriano es lento; en condiciones óptimas de cultivo (temperatura 37 °C, pH = 7) el tiempo de duplicación es de 15-18 h, tardando de una a 3 semanas en aparecer colonias visibles en medios de cultivo sólidos3,4.
La tuberculosis más frecuente es la pulmonar, por lo que la mayoría de las muestras procederán del aparato respiratorio; pero la tuberculosis puede afectar a casi todos los órganos. En la actualidad la tuberculosis extrapulmonar representa 22-39%, por lo que se puede recibir en el laboratorio un gran diversidad de muestras4,5. La calidad, cantidad y representatividad de la muestra condicionarán la posibilidad de realizar el diagnóstico.
Obtención y transporte de muestras clínicas
Es necesario enviar las muestras en frasco estéril, transportarlas lo más rápido posible al laboratorio y siempre que sea posible conservarlas a 4 °C. En caso de pacientes ambulatorios se puede almacenar las 3 muestras en una nevera (esputos/orinas) y remitirlas juntas al laboratorio3,4.
Muestras respiratorias
El esputo es la muestra más frecuente y epidemiológicamente la más importante. Se enviarán al laboratorio 3 esputos recogidos en 3 días consecutivos por la mañana, en ayunas, y en cantidad de 5-10 ml; volúmenes inferiores pueden disminuir la rentabilidad de la muestra. Cuando el enfermo no puede espectorar se podrá recurrir a inducir el esputo (aerosol de solución salina, durante 15-20 min) o a la realización de una fibrobroncoscopia, que permite realizar broncoaspirados selectivos, lavados broncoalveolares y biopsias de las lesiones bronquiales. Es importante procesar los esputos obtenidos tras la broncoscopia, ya que tienen una buena rentabilidad.
El estudio del jugo gástrico puede ser útil en el diagnóstico de la tuberculosis pediátrica; se debe obtener matinalmente en 3 días consecutivos y procesarse con rapidez. Si se va a tardar más de 2 h se debe neutralizar con carbonato sódico.
Orinas
Se debe enviar 3 orinas matinales completas durante 3 días consecutivos, cuya cantidad no debe ser inferior a 70 ml.
Líquidos orgánicos
En los líquidos cefalorraquídeos, pleurales, sinoviales, pericárdicos o ascíticos es necesario enviar al laboratorio la mayor cantidad posible, ya que la concentración de micobacterias en este tipo de muestra es muy baja. Es muy importante recordarlo cuando existe una alta sospecha de meningitis tuberculosa, dada la gravedad de la enfermedad y la relevancia de la realización de un diagnóstico microbiológico de certeza.
Biopsias
Siempre que se biopsie un órgano en el que exista sospecha de tuberculosis deberá enviarse una muestra al laboratorio de microbiología y para evitar su desecación se debe añadir unas gotas de agua destilada. En el caso de sospecha de tuberculosis intestinal se debe recurrir, si es posible, a biopsiar la lesión, ya que la rentabilidad del procesamiento de las heces es muy baja.
Hemocultivos
Normalmente sólo deben ser utilizados en pacientes inmunodeprimidos en los que se sospeche enfermedad diseminada, fundamentalmente en pacientes con sida con un número de CD4 inferior a 50/µl. Generalmente su rentabilidad es superior en el diagnóstico de otras infecciones por micobacterias (especies del complejo M. avium-intracellulare, M. genavense) que para la tuberculosis. Los sistemas utilizados con mayor rentabilidad son los de lisis centrifugación y el BACTEC 460TB (viales BACTEC13A). También se han comunicado buenos resultados con el sistema automatizado ESP Culture II-Myco6.
Todas las muestras recibidas para estudio de tuberculosis se deben manipular como si contuvieran micobacterias. Los Centers for Disease Control and Prevention (CDC) y los National Institutes of Health recomiendan el empleo de prácticas de bioseguridad de nivel 2 para la preparación de frotis acidorresistentes y el cultivo de las muestras7.
Técnicas de diagnóstico
Observación microscópica
Las técnicas utilizadas para la observación de las micobacterias se basan en su ácido-alcohol resistencia; es necesario recordar que esta característica es compartida parcialmente por otras bacterias, como especies de Nocardia, Legionella micdadei, Rhodococcus sp. y quistes de Cryptosporidium sp., Isospora sp., Cyclospora sp. y Microsporidea, aunque presentan menos estabilidad en la coloración. Las 2 tinciones más utilizadas son la clásica de Zhiel-Neelsen y la tinción con fluorocromos (auramina 0). Esta última permite una lectura más rápida (2-3 min) y se recomienda cuando el número de muestras a examinar es superior a diez diarias; también hay que tener en cuenta que los frotis teñidos por auramina pueden reteñirse por Zhiel-Neelsen, lo que posibilita la diferenciación de la morfología bacteriana. Esta tinción permite distinguir ciertas características morfológicas, pero no permite diferenciar con certeza las especies de micobacterias. M. Kansasii puede sospecharse por su mayor tamaño y apariencia de bandas cruzadas. Algunas micobacterias de crecimiento rápido (cepas del complejo M. fortuitum) pueden teñirse mal con auramina 0, teniendo que recurrir a la tinción clásica4.
Las recomendaciones internacionales más utilizadas para la emisión de resultados se exponen en la tabla 18. Esta cuantificación es importante, ya que la reducción del número de bacilos ácido-alcohol resistentes (BAAR) orienta sobre la eficacia del tratamiento antituberculoso.
El hallazgo de BAAR en las muestras clínicas es la primera evidencia de la presencia de una micobacteria. Es el procedimiento más fácil y rápido que permite un diagnóstico presuntivo y el reconocimiento de los pacientes bacilíferos, lo que le confiere un gran valor epidemiológico. Una tinción negativa nunca excluye una tuberculosis. Para que una tinción sea positiva es necesario que el número de BAAR en el esputo sea de 5.000-10.000/ml, lo que hace que la sensibilidad de la técnica sea limitada, del 22-80% según las series4, y se encuentre influida por diversos factores (tipo de muestra, población estudiada, etc.). En tuberculosis primarias y extrapulmonares, su sensibilidad es muy baja9. Dado que la eliminación de bacilos puede ser discontinua, se recomienda el procesamiento de varias muestras para aumentar la sensibilidad de la observación microscópica directa6.
La especificidad de la tinción de BAAR es bastante elevada; la presencia de micobacterias no tuberculosas en el agua con la que se realiza la tinción puede ser causa de falsos positivos. También puede existir transferencia de micobacterias entre distintos frotis, así como contaminación en el aceite de inmersión2.
Independientemente del resultado de la observación microscópica, se debe siempre efectuar el cultivo de las muestras clínicas para realizar un diagnóstico de certeza de tuberculosis.
Cultivo e identificación del complejo M. tuberculosis
La técnica de cultivo es más sensible que la microscopia para el diagnóstico, ya que permite detectar 10 bacterias/ml de muestra concentrada. Asimismo, asegurar la negativización de los cultivos es imprescindible para el seguimiento de la enfermedad y el control de la eficacia del tratamiento. La realización del cultivo es fundamental para el aislamiento de la bacteria, que
permitirá, si fuera necesario, estudios de resistencia a fármacos y/o estudios de tipificación genética6.
Las muestras remitidas para su estudio al laboratorio de micobacterias se pueden dividir en dos grupos3,4,10:
1. Muestras procedentes de lugares estériles, como líquidos cefalorraquídeos, pleurales, peritoneales, pericárdicos y biopsias de tejidos. Éstas pueden sembrarse directamente en los medios de cultivo. Si el volumen es grande pueden requerir una concentración previa.
2. Muestras procedentes de lugares en los que existe flora comensal (esputos, orinas etc.) que se multiplica más rápido que las micobacterias, por lo que puede impedir el crecimiento de las micobacterias. Este tipo de muestras debe ser sometido a un proceso de homogeneización (descontaminación y posterior neutralización-concentración). Los métodos más utilizados
son el de Tacquet y Tison (laurilsulfato sódico), el de NALC-NaOH y el que utiliza ácido oxálico, que elimina P. aeruginosa, para enfermos con fibrosis quística2,10. El concentrado final puede utilizarse para tinciones ácido alcohol-resistentes, para cultivo o, si fuera necesario, para la amplificación de ácidos nucleicos. La elección de cualquiera de estos sistemas dependerá sobre todo del tipo de medio de cultivo líquido a utilizar y/o de la posible realización de técnicas moleculares, ya que algunos reactivos pueden retrasar el crecimiento de las micobacterias o interferir en las reacciones de amplificación11.
La gran variedad de medios permite a cada laboratorio elegir los que mejor se adapten sus necesidades y a la población atendida. Existen 2 grupos de medios de cultivo: sólidos y líquidos. En general, se admite que el uso combinado de un medio sólido y otro líquido es idóneo para la realización de aislamientos primarios, buscando rapidez y sensibilidad. En condiciones normales, los cultivos deben incubarse a 35-37 °C e incubarse durante 6-8 semanas. Se acepta como control de calidad de buen funcionamiento de un laboratorio una contaminación entre el 3-5% de los cultivos en medio sólido. Un rango menor indicaría una descontaminación demasiado severa y un rango superior al 5% sería debido a una descontaminación suave. No existe consenso en cuanto a posibles rangos de contaminación en los distintos medios líquidos utilizados en la actualidad3.
Medios de cultivos sólidos
Los más comúnmente usados son los que utilizan como base huevo coagulado, como el Löwenstein-Jensen y el de Coletsos. Este último facilita el crecimiento de M. bovis y de cepas disgónicas de M. tuberculosis. También se puede utilizar medios con base de agar (Middlebrook 7H10, 7H11). Si se dispone de estufas con atmósfera de 5-10% de CO2, es conveniente mantener los tubos al menos 5-7 días (esto es importante en los medios con agar). Los tubos deben leerse, al menos una vez por semana, aunque es deseable realizar 2 lecturas semanales. Una ventaja de estos medios es que permiten apreciar la morfología colonial, por lo que se puede observar características de la colonia (rugosidad, pigmentación, etc.) que son ya datos orientativos para su posterior identificación10. En el medio de Löwenstein-Jensen las colonias de M. tuberculosis empiezan a observarse a las 2-3 semanas de incubación. La mayor desventaja de estos medios es la lentitud de crecimiento de las micobacterias, por lo que en los últimos años han tenido un gran desarrollo los medios líquidos.
Medios de cultivos líquidos
Su mayor ventaja es que acortan en 2-3 semanas la detección de las micobacterias, y permiten un mayor número de aislamientos de algunas micobacterias no tuberculosas, como las pertenecientes al complejo M. avium-intracellulare. Actualmente los medios líquidos se pueden dividir en dos grupos, que exponemos a continuación.
Medios de lectura semiautomática. Estos sistemas requieren una gran manipulación de los viales durante todo el proceso y la lectura se realiza de forma manual. El más ampliamente utilizado es el sistema BACTEC 460TB, comercializado a finales de los años setenta, que emplea viales de medio líquido Middlebrook 7H12, una mezcla antibiótica selectiva PANTA (polimixina B, azlocidina, ácido nalidíxico, trimetroprim y anfotericina B) y un suplemento de enriquecimiento de estearato de polioxietileno (POES). El medio contiene ácido palmítico marcado con 14C radiactivo del que las micobacterias, al crecer, liberan 14CO2. Éste es detectado en el BACTEC 460TB y traducido a un índice de crecimiento (escala 0-999), que es proporcional a la cantidad de micobaterias que se están multiplicando. Este sistema es más sensible que los tradicionales6,12,13. Presenta como inconveniente la utilización de isótopos radiactivos y que se pueden producir falsos positivos por contaminación de los frascos a partir de las agujas del aparato de lectura deficientemente esterilizadas14. En la actualidad, está comercializado el sistema Mycobacteria Growth Indicator (MGIT), que utiliza el medio de Middlebrook 7H9 y la mezcla antibiótica PANTA, con un suplemento de enriquecimiento OADC (ácido oleico, albúmina, dextrosa y catalasa); como sustrato utiliza un compuesto de rutenio. Durante el crecimiento bacteriano se consume oxígeno y el compuesto de rutenio emite fluorescencia, que se detecta en una lámpara de Wood. Tiene menor sensibilidad que el anterior, pero es una buena alternativa para laboratorios pequeños15,16.
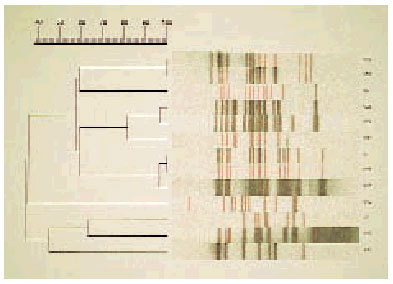
Medios de lectura automática (fig. 2). En los últimos años se han diseñado sistemas de cultivo líquido no radiométricos. Todos ellos son métodos no invasivos (no utilizan agujas en la detección), con escasa manipulación de viales (una vez inoculados se almacenan en un incubador específico) y lectura continua totalmente automatizada. La principal diferencia está en el sistema de detección. Los más utilizados son: BACTEC 9000MB System (la base de la detección es la descrita en el sistema MGIT)7,18, ESPII-Myco (detección basada en la disminución de presión por el consumo de oxígeno debido al crecimiento de las bacterias)19 y sistema MB-BacT (detección mediante colorimetría de la producción de CO2 del crecimiento bacteriano).
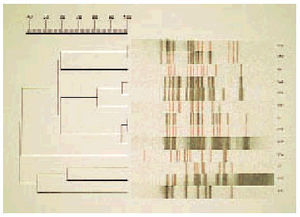
Fig. 2. Análisis automatizado. Dendograma representativo del estudio mediante RFLP-IS6110 de 13 cepas de M. tuberculosis. Las cepas V10 y V20, así como la V1 y V52, presentan patrones idénticos.
Identificación del complejo M. tuberculosis
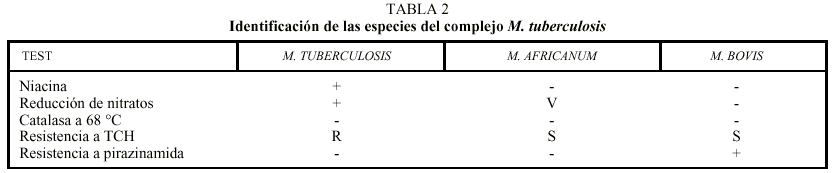
Tradicionalmente, las micobacterias aisladas en cultivo se han identificado sobre todo por sus características morfológicas en el medio de Löwenstein-Jensen (velocidad de crecimiento, rugosidad y pigmentación) y metabólicas. Se confirma mediante pruebas bioquímicas, el test de la niacina, que pone de manifiesto la capacidad para producir ácido nicotínico y es la prueba fundamental para la identificación de M. tuberculosis, aunque ésta no debe basarse nunca exclusivamente en esta prueba10. Las pruebas bioquímicas necesarias para la correcta identificación del complejo M. tuberculosis se exponen en la tabla 2.
El desarrollo de la biología molecular ha permitido identificar secuencias de ácido desoxirribonucleico (ADN) y de ácido ribonucleico (ARN) específicas de las distintas especies de micobacterias. Para hibridar con estas secuencias se han preparado sondas genéticas marcadas con sustancias cromógenas, que proporcionan resultados en menos de una hora. Actualmente disponemos de sondas frente al complejo M. tuberculosis, M. avium, M. intracellulare, M. kansasii y M. gordonae (Accuprobe.Gen-Probe Inc.) que se pueden utilizar
tanto sobre colonias crecidas en medios sólidos como sobre cultivos positivos en medios líquidos. El límite de detección de estas sondas es de, aproximadamente, 106 unidades formadoras de colonias (UFC)/ml20. El principal inconveniente de la sonda para el complejo M. tuberculosis es que no diferencia entre las 3 especies del complejo. La sonda tiene una alta especificidad, pero se han descrito falsos positivos con M. terrae y M. celatum3.
Técnicas de amplificación de ácidos nucleicos
Los problemas inherentes a la lentitud del cultivo de las micobacterias han hecho que las técnicas de biología molecular que permiten la amplificación de secuencias de ADN y de ARN específicas del complejo M. tuberculosis se desarrollen ampliamente en el diagnóstico de la tuberculosis, permitiendo un diagnóstico rápido (5-8 h). Estas técnicas presentan problemas en su evaluación, siendo necesaria, en la actualidad, una estrecha relación entre el clínico y el microbiólogo para concretar las situaciones en las que se debe utilizar estas técnicas de amplificación. Es importante conocer con precisión las ventajas y limitaciones de cada sistema de amplificación6.
En general, todas la técnicas de amplificación de ácidos nucleicos comportan 3 pasos fundamentales.
Preparación de la muestra
Este paso está dirigido a eliminar posibles inhibidores existentes en las muestras clínicas, así como a conseguir una extracción eficiente de los ácidos nucleicos de las micobacterias. La lisis puede ser mecánica (sonicación), física (calor) y química (detergentes, proteasas); pueden utilizarse uno o varios procedimientos, ya que la pared celular de las micobacterias es difícil de lisar.
Amplificación
Proceso en el que se producen numerosas copias del ácido nucleico diana. Existen técnicas que amplifican el ARN, y otras el ADN, utilizando diversas dianas de amplificación (IS 6110, rRNA 16s) y distintas enzimas (transcriptasa inversa/ARN-polimerasa/Taq polimerasa/ADN-ligasa/ADN-polimerasa). Según el protocolo utilizado variarán el número de ciclos y las condiciones de temperatura.
Detección del producto amplificado
Se puede realizar mediante electroforesis en geles de agarosa (generalmente con menor especificidad), técnicas de hibridación en distintos tipos de soporte (tipo enzimoinmunoanálisis), etc.6.
Los primeros ensayos utilizaban muy diversos protocolos, con reactivos de muy distinta fabricación y con variedad de controles de calidad, y obtuvieron resultados heterogéneos según los distintos laboratorios21,22. Esto hizo imposible la realización de análisis globales sobre la utilización de estas técnicas en el diagnóstico de la tuberculosis. En la actualidad disponemos en el mercado de una gran variedad de técnicas que permiten trabajar de forma estandarizada. Las más utilizadas y, por tanto, de las que existe más experiencia en el diagnóstico clínico son las que se citan a continuación.
Amplificación mediada por transcripción (TMA). (AMTD-2. GenProbe Inc). Utiliza un proceso isotérmico y autocatalítico, diseñado para amplificar el ARN ribosomal 23S micobacteriano (utiliza ARN-polimerasa/transcriptasa inversa). La detección específica se realiza mediante una sonda ADN marcada con un ester de acridina quimioluminiscente. A través de un proceso químico (que protege la hibridación) se eliminan las sondas no hibridadas. Los híbridos ARNr-ADN, específicos de complejo M. tuberculosis, son detectados en un luminómetro. Es un sistema manual de fácil aplicación en cualquier laboratorio clínico23,24.
Reacción en cadena de la ligasa (LCR) (LCx.Abbot Lab). La LCR utiliza 4 iniciadores diseñados para acotar la región a amplificar; así, las sondas pueden unirse enzimáticamente (mediante la ADN-ligasa) previa actuación de la ADN-polimerasa, para formar el producto amplificado. Se utiliza como diana el gen que codifica para la proteína antigénica b, específica del complejo M. tuberculosis. La detección de los amplicones tiene lugar mediante un enzimoinmunoanálisis de micropartículas (MEIA), tras generar el producto amplificado una molécula fluorescente. Es un sistema semiautomatizado23,25,26.
Reacción en cadena de la polimerasa (PCR) Amplicor© (Roche Systems). La diana de amplificación es un segmento de 584 pares de bases (pb) ubicado en el gen que codifica para el ARNr 16s, común a todo el género Mycobacterium. La detección se lleva a cabo con sondas específicas para el complejo M. tuberculosis que, una vez unidas al producto amplificado, darán lugar a una reacción colorimétrica. Esta técnica incluye controles internos para detectar inhibidores de la amplificación27,28.
Las evaluaciones de todas estas técnicas sobre muestras clínicas con tinción de auramina positiva ofrecen muy buenos resultados en cuanto a la sensibilidad (> 98%) y la especifidad (> 98%). En las muestras de origen respiratorio, cuando la tinción de auramina es negativa existe una mayor variabilidad en los estudios; en general presentan mayor sensibilidad las técnicas que amplifican ARN (80-85%) que las que amplifican ADN (53-72%)23-27.
En las series que estudian muestras extrapulmonares aún es mayor la dispersión de resultados, obteniéndose sensibilidades entre el 71-78% con las técnicas que amplifican ADN y entre el 65-77% con las que amplifican ARN23,24,26,28,29.
Estos datos indican que una amplificación negativa en una muestra con tinción de auramina negativa no descarta el diagnóstico de tuberculosis.
Existe una dificultad añadida en la interpretación de las amplificaciones positivas con cultivo negativo, que intentan justificarse con valoraciones clínicas. Sin embargo, aparece en casi todos los estudios un 1-5% que debe ser asumido como falsos positivos6.
En la actualidad, las técnicas de amplificación de ácidos nucleicos son un apoyo en el diagnóstico de la tuberculosis, pero no desplazan a las técnicas clásicas. Las limitaciones y el elevado coste de esta tecnología está siendo un obstáculo para su introducción en la rutina de los laboratorios de micobacterias.
Técnicas serológicas
Los principales tipos de antígenos de micobacterias utilizados para el diagnóstico serológico mediante técnicas de enzimoinmunoanálisis son de naturaleza proteica (PPD, antígeno 5) o de naturaleza lipídica (PGL-tb 1, SL I, SL IV)3. Existe comercializado un equipo que utiliza el denominado antígeno 60, que es un complejo proteínico-lipopolisacárido procedente del citoplasma y de la membrana celular de M. bovis BCG y es común a M. tuberculosis, M. bovis y otras micobacterias3. Estas técnicas serológicas, que parecían prometedoras en los estudios preliminares, resultaron ser finalmente decepcionantes en la práctica clínica. En la actualidad no se reconoce utilidad diagnóstica clara a ninguna determinación serológica.
Estudios de sensibilidad a fármacos antituberculosos
Es bien sabido que en el genoma de M. tuberculosis se producen mutaciones espontáneas con relativa frecuencia. La incidencia natural de mutantes dentro de una población bacteriana varía para los diferentes quimioterápicos: para rifampicina aparece una mutante resistente entre 108 bacilos, para isoniacida sería entre105-106 bacilos. Para evitar la selección de cepas resistentes el tratamiento de la tuberculosis se realiza con al menos 3 quimioterápicos4,5. Los fármacos de primera línea utilizados en el tratamiento de la tuberculosis son: isoniacida (H), rifampicina (R), etambutol (E), pirazinamida (Z) y estreptomicina (S). Los de segunda línea son: ácido p-amino-salicílico, etionamida, cicloserina, capreomicina, kanamicina, amikacina, ofloxacino y rifabutina4.
La detección de cepas resistentes es otra de las funciones importantes de los laboratorios de micobacterias. Las pruebas de sensibilidad se realizan con dos finalidades distintas:
1. Estudios con fines epidemiológicos, cuyo objetivo es el conocimiento de las resistencias primarias; se realizan periódicamente y se coordinan desde los programas de control de tuberculosis correspondientes a cada zona.
2. Estudios de sensibilidad individualizados. Se realizan en cepas procedentes de pacientes pertenecientes a grupos concretos previamente definidos en los programas de control. Aunque la elección de estos grupos depende de cada país y de su problemática respecto a la tuberculosis, en general se admite la realización del antibiograma en los siguientes casos:
Pacientes previamente tratados.
Pacientes en los que el esputo con baciloscopia positiva tras una inicial negativización vuelve a positivizarse.
Cuando el examen microscópico no se negativiza tras los 2-3 primeros meses de tratamiento.
Cuando el cultivo de esputo no se negativiza a los 6 meses de tratamiento.
Pacientes infectados por cepas resistentes a algún fármaco.
Todos los pacientes infectados con el VIH en zonas de alta incidencia de infección por el VIH3.
Todos los pacientes menores de 15 años.
El programa de control de la tuberculosis de la Comunidad Autónoma Gallega incluye también a los pacientes ingresados en penitenciarias, inmigrantes, contactos con infectados por cepas resistentes, usuarios de drogas por vía parenteral y todos los infectados por el VIH5,6.
Métodos de antibiograma de M. tuberculosis
Se pueden realizar a partir de la muestra recibida en el laboratorio, cuando en ella se observen abundantes bacilos acidorresistentes en el examen microscópico (método directo), o a partir de la cepa aislada en el cultivo (método indirecto). Los resultados del método directo son más rápidos, pero sólo son orientativos, debiéndose confirmar por el método indirecto5,9.
El método de referencia clásico es el de las proporciones4,10, realizado sobre medios sólidos como el Löwenstein-Jensen o en el medio de base de agar 7H10, a los que se incorporan los antibióticos en concentraciones críticas previamente definidas. Para la lectura es necesaria la visualización de colonias, por lo que, dado el metabolismo lento de M. tuberculosis, los resultados tardan de 21 a 28 días. Esta lentitud es un gran inconveniente para su aplicación en casos clínicos concretos. La dificultad al trasladar este método a medios líquidos, más rápidos, es la necesidad de redefinir todos los parámetros del método: inóculo bacilar, concentración crítica del fármaco y tiempo de lectura3,6.
En la actualidad, de los medios líquidos semiautomatizados, sólo el sistema radiométrico BACTEC 460 permite obtener resultados concordantes con los sistemas de referencia, para los fármacos de primera línea, en un tiempo muy inferior (5-10 días) y es el único aprobado por la FDA4,30. Otros sistemas líquidos más automati-
zados y de más sencillo manejo (ESP II/ MBBact-alert/BACTEC-MGIT 960) están en avanzada fase de aprobación6.
La aparición de cepas multirresistentes ha aumentado la necesidad de disponer de métodos más rápidos para detectar la resistencia a los fármacos de primera línea31.
Se están estudiando técnicas que detecten genes de resistencia, por tanto independientes del crecimiento bacteriano. Los estudios más avanzados están realizados sobre la detección de resistencia a rifampicina, ya que las mutaciones se encuentran principalmente en un solo gen (rpoB). Existe un sistema ya comercializado (INNO-lipa) que utiliza una PCR y posterior hibridación en tiras de nitrocelulosa que llevan incorporadas sondas que cubren la totalidad del gen rpoB, informando hasta del 86% del total de las mutaciones responsables de la resistencia a rifampicina, en caso de existir. Es una técnica rápida, que se correlaciona bien con los métodos tradicionales y que permite sospechar que la cepa es multirresistente, ya que el 90% de las cepas resistentes a rifampicina lo son también a isoniacida30. En los últimos años se han descrito y reconocido genéticamente mutaciones de resistencia a otros fármacos (H, E, Z, S), por lo que es posible que surjan nuevas técnicas rápidas6.
Tipificación molecular. Técnicas de restricción-hibridación (RFLP)
En el estudio de la epidemiología de la tuberculosis puede ser importante comparar cepas de M. tuberculosis aisladas de los enfermos (y en su caso del ambiente). Históricamente, los únicos sistemas para subtipificar aislamientos de M. tuberculosis eran marcadores fenotípicos, como la resistencia a antimicrobianos y la fagotipificación, ambos sistemas de bajo poder discriminativo32. Actualmente disponemos de marcadores genotípicos más eficaces que estudian directamente el genoma bacteriano. Se considera que un marcador tiene un buen poder discriminativo cuando la probabilidad de que 2 cepas no relacionadas sean consideradas como idénticas sea menor del 5%. Además, un marcador debe ser rápido, simple, estable, reproducible y debe ser capaz de tipificar la práctica totalidad de las cepas estudiadas. El marcador genético de referencia en la actualidad es la técnica RFLP-IS611033.
Las técnicas de restricción-hibridación consisten en una extracción y la restricción del ADN de las cepas con una enzima de alta frecuencia de corte, separación por electroforesis de los fragmentos en función de su peso molecular, transferencia a una membrana e hibridación con una sonda complementaria de uno de los elementos repetidos. De este modo se obtienen patrones con un número discreto de bandas, que son fácilmente comparables entre sí34,35. Para comparar un número elevado de aislamientos se han desarrollado sistemas automatizados (fig. 1).
El genoma de M. tuberculosis contiene, en general, un número elevado y variable (5-20) de copias de la secuencia de inserción IS6110, las cuales se encuentran en posiciones variables, lo que proporciona a este marcador un poder discriminativo elevado. Existe un protocolo estandarizado, y aceptado internacionalmente, que permite comparar los resultados obtenidos en diferentes laboratorios, por lo que las bases de datos a gran escala permiten seguir la diseminación de todo el mundo36. En cepas con menos de 6 copias de IS6110 el poder es menor. Este marcador genético tiene como inconveniente la complejidad de la técnica y la necesidad de una cantidad apreciable de ADN, por lo que se realiza sobre cultivos de micobacterias32.
Las principales aplicaciones de la genotipificación de cepas de M. tuberculosis34-36 son: a) estudios epidemiológicos poblacionales; b) diferenciación entre recidiva y reinfección exógena; c) control de la diseminación de cepas multirresistentes, y d) detección de contaminaciones cruzadas y otros errores del laboratorio.
En la actualidad se están estudiando técnicas complementarias y alternativas, estudios de polimorfismo de la región DR mediante el análisis de los oligonucleótidos espaciadores (spoligotyping) y las técnicas de PCR basadas en iniciadores complementarios de secuencias repetidas, con el fin de encontrar técnicas más simples y que se puedan aplicar directamente sobre muestras clínicas38.