Los microsatélites son secuencias altamente polimórficas de ADN constituidas por repeticiones cortas regularmente de 1-7 pares de bases y con más del 70% de heterocigocidad. El objetivo del artículo fue obtener las frecuencias alélicas de 5 loci por electroforesis capilar y compararlas con la base de datos Combined DNA Index System (CODIS) para determinar la eficacia de la técnica en la determinación ancestral.
Material y métodosSe extrajo el ADN de 50 individuos no relacionados (G1). Se amplificaron 6 loci mediante PCR. Se identificó la longitud de los marcadores por electroforesis capilar y las frecuencias se compararon con las reportadas en CODIS (G2) para población latina.
ResultadosLas frecuencias de los polimorfismos TH01 y D2S1338 fueron las únicas similares a lo reportado en CODIS. El número de alelos del FGA fue distinto en ambos grupos, G1 no presentó 4 alelos reportados. El polimorfismo D18S51 en G1, presentó un alelo de 10.2 repeticiones no reportado en CODIS, y careció de 4 alelos. Para D16S539, los valores de nuestra población fueron distintos a los reportados, los alelos 7 y 17 presentes en nuestra población no están reportados en CODIS. Los alelos 14 y 15 reportados en CODIS no se observaron en nuestra población.
ConclusionesEn la comparación de las frecuencias de los polimorfismos en una muestra de origen mestizo mexicana con otra población latina, las diferencias encontradas nos muestran la necesidad de crear bases de datos de referencia de nuestra población, teniendo en cuenta el mestizaje que nos caracteriza.
Microsatellites are highly polymorphic DNA sequences, consisting of short repeats, regularly from 1 to 7 base pairs, and over 70% of heterozygosity. The objective of this study was to obtain allele frequencies of 5 loci by capillary electrophoresis and comparing them with the Combined DNA Index System database (CODIS) to determine the effectiveness of the technique in the ancestral determination.
Material and methodsDNA from 50 unrelated individuals (G1) was extracted. Six loci were amplified by PCR. The lengths of the markers were identified by capillary electrophoresis and the frequencies were compared with those reported in CODIS (G2) for Latin-Americans.
ResultsThe frequencies of the polymorphisms, TH01 and D2S1338, were the only ones similar to those reported in CODIS. The number of alleles of FGA was different in both groups, and G1 did not have 4 of the reported alleles. The D18S51 polymorphism in G1 presented with a 10.2 allele unreported in CODIS, and lacked 4 alleles. For D16S539, our population values were different from those reported, 7 and 17 alleles were present in our population, but were not reported in CODIS. Moreover, alleles 14 and 15 reported in CODIS were not observed in our population.
ConclusionsThe results of our study enable us to compare the frequencies of the polymorphisms in a sample of Mexican with other Latin Americans. The differences suggest the need to create databases of reference for our population, taking into account the race mixture that characterises us.
En su libro El origen de las especies, Charles Darwin escribió: «Probablemente todos los seres orgánicos que hayan vivido nunca en esta tierra han descendido de alguna forma primordial».1 Es así que nuestro ADN puede ser prácticamente idéntico al de otras personas, sin embargo, hay regiones heredadas que pueden variar de una persona a otra; estas secuencias se denominan «polimorfismo»2.
Las regiones polimórficas del ADN tienen un alto poder de discriminación, por ejemplo permiten con gran fiabilidad excluir a un individuo que puede ser falsamente acusado en un crimen3. El tamaño del genoma humano es de aproximadamente 3.2Gb, de este, el 0.1% es diferente entre las personas, lo que permite la variabilidad genética entre grupos étnicos4.
Los microsatélites (o STR por sus siglas en inglés: short tandem repeat) son repeticiones cortas en tándem y son un tipo de polimorfismos con secuencias repetidas de 2-7 pares de bases5, que varían en longitud entre los individuos y generalmente existen muchos alelos diferentes dentro de una población6.
Estos loci son propensos a mutaciones debido a que son susceptibles de deslizarse durante la replicación del ADN7. Esta tasa alta de mutación ha sido un factor clave en la determinación de la capacidad informativa de los STR en los estudios genéticos. Han sido utilizados ampliamente durante los últimos 10 años para investigar la variación genética a través de poblaciones en muchas especies8.
La diversidad genética (entre individuos de un mismo grupo o entre grupos diferentes) puede ser evaluada a partir de la distribución de las frecuencias alélicas de los marcadores genéticos. Las variaciones en estas secuencias permiten estimar el grado de divergencia entre las poblaciones, cuya variabilidad puede deberse a diversos factores9,10.
En medicina forense los STR desempeñan un papel importante. Las bases de datos de ADN tanto de EE. UU. (Combined DNA Index System) como de países europeos (European Standard Set, UK Core Loci, Interpol Standard Set of Loci) se basan únicamente en estos loci para crear huellas genéticas11. La naciente comunidad de la genealogía genética utiliza estos loci para desarrollar bases de datos que contienen linajes de cientos de miles de individuos12.
El marcador TH01 es una repetición simple tetranucleótida (TCAT) con alelos que van desde 3 a 14 repeticiones, localizada en el intrón 1 (es decir, 01) del gen de la tirosina hidroxilasa en 11p15.513.
FGA es una repetición compuesta tetranucleótida (CTTT)n flanqueada a ambos lados por repeticiones degeneradas (TTCC)n que se encuentra en el tercer intrón del locus de la cadena alfa del fibrinógeno humano en 4q31.3. Los alelos reportados varían en tamaño desde 12.2 a 51.2, que abarca más de 35 repeticiones. Una deleción 2bp, desde la pérdida de un CT, en la región justo antes del motivo de repetición central es responsable de las microvariantes alélicas X.2 que son muy frecuentes en este sistema STR13.
D18S51 es una repetición sencilla AGAA en el brazo largo del cromosoma 18. Los alelos varían de tamaño de 5.3 a 40 repeticiones. Más de 70 alelos han sido reportados, siendo uno de los más polimórficos de uso común13.
D16S539 es una repetición simple de 4 nucleótidos (GATA) que van de 4 a 17 unidades localizado en 16q24.113. D2S1338 es una repetición de tetranucleótidos compuesta que se encuentra en el brazo largo del cromosoma 2. Los alelos van desde 10 a 31 repeticiones13.
MetodologíaSe colectaron muestras de sangre de 50 individuos sanos, donadores recurrentes, no relacionados entre ellos, nacidos en México y de padres mexicanos (G1)
El ADN se extrajo mediante el ensayo comercial High Pure PCR Template Preparation Kit V. 20 (Roche Mannheim Alemania, n.o de catálogo: 11796828001).
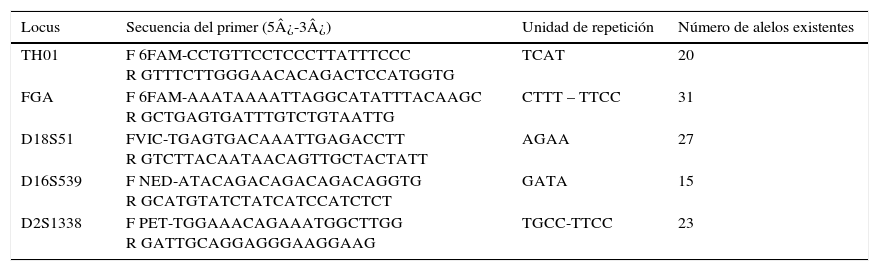
Las secuencias se amplificaron por PCR punto final. El iniciador sentido se marcó en el extremo 5’ con 6-FAM para tirosin hidroxilasa (TH01), cadena alfa de fibrinógeno (FGA) y amelogenina, el D18S51 con PET, el D16S539 con NED y el D2S1338 con VIC, para su posterior identificación por electroforesis capilar (tabla 1).
Descripción de las secuencias de los iniciadores para los STR autosómicos utilizados. Se indica la unidad de repetición y el número de alelos existentes
| Locus | Secuencia del primer (5¿-3¿) | Unidad de repetición | Número de alelos existentes |
|---|---|---|---|
| TH01 | F 6FAM-CCTGTTCCTCCCTTATTTCCC R GTTTCTTGGGAACACAGACTCCATGGTG | TCAT | 20 |
| FGA | F 6FAM-AAATAAAATTAGGCATATTTACAAGC R GCTGAGTGATTTGTCTGTAATTG | CTTT – TTCC | 31 |
| D18S51 | FVIC-TGAGTGACAAATTGAGACCTT R GTCTTACAATAACAGTTGCTACTATT | AGAA | 27 |
| D16S539 | F NED-ATACAGACAGACAGACAGGTG R GCATGTATCTATCATCCATCTCT | GATA | 15 |
| D2S1338 | F PET-TGGAAACAGAAATGGCTTGG R GATTGCAGGAGGGAAGGAAG | TGCC-TTCC | 23 |
Fuente: Buttler y Hill13.
Las amplificaciones se realizaron individualmente para cada par de iniciadores, en un volumen de reacción de 10uL usando una mezcla maestra que contenía 7.5U de Hot Start Master Mix (Qiagen, Cat. 203445), 0.5uM de cada par de iniciadores, 50ng de ADN y se ajustó el volumen final con agua grado biología molecular. Para amplificación se usaron las siguientes condiciones: 95°C por 10min, 32 ciclos: 94°C por 1min, 55°C por 1min, 72°C por 1min, 60°C por 45min.
La electroforesis capilar se llevó a cabo en un secuenciador Abi 3500, con un capilar de 50cm, usando el polímero POP 7 y determinando el peso molecular mediante GS 600 LIZ.
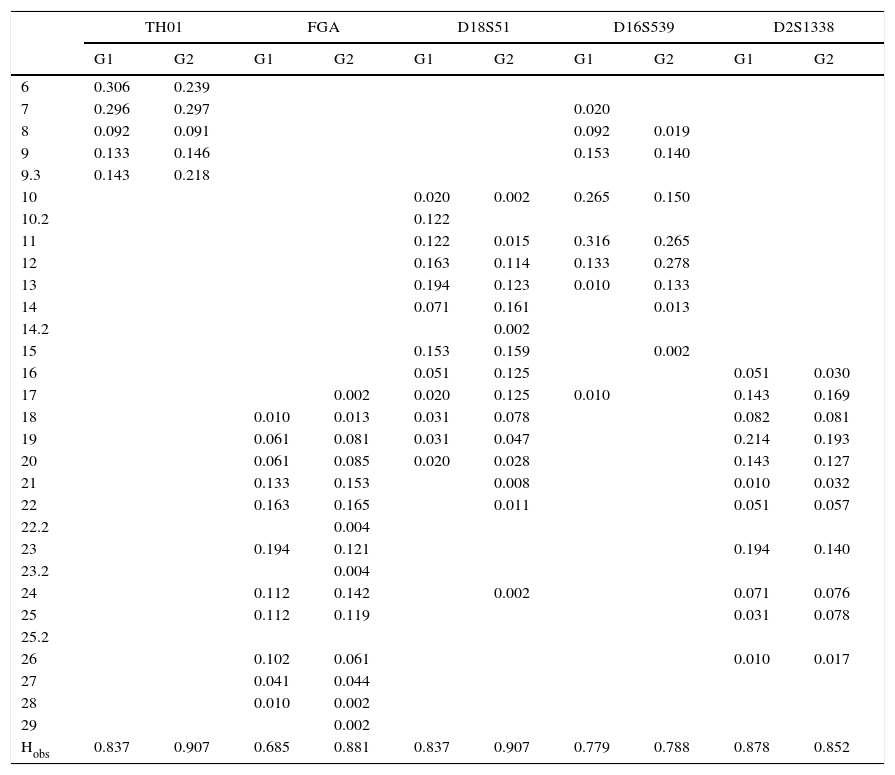
La longitud de los marcadores se comparó con las frecuencias reportadas en Combined DNA Index System (CODIS) para población latina (G2) (tabla 2).
Frecuencias de los marcadores analizados en donadores recurrentes comparadas con los datos reportados en la base de datos CODIS
| TH01 | FGA | D18S51 | D16S539 | D2S1338 | ||||||
|---|---|---|---|---|---|---|---|---|---|---|
| G1 | G2 | G1 | G2 | G1 | G2 | G1 | G2 | G1 | G2 | |
| 6 | 0.306 | 0.239 | ||||||||
| 7 | 0.296 | 0.297 | 0.020 | |||||||
| 8 | 0.092 | 0.091 | 0.092 | 0.019 | ||||||
| 9 | 0.133 | 0.146 | 0.153 | 0.140 | ||||||
| 9.3 | 0.143 | 0.218 | ||||||||
| 10 | 0.020 | 0.002 | 0.265 | 0.150 | ||||||
| 10.2 | 0.122 | |||||||||
| 11 | 0.122 | 0.015 | 0.316 | 0.265 | ||||||
| 12 | 0.163 | 0.114 | 0.133 | 0.278 | ||||||
| 13 | 0.194 | 0.123 | 0.010 | 0.133 | ||||||
| 14 | 0.071 | 0.161 | 0.013 | |||||||
| 14.2 | 0.002 | |||||||||
| 15 | 0.153 | 0.159 | 0.002 | |||||||
| 16 | 0.051 | 0.125 | 0.051 | 0.030 | ||||||
| 17 | 0.002 | 0.020 | 0.125 | 0.010 | 0.143 | 0.169 | ||||
| 18 | 0.010 | 0.013 | 0.031 | 0.078 | 0.082 | 0.081 | ||||
| 19 | 0.061 | 0.081 | 0.031 | 0.047 | 0.214 | 0.193 | ||||
| 20 | 0.061 | 0.085 | 0.020 | 0.028 | 0.143 | 0.127 | ||||
| 21 | 0.133 | 0.153 | 0.008 | 0.010 | 0.032 | |||||
| 22 | 0.163 | 0.165 | 0.011 | 0.051 | 0.057 | |||||
| 22.2 | 0.004 | |||||||||
| 23 | 0.194 | 0.121 | 0.194 | 0.140 | ||||||
| 23.2 | 0.004 | |||||||||
| 24 | 0.112 | 0.142 | 0.002 | 0.071 | 0.076 | |||||
| 25 | 0.112 | 0.119 | 0.031 | 0.078 | ||||||
| 25.2 | ||||||||||
| 26 | 0.102 | 0.061 | 0.010 | 0.017 | ||||||
| 27 | 0.041 | 0.044 | ||||||||
| 28 | 0.010 | 0.002 | ||||||||
| 29 | 0.002 | |||||||||
| Hobs | 0.837 | 0.907 | 0.685 | 0.881 | 0.837 | 0.907 | 0.779 | 0.788 | 0.878 | 0.852 |
El polimorfismo de la TH01 presentó el mismo número de repeticiones en ambos grupos, sin embargo, las frecuencias variaron en cada una de ellas. El alelo con 6 repeticiones fue el más frecuente en G1 (0.306), mientras que el de 7 lo fue en G2 (0296). En ambos casos el alelo con 10 repeticiones fue el menos frecuente (0.031 y 0.0085 respectivamente).
El número de alelos del FGA fue distinto en ambas poblaciones, el G1 no presento 4 alelos reportados para población latina (17, 22.2, 23.2 y 29), en ninguno de los 2 grupos se observó el alelo de 11 repeticiones que sí ha sido reportado en otras poblaciones, por ejemplo africanos, asiáticos o caucásicos. El alelo de 23 repeticiones fue el más frecuente en G1 (0.194), mientras que los alelos 18 y 28 fueron los menos prevalentes (0.01 en ambos casos). Comparando los datos de G2 el alelo de 22 es el más frecuente (0.1653) y los alelos de 17, 28 y 29 repeticiones son los menos frecuentes (0.0021 en los 3 casos).
El polimorfismo D18S51 en G1 presentó una alelo de 10.2 repeticiones no reportado en CODIS con una frecuencia de 0.1224, asimismo, careció de 4 alelos reportados para población latina (14.2, 21, 22,24). Las frecuencias observadas en G1 y G2 para cada alelo fueron distintas. El alelo más frecuente en ambos grupos fue el de 13 repeticiones (0.1939 y 0.1229 respectivamente), mientras que el menos frecuente fue el de 20 y 17 para G1 (0.02) y para G2 fueron 10, 14.2 y 24 (0.0021 en todos los casos).
Para el marcador D16S539, los valores de nuestra población fueron muy distintos a los reportados en CODIS. Los alelos de 7 y 17 repeticiones presentes en nuestra población no están reportados en CODIS para latinos. Por otra parte, los alelos 14 y 15 reportados en CODIS no se observaron en nuestra población. Los alelos más frecuentes fueron el de 11 (0.316) en G1 y de 12 en G2 (0.278). Los menos frecuentes fueron los de 13 y 17 (0.010) en G1 y 15 (0.0021) en G2.
En el caso del marcador D2S1338 se observó el mismo número de alelos en las mismas frecuencias reportadas por CODIS para la población latina. El alelo 19 (0.214 y 0.193) fue el más frecuente en ambos casos. El alelo menos frecuente en G2 fue el de 26 (0.0169), para G1 además del 26 también fue el de 21 (0.0102).
DiscusiónCuando un individuo es el resultado de una mezcla de ascendencias representa un mosaico genético.
Dentro del proyecto de los 1,000 genomas, la tipificación mexicana (MXL) corresponde a una muestra de Los Ángeles, California. Sin embargo la población hispana incluye los diversos grupos étnicos de América Latina; aunque las contribuciones genéticas significativas se pueden remontar hasta los mismos grupos indígenas, incluye también ascendencia europea y de poblaciones de África occidental. Dentro de esta amalgama genética cada población latina estará determinada por su propio origen étnico. Esta composición génica implica una comprensión más profunda de la evolución humana, ya que la diversidad genética entre las poblaciones latinas es particular en cada caso.
La población mexicana se conoce por ser una mezcla de múltiples características que desciende principalmente de linajes indígenas mesoamericanos y europeos, principalmente españoles, pero también tiene un origen africano que se deriva en gran parte de la trata de esclavos transatlántica, principalmente de África occidental, desde Senegal a Nigeria y Angola; por ejemplo, los mexicanos en promedio tienen menor ascendencia africana que los brasileños14.
El uso de marcadores de longitud variable es una estrategia viable, capaz de diferenciar el ADN de distintos individuos; y dado que la probabilidad de su coincidencia al azar es superior a 1/1x1015, lo convierte en una metodología aplicable en otras investigaciones, por ejemplo para la identificación de ADN fetal a partir de sangre periférica materna.
Nuestros resultados son diferentes a las frecuencias reportadas en la base CODIS para latinos; creemos que nuestra población de estudio es muy homogénea al tratarse únicamente de mexicanos con padres mexicanos, sin embargo, esto solo refleja las características de una muestra de habitantes de la Ciudad de México; aunque no intencional, sí presenta un sesgo, lo que a su vez nos obliga a tratar de obtener una muestra representativa de la población mexicana.
A diferencia de otras regiones del mundo en donde el colonialismo europeo dio lugar a una disminución significativa de la población nativa, en México los indígenas no solo contribuyeron en el proceso de mestizaje, sino que actualmente constituyen un segmento importante de la población15. Estudios recientes han mostrado que el promedio de ancestría indígena es del 53%, mientras que el 42 y 5% son de origen europeo y africano, respectivamente16.
En 2014 Moreno-Estrada et al.17 presentaron los resultados de la genotipificación de 1,000 personas, representada por 20 grupos indígenas y 11 poblaciones mestizas, sin embargo, la creación de una base de datos mexicana debería incluir, además de los grupos endémicos, a aquellos que comparten el territorio con zonas urbanas, ya que su aporte genético da lugar a las nuevas poblaciones mestizas metropolitanas, de esta forma podríamos evaluar con mucha mayor resolución el impacto de los componentes indígenas en la población.
ConclusiónEn este trabajo presentamos los resultados de un estudio preliminar que nos permitió comparar las frecuencias de los polimorfismos de amplitud variable en una muestra de origen mestiza mexicana con otra población latina. Las diferencias encontradas aquí nos muestran la necesidad de crear bases de datos de referencia de nuestra población, teniendo en cuenta el mestizaje que nos caracteriza.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.