En la provincia del Chaco, el agua subterránea representa una fuente alternativa, y muchas veces única, para el consumo humano; esta es utilizada en el 14% de los hogares. A pesar de que se reconoce el riesgo de la exposición al agua contaminada, la prevalencia de los diferentes patotipos de Escherichia coli en ambientes acuáticos no ha sido bien caracterizada. E. coli enteroagregativo (ECEA) es un patógeno emergente cuya importancia en la salud pública mundial se incrementó y quedó claramente establecida en los últimos años. El objetivo del presente trabajo fue detectar la presencia de ECEA típico mediante el reconocimiento de los factores de virulencia aap, AA probe y aggR por reacción en cadena de la polimerasa, en fuentes de agua subterráneas de la provincia del Chaco. Se identificó E. coli en 36 (38,7%) de las 93 muestras estudiadas, provenientes de diferentes localidades. De esos 36 aislamientos, se identificaron 6 (16,7%) portadores de los genes de ECEA, lo que representa una prevalencia del 6,4% considerando las 93 fuentes de agua subterránea estudiadas. De esos 6 aislamientos, 3 eran portadores del gen aap, 2 del gen AA probe y uno de la combinación aggR/aap. El presente trabajo representa el primer aporte en el estudio de la presencia y distribución de genes de virulencia de ECEA en fuentes de agua subterránea de la región.
Groundwater is an important source of drinking water for many communities in Northern Argentina; particularly, in the province of Chaco, where about 14% of households use this natural resource. Enteroaggregative Escherichia coli is an emerging pathogen whose global importance in public health has increased in recent years. Despite the significant risk of disease linked to contaminated water exposure, the prevalence of E. coli pathotypes in aquatic environments is still not so well defined. The aim of the present study was to detect the presence of typical enteroaggregative E. coli through the recognition of its virulence factors aap, AA probe and aggR by molecular techniques. A total of 93 water samples from different small communities of Chaco were analyzed. E. coli was identified in 36 (38.7%) of the tested samples. Six strains isolated from different samples harbored the studied genes. Of these 6 isolates, 3 carried the aap gene, 2 the AA probe and the last one the combination of aap/aggR genes. The prevalence of E. coli isolates harboring enteroaggregative virulence genes in groundwater sources was 6.4%. This work represents the first contribution to the study of the presence and distribution of virulence genes of EAEC in groundwater sources in this region of Argentina.
El acceso al agua potable es esencial para la vida. Sin embargo, debido al crecimiento de la población, el incremento de la industrialización y el cambio climático, la escasez de fuentes de agua para consumo libres de contaminantes es uno de los mayores problemas que enfrenta la población mundial. Se estima que para el año 2025, el 60% de la población mundial sufrirá problemas de escasez de agua. Entre los diferentes contaminantes del agua (compuestos orgánicos, inorgánicos y microbios), la Organización Mundial de la Salud considera los microbios como la principal amenaza, tanto en países desarrollados como en vías de desarrollo4.
En la provincia del Chaco, solamente el 58,6% de los hogares tienen acceso al sistema de distribución del agua potable dentro de la vivienda, mientras que en el 17,9% de los hogares el acceso a la red pública tiene lugar fuera de aquella. El resto de los hogares (23,5%) se abastecen de agua a partir de fuentes subterráneas, superficiales, de lluvia o bien por trasporte por cisternas. A su vez, solo el 26,4% de los hogares tienen cloaca conectada a la red pública. En el resto de los hogares, la descarga de los efluentes cloacales se realiza a través de cámara séptica, pozo ciego o a excavaciones directas en la tierra14.
Poseemos limitados conocimientos sobre los principios que gobiernan la prevalencia y la patogénesis de los microorganismos presentes en las fuentes de agua; esta carencia se puede atribuir a varios factores. En primer lugar, a la dificultad que plantea la detección, identificación y cuantificación de los microorganismos en el agua, lo que requiere muchas veces la combinación de métodos clásicos y moleculares. En segundo lugar, a que la virulencia de los patógenos presentes en los ambientes acuáticos depende de las condiciones ambientales y del tratamiento que reciba el agua de la fuente antes de destinarla al consumo humano. Finalmente, cabe señalar que la transmisión de los patógenos al ser humano a través del agua es un proceso complejo que depende, entre otros, de la dosis infectiva, del microorganismo involucrado y del estado inmunitario del ser humano3.
La calidad bacteriológica del agua procedente de fuentes subterráneas, que están naturalmente protegidas, es generalmente mejor que aquella procedente de fuentes superficiales no protegidas. No obstante, existen informes de brotes por patógenos fecales originados a partir de estas fuentes10,23. La contaminación microbiológica del agua subterránea se atribuye principalmente a la infiltración de agua contaminada con materia fecal humana o de animales. Este proceso está en relación con un inadecuado diseño, ubicación, construcción, operación o mantenimiento de los pozos y perforaciones utilizados para acceder al agua, como también con las características geológicas y las condiciones climáticas. Complementariamente, las bombas manuales utilizadas para la extracción de agua pueden actuar como reservorios microbianos y como fuente de contaminación del agua subterránea10,21,25.
Para la determinación de la calidad bacteriológica del agua destinada al consumo humano se recurre a ciertos indicadores. El papel tradicional de estos es detectar la contaminación fecal del agua y de allí inferir la probable presencia de patógenos y el consiguiente riesgo para la salud. Así, el recuento de heterótrofos y de coliformes totales da idea de la calidad general del agua, en tanto que E. coli y los enterococos son indicadores de contaminación fecal. Asimismo, la presencia de un patógeno determinado no se puede asociar normalmente al concepto de indicador, dado que su ausencia no es indicativa de la ausencia de otro microorganismo patógeno. Entre los patógenos bacterianos diseminados a través del agua, los causantes de cuadros entéricos como E. coli, Vibrio cholerae y Salmonella spp. son usualmente transmitidos al ser humano por consumo de agua y de alimentos con ellos contaminados15,25,31.
La especie E. coli es un bacilo gram negativo que pertenece a la familia Enterobacteriaceae. Coloniza el tracto gastrointestinal del recién nacido a las pocas horas de vida y, a partir de ese momento, huésped y microorganismo obtienen mutuo beneficio. Sin embargo, existen clones de este microorganismo que han adquirido diferentes factores de virulencia que les permiten adaptarse a nuevos nichos y causar un amplio espectro de enfermedades35. El papel de E. coli como patógeno entérico aumentó con la aparición de E. coli O157:H7. Las cepas de E. coli causantes de enteritis se han clasificado en 5 grupos principales, a los cuales se les agrega E. coli de adherencia difusa (ECAD). Cada uno de ellos engloba diferentes serotipos, factores de virulencia y mecanismos de patogenia. Así, según los genes de virulencia presentes en cada cepa, se pueden clasificar como: E. coli enteropatógeno (ECEP), E. coli enteroinvasivo (ECEI), E. coli enterotoxigénico (ECET), E. coli enterohemorrágico (ECEH) y E. coli enteroagregativo (ECEA)13,18,19. El principal reservorio de ECEA, ECEI y ECAD es el ser humano. El elevado inóculo requerido para el desarrollo de la infección por ECEA indica el agua y los alimentos como vehículos de este patotipo12,22,34.
A pesar de que se reconoce el riesgo que entraña la exposición al agua contaminada, la prevalencia de los diferentes patotipos de E. coli en ambientes acuáticos no ha sido bien caracterizada33.
Las cepas de E. coli diarreogénicas representan una de las principales causas de diarreas en países desarrollados así como en vías de desarrollo. Sin embargo, se desconoce el real impacto de estas cepas patogénicas como causales de enfermedad diarreica en el mundo entero, dado que la mayoría de los laboratorios clínicos no tienen implementadas técnicas para su detección.
Dentro de este grupo de cepas diarreogénicas, E. coli enteroagregativo se presenta como un patógeno emergente y genéticamente heterogéneo9,11, el cual fue descrito por primera vez en 1987 por Nataro et al.28. Actualmente se considera a este patotipo como un problema de salud pública, sin distinción entre países desarrollados y en vías de desarrollo. Es agente causal de diarrea persistente, tanto en población adulta como pediátrica, y también está relacionado con el retardo del crecimiento en este último grupo etario6,28. Su mecanismo de patogénesis no está completamente dilucidado. Se postula que coloniza la mucosa intestinal, predominantemente la colónica. Allí la bacteria exacerba la producción de mucus, que la atrapa en un biofilm, produciendo daños a la mucosa y secretando enterotoxinas y citotoxinas29,35.
A pesar de que se han descrito gran diversidad de toxinas, adhesinas y proteínas involucradas en la patogénesis de ECEA, los genes que las codifican son muy variables y ninguno de ellos fue encontrado en todos los aislamientos de este patotipo16,35.
El rasgo que define a las cepas de ECEA es su capacidad de adherirse en cultivos a células epiteliales HEp-2 con una apariencia de ladrillos apilados. Si bien el cultivo celular constituye la técnica de referencia para la identificación de estos microorganismos, este procedimiento resulta engorroso y no es fácil implementarlo en laboratorios de mediana complejidad. Las técnicas moleculares permiten la detección rápida del grupo heterogéneo de genes de virulencia presentes en el aislamiento y constituye, además, una herramienta epidemiológica6,9,29. La presencia del plásmido pAA, donde se encuentran codificados el mayor número de genes de virulencia, permite diferenciar entre cepas típicas y atípicas de este patotipo. Las cepas portadoras de este plásmido representan un importante subgrupo considerado como cepas típicas de ECEA16,35. Entre los genes más estudiados de este patotipo se encuentran el aggR (activador transcripcional), el aap (que codifica a la dispersina), el AA probe (que codifica una proteína de membrana externa, la cual forma parte de un sistema de proteínas transportadoras) y el astA (que codifica la enterotoxina)7,16.
La ECEA fue informada como agente causal de cuadros de diarrea tanto aguda como persistente en diferentes países latinoamericanos, como Chile, Brasil y Perú34. En nuestro país, Rüttler et al.32 detectaron la presencia de ECEA en un brote de diarrea aguda en niños menores de 2 años en la ciudad de Mendoza. Datos de la región del noreste de Argentina (NEA) señalan a ECEA como el patotipo más frecuente, junto con ECET, los que fueron recuperados de muestras de pacientes pediátricos con diarrea aguda en la ciudad de Corrientes. Esos mismos patotipos fueron los más prevalentes en la población adulta de esa ciudad8,24. E. coli enteroagregativo O104:H4 fue la cepa responsable del brote que se inició en mayo del 2011 en Alemania. En ese brote se informó un total de 3.842 casos, que incluyeron a 2.987 individuos con gastroenteritis, con 18 defunciones, y 855 casos de síndrome urémico hemolítico que derivaron en 35 defunciones. Los análisis moleculares demostraron que la cepa causante del brote albergaba una combinación inusual de genes de virulencia provenientes de 2 patotipos hasta ese momento diferentes: ECEA y E. coli productora de toxina Shiga (STEC)27.
Los genes estudiados mediante el protocolo de PCR utilizado en este trabajo fueron aggR, activador transcripcional de varios genes de virulencia de este patotipo; AA probe (también conocido como CVD432), que codifica una proteína de membrana externa, la cual forma parte de un sistema de proteínas transportadoras, y finalmente aap, que codifica la dispersina6,26.
El objetivo del presente trabajo fue detectar por la técnica de PCR, mediante el reconocimiento de los factores de virulencia aggR, aap y AA probe, la presencia de E. coli enteroagregativo en fuentes de agua subterráneas de la provincia del Chaco, así como evaluar el papel de estas fuentes subterráneas en la transmisión de ECEA.
Materiales y métodosToma de muestras: las muestras de agua procedieron de fuentes de agua subterráneas a las cuales se accedió a través de los pozos o perforaciones existentes en los hogares. Solo se tomaron muestras en aquellos hogares donde el agua subterránea es la única fuente disponible para el consumo, por lo que su contaminación representa un riesgo para la salud. Las muestras fueron recolectadas en botellas estériles de 150-250 cm3 de capacidad, dejando un espacio aéreo para facilitar la homogeneización antes del estudio, y se mantuvieron refrigeradas hasta su llegada al laboratorio, donde se procesaron2,31.
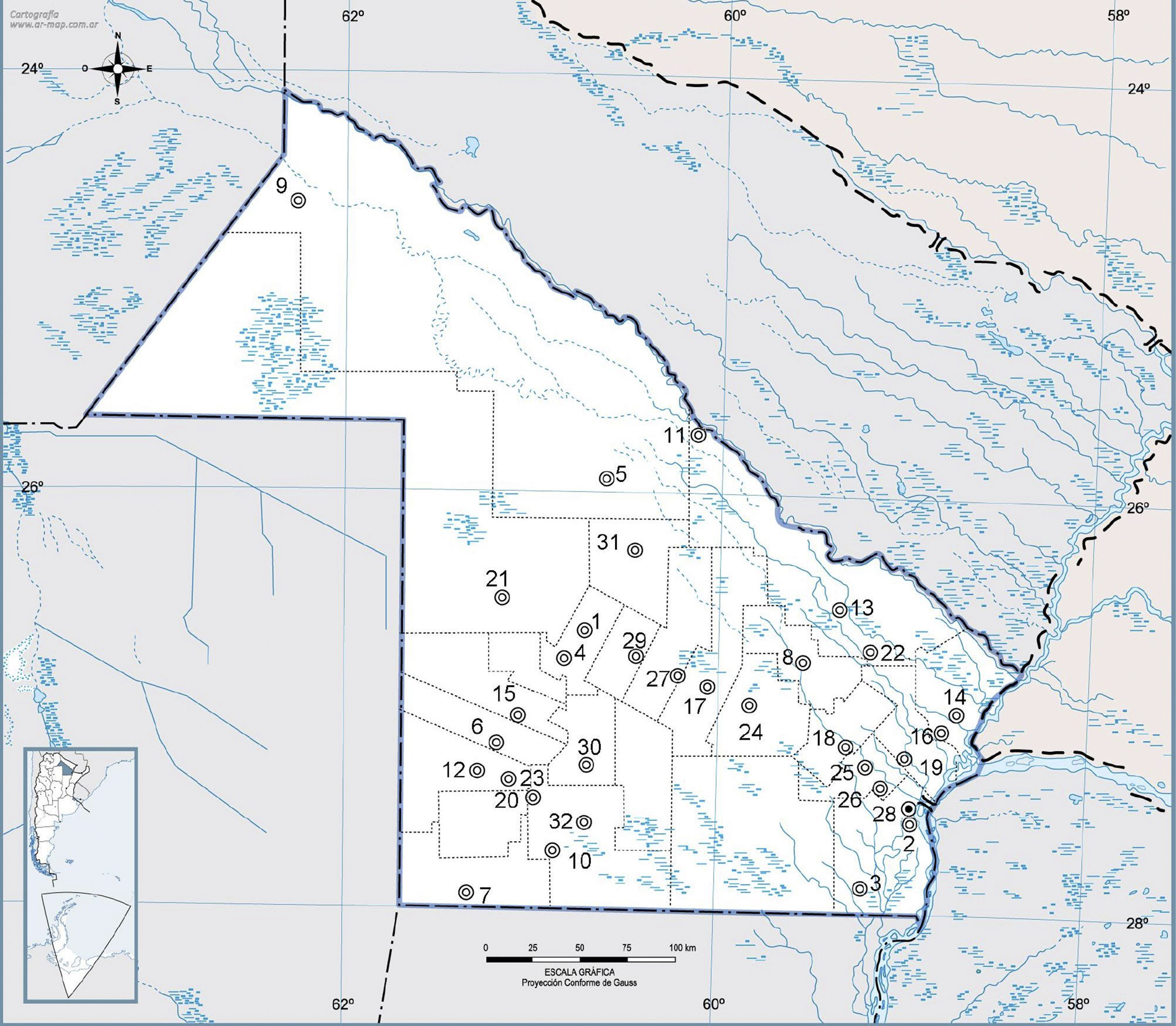
Las muestras fueron tomadas en las siguientes localidades de la provincia (fig. 1): Avia Terai, Barranqueras, Basail, Campo Largo, Castelli, Charata, Chorotis, Colonias Unidas, Comandancia Frías, Coronel Du Graty, Fortín Lavalle, General Pinedo, General San Martín, La Leonesa, Las Breñas, Las Palmas, Machagai, Makallé, Margarita Belén, Mesón de Fierro, Pampa del Infierno, Pampa Almirón, Pampa Landriel, Presidencia de La Plaza, Puerto Bastiani, Puerto Tirol, Quitilipi, Resistencia, Sáenz Peña, San Bernardo, Tres Isletas y Villa Ángela.
Localización geográfica de las fuentes de agua subterráneas evaluadas.
1: Avia Terai; 2: Barranqueras; 3: Basail; 4: Campo Largo; 5: Castelli; 6: Charata; 7: Chorotis; 8: Colonias Unidas; 9: Comandancia Frías; 10: Coronel Du Graty; 11: Fortín Lavalle; 12: General Pinedo; 13: General San Martín; 14: La Leonesa; 15: Las Breñas; 16: Las Palmas; 17: Machagai; 18: Makallé; 19: Margarita Belén; 20: Mesón de Fierro; 21: Pampa del Infierno; 22: Pampa Almirón; 23: Pampa Landriel; 24: Presidencia de La Plaza; 25: Puerto Bastiani; 26: Puerto Tirol; 27: Quitilipi; 28: Resistencia; 29: Sáenz Peña; 30: San Bernardo; 31: Tres Isletas; 32: Villa Ángela.
Cultivo e identificación bioquímica: las muestras se procesaron por la técnica de enriquecimiento directo de 100ml en mismo volumen de medio EC (EC Medium. Acumedia Manufacturers) doble concentración, lo cual brindó información cualitativa sobre la presencia o ausencia del microorganismo objeto de estudio. Las botellas inoculadas con las muestras previamente agitadas se incubaron a 35±0,5°C y se inspeccionaron a las 24 y 48h. A partir de las botellas donde se evidenció desarrollo bacteriano se realizaron repiques en placas de agar eosina azul de metileno (EMB Britania), las que se incubaron a 35±0,5°C durante 24h para la recuperación de E. coli. Todos los aislamientos fueron identificados mediante las siguientes pruebas bioquímicas clásicas: producción de H2S y gas, utilización de glucosa, lactosa, sacarosa y citrato, producción de ureasa e indol, descarboxilación de aminoácidos, producción de oxidasa y reacción de orto-nitrofenil galactopiranósido, motilidad y producción de fenilalanina desaminasa2.
En lugar de emplear la técnica de filtración de membrana, se recurrió al enriquecimiento directo; esto obedeció al desconocimiento de la concentración bacteriana inicial en la muestra de agua, que podría producir un sobrecrecimiento de colonias y dificultar la detección de E. coli2.
Caracterización genotípica: las cepas identificadas bioquímicamente como E. coli se sometieron a amplificación mediante PCR múltiple (mPCR). Se utilizaron 3 pares de iniciadores que detectan 3 genes codificantes de factores de virulencia de ECEA: aap, aggR y AA probe. La secuencia de los oligonucleótidos utilizados y el protocolo de PCR son los descritos por Cerna et al.6. Se incluyó la cepa E. coli 17-2, provista por el Servicio de Fisiopatogenia del INEI-ANLIS «Dr. Carlos G. Malbrán», como control positivo6.
Se definió como muestra positiva aquella en la cual se recuperaron cepas bioquímicamente identificadas como E. coli. Por cada muestra positiva se analizaron por PCR de manera separada 2 de estas cepas. La lisis bacteriana se realizó preparando una suspensión bacteriana en solución de tritón al 1% en tubos de polipropileno. Los tubos se colocaron en un baño a 100°C durante 10min, luego se centrifugó durante 1min a 10 000rpm. A partir de los templados obtenidos, se realizó una dilución 1/10 para emplearla en la mPCR. La electroforesis se efectuó en gel de agarosa al 2,0%, posteriormente los geles se tiñeron con GelRed® y se tomó registro fotográfico bajo luz UV. El análisis de las bandas se realizó con el programa UVI-Pro. El análisis de los resultados se realizó a través del programa EpiInfo6.
La selección de 2 cepas de E. coli por muestra positiva para su estudio molecular se realizó por razones operativas25,33. Las técnicas moleculares aplicadas al control de aguas complementan las limitaciones de los métodos basados en cultivo, pero requieren estandarización y validación3,17. Por el contrario, las técnicas de detección de indicadores bacterianos basadas en su cultivo o cuantificación se encuentran estandarizadas. Esta estandarización permite asegurar la calidad higiénica del agua de bebida1-3.
Resultados y discusiónEn la provincia del Chaco, el 14% de los hogares se abastecen de agua a partir de fuentes subterráneas ubicadas tanto dentro como fuera del terreno, y el agua es utilizada directamente, sin tratamiento previo14.
Se analizaron 93 muestras procedentes de fuentes subterráneas destinadas al consumo humano de diferentes localidades de la provincia del Chaco. Se identificó E. coli en 36 (38,7%) de las muestras estudiadas. El Código Alimentario Argentino, en su capítulo xii, establece la ausencia de E. coli en 100ml como uno de los criterios microbiológicos del agua destinada al consumo humano1. En el presente trabajo se determinó que el 38,7% no cumplía con este criterio, por lo que esas muestras se deberían considerar no aptas para consumo humano. Si bien no se detectó la presencia de este microorganismo en el 61,3% restante, al no haberse determinado la totalidad de los parámetros microbiológicos de potabilidad, no se puede dictaminar que esas muestras sean bacteriológicamente aptas para el consumo humano1. En las fuentes subterráneas de agua, la ausencia del indicador de contaminación fecal indica que es improbable la ocurrencia de patógenos. Por el contrario, tan pronto como se detecten estos indicadores, la probabilidad de ocurrencia de patógenos se incrementa significativamente31. En estas fuentes la sobrevida de las bacterias aumenta por las bajas temperaturas, la humedad del suelo, el pH neutro o alcalino y la presencia de carbono orgánico5.
Por mPCR se encontró que 6 de las 36 cepas de E. coli aisladas (16,7%) portaban los genes de virulencia de ECEA estudiados, lo que representa una prevalencia del 6,4% en el total de fuentes de agua subterránea analizadas. De estos 6 aislamientos, 3 fueron portadores del gen aap, 2 del gen AA probe y la sexta portaba la combinación aggR/aap. La técnica de PCR utilizada en el presente trabajo permitió detectar la presencia de 3 genes de virulencia de ECEA presentes en el plásmido AA, es decir, permitió detectar genéticamente las cepas típicas de ECEA, las que representan el grupo mayoritario de este patotipo6,16.
Otros autores detectaron el gen AA probe solamente en cepas de ECEA; el gen aggR se detectó en el 80% de las cepas de este patotipo, en tanto que el gen aap también se detectó en cepas no patogénicas de E. coli16,26. Por otro lado, ninguno de los 3 genes estudiados se detectó en los otros 5 patotipos de E. coli. Todos los trabajos en los cuales se determinó la distribución de los genes de virulencia se realizaron a partir de muestras de pacientes de diferentes grupos etarios, con diarrea o sin esta. En la literatura consultada no se encontraron trabajos referidos al estudio de la distribución de los genes de este patotipo en cepas de origen ambiental6,16,26.
Masters et al.22 identificaron al gen astA, que codifica la enterotoxina termoestable, como el más prevalente de todos los genes productores de toxina de los diferentes patotipos de E. coli estudiados en fuentes superficiales de agua de Australia, y relacionaron dicho gen con ECEA. Sin embargo, el citado gen también fue detectado en ECET y la toxina termoestable también es producida por los patotipos ECET, ECEH y ECEP. Sidhu et al.33 detectaron la presencia de ECEA con una frecuencia del 23% en períodos secos y del 12% en períodos húmedos en fuentes superficiales de Brisbane, Australia, mediante la detección de los genes aggR y astA, que codifica para la enterotoxina termoestable. Orsi et al.30 identificaron ECEA como el patotipo más prevalente en fuentes de agua superficiales de San Pablo, Brasil, a través del uso de la sonda AA probe.
Al igual que en el trabajo de Sidhu et al.33, en la presente investigación se utilizó al gen aggR para la identificación de ECEA en las muestras de agua subterránea. Sin embargo, no se realizó la detección del gen astA por ser un gen compartido por otros patotipos de E. coli33.
En la revisión bibliográfica realizada no se encontraron trabajos que hayan abordado la detección de genes de este patotipo en fuentes de agua subterráneas. No obstante, sí se halló un estudio que lo detectó como agente causal de diarrea en la India, asociado al consumo de agua procedente de esta clase de fuente13.
Considerando que el agua puede actuar como vehículo de las cepas de ECEA y ante los datos de prevalencia de este patotipo en la población de la región NEA, se hace evidente que su distribución en el ambiente requiere más estudios8,24. Los conocimientos generados son importantes en términos de salud pública, principalmente si se considera que cada fuente subterránea abastece a una familia y que los casos de infecciones por patógenos presentes en el agua raramente son comunicados15,23.
La combinación de técnicas utilizadas en el presente trabajo, esto es, el cultivo, la identificación bioquímica y la mPCR para la detección de los genes presentes en el plásmido pAA, permitió la detección de las cepas típicas de ECEA viables. Asimismo, por la heterogeneidad de este patotipo resulta difícil encontrar genes del cromosoma que permitan su diferenciación de otros patotipos de E. coli. Ante la detección de cepas típicas se deberían continuar los estudios para evaluar la presencia o ausencia de las cepas atípicas de ECEA, como también de cepas viables no cultivables, las que mantienen todo su potencial patogénico. Diferentes investigaciones confirman la persistencia de estas últimas cepas de E. coli en el ambiente15,20.
La identificación de cepas de E. coli que poseen genes compatibles con ECEA en las fuentes de agua subterránea aquí estudiadas requiere su confirmación a través de la técnica de referencia, el cultivo celular. Para ello, las cepas serán derivadas al laboratorio de referencia. Dada la heterogeneidad de este patotipo, la presencia de determinados genes de virulencia no necesariamente indica que la cepa es patogénica, a menos que posea la combinación adecuada de genes de virulencia para producir enfermedad en un determinado huésped22,26,34. Sin embargo, este análisis escapa al objetivo del presente trabajo.
Las técnicas basadas únicamente en cultivos resultan insuficientes para la detección de este patógeno en las fuentes de agua y no permiten garantizar la protección de la salud17. De allí que la combinación de la técnica de cultivo y los métodos moleculares representen una alternativa válida en la detección de patógenos presentes en esta clase de fuentes de agua3,20.
En los países desarrollados, la protección contra las enfermedades transmitidas por el agua representa una meta de salud pública que ya lleva décadas. Con tal fin se establecieron diversas estrategias basadas en los principios de barreras múltiples, como lo son la protección de las fuentes de agua de la contaminación microbiológica, el tratamiento para la remoción y/o inactivación de patógenos y el desarrollo de criterios operativos para evitar la contaminación en la extracción y distribución de agua21. En tanto, en la provincia del Chaco se hace evidente la necesidad del trabajo conjunto para el logro de los denominados Objetivos de Desarrollo Sostenible, donde en el objetivo 6 se establece la necesidad de «asegurar la disponibilidad y la gestión sostenible de agua y saneamiento para todos».
En el presente trabajo se determinó una prevalencia de cepas típicas de ECEA del 6,4%, de modo que estas fuentes de agua pueden actuar como reservorio y vehículo de dichas cepas. Ante los resultados obtenidos, no se debe subestimar la presencia de un potencial riesgo para la salud en las personas que consumen de las fuentes de agua estudiadas.
Considerado ECEA como un patógeno emergente, el presente estudio revela claramente la necesidad de desarrollar nuevas líneas de investigación tendientes a esclarecer el impacto que tiene para la salud pública la presencia de cepas de E. coli portadoras de genes de virulencia en fuentes de agua destinadas al consumo humano en nuestra región.
Este trabajo representa el primer aporte en el estudio de la presencia y distribución de los genes de virulencia de ECEA en fuentes de agua subterránea de la región.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.