El complejo Burkholderia cepacia está formado por 22 especies conocidas como patógenos oportunistas en personas inmunocomprometidas, especialmente en aquellas con fibrosis quística. También se aíslan de infecciones nosocomiales y son difíciles de erradicar debido a su capacidad intrínseca para resistir una gran variedad de antibióticos. En general, estas especies presentan genomas de gran tamaño (hasta 9 Mpb) divididos en 2-5 replicones. Esta característica aporta una gran versatilidad metabólica, que se considera importante para habitar el suelo, el agua, las plantas, incluso los nódulos en leguminosas. Algunas especies del complejo B. cepacia exhiben actividades benéficas, como biorremediación, biocontrol y promoción del crecimiento vegetal. No obstante, debido a su papel en infecciones de humanos, su uso en la agricultura está restringido. El complejo B. cepacia es un tema constante de estudio debido a su impacto en el sector salud y su potencial en la agricultura. En este trabajo se examina la historia del complejo B. cepacia y se revisa la información reciente relacionada con este grupo de bacterias.
The Burkholderia cepacia complex is a group of 22 species, which are known as opportunistic pathogens in immunocompromised people, especially those suffering from cystic fibrosis. It is also found in nosocomial infections and is difficult to eradicate due to intrinsic resistance to several antibiotics. The species have large genomes (up to 9 Mbp), distributed into 2-5 replicons. These features significantly contribute to genome plasticity, which makes them thrive in different environments like soil, water, plants or even producing nodules in legume plants. Some B. cepacia complex species are beneficial in bioremediation, biocontrol and plant-growth promotion. However, because the B. cepacia complex is involved in human infection, its use in agriculture is restricted. B. cepacia complex is being constantly studied due to the health problems that it causes and because of its agricultural potential. In this review, the history of B. cepacia complex and the most recently published information related to this complex are revised.
Burkholderia sensu lato se encuentra constituida por más de 100 especies que habitan diferentes ambientes, como el agua y el suelo. Asimismo, pueden establecer simbiosis con plantas, ya sea como endófito o desarrollando nódulos en leguminosas. También se han descrito especies que pueden comportarse como patógenas de humanos (algunas con carácter de oportunistas), plantas y animales. Recientemente, sugerimos que Burkholderia está constituida por diferentes grupos filogenéticos, los cuales podrían representar nuevos géneros bacterianos37,38. Análisis filogenómicos basados en el análisis de secuencias de genomas condujeron a la propuesta de separar el género en Burkholderia, Paraburkholderia y Caballeronia26,74. Sin embargo, existe cierta resistencia en parte de la comunidad científica para aceptar la división del género24. El subcomité para la taxonomía de Rhizobium y Agrobacterium del International Committee on Systematics of Prokaryotes (ICSP), durante su reunión en Budapest del 25 de agosto del 2016, hizo notar que existen razones válidas para dividir a Burkholderia; no obstante, establece que se requiere un estudio filogenómico a gran escala para definir este grupo bacteriano21. Se realizó un estudio de este tipo mediante la comparación de 106 proteínas conservadas en 92 especies de Burkholderia10. El análisis mostró 5 linajes que corresponden a Burkholderia, Paraburkholderia, Caballeronia, Robbsia andropogonis y Paraburkholderia rhizoxinica. R. andropogonis también fue propuesta como un nuevo género bacteriano recientemente56. Con esta nueva evidencia, es posible que el ICSP defina la situación del género Burkholderia sensu lato en un tiempo corto.
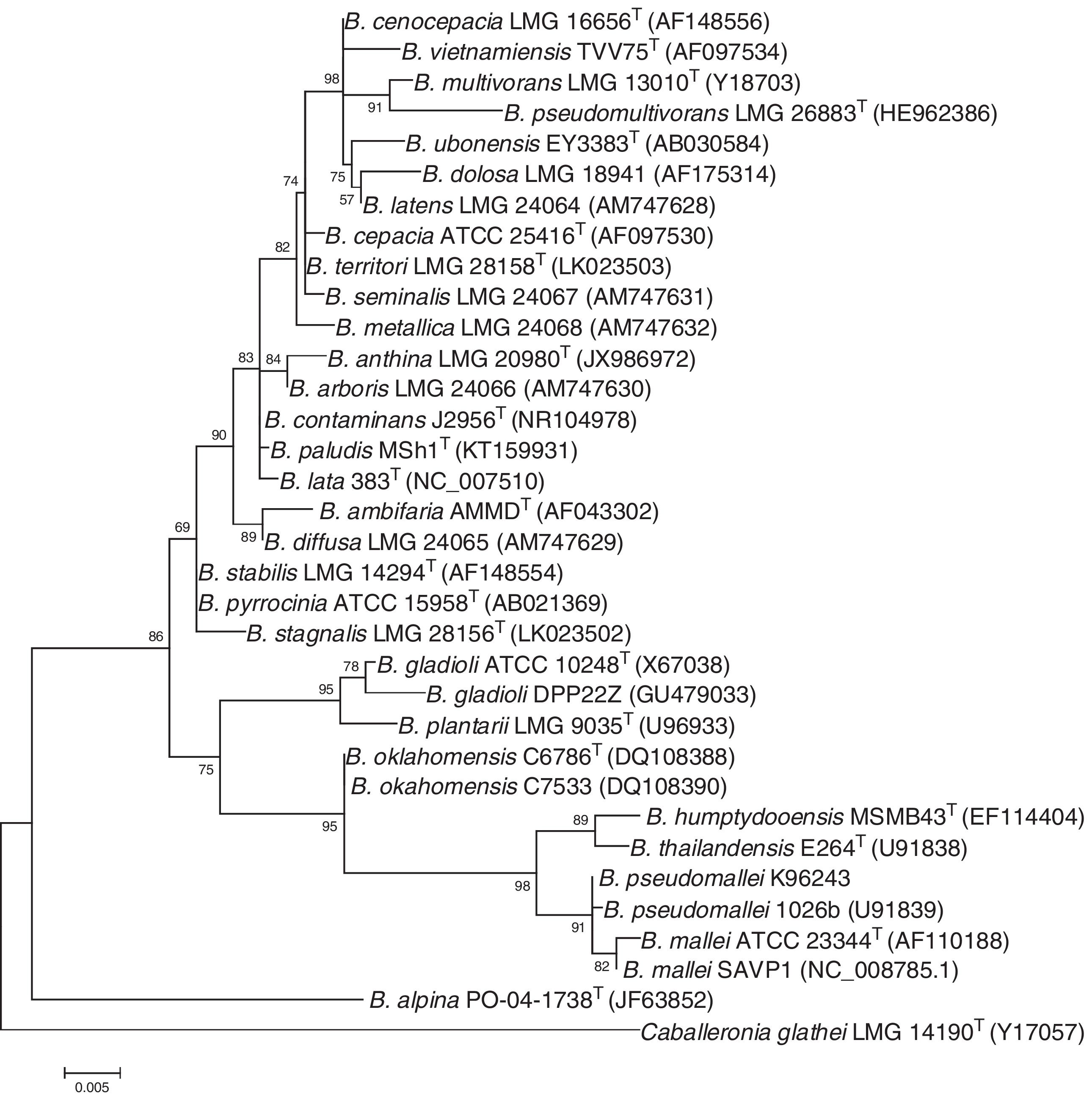
TaxonomíaEl género Burkholderia fue propuesto en 199288, reclasificando 7 especies de Pseudomonas: Pseudomonas cepacia, Pseudomonas mallei, Pseudomonas pseudomallei, Pseudomonas plantarii, Pseudomonas caryophylli, Pseudomonas pickettii y Pseudomonas solanacearum. Posteriormente, Burkholderia pickettii y Burkholderia solanacearum fueron transferidas al género Ralstonia89. Unos años después, se estudió la estructura taxonómica de Burkholderia cepacia, especie tipo del género, analizando 128 cepas de los géneros Pseudomonas, Burkholderia y Ralstonia81. Utilizando una batería de pruebas fenotípicas y genotípicas, se demostró que B. cepacia se encontraba constituida por 5 especies genómicas, las cuales fueron denominadas genomovares I a V. El genomovar agrupa cepas que comparten características fenotípicas similares, pero que genotípicamente son diferentes80. En conjunto con la descripción de los genomovares de B. cepacia, se propuso que estos formaran parte del complejo B. cepacia81. En el momento presente, los diferentes genomovares han sido descritos como nuevas especies y algunas otras nuevas especies han sido incluidas en el complejo B. cepacia, el cual está formado por 22 especies (tabla 1). Las especies del complejo B. cepacia forman un grupo muy estable dentro del género Burkholderia cuando se analiza filogenéticamente el fragmento 16S del gen rrs. Aunado a esto, un número importante de especies del complejo B. cepacia están siendo caracterizadas para nombrarlas formalmente como nuevas especies24,84. En la actualidad, este grupo está relacionado filogenéticamente con el grupo Burkholderia pseudomallei y con algunas especies patógenas de plantas26 (fig. 1).
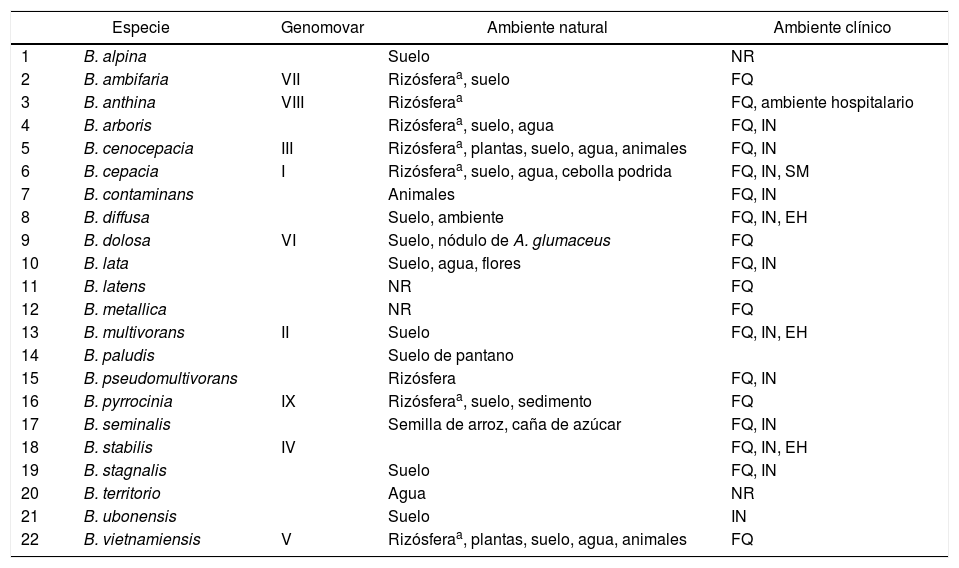
Lista de especies del complejo Burkholderia cepacia descritas hasta 2017
| Especie | Genomovar | Ambiente natural | Ambiente clínico | |
|---|---|---|---|---|
| 1 | B. alpina | Suelo | NR | |
| 2 | B. ambifaria | VII | Rizósferaa, suelo | FQ |
| 3 | B. anthina | VIII | Rizósferaa | FQ, ambiente hospitalario |
| 4 | B. arboris | Rizósferaa, suelo, agua | FQ, IN | |
| 5 | B. cenocepacia | III | Rizósferaa, plantas, suelo, agua, animales | FQ, IN |
| 6 | B. cepacia | I | Rizósferaa, suelo, agua, cebolla podrida | FQ, IN, SM |
| 7 | B. contaminans | Animales | FQ, IN | |
| 8 | B. diffusa | Suelo, ambiente | FQ, IN, EH | |
| 9 | B. dolosa | VI | Suelo, nódulo de A. glumaceus | FQ |
| 10 | B. lata | Suelo, agua, flores | FQ, IN | |
| 11 | B. latens | NR | FQ | |
| 12 | B. metallica | NR | FQ | |
| 13 | B. multivorans | II | Suelo | FQ, IN, EH |
| 14 | B. paludis | Suelo de pantano | ||
| 15 | B. pseudomultivorans | Rizósfera | FQ, IN | |
| 16 | B. pyrrocinia | IX | Rizósferaa, suelo, sedimento | FQ |
| 17 | B. seminalis | Semilla de arroz, caña de azúcar | FQ, IN | |
| 18 | B. stabilis | IV | FQ, IN, EH | |
| 19 | B. stagnalis | Suelo | FQ, IN | |
| 20 | B. territorio | Agua | NR | |
| 21 | B. ubonensis | Suelo | IN | |
| 22 | B. vietnamiensis | V | Rizósferaa, plantas, suelo, agua, animales | FQ |
Las referencias de cada especie se encuentran en el sitio http://www.bacterio.net/-allnamesac.html, List of Prokaryotic names with Standing in Nomenclature (LPSN).
FQ: muestras de pacientes con fibrosis quística; IN: muestras de pacientes con infecciones nosocomiales; EH: muestras de equipo de hospitales; SM: muestras de soluciones de uso médico; NR: no reportada.
Árbol filogenético basado en la comparación de las secuencias del gen de la subunidad 16S del ARN ribosomal de especies propuestas del género Burkholderia, Caballeronia y Paraburkholderia. El alineamiento de las secuencias se determinó con el programa MUSCLE y el árbol se obtuvo mediante el método de máxima verosimilitud. La barra representa el número de sustituciones esperadas por sitio bajo el modelo GTR + G. Entre paréntesis se indican los números de acceso de las secuencias en la base de datos NCBI.
La distribución del complejo B. cepacia en el ambiente es amplia; se ha indicado que debido a sus grandes genomas y a la presencia de múltiples secuencias de inserción27, sus miembros pueden colonizar ambientes naturales como suelo, agua, rizósfera o semillas (tabla 1), y recientemente se descubrió que también pueden formar nódulos en la leguminosa Stylosanthes, la cual se encuentra en el Amazonas20. Anteriormente, se identificó B. cepacia genomovar VI (actualmente Burkholderia dolosa, tabla 1), aislado de nódulos de Alysicarpus glumaceus (Vahl) DC en Senegal, pero no se demostró que formara nódulos en dicha especie vegetal82. Aparentemente, la presencia del complejo B. cepacia en nódulos de leguminosas es considerada más como un contaminante que especies con la capacidad para nodular, es decir, serían bacterias asociadas al nódulo (nodule associated bacteria)63. En diferentes países de Europa, Asia y América se ha reportado la presencia de especies del complejo asociadas a plantas de interés agrícola, como maíz, arroz, agave y café, entre otras35,36,90. Además de haberse demostrado en suelos agrícolas, la presencia del complejo B. cepacia se ha observado en suelos urbanos, principalmente en jardines y campos de golf, en 3diferentes estados de Estados Unidos65. En Bélgica, se localizó el complejo B. cepacia en muestras de agua y suelo de viviendas y jardines de pacientes con fibrosis quística (FQ), lo cual indicaría una posible vía de propagación entre el ambiente y el humano85. Un dato interesante es que 2cepas de Burkholderia contaminans y una de Burkholderia lata aisladas de Phaseolus vulgaris L. y Pithecellobium sp., respectivamente, resultaron capaces de fijar nitrógeno en vida libre19, lo que representa una novedad, ya que por primera vez se reportaron especies del complejo B. cepacia diazótrofas diferentes de Burkholderia vietnamiensis. Por otro lado, el complejo B. cepacia se ha identificado en ambientes hospitalarios y en infecciones nosocomiales (tabla 1). Entre ellas, Burkholderia metallica y Burkholderia latens han sido aisladas exclusivamente de ambientes clínicos1,45, en tanto que Burkholderia territori y Burkholderia paludis han sido detectadas en muestras ambientales de agua25,67. El resto de las especies se han identificado en ambos tipos de muestras (tabla 1).
PatogenicidadEn los años 70, P. (Burkholderia) cepacia emergió como un patógeno oportunista de personas inmunocomprometidas29. Las infecciones que causa el complejo B. cepacia se asocian principalmente con personas que presentan FQ, una alternación genética en la cual las vías respiratorias bajas se obstruyen con un moco deshidratado, de consistencia viscosa. Estas condiciones constituyen un ambiente ideal en el cual se pueden establecer infecciones crónicas que impiden el normal funcionamiento del pulmón, ocasionando una neumonía, que puede a su vez derivar en una septicemia y, en algunas ocasiones, en la muerte del paciente. Este deterioro progresivo y rápido de los pacientes fue denominado el «síndrome cepacia», caracterizado por una neumonía necrosante, bacteriemia y sepsis48.
Algunas especies del complejo B. cepacia son significativamente importantes como patógenos de humanos debido a que tienen un alto grado de resistencia a los antibióticos, una excepcional transmisibilidad en ambientes nosocomiales y gran capacidad para sobrevivir intracelularmente49,71,73. La caracterización de los determinantes de patogenicidad en el complejo B. cepacia ha llevado a describir diferentes marcadores, por ejemplo, porinas y homoserinas lactonas, o factores como la amidasa, una proteína involucrada en el metabolismo de los aminoácidos, que promueve la supervivencia y persistencia de la bacteria in vivo8. También, se ha mostrado que el marcador epidémico de cepas de B. cepacia (BCESM) es parte de una isla genómica que contiene genes que codifican proteínas relacionadas con la virulencia y el metabolismo8. Otro factor de patogenicidad encontrado en Burkholderia cenocepacia es el sistema de secreción tipo IV (SST4)4. El SST4 está involucrado en la translocación de macromoléculas a través de la membrana celular y representa un mecanismo ampliamente distribuido para la transmisión de resistencia a antibióticos y de factores de virulencia en bacterias patógenas3. Es claro que la virulencia del complejo B. cepacia es multifactorial, pero se ha puesto de manifiesto que los factores de quorum sensing, la adquisición de hierro a través de sideróforos y la biosíntesis de lipopolisacáridos son requeridos para una patogenicidad completa79.
EpidemiologíaLa primera evidencia de una infección por B. cepacia en pacientes con FQ apareció en los años 70 y para 1984 se documentó la gran prevalencia de esta especie entre pacientes que recibieron tratamiento en un hospital en Canadá48, observándose también que había transmisión persona-persona e incluso el ambiente podía funcionar como un reservorio del complejo B. cepacia17. Estudios epidemiológicos efectuados en los años 90 demostraron que la especie de mayor prevalencia en el mundo en pacientes con FQ es B. cenocepacia60. Un análisis de la secuencia del gen recA permitió la división de esta especie en 4 grupos (IIIA a IIID), pero clínicamente los más importantes son los grupos IIIA y IIIB57. En el grupo IIIA existen linajes epidémicos que han causado infecciones devastadoras en Canadá, Inglaterra y Europa, entre ellas las cepas ET12, Midwest clone, PHDC, CZ1 y ST3227,30,57,59,77,83. Por otro lado, en Estados Unidos, el grupo más problemático ha sido el IIIB14,18, pero Burkholderia multivorans también ha emergido como un importante patógeno en FQ7. En España y Argentina, B. contaminans se ha encontrado con gran frecuencia47,50,64, y Burkholderia pyrrocinia mostró alta prevalencia en Italia11. En Brasil, se han detectado las especies de B. cenocepacia IIIA y IIIB, B. vietnamiensis, B. multivorans y B. cepacia86. En México se ha reportado B. cepacia en un caso de un paciente con enfermedad granulomatosa crónica40, así como B. cenocepacia IIIB y Burkholderia stabilis en sangre de pacientes en cuidado intensivo87.
Una herramienta de gran valor que ha sido utilizada para el estudio de la epidemiología del complejo B. cepacia es la tipificación de secuencias multilocus (MLST), la cual utiliza la comparación de secuencias nucleotídicas de 7 genes del tipo «housekeeping»61. La revaluación del complejo B. cepacia mediante MLST ha conducido a la agrupación de algunos linajes epidémicos y otros resultaron ser linajes independientes; también ha llevado a una identificación más sencilla de los aislados a nivel de especie28. Se ha indicado que una divergencia del 3% en las secuencias concatenadas puede servir como un valor para distinguir entre especies del complejo B. cepacia y ser equivalente a la definición de especie utilizando el 70% en hibridación DNA-DNA84. Haciendo uso de la base de datos PubMLST (http://pubmlst.org/bcc/), se observó que existen al menos 16 nuevas especies que esperan una descripción formal84. Aun cuando el uso de la metodología MLST ha sido de gran relevancia para el estudio del complejo B. cepacia, en la actualidad el incremento en la disponibilidad de genomas secuenciados ha hecho posible estudiar polimorfismos en Burkholderia, aunque hasta ahora solo en B. pseudomallei33 y B. dolosa55. Es evidente que el complejo B. cepacia es un grupo de importancia clínica que aún requiere mayor cantidad de estudios para conocer su diversidad; en tal sentido, la secuenciación de genomas permitirá una mayor comprensión de la epidemiología y la estructura poblacional.
Potencial agrobiotecnológicoEl complejo B. cepacia es bien conocido como un grupo de patógenos oportunistas que amenaza la vida de personas que presentan la enfermedad hereditaria FQ, pero también estas especies han sido estudiadas por sus aplicaciones biotecnológicas, como es la promoción del crecimiento vegetal (plant growth promoting bacteria [PGPB]), el biocontrol de fitopatógenos y la biorremediación. Durante mucho tiempo se consideró a B. vietnamiensis como la única especie fijadora de nitrógeno del complejo B. cepacia43, pero B. lata y B. contaminans también son fijadoras de nitrógeno19. La capacidad de B. vietnamiensis como PGPB se ha confirmado en experimentos de inoculación en arroz y caña de azúcar, en los cuales se detectó un incremento significativo en el peso seco de las plantas, aunque no se observó una diferencia en el contenido de nitrógeno en arroz, pero sí en la caña de azúcar, indicando que la fijación de nitrógeno es el mecanismo promotor de crecimiento78. Por otro lado, el papel para solubilizar fosfatos ha sido mostrado cuando se determinó el contenido de fosfato en la planta Lycopodium cernuum L. inoculada con B. cepacia42. La cepa MSSP de Burkholderia sp. (identificada como cepa del complejo B. cepacia, número de acceso AY551271), en un consorcio multiespecies o individualmente, promueve el crecimiento de Cajanus cajan (L.) Millsp.68. Otro ejemplo de PGPB se ha observado en la inoculación de B. multivorans WS-FJ9 en Populus euramericana Guinier54. En lo que respecta al control biológico, diferentes especies del complejo B. cepacia han sido analizadas en la producción de compuestos antimicrobianos, con potencial uso en la agricultura (material suplementario).
Otro aspecto importante en el complejo B. cepacia es su actividad para degradar compuestos complejos. En experimentos in vitro se ha observado que algunas especies pueden crecer en cianuro, polihidroxialcanoatos, cristal violeta, metil-paratión, tricloro-etileno, α y β endosulfanos o ácido 2,4-diclorofenoxiacético, entre otros2,5,15,32,39,46,76. También se ha determinado la capacidad de B. cepacia para degradar compuestos en presencia de plantas, como es el caso de la cepa L.S.2.4 inoculada en Lupinus luteus L. y en presencia de tolueno9. Debido a estas características importantes en la agrobiotecnología, en los años 90, varias cepas del complejo B. cepacia fueron registradas en los Estados Unidos para su uso como agentes de biocontrol de hongos fitopatógenos, tal es el caso de los productos Deny® (liquid and peat moss-based formulations of B. cepacia, CCT Corp., Carlsbad, CA, EE. UU.) y Blue Circle® (Stine Seed Farm, Stine Microbial Products), propuestos por la compañía Lubrizol6. También la cepa Ral-3 de Burkholderia ambifaria fue propuesta como promotora del crecimiento vegetal por la compañía AgBiologicas, una rama de AGRIUM6, pero debido a la relación de este grupo bacteriano con infecciones en pacientes con FQ, los productos comerciales fueron retirados del mercado y la Agencia de Protección Ambiental de los Estados Unidos estableció una moratoria para productos que contuvieran miembros del complejo B. cepacia (https://www.gpo.gov/fdsys/pkg/FR2004-09-29/pdf/04-21695.pdf). De acuerdo con la bioética, las nuevas biotecnologías deben ser examinadas cuidadosamente, debido a que pueden tener un impacto negativo en el mundo66. Aun así, se ha propuesto sí se debería tener una opción abierta sobre el uso biotecnológico del complejo B. cepacia, esto debido a las diferencias entre las mismas especies e incluso las cepas del complejo B. cepacia en cuanto a su transmisibilidad e impacto clínico sobre pacientes con FQ16. En este mismo sentido, B. multivorans WS-FJ9 fue analizada para determinar la seguridad de su uso como PGPB54. Para ello, se llevaron a cabo pruebas de patogenicidad en cebolla, tabaco y alfalfa, y se detectaron lesiones menores que no progresaron con el paso del tiempo, por lo cual los autores mencionaron el uso potencial de esta bacteria como agente de biocontrol. Algo similar sucedió con B. pyrrocinia JK-SH007 para su uso como agente de biocontrol: se determinó la inocuidad de la cepa en experimentos de inoculación en cebolla y alfalfa72. No obstante, debe tomarse en cuenta una posible transferencia de material genético entre las especies del complejo B. cepacia, dado que el suelo es considerado un reservorio de especies de este grupo bacteriano, que puede producir infecciones en humanos, como se mencionó anteriormente.
Producción de compuestos extracelularesComo parte de la versatilidad metabólica que le permite al complejo B. cepacia adaptarse a diversos ambientes, se ha reportado la producción de compuestos antimicrobianos que inhiben el crecimiento de diversos patógenos de plantas y humanos. Se han descrito diferentes péptidos con actividad antifúngica, como las cepacidinas, glidobactinas y altericidinas producidas por B. cepacia51,52,75, un oligopéptido no ribosomal, y la occidiofungina, producida por B. contaminans13,44. Adicionalmente, se conoce la producción por B. cepacia de compuestos del tipo de los sideróforos y pigmentos de la familia de las fenazinas con actividad contra hongos fitopatógenos12,23. El compuesto CF66I, del que solo se sabe que contiene enlaces amida, un ácido graso a-metil, bromina y unas unidades estructurales como el CH2CH2O, es producido por B. cepacia y presenta propiedades inhibitorias contra hongos filamentosos y levaduras53,70. Los compuestos con actividad antibacteriana reportados en el complejo B. cepacia corresponden a moléculas de diferente naturaleza. Las cepacinas A y B son antibióticos acetilénicos producidos por B. cepacia que inhiben el crecimiento de bacterias Gram negativas y positivas69. Los policétidos llamados enaciloxinas producidos por B. ambifaria presentan actividad contra especies del complejo B. cepacia, Acinetobacter baumannii y Pseudomonas aeruginosa58. Asimismo, se ha reportado actividad inhibitoria de Xanthomonas campestris mediada por sideróforos producidos por B. cepacia22. Un aislado de Burkholderia ubonensis produce un compuesto tipo bacteriocina denominado bacteriocin-like inhibitory substance (BLIS). El compuesto BLIS tiene actividad contra el patógeno causante de meliodosis, B. pseudomallei62. Finalmente, una bacteriocina tipo lectina (LlpA) codificada en el genoma de B. cenocepacia AU1054 presenta actividad contra B. ambifaria, Burkholderia anthina, B. cenocepacia, B. contaminans y B. metallica41. Diferentes cepas de B. cenocepacia tienen en su genoma homólogos para la producción de la bacteriocina LlpA. El mecanismo de acción de esta bacteriocina, aún desconocido, representa un campo por explorar en la búsqueda de nuevos antibióticos producidos por el complejo B. cepacia. La bioinformática también ha sido aplicada en el complejo B. cepacia para la búsqueda de compuestos antimicrobianos. Recientemente se describió la especie B. paludis aislada de la superficie de un pantano en una reserva forestal del sudeste de Malasia67. El genoma de la cepa B. paludis MSh1T tiene 43 genes relacionados con la producción de metabolitos secundarios que podrían estar implicados en la producción de compuestos con actividad antimicrobiana, como policétidos, péptidos no ribosomales y bacteriocinas. Estos podrían ser compuestos nuevos o no estudiados hasta el presente. Otro análisis bioinformático sobre la búsqueda de péptidos no ribosomales potencialmente involucrados en el biocontrol o útiles como compuestos farmacéuticos en especies del complejo B. cepacia reveló la posible síntesis de lipopéptidos como xylocandina A1, A2, B1 y B2; cepaciadina A1 y A2; burkholdinas; occidiofunina; burkholdina/occidiofungina y sideróforos, entre ellos ornibactina, pioquelina, cepaciaquelina34.
Desafortunadamente, una limitante para la producción a nivel industrial de estos compuestos antimicrobianos es la relación del complejo B. cepacia con las infecciones en pacientes con FQ. Sin embargo, con el uso de la ingeniería genética, los genes que codifican las rutas biosintéticas de los compuestos antimicrobianos podrían transferirse a organismos que los expresen y que puedan utilizarse en procesos biotecnológicos, sin riesgo alguno.
Conclusiones y perspectivasEl complejo B. cepacia comprende un grupo de bacterias de gran valor en el campo de la agrobiotecnología. Sin embargo, debido a su carácter de patógeno oportunista, su uso está limitado a ensayos en el laboratorio. Tomando esto en consideración, en 2016 Eberl y Vandamme31 propusieron como alternativa la posibilidad de reemplazar al complejo B. cepacia por especies del recientemente propuesto género Paraburkholderia, en particular por Paraburkholderia phenazinium, Paraburkholderia megapolitana y Paraburkholderia bryophila, debido a que estas muestran actividad antifúngica. No obstante, e independientemente de la posibilidad de utilizar a este grupo de bacterias con fines biotecnológicos, es importante llevar a cabo una caracterización exhaustiva del microorganismo, en la que se incluyan pruebas con líneas celulares o en ratones, para determinar el potencial patogénico de las cepas31.
FinanciaciónEl trabajo de PES es financiado por los proyectos SIP 2015-0358 y SIP 2016-0077 del Instituto Politécnico Nacional.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
PES y JAIG agradecen los estímulos de EDI, COFAA y SNI. FURR cuenta con una beca CONACyT.