Se estudiaron 28 aislamientos obtenidos de muestras clínicas de perros e identificados por espectrometría de masas (MALDI-TOF) como Staphylococcus pseudintermedius; el objetivo fue evaluar la sensibilidad a los antimicrobianos por el método de difusión y establecer la relación clonal entre aislamientos por electroforesis en campo pulsado (PFGE). La resistencia a meticilina se evaluó mediante PCR por amplificación del gen mecA y se observó en 3/28 aislamientos (10,7%). Quince aislamientos (53,6%) presentaron resistencia a alguno de los antibióticos ensayados y 11 de ellos (39,3%) presentaron resistencia múltiple (resistencia a 3 o más familias de antibióticos). Once aislamientos (39,3%) presentaron resistencia a eritromicina, debido a la presencia de metilasa ribosomal ermB, y no se detectó resistencia inducible a clindamicina. Por PFGE se pudieron diferenciar 27 tipos clonales, lo cual demuestra gran diversidad clonal. Se destaca el hallazgo de aislamientos de S. pseudintermedius multirresistentes como una eventual problemática a considerar en el diagnóstico veterinario de laboratorio, el tratamiento de las infecciones caninas y el ámbito de la salud pública.
Twenty-eight strains isolated from dog clinical samples identified as Staphylococcus pseudintermedius by mass spectrometry (MALDI-TOF) were studied to assess antimicrobial susceptibility by the diffusion method and clonal relationship by pulsed field gel electrophoresis (PFGE). Methicillin resistance (3/28 isolates; 10,7%) was evaluated by mecA PCR. Fifteen strains (53.6%) were resistant to at least one of the antibiotics tested, and eleven of them (39.3%) showed multiple resistance (3 or more antimicrobial families). Eleven isolates (39.3%) were resistant to erythromycin due to the presence of ribosomal methylase ermB, whereas clindamycin inducible resistance was not detected. Twenty-seven (27) clonal types were differentiated by PFGE, suggesting high clonal diversity. We emphasize that the finding of multiresistant S. psedintermedius strains is an emerging problem to be considered in veterinary diagnostic laboratory treatment of canine infections and in public health settings.
El género Staphylococcus incluye una variedad de especies que participan como patógenos oportunistas en diferentes enfermedades estudiadas por la medicina veterinaria.
Entre los estafilococos coagulasa positivos, Staphylococcus pseudintermedius fue reclasificado y es una especie de difícil diferenciación de Staphylococcus intermedius y Staphylococcus delphini, dentro del llamado «grupo Staphylococcus intermedius»24. La revisión de esta clasificación permitió asumir que la mayoría de los estafilococos aislados de caninos e identificados como S. intermedius son S. pseudintermedius14. Para poder diferenciarlos, se han utilizado distintos métodos fenotípicos convencionales23, espectrometría de masas10 y métodos genotípicos1.
S. pseudintermedius es un microorganismo comensal de piel y mucosas en los caninos, y entre las infecciones más frecuentes se describen las que produce en piel, oído, vías urinarias y hueso2.
Así como se ha observado un aumento de la resistencia a la meticilina en Staphylococcus aureus aislados de animales de compañía, lo mismo se ha observado en S. pseudintermedius29. Esta resistencia se notifica progresivamente en diferentes países, con la consiguiente dificultad en el tratamiento de piodermas y otitis recidivantes16,33.
La emergencia de resistencia a la meticilina en S. psedintermedius hizo que se revieran y elaboraran nuevas normas para evaluar la sensibilidad de este coco coagulasa positivo en animales. Hasta el año 2004, el Clinical and Laboratory Standards Institute (CLSI) recomendaba interpretar las pruebas de sensibilidad para los estafilococos coagulasa positivos de medicina veterinaria como «Staphylococcus especie no S. aureus»5, pero en 2008 recomienda interpretarlos según el criterio empleado para S. aureus6. Recientemente, las normas CLSI 2013 establecieron puntos de corte específicos para S. pseudintermedius8.
El objetivo del presente trabajo fue realizar un estudio retrospectivo de aislamientos de S. pseudintermedius recuperados de muestras de caninos, evaluando la resistencia antimicrobiana y la relación genética entre aislamientos.
Materiales y métodosOrigen de los aislamientosSe analizaron 28 aislamientos recuperados de infecciones de caninos registradas en el período 2004-2013; 12 se obtuvieron de hisopados óticos, 8 de hisopados vaginales, 4 de punciones de piel, 1 de orina, 1 de líquido pericárdico, 1 de secreción conjuntival y 1 de lavado nasal.
Identificación por pruebas bioquímicas y espectrometría de masasEn el Laboratorio de Diagnóstico e Investigaciones Bacteriológicas de la Facultad de Ciencias Veterinarias de la Universidad Nacional de La Plata se procedió a la identificación de los aislamientos. Se realizaron las siguientes pruebas bioquímicas: coagulasa; hidrólisis de la arginina; l-pirrolidonilarilamidasa (PYR); Voges Proskauer (VP) y fermentación de trehalosa, manitol, xilosa, celobiosa, arabinosa, maltosa, lactosa, sacarosa y rafinosa24. La identidad de los aislamientos se confirmó por espectrometría de masas con el equipo Bruker Daltonics Microflex LT, usando el software MALDI Biotyper 3.1 (Bruker Daltonics, Bremen, Alemania) y ácido α-ciano-4-hidroxicinámico como matriz. Los ensayos fueron realizados por duplicado.
Pruebas de sensibilidad antimicrobianaLa sensibilidad a los antimicrobianos se evaluó por el método de difusión en agar según normas M2-A11 del CLSI7. Se utilizaron discos de oxacilina 1μg, cefoxitina 30μg, eritromicina 15μg, clindamicina 2μg, tetraciclina 30μg, teicoplanina 30μg, ciprofloxacina 5μg, gentamicina 10μg, nitrofurantoína 30μg, rifampicina 5μg, cloranfenicol 30μg y trimetoprima-sulfametoxazol 25μg (Laboratorio Britania, Buenos Aires, Argentina). Los resultados del método de difusión con oxacilina, cefoxitina, eritromicina, clindamicina, tetraciclina, gentamicina, cloranfenicol y trimetoprima/sulfametoxazol se interpretaron según las normas CLSI para aislamientos de animales Vet01-S28, mientras que con el resto de los antimicrobianos los resultados se interpretaron según las normas CLSI M100-S249 para aislamientos de humanos, por no disponer de puntos de corte para aislamientos de animales. Se evaluó el fenotipo de resistencia a macrólidos utilizando la prueba del D-test, según lo describen Steward et al. para S. aureus26.
Métodos molecularesLa presencia de los genes mecA, ermB, msrA, lnuA, lnuB y lnuC se determinó por PCR según condiciones previamente descritas11,27,28,31,35. La caracterización del SCCmec se realizó por PCR multiplex18.
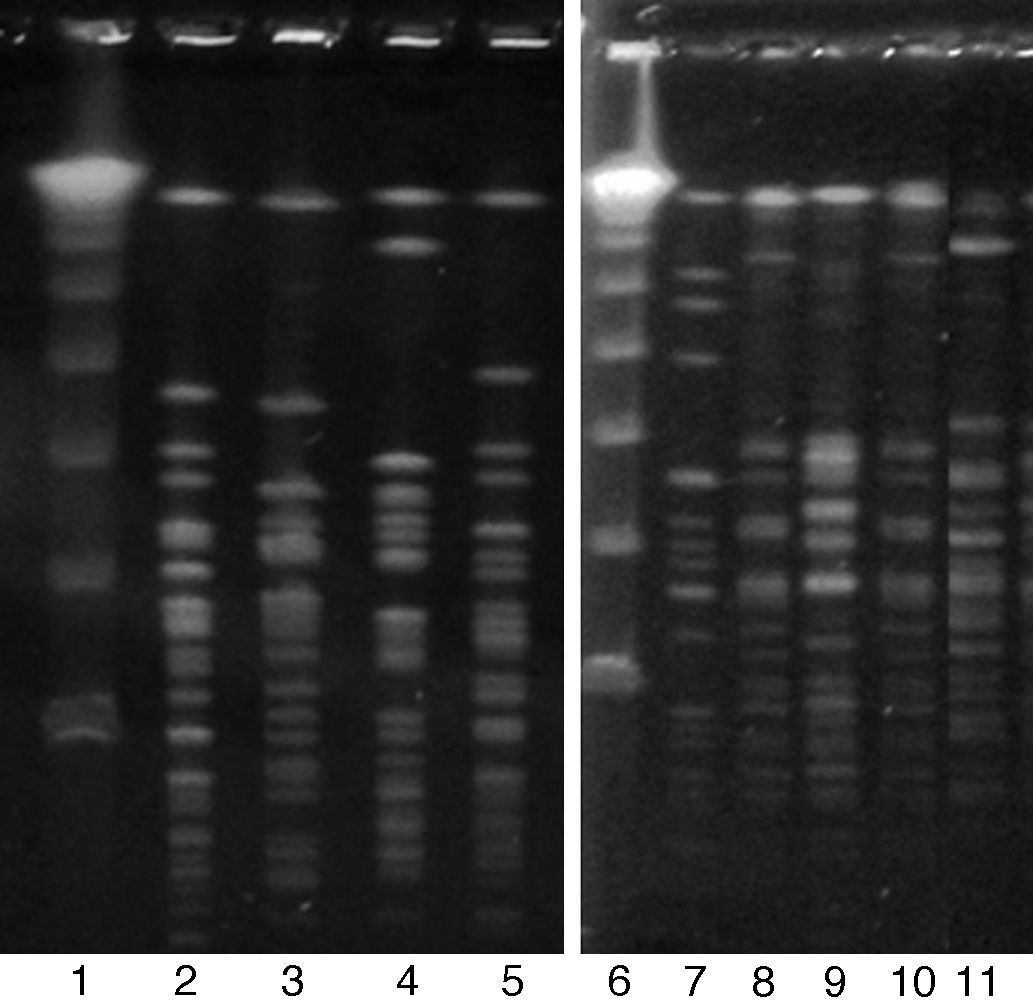
La relación clonal entre aislamientos se estableció por electroforesis en campo pulsado (PFGE). Se prepararon discos de agarosa con el ADN cromosómico y se realizó la restricción con la enzima SmaI4. Los fragmentos de ADN se separaron en geles de agarosa al 1% usando un aparato CHEF- DR III (Biorad Laboratorios, Richmond, CA, EE. UU.) con las siguientes condiciones de corrida: 6V/cm y pulsos de 2 a 20 s durante 20h, a 11,3°C. Como marcador de peso molecular se utilizó Lambda Ladder (New England Biolabs, Ipswich, MA, EE. UU.). La relación entre los aislamientos se estableció usando el criterio de Tenover28.
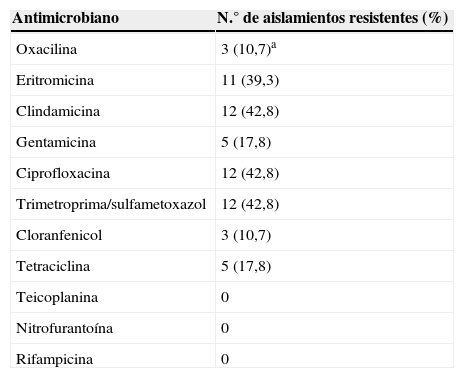
ResultadosLas pruebas bioquímicas permitieron identificar a los aislamientos como S. pseudintermedius; este resultado se confirmó por espectrometría de masas. De los 28 aislamientos de S. pseudintermedius estudiados, 13 (46,4%) fueron sensibles y 15 (53,6%) presentaron resistencia a al menos uno de los antibióticos ensayados. Todos los aislamientos fueron sensibles a nitrofuranos, teicoplanina y rifampicina. En la tabla 1 se puede observar el porcentaje de resistencia a los antimicrobianos ensayados y en la tabla 2 los perfiles fenotípicos. Tres de los aislamientos fueron definidos como resistentes a meticilina debido a la presencia del gen mecA. Mediante el método de difusión, estos aislamientos presentaron resistencia a oxacilina, pero sensibilidad a cefoxitina. Todos los aislamientos mecA negativos fueron sensibles a oxacilina y cefoxitina.
Resistencia a los antimicrobianos de 28 aislamientos de S. pseudintermedius de caninos
| Antimicrobiano | N.° de aislamientos resistentes (%) |
|---|---|
| Oxacilina | 3 (10,7)a |
| Eritromicina | 11 (39,3) |
| Clindamicina | 12 (42,8) |
| Gentamicina | 5 (17,8) |
| Ciprofloxacina | 12 (42,8) |
| Trimetroprima/sulfametoxazol | 12 (42,8) |
| Cloranfenicol | 3 (10,7) |
| Tetraciclina | 5 (17,8) |
| Teicoplanina | 0 |
| Nitrofurantoína | 0 |
| Rifampicina | 0 |
a Estos 3 aislamientos fueron sensibles a cefoxitina.
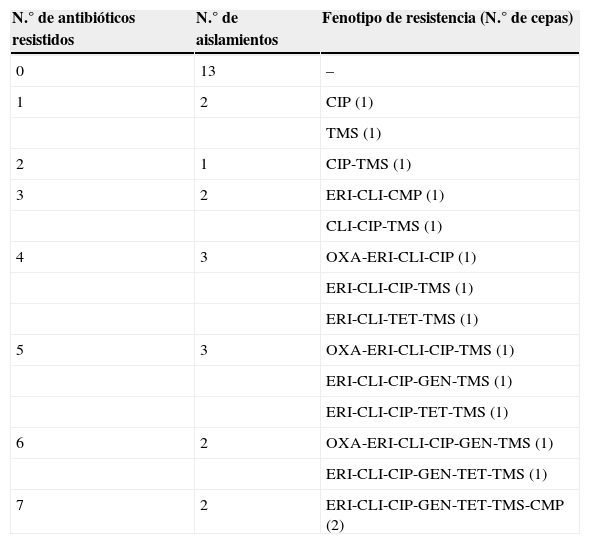
Perfiles de resistencia antimicrobiana de S. pseudintermedius en muestras clínicas de origen canino
| N.° de antibióticos resistidos | N.° de aislamientos | Fenotipo de resistencia (N.° de cepas) |
|---|---|---|
| 0 | 13 | – |
| 1 | 2 | CIP (1) |
| TMS (1) | ||
| 2 | 1 | CIP-TMS (1) |
| 3 | 2 | ERI-CLI-CMP (1) |
| CLI-CIP-TMS (1) | ||
| 4 | 3 | OXA-ERI-CLI-CIP (1) |
| ERI-CLI-CIP-TMS (1) | ||
| ERI-CLI-TET-TMS (1) | ||
| 5 | 3 | OXA-ERI-CLI-CIP-TMS (1) |
| ERI-CLI-CIP-GEN-TMS (1) | ||
| ERI-CLI-CIP-TET-TMS (1) | ||
| 6 | 2 | OXA-ERI-CLI-CIP-GEN-TMS (1) |
| ERI-CLI-CIP-GEN-TET-TMS (1) | ||
| 7 | 2 | ERI-CLI-CIP-GEN-TET-TMS-CMP (2) |
OXA: oxacilina, TMS: trimetoprima/sulfametoxazol; CIP: ciprofloxacina; ERI: eritromicina; CLI: clindamicina; CMP: cloranfenicol; GEN: gentamicina; TET: tetraciclina.
Se caracterizó el cassette cromosómico del estafilococo SCCmec en los 3 aislamientos resistentes a meticilina, este resultó ser una variante del SCCmecIII (datos no mostrados).
Doce aislamientos (42,8%) presentaron resistencia a macrólidos y lincosamidas, y 11 aislamientos (39,3%) con fenotipo MLSb constitutivo (macrólidos, lincosamidas y estreptograminas B) presentaron resistencia a eritromicina y clindamicina, y dieron positiva la PCR para el gen ermB y negativa para los genes msrA, lnuA, lnuB y lnuC. Un aislamiento presentó sensibilidad a eritromicina y resistencia a clindamicina y lincomicina, fenotipo compatible con la presencia de lincosamidanucleotidiltransferasa, con PCR negativa para lnuA, lnuB, lnuC y demás genes ensayados.
Los 28 aislamientos de S. pseudintermedius se pudieron diferenciar en 27 tipos clonales por SmaI PFGE (fig. 1). Solo 2 aislamientos se encontraron genéticamente relacionados (fig. 1, calles 8 y 10).
DiscusiónS. pseudintermedius forma parte de la microbiota de animales sanos y habita la piel, el folículo piloso y las mucosas de nariz y ano19.
En 2006, Van Hoovels et al. reportaron el primer caso de infección por S. pseudintermedius en humanos, por lo que propusieron incluir esta especie en la base de datos de los sistemas comerciales de identificación30. Estudios recientes de Decristophoris et al.10 aportan evidencias del alto poder discriminatorio que tiene la espectrometría de masas para diferenciar Staphylococcus del grupo SIG (S. intermedius, S. pseudintermedius y S. dephini) al comparar esta técnica con la secuenciación del gen hps60 de las 3 especies, por lo que esta nueva metodología podría ser una herramienta útil para la diferenciación entre especies semejantes.
En un trabajo realizado en caninos en Noruega, Osland et al.21 observaron gran diversidad clonal entre los aislamientos de S. pseudintermedius sensibles a la meticilina, pero reportaron alta clonalidad entre los aislamientos de S. pseudintermedius resistentes a meticilina. En nuestro estudio los aislamientos mostraron gran diversidad genética, ya que los 28 aislamientos se diferenciaron en 27 tipos clonales, incluso los aislamientos resistentes a meticilina.
La resistencia a meticilina en S. pseudintermedius emergió en 2006 y aumentó en los últimos años; hoy representa un problema importante en salud animal3,29. En nuestro estudio solo 3 aislamientos (10,7%) fueron resistentes a meticilina, lo cual difiere de lo hallado por Feng et al. en el sur de China, que encontraron 48% de aislamientos resistentes a este antimicrobiano en S. pseudintermedius de perros13. El hallazgo de aislamientos resistentes a meticilina portadores del gen mecA alerta sobre la circulación de este mecanismo en mascotas de nuestro medio. Si bien se han detectado S. pseudintermedius resistentes a meticilina en caninos de otros países2, este sería el primer reporte en nuestro país.
El hallazgo del cassette cromosómico SCCmec variante del tipo iii coincide con lo encontrado por Ishihara et al.16 en un trabajo realizado en hospitales veterinarios, donde el SCCmec tipo II-III representó el 85,2% de los aislamientos de S. pseudintermedius. McCarthy et al.17 encontraron que la diseminación de S. pseudintermedius resistente a meticilina en Estados Unidos se debió al clon ST68 SCCmecV, mientras que en Europa se asoció al clon ST71 SCCmecII-III. Ambos clones continúan diseminándose globalmente.
Por otro lado, recientemente Paul et al. documentaron la presencia de S. pseudintermedius en personal veterinario en contacto con pequeños animales, a pesar de que esta especie no forma parte de la flora normal del hombre22. Por lo tanto, se debería considerar a S. pseudintermedius resistente a meticilina como un agente zoonótico emergente.
Al igual que en S. aureus, la resistencia a meticilina en S. pseudintermedius está mediada por el gen mecA, por lo que la prueba de oro para detectar la resistencia a la meticilina en los estafilococos es la PCR21. Sin embargo, en el diagnóstico de laboratorio veterinario de rutina se utilizan pruebas fenotípicas, como el método de difusión de Kirby Bauer con discos de oxacilina y cefoxitina. La interpretación de las pruebas de difusión con disco para detectar resistencia a meticilina en S. pseudintermedius presenta controversias. El CLSI no tiene punto de corte establecido para estafilococos coagulasa positivos distintos de S. aureus en aislamientos de humanos. En 2009, Schissler et al. evaluaron la utilidad de los puntos de corte del CLSI para resistencia a meticilina en aislamientos caninos de S. pseudintermedius y demostraron que el disco de cefoxitina no es apropiado para detectar resistencia a meticilina en aislamientos de S. pseudintermedius25. Otros autores coincidieron en que el disco de cefoxitina no es apto para detectar la emergencia de resistencia a meticilina en aislamientos de S. pseudintermedius de perros3,32,33. Recientemente, en las normas CLSI 2013 para uso veterinario Vet01-S28 se incluyó el punto de corte para oxacilina en S. pseudintermedius, y además se destaca que la cefoxitina no tiene valor predictivo del gen mecA. En el presente estudio, en ninguno de los aislamientos en los que se detectó el gen mecA se observó resistencia a cefoxitina; sin embargo, el disco de oxacilina permitió detectarlos eficientemente, tanto interpretando con los puntos de corte de 2004 y 2008 para humanos como con los de 2013 para animales. La inferencia de resistencia a meticilina con cefoxitina traerá aparejados errores muy mayores o de falsa sensibilidad, lo que conduciría erróneamente al tratamiento con antibióticos β-lactámicos.
El alto porcentaje de resistencia a macrólidos detectado (11/28 aislamientos; 39,3%) se debió al mecanismo de metilación ribosomal y correlacionó en todos los casos con la presencia del gen ermB. No se detectó resistencia inducible a clindamicina ni eflujo, sin embargo, debido al alto porcentaje de resistencia a clindamicina (42,8%), debería evitarse el uso de aquella en el tratamiento empírico de piodermas en perros. Faires et al.12 describen un 76% de resistencia a macrólidos en un estudio realizado en Canadá, con predominio del fenotipo MLSb constitutivo, lo cual coincide con nuestros resultados. En una revisión reciente34 se describe que la metilación ribosomal debida a la presencia del gen ermB es el mecanismo de resistencia a macrólidos y lincosamidas prevalente en S. pseudintermedius.
Un alto porcentaje de aislamientos de S. pseudintermedius presentó resistencia a por lo menos uno de los agentes ensayados (15/28; 53,6%) y 11 de estos (39,3%) presentaron multirresistencia (resistencia a 3 o más familias de antimicrobianos). Es alarmante el alto porcentaje de resistencia a ciprofloxacina (42,8%) y trimetoprima/sulfametoxazol (42,8%), detectado en este estudio, ya que estos son antibióticos ampliamente usados en medicina humana. Varios autores describen la multirresistencia en aislamientos de S. pseudintermedius resistentes a meticilina. Bemis et al. encontraron que más del 90% de los aislamientos de S. pseudintermedius resistentes a meticilina que estudiaron presentaban resistencia a por lo menos 4 antibióticos no β-lactámicos3. Huerta et al. compararon la resistencia a meticilina y multirresistencia de S. pseudintermedius entre perros urbanos y callejeros, y encontraron mayor proporción de resistencia en la población urbana15.
Un estudio reciente evaluó los cambios en el genoma asociados a la rápida aparición y evolución de multirresistencia en S. pseudintermedius. Dicho estudio demostró que el uso de unos pocos antibióticos podría ser suficiente para seleccionar clones pandémicos con elementos como el transposón Tn5405-like, que confieren multirresistencia17.
La identificación correcta de la especie de estafilococo coagulasa positivo tiene importancia en la clínica veterinaria, tanto por la detección de resistencia a meticilina como para la prescripción del tratamiento, ya que las cefalosporinas son los antimicrobianos de elección para tratar patologías caninas causadas por S. pseudintermedius.
La emergencia de resistencia a antimicrobianos en S. pseudintermedius provenientes de animales y su hallazgo en casos humanos resalta la necesidad del uso prudente de tales fármacos y su vigilancia, según lo aconseja la Organización Mundial de Sanidad Animal en el código sanitario para los animales terrestres, en relación con la prescripción y normas de buena usanza en medicina veterinaria20.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.