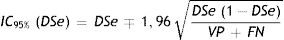Cercospora kikuchii, fitopatógeno usual en plantas de soja, ocasiona deterioro en las cosechas. Su identificación precoz y correcta evitaría el uso indebido de plaguicidas y permitiría iniciar un tratamiento adecuado. Una técnica rápida, económica y de fácil ejecución es el Dot blot, capaz de reconocer la presencia de una proteína específica del género conocida como CFP (Cercosporin Facilitator Protein). El objetivo de este trabajo fue validar dicha técnica para garantizar la fiabilidad del resultado. Para ello, se procesaron 29 plantas de soja infectadas y 31 plantas sanas teniendo en cuenta una confianza deseada del 95% y un error permitido del 5%. La técnica presentó una sensibilidad diagnóstica del 93,3% y una especificidad diagnóstica del 96,7%. La eficacia fue del 95% y los valores predictivos positivo y negativo del 96,6 y el 93,5%, respectivamente. Estos resultados la postulan como una herramienta útil para detectar precozmente C. kikuchii en plantas de soja.
Cercospora kikuchii is a common pathogen in soybean plants that causes crop spoilage. Its early and precise identification would prevent the misuse of pesticides and allow the initiation of an appropriate treatment. A quick, economical and easy-to-execute technique is the Dot blot, capable of recognizing the presence of a genus-specific protein called CFP (Cercosporin Facilitator Protein). The objective was to validate this technique to guarantee the reliability of the results. For that purpose, 29 infected soybean plants and 31 healthy plants were processed, taking into account a 95% desired confidence level and a permissible error of 5%. The technique provided a diagnostic sensitivity of 93.3% and a diagnostic specificity of 96.7%. The efficiency was 95% and positive and negative predictive values were 96.6% and 93.5%, respectively. These results postulate it as a useful resource for the early detection of C. kikuchii in soybean plants.
En Argentina, la soja es el principal producto de la agricultura nacional2. Sin embargo, su cultivo se encuentra limitado por las enfermedades de fin de ciclo (EFC) debido a que estas provocan madurez anticipada de la planta y reducción del rendimiento o de la calidad de las semillas, lo que genera graves deterioros en las cosechas y pérdidas en la producción12. Entre las EFC que presentan mayor prevalencia e incidencia en las principales áreas sojeras del país se encuentra el tizón de la hoja y mancha púrpura de la semilla, enfermedad ocasionada por el hongo Cercospora kikuchii3.
La patogenicidad de este hongo se asocia a la producción de la exotoxina cercosporina, responsable de muchos de los síntomas de la enfermedad. La cercosporina ha sido aislada in vitro de al menos 34 especies de Cercospora, entre las que se encuentra Cercospora sojina. La producción y la exportación de cercosporina hacia el exterior del hongo están reguladas por la expresión del gen cfp, que codifica la proteína denominada Cercosporin Facilitator Protein (CFP)1.
Debido a que este patógeno se encuentra presente en estadios tempranos del cultivo en forma latente y asintomática, resulta indispensable disponer de métodos de diagnóstico precoces y certeros, que contribuyan a generar medidas de control efectivas, para optimizar los recursos y reducir los efectos negativos sobre el medio ambiente por el uso indebido de plaguicidas. Asimismo, dichos métodos permitirían originar información respecto de la interacción patógeno-hospedante8.
Hasta el momento, no existen en el mercado métodos de detección temprana de este patógeno. En la actualidad, su diagnóstico se realiza cuando la planta ya presenta los síntomas característicos en las etapas avanzadas, utilizando métodos tradicionales (aislamiento, cultivo del hongo, observación de características macro y microscópicas) que requieren de personal especializado y prolongados períodos de tiempo (semanas).
Por ello, el desarrollo de un método de detección precoz de Cercospora en plantas de soja es de gran necesidad. Entre las principales técnicas disponibles que permiten complementar o reemplazar los procedimientos de laboratorio tradicionales se encuentran las inmunoquímicas y las moleculares8.
Latorre Rapela7 desarrolló y optimizó un método de detección precoz para el género Cercospora, capaz de reconocer la presencia de la proteína CFP, específica del género. Dicho método es muy versátil y aplicable al diagnóstico de un gran número de muestras y se basa en la técnica de Dot-enzyme linked immunosorbent assay (Dot-ELISA). Esta metodología consiste en un ELISA rápido, que utiliza como soporte sólido papel de nitrocelulosa, caracterizado por su excelente capacidad para adsorber proteínas13.
Para su correcta aplicación, debe efectuarse su validación diagnóstica, evaluando dicha metodología en un número considerable de plantas de soja enfermas y sanas, según el intervalo de confianza (IC) deseado y el error estimado. Entre los principales indicadores estadísticos se deben determinar la sensibilidad y la especificidad, la eficacia y los valores predictivos positivo (VPP) y negativo (VPN)4.
El objetivo del presente trabajo fue validar la técnica de Dot blot para la detección precoz de C. kikuchii en plantas de soja, desarrollada previamente en el laboratorio de Microbiología General de la Facultad de Bioquímica y Ciencias Biológicas de la Universidad Nacional del Litoral.
Para tal fin, se trabajó con 29 muestras positivas (hojas de plantas infectadas experimentalmente con C. kikuchii NBRC 6711, cepa perteneciente a la colección japonesa NITE Biological Resource Center [NBRC]) y con 31 muestras negativas (hojas de plantas sanas), acorde con lo establecido para una sensibilidad diagnóstica (DSe) y especificidad diagnóstica (DSp) estimada del 98%, con una confianza deseada del 95% y un error permitido del 5%6.
Las muestras positivas se obtuvieron a partir de plantas de soja cultivadas e inoculadas bajo condiciones de crecimiento controladas. El cultivo de las plantas se llevó a cabo utilizando semillas RAS16, grupo de madurez 5, que a simple vista no presentaban daño mecánico por almacenamiento o enfermedad. Para el pregerminado de las semillas, estas se desinfectaron con hipoclorito de sodio al 0,5% y luego se colocaron en bandejas plásticas que contenían papel de filtro embebido en agua sobre una capa de algodón, a fin de mantener las condiciones de humedad apropiadas. Estas bandejas se mantuvieron bajo condiciones de oscuridad continua a una temperatura de 25±0,2°C durante 96h, en una incubadora modelo I-501PF (INGELAB, Argentina). Luego, las semillas pregerminadas se dispusieron en macetas plásticas de 300ml, estas macetas contenían tierra-arena-vermiculita en proporción 2:1:1, a 1,5cm de la superficie. Las plantas continuaron su desarrollo en la incubadora bajo condiciones alternadas de luz (16h luz/8h oscuridad), a la temperatura ya citada, y durante su crecimiento fueron fertilizadas con solución de Hoagland o solución nutritiva de Jensen11. Una vez que las plantas alcanzaron el estado fenológico V3, se inocularon con una suspensión de C. kikuchii NBRC 6711.
Para la preparación del inóculo, el hongo se sembró en 5 tubos que contenían agar papa dextrosa; estos se incubaron durante 4 días a 25±0,2°C, bajo ciclos alternados de luz (16h luz/ 8h oscuridad). Transcurrido dicho tiempo, se agregaron 5ml de agua destilada estéril a cada tubo y se procedió a desprender las estructuras fúngicas mediante un raspado suave de la superficie del cultivo con ansa. La suspensión se cuantificó mediante un recuento en placa en superficie y la concentración del inóculo se ajustó en 104UFC/ml.
Para la inoculación se seleccionaron las hojas trifoliadas correspondientes al primer y segundo par de hojas completamente desplegadas. Se asperjaron con un volumen de 2,5ml del inóculo por planta, humedeciendo ambas caras de las hojas. Luego, se cubrieron con bolsas plásticas transparentes y perforadas, previamente humedecidas en su interior, y así se mantuvieron durante 24h a fin de proveer las condiciones de humedad y mojado foliar adecuadas para estimular el proceso de infección7.
Una vez inoculadas, las plantas se colocaron nuevamente en la incubadora manteniendo las condiciones de temperatura y ciclos de luz/oscuridad empleados para su crecimiento.
Transcurridos 4 días, se recolectaron las hojas trifoliadas y se lavaron 3 veces con agua estéril para eliminar restos de inóculo adheridos a la superficie foliar. Se observaron a simple vista en busca de lesiones macroscópicas y con lupa estereoscópica (BOECO Germany) en busca de lesiones no visibles a simple vista. Las zonas de tejido lesionado se cortaron con un bisturí y, posteriormente, se realizó la extracción de las proteínas totales siguiendo el método propuesto por Rollins et al.14.
El tiempo de muestreo de 4 días se seleccionó teniendo en cuenta los resultados logrados por Latorre Rapela7, y la técnica de Dot blot se llevó a cabo según el diseño previamente descrito por este mismo autor. A modo de resumen, sobre una membrana de nitrocelulosa se depositaron los extractos proteicos solubles puros y cantidades decrecientes de aquellos (1/2, 1/8, 1/32 y 1/64). Posteriormente, la membrana se secó a temperatura ambiente y se bloqueó con solución salina de fosfatos (PBS)-leche 5%. Después de 3 ciclos de lavado con PBS-Tween 20 (PBS-T) durante 5min, se incubó con suero de conejo anti-CFP diluido 1/500 en PBS-leche al 1%. Luego de una nueva etapa de lavado (3 veces), se incubó con suero anti-IgG total de conejo conjugado con peroxidasa, diluido 1/500 con PBS-leche al 1%. Tras otra etapa de lavado (3 veces), se agregó como reactivo revelador una solución de tetraclorhidrato de 3,3′-diaminobenzidina y peróxido de hidrógeno (Invitrogen Argentina S.A.). La reacción se detuvo con una etapa de lavado con agua. Todos los pasos de incubación se realizaron en agitación moderada. Como control positivo se utilizó el extracto proteico de C. kikuchii NBRC 6711 y como controles de especificidad de género, los extractos proteicos de Penicillium sp. y Alternaria sp.
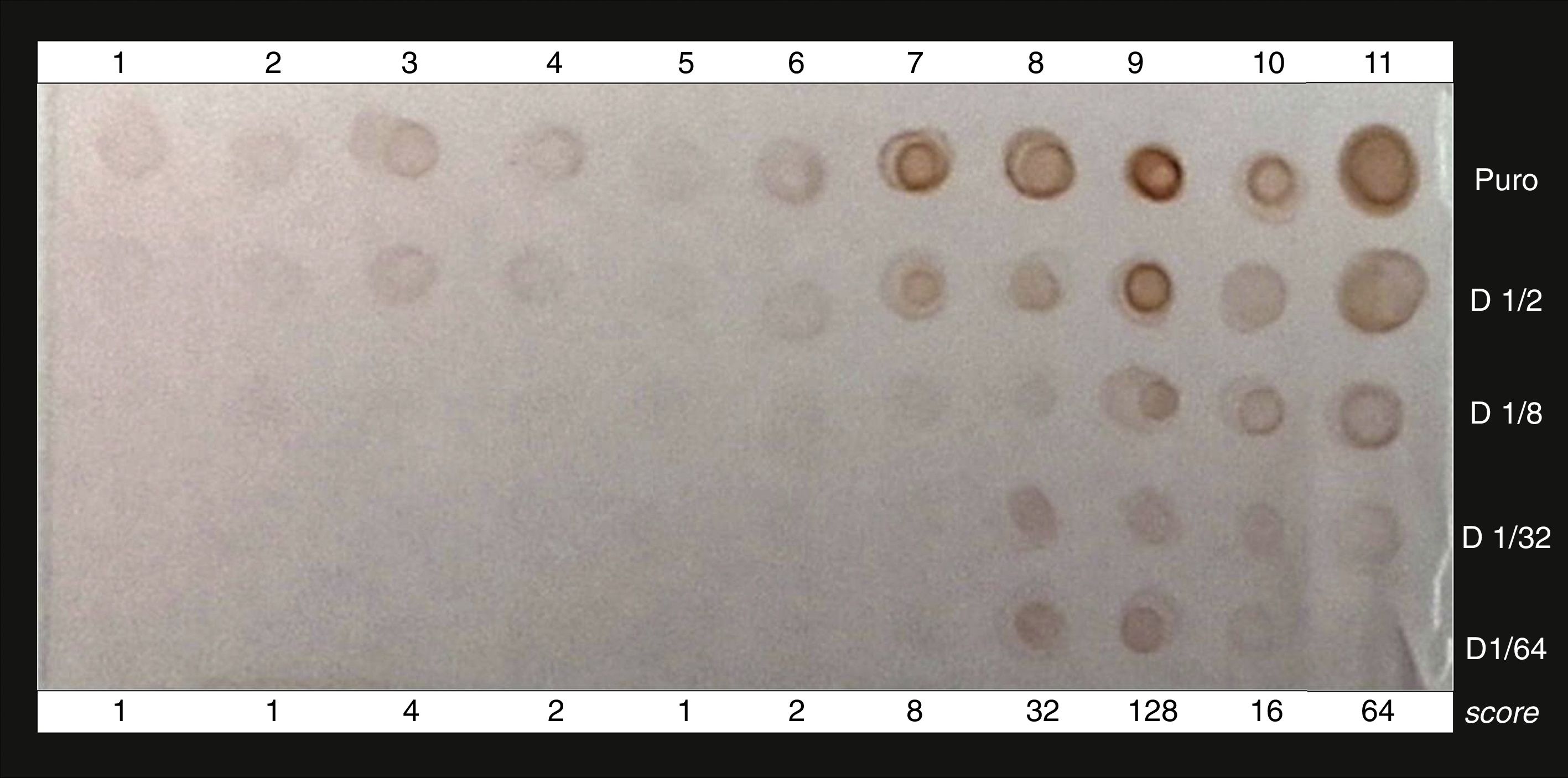
Para facilitar la lectura visual y disminuir la subjetividad de las observaciones, se estableció un score o puntaje asignando valores a las señales obtenidas y multiplicándolos entre sí. Cada señal obtenida sobre la membrana se consideró que tenía una base numérica igual a 4, 2 o 1, dependiendo de si la señal era muy fuerte, tenue/baja o no visible, respectivamente. Luego, los puntajes asignados a cada una de las 5 observaciones correspondientes a cada muestra (la original y sus 4 diluciones) se multiplicaron y el resultado se consideró su score final. Por ejemplo, en la figura 1, para la muestra correspondiente a la calle 7, el score se determinó de la siguiente manera, 4×2×1×1×1 = 8.
Dot blot aplicado a extractos de plantas sanas e infectadas con Cercospora kikuchii NBRC 6711. Calles 4, 5 y 6: plantas sanas. Calles 7, 8, 9 y 10: plantas inoculadas con la cepa de referencia C. kikuchii NBRC 6711. Calle 1: control negativo (extracto de Penicillium sp.). Calle 2: control negativo (extracto de Alternaria sp.). Calle 3: falso positivo. Calle 11: control positivo (extracto de C. kikuchii NBRC 6711).
Se definió una línea de corte a través de la curva ROC utilizando el programa MedCalc y se consideró un score ≤ 2 para las plantas sanas y uno ≥ 4 para las plantas enfermas. Para el análisis estadístico de los resultados se utilizó una tabla de contingencia de las variables enfermedad y técnica de detección o de diagnóstico10.
Los parámetros de validación evaluados fueron:
- a.
DSe=VP/(VP+FN).
- b.
DSp=VN/(VN+FP).
- c.
Eficacia=(VP+VN)/(VP+VN+FP+FN).
- d.
VPP=VP/(VP+FP).
- e.
VPN=VN/(VN+FN)
El VP correspondió al número de los verdaderos positivos, el VN al de los verdaderos negativos, el FP al de los falsos positivos y el FN al de los falsos negativos.
Se calcularon los intervalos de confianza para la DSe y DSp empleando las ecuaciones 1 y 2, considerando una confianza del 95%:
A manera de ejemplo, se muestra en la figura 1 el Dot blot aplicado a extractos de plantas sanas e infectadas con C. kikuchii NBRC 6711.
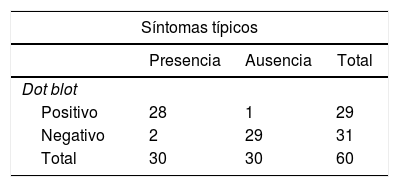
De las 29 muestras de plantas enfermas analizadas, solo 2 presentaron un score ≤ 2, lo que indica que hubo solo 2 falsos negativos con este método, y de las 31 muestras de plantas sanas, solo una arrojó un resultado falso positivo, al presentar un score ≥ 4 (fig. 1, calle 3), estos resultados se volcaron en la tabla 1.
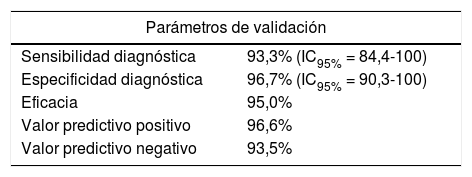
Con los resultados obtenidos se calcularon luego los parámetros de validación antes mencionados, que se expresan en la tabla 2.
Las estimaciones de la DSe (proporción de plantas que se sabe que están infectadas y que dan positivo en una prueba) y de la DSp (proporción de plantas que se sabe que no están infectadas y que dan negativo en una prueba) son los indicadores principales del rendimiento durante la validación de una prueba5.
La técnica de Dot blot empleada presentó una DSe del 93,3% (IC95%=84,4-100) y una DSp de 96,7% (IC95%=90,3-100), valores inferiores al 98% estimado. Sin embargo, dichos valores sí están comprendidos dentro del IC seleccionado para la validación de la técnica (95%).
Estas estimaciones son la base del cálculo de otros parámetros, como los VPP y los VPN, a partir de los cuales se realizan inferencias sobre los resultados de la técnica5. El VPP del 96,6% y el VPN del 93,5% indican que la probabilidad de que la planta esté realmente infectada (VPP) o realmente sana (VPN) es muy alta cuando se aplica esta técnica diagnóstica y los scores antes señalados.
La eficacia de la prueba fue del 95%, lo que revela que la probabilidad de que el patógeno sea detectado correctamente es también elevada15.
Al comparar estos resultados con los obtenidos por Peretti Canale et al.11 para la técnica de PCR, se puede observar que ambas metodologías presentaron parámetros de validación similares. No obstante, el Dot blot es una prueba de diagnóstico económica y de fácil ejecución en laboratorios de baja complejidad13. Al generar un precipitado coloreado, también podría ser utilizada en el campo como un método de screening9, sin la necesidad de disponer de equipos específicos.
Por lo tanto, el método de Dot blot descrito y validado es una metodología útil para la detección de C. kikuchii en plantas de soja, ya que ofrece resultados positivos y negativos que permiten confirmar o descartar la presencia de este patógeno con un elevado nivel de confianza en etapas tempranas de infección.
Además, esta metodología podría ser utilizada para la detección de otras especies de Cercospora productoras de cercosporina, como es el caso de C. sojina. Cabe destacar que durante la optimización del método, Latorre Rapela obtuvo similares resultados para ambas especies7.
Los resultados alcanzados en el presente trabajo son los primeros de su tipo en Argentina para el diagnóstico de esta importante enfermedad de fin de ciclo de la soja y brindan la posibilidad de contar con una valiosa herramienta para el perfeccionamiento de los procedimientos fitosanitarios actuales, ya que el diagnóstico precoz de las enfermedades, de forma rápida y precisa, evita el uso desmedido o incorrecto de compuestos químicos como plaguicidas.
Responsabilidades éticasProtección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Derecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
FinanciaciónEste trabajo se realizó en el marco del Proyecto CAI+D 2011, financiado por Universidad Nacional del Litoral, Argentina y del Proyecto Federal de Innovación Productiva PFIP 2009-1 evaluado/financiado por COFECYT.
Conflicto de interesesLos autores declaran que no existen conflictos de intereses.
Se agradece a la Dra. Roxana Maumary por la desinteresada provisión de las semillas utilizadas.