Se describe por primera vez en Argentina el aislamiento de Salmonella enterica no Typhi fermentadora de lactosa y lisina decarboxilasa negativa; este se obtuvo del hemocultivo de una paciente con lupus eritematoso sistémico. El microorganismo fue identificado como S. enterica (score: 2,148) mediante espectrometría de masas; el sistema VITEK® 2 lo identificó como grupo Salmonella (discriminación: 86%) y las pruebas bioquímicas como complejo Citrobacterfreundii. La reacción serológica con el antisuero polivalente OMA para el antígeno somático fue positiva. En el Centro Nacional de Referencia ANLIS Dr. Carlos G. Malbrán, se confirmó su identidad como Salmonella subesp. I 4,[5],12:i:- atípica. La espectrometría de masas resultó especialmente útil en la identificación microbiana en presencia de atipias; en estos casos, las pruebas bioquímicas pueden llevar a una identificación errónea. Se advierte sobre la circulación de esta cepa en Argentina.
We describe here the first isolation in Argentina of a lactose-fermenting and lysine decarboxylase-negative non-Typhi Salmonella enterica from a blood culture of a patient with systemic lupus erythematosus. The microorganism was identified as S. enterica (score: 2.148) by mass spectrometry, as “Salmonella group” (discrimination: 86%) by the VITEK® 2 system and as Citrobacter freundii complex by biochemical tests. The serological test using the OMA antiserum for the somatic antigen was positive. The identification was confirmed as Salmonella subsp. I 4,[5],12:i:- atypical at the National Reference Center ANLIS Dr. Carlos G. Malbrán. Mass spectrometry is especially useful in microbial identification in the presence of atypia, while biochemical tests can lead to misidentification in these cases. This study warns about the circulation of this strain in Argentina.
Las serovariedades de Salmonella enterica, excluidas Salmonella Typhi y Salmonella Paratyphi, habitualmente causan gastroenteritis en humanos que consumen alimentos contaminados. En pacientes con alteraciones de la inmunidad celular (enfermedades del tejido conectivo, SIDA, otras) y hemoglobinopatías, estas bacterias pueden causar infecciones extraintestinales que no son precedidas por diarrea11,15.
Esta es la primera comunicación en Argentina de una bacteriemia humana causada por Salmonella subesp. I 4,[5],12:i:-, con la atipia de ser lactosa positiva y lisina decarboxilasa negativa, en una paciente con lupus eritematoso sistémico (LES). La paciente, una mujer de 60 años, residía en la Ciudad Autónoma de Buenos Aires (Argentina) y carecía de antecedentes conocidos de viajes. Ingresó en el Servicio de Urgencias de un hospital de esa ciudad con el antecedente de fiebre diaria (38°C) durante los 2 meses previos a la consulta, astenia y adinamia. También refirió artralgias, alopecia, úlceras orales y dificultad para deambular por debilidad en la pantorrilla derecha.
A su ingreso hospitalario, la paciente presentó los siguientes parámetros: tensión arterial 140/90mmHg; frecuencia cardíaca 115lpm; frecuencia respiratoria 20rpm; saturación de O2 99%; temperatura 36,8°C. Se encontraba lúcida, con pupilas simétricas reactivas, movilización pasiva y activa conservada de los 4 miembros y buena mecánica ventilatoria. Se detectó abdomen blando, sin visceromegalias palpables, con petequias palatales, piel descamativa y alopecia occipital.
Los análisis de sangre realizados al ingreso mostraron los siguientes resultados: hematocrito 26%, hemoglobina 8,1g/dl, VCM 86,8fl, recuento de glóbulos blancos 3.380/mm3 (81% neutrófilos, 10% linfocitos), plaquetas 100.000/mm3, eritrosedimentación 105mm, glucemia 93mg/dl, urea 29mg/dl. Se decide su internación en el Servicio de Clínica Médica para su estudio y tratamiento.
Durante las primeras 12h de internación, se constató en la paciente fiebre persistente y se procedió a tomar muestras para cultivo bacteriológico de esputo y urocultivo, que fueron negativos, y hemocultivos, con el aislamiento de S. enterica (excluida S. Typhi) en uno de 2 frascos. Dicho microorganismo se identificó por espectrometría de masas, sumada a la no aglutinación con el antisuero anti-Vi. Se realizó tratamiento con ceftriaxona 1g/día durante 14 días, con cesación de los registros febriles.
Entre los estudios realizados, se destacan los siguientes hallazgos: disminución de factores del complemento (C3: 45mg/dl y C4: 5mg/dl), anticuerpo (Ac.) anti-ADN nativo positivo, Ac. anti-SM 116U, Ac. anti-RNP 154U, Ac. anti-Ro/SS-A 25U, Ac. anti-La/SS 27U, factor reumatoideo 12U, Ac. anti-CCP 40U y FAN 1/320 (con patrón homogéneo), tricitopenia (disminución de glóbulos blancos, rojos y plaquetas), TSH 31,6μUI/ml, T4L 0,8ng/dl. El sedimento urinario mostró hematuria dismórfica.
Se realizó un ecocardiograma, que mostró derrame pericárdico; una biopsia renal, con glomerulonefritis lúpica tipo IV; y un electromiograma, que evidenció mononeuropatía. Se diagnosticó LES, hipotiroidismo y anomalía estructural de la vía biliar.
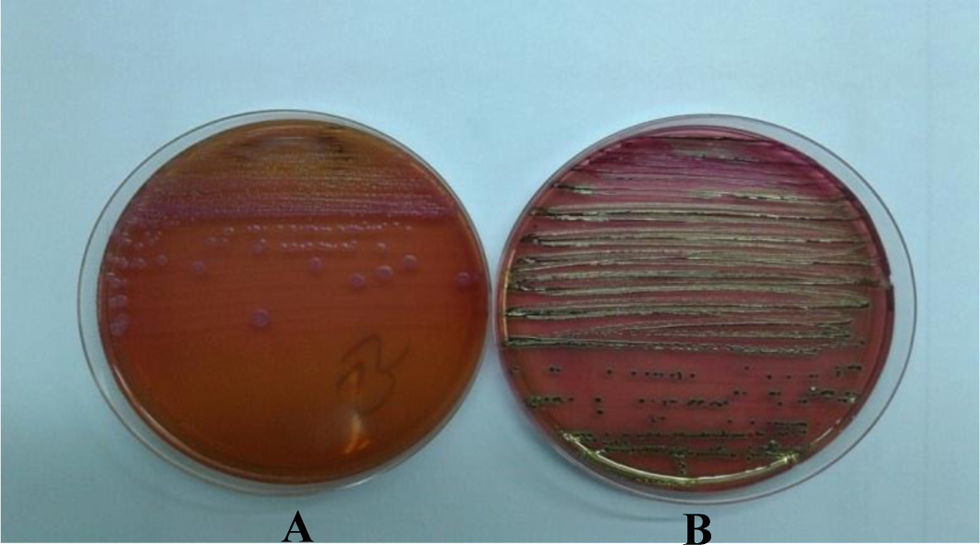
Para el estudio bacteriológico, se tomaron 2 muestras de sangre con las que se inocularon 2 frascos de hemocultivos, aeróbico y anaeróbico (método automatizado, BACTEC™, BD, Sparks, MD, EE. UU.). El frasco aeróbico se detectó positivo a las 12h 47min. En la coloración de Gram, se observaron bacilos negativos. En el subcultivo, a las 24h de incubación en atmósfera aeróbica en el medio agar EMB de Levine, se obtuvo desarrollo de colonias fermentadoras de lactosa, con brillo metálico, y en el agar Salmonella-Shigella (SS), las colonias fueron rosadas.
El aislado fue identificado como S. enterica (score: 2,148) por el método de espectrometría de masas MALDI-TOF MS Biotyper® 3 (Bruker Daltonics, Bremen, Alemania). El sistema VITEK® 2 (BioMérieux, Marcy-l’Étoile, Francia) identificó el aislado como grupo Salmonella (discriminación: 86%). Además, se realizó la identificación manual mediante las pruebas bioquímicas convencionales, cuyos resultados se describen a continuación. TSI: pico y fondo ácidos con gas, sin producción de SH2; ONPG: positiva; IMViC: −+−+; producción de ureasa (técnica de Christensen): negativa; decarboxilación de aminoácidos (técnica de Möeller): negativa para lisina, positiva para ornitina y arginina; movilidad: positiva; producción de SH2 en el medio SIM y LIA; PYR y utilización de malonato: negativos.
Se evaluó la utilización de hidratos de carbono en el medio base de Andrade; los resultados fueron positivos para lactosa, arabinosa, xilosa, galactosa y sorbitol, y negativos para ramnosa, rafinosa y trehalosa. La reacción serológica con el antisuero polivalente OMA (Bio Rad, Marnes-la-Coquette, Francia) para el antígeno somático fue positiva.
El aislamiento se derivó al Laboratorio Nacional de Referencia, Servicio de Enterobacterias, del Departamento de Bacteriología del INEI-ANLIS Dr. Carlos G. Malbrán, en donde fue identificado como Salmonella subesp. I 4,[5],12:i:- y confirmado por la técnica de PCR múltiple para serotipificación somática y flagelar, que dio por resultado la misma fórmula antigénica (según Herrera-León et al.9,10 y Echeita et al.7). Por otra parte, el aislado pertenece a Salmonella subesp. I o enterica con la atipia de ser lactosa positiva y no decarboxilar lisina.
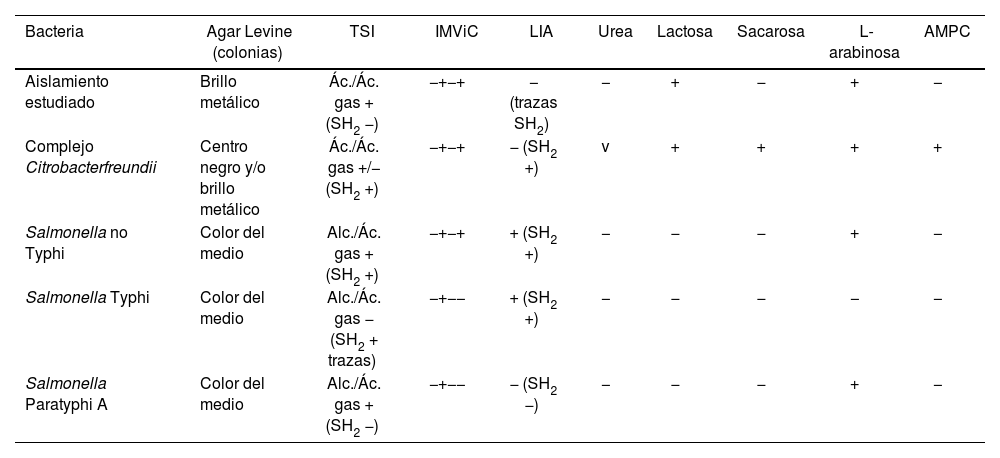
En la tabla 1 se muestran las principales pruebas que habitualmente se utilizan para diferenciar Salmonella no Typhi de las bacterias con las que, en este caso, podría confundirse.
Comparación de las pruebas bioquímicas del aislado frente al complejo Citrobacterfreundii, Salmonella enterica noTyphi, S. Typhi y S. Paratyphi A
| Bacteria | Agar Levine (colonias) | TSI | IMViC | LIA | Urea | Lactosa | Sacarosa | L-arabinosa | AMPC |
|---|---|---|---|---|---|---|---|---|---|
| Aislamiento estudiado | Brillo metálico | Ác./Ác. gas + (SH2 −) | −+−+ | − (trazas SH2) | − | + | − | + | − |
| Complejo Citrobacterfreundii | Centro negro y/o brillo metálico | Ác./Ác. gas +/− (SH2 +) | −+−+ | − (SH2 +) | v | + | + | + | + |
| Salmonella no Typhi | Color del medio | Alc./Ác. gas + (SH2 +) | −+−+ | + (SH2 +) | − | − | − | + | − |
| Salmonella Typhi | Color del medio | Alc./Ác. gas − (SH2 + trazas) | −+−− | + (SH2 +) | − | − | − | − | − |
| Salmonella Paratyphi A | Color del medio | Alc./Ác. gas + (SH2 −) | −+−− | − (SH2 −) | − | − | − | + | − |
Se realizó el antibiograma por el método automatizado Phoenix™ (BD, Sparks, MD, EE. UU.) utilizando el panel para bacilos gram negativos Nmic 406. La bacteria fue sensible a ampicilina+sulbactam (CIM=8/4μg/ml), piperacilina+tazobactam (CIM≤4/4μg/ml), cefazolina (CIM≤2μg/ml), ceftriaxona (CIM≤1μg/ml), imipenem (CIM≤0,25μg/ml), meropenem (CIM≤0,5μg/ml), ertapenem (CIM≤0,25μg/ml), amicacina (CIM≤8μg/ml) y trimetoprima/sulfametoxazol (CIM≤0,5/9,5μg/ml). Asimismo, presentó sensibilidad intermedia a ciprofloxacina (CIM=0,5μg/ml) y fue resistente a ampicilina (CIM>16μg/ml) y gentamicina (CIM>8μg/ml); los resultados fueron interpretados siguiendo las recomendaciones del Clinical and Laboratory Standards Institute5.
Salmonella subesp. I 4,[5],12:i:-, por sus características genotípicas, bioquímicas y fenotípicas, se considera una variante monofásica de Salmonella Typhimurium. Cabe destacar que esta serovariedad y Salmonella ser. Enteritidis son las más frecuentemente aisladas en humanos en nuestro país1,6,13.
Por lo general, en el laboratorio clínico, la identificación de Salmonella se realiza mediante pruebas bioquímicas seleccionadas a partir de la observación de colonias no fermentadoras de lactosa en el medio de agar EMB de Levine o en agar SS (o en ambos). La decarboxilación de lisina es, habitualmente, una prueba clave, puesto que permite excluir al complejo Citrobacterfreundii y a Proteus spp. Las pruebas de ureasa y detección rápida de galactosidasa (utilizando, por ejemplo, ONPG), también son muy usadas en el diagnóstico rutinario para descartar Proteus spp. o C. freundii. Asimismo, se confirma la identificación a nivel de género mediante una reacción serológica que determina el antígeno somático “O” y, en el caso de sospecha de Salmonella ser. Typhi, además de las características fenotípicas diferenciales con el resto de las serovariedades de Salmonella, se prueba con el antisuero anti-Vi. La identificación a nivel de serovariedad queda limitada a un laboratorio de referencia.
Actualmente, los laboratorios de mediana complejidad cuentan con métodos automatizados para la identificación microbiana, como VITEK® (bioMérieux) o Phoenix™ (BD), entre otros. En algunos laboratorios, se realiza espectrometría de masas. En este caso, la espectrometría de masas identificó rápidamente este aislamiento como Salmonella, lo que demuestra la robustez de este método frente a las pruebas bioquímicas tradicionales, que por sí solas no hubieran permitido identificarlo correctamente. Dada la morfología cultural en el agar EMB de Levine y en el agar SS (colonias fermentadoras de lactosa), sumado a la ausencia de lisina decarboxilasa y al resto de las características fenotípicas, el aislado hubiera sido identificado erróneamente como complejo C.freundii (figs. 1 y 2).
Los pacientes con LES suelen tener infecciones extraintestinales por S. enterica (no Typhi) por defecto en la inmunidad celular, hecho que debería tenerse en cuenta a la hora de interpretar la identificación fenotípica convencional de una enterobacteria aislada de hemocultivo en estos pacientes11,15.
A mediados de la década de los noventa, S. enterica serovar 4,[5],12:i:- emergió como patógeno humano en Europa y luego fue incrementándose su incidencia, asociada al consumo de productos porcinos, a diferencia de las otras serovariedades de S. enterica, que se encuentran con más frecuencia en aves de corral y huevos3,12,15. Esta serovariedad puede causar enterocolitis y septicemia en porcinos8.
En los años subsiguientes, se documentaron brotes por esta serovariedad en humanos, con aislados recuperados de materia fecal o sangre en España, Francia, Tailandia, Taiwán, EE. UU. y Brasil; recientemente, también se describió un aislamiento con la atipia de fermentar lactosa (operón lac +)2,4,6,12–14.
Dada la atipia en la identificación de Salmonella, es de interés realizar la secuenciación del genoma completo (plataforma Illumina) para la caracterización genotípica y el estudio de la relación filogenética con los aislamientos circulantes en Argentina de Salmonella monofásica 4,[5],12:i:- bajo vigilancia en el marco de la Red Nacional (Red Nacional de Diarreas) y Regional (Red PulseNet América Latina y El Caribe).
Se advierte sobre la circulación de S. enterica serovar 4,[5],12:i:- en Argentina, y, en particular, de esta cepa con atipia en la fermentación de lactosa (lactosa +) y la no decarboxilación de la lisina, cuya interpretación puede conducir a una identificación equivocada.
Se destaca la utilidad de los métodos automatizados y la espectrometría de masas (MALDI-TOF) para la identificación segura de estos aislados, lo que posibilita un diagnóstico y tratamiento adecuado de la enfermedad infecciosa. Asimismo, cabe notar la importancia de asociar el proceso de identificación con la enfermedad de base del paciente y el contexto epidemiológico, pues si en el caso de esta paciente solo se hubiera podido contar con los resultados de las pruebas fenotípicas convencionales, esa hubiera sido la única llamada de atención para sospechar de una infección por Salmonella.
FinanciaciónEl presente trabajo ha sido financiado con fondos del Proyecto UBACYT 2018 Modalidad I: código 20020170100109BA.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.