Las enfermedades de poscosecha en zanahoria son uno de los principales problemas durante su almacenamiento, afectan la vida de anaquel y ocasionan pérdidas económicas considerables. En este estudio se analiza la pudrición bacteriana en 10 muestras de zanahorias comerciales aparentemente sanas adquiridas en supermercados y mercados locales de Texcoco, Estado de México, México. Las zanahorias de 7 muestras desarrollaron pudrición bacteriana a los 5días de incubación en cámara húmeda a 28°C, y las zanahorias baby (industrializadas) mostraron la descomposición más grave. De las zanahorias con síntomas de pudrición se aislaron 23 cepas bacterianas. Para identificar y caracterizar a los agentes causales de la pudrición, las cepas se inocularon en zanahorias sanas en ensayos de laboratorio. De las 23 cepas analizadas, 8 causaron pudrición en el tejido de zanahoria. La caracterización bioquímica y molecular por secuenciación del gen 16S del ARNr identificó a estas 8 cepas como Gluconobacter cerinus (cepas 1032.2, 1059 y 1070.1), Gluconobacter kondonii (1027.1), Gluconobacter wancherniae (1033.1) y Paenibacillus polymyxa (cepas 1074.2, 1076 y 1077). Estas bacterias mostraron virulencia en otras pruebas de patogenicidad vegetal; las cepas del género Gluconobacter indujeron reacción de hipersensibilidad en hojas de tabaco y las cepas de P.polymyxa mostraron actividad pectinolítica en tubérculo de papa. Esta investigación reporta por primera vez a G.cerinus,G.kondonii, G.wancherniae y P.polymyxa como agentes causales de pudrición en zanahorias comerciales.
Postharvest diseases of carrots are one of the most important concerns in carrot storage, as they affect the shelf life of carrots and cause significant financial losses. In this study, ten commercial carrot samples from supermarkets and local markets in Texcoco, State of Mexico, Mexico, were analyzed for bacterial spoilage after incubation at 28°C in a humidity chamber. Carrots from seven samples developed bacterial spoilage after five days of incubation, of which baby carrots (industrially processed) showed the most severe rot. Twenty-three bacterial strains were isolated from the tissues of spoiled carrots. To identify and characterize the spoilage pathogens, the isolated strains were inoculated into healthy carrots in laboratory tests. Of the 23 strains analyzed, eight caused carrot tissue spoilage. Biochemical and molecular characterization by 16S ribosomal RNA gene sequencing identified Gluconobacter cerinus (1032.2, 1059 and 1070.1), G.kondonii (1027.1), G.wancherniae (1033.1) and Paenibacillus polymyxa (1074.2, 1076 and 1077) as the causal agents of the carrot rot evaluated in this study. In addition, these bacteria showed virulence in other plant pathogenicity tests; Gluconobacter strains induced a hypersensitivity reaction in tobacco leaves and Paenibacillus strains showed pectolytic activity in potato tubers. This study is the first to report G.cerinus, G.kondonii, G.wancherniae and P.polymyxa as causal agents of commercial carrot rot.
La zanahoria (Daucus carota L.), familia Apiaceae, es una de las hortalizas con mayor demanda comercial. Como parte de la dieta humana, se considera una fuente importante de hidratos de carbono, fibra, antioxidantes y carotenoides. Además, contribuye a la prevención de procesos degenerativos, como la arteriosclerosis, las cataratas y la enfermedad de Alzheimer, incluso reduce el riesgo de sufrir algunos tipos de cáncer36. En México, la zanahoria ocupa el sexto lugar entre las hortalizas con mayor producción (355.441t); el primer estado productor es Guanajuato (91,376t), seguido de Puebla (88,902t), Zacatecas (72,692t) y Estado de México (26,692t)28. Las mayores pérdidas económicas en la producción de zanahoria ocurren durante el proceso de almacenamiento, transporte y comercialización. Se consigna que uno de los problemas más importantes del almacenamiento de zanahoria a largo plazo son las pudriciones ocasionadas por bacterias u hongos, las que disminuyen los estándares de calidad y causan pérdidas económicas considerables16,34.
Las especies bacterianas reportadas con mayor frecuencia como causantes de pudrición blanda en zanahorias, antes y después de la cosecha, son Dickeya chrysanthemi, Pectobacterium carotovorum, Pseudomonas fluorescens, Pseudomonas marginalis y Pseudomonas viridiflava14. En el caso de los géneros Dickeya y Pectobacterium, suelen causar pérdidas importantes a temperatura ambiente tanto en el campo como en el almacenamiento8,14. Las pudriciones blandas de zanahoria en poscosecha se han atribuido al manejo inadecuado de plagas en los cultivos y a las heridas en las raíces, ocasionadas por los daños mecánicos durante la eliminación de hojas, el lavado y el transporte de esta hortaliza, lo cual aumenta la propensión a la infección y al desarrollo de microorganismos patógenos34.
Por lo expuesto, se hipotetiza que las zanahorias comerciales están contaminadas por bacterias que afectan la calidad sanitaria de esta hortaliza. El objetivo del presente estudio fue evaluar la sanidad de las zanahorias que adquiere el consumidor en supermercados y mercados locales de Texcoco (Estado de México, México), así como identificar y caracterizar a aquellos agentes que causan pudrición en zanahorias aparentemente sanas.
Materiales y métodosMuestreo de zanahorias comerciales sanasEntre abril y septiembre de 2023 se realizaron seis muestreos. Estas zanahorias constituyeron 10 muestras, identificadas como OZ1 a OZ10 (tabla 1). Las muestras consistieron en zanahorias baby con empaque comercial y zanahorias estándar (variedad Nantes) con empaque o a granel. Los empaques comerciales fueron bolsas de polietileno selladas sin perforaciones, mientras que las zanahorias sin empaque se encontraban a granel, totalmente expuestas al consumidor. En los supermercados, las zanahorias se muestrearon de anaqueles con temperatura de 18-20°C, mientras que en los mercados locales, las muestras estaban a temperatura ambiente (∼25°C).
Calidad fitosanitaria de zanahorias comerciales en Texcoco, México
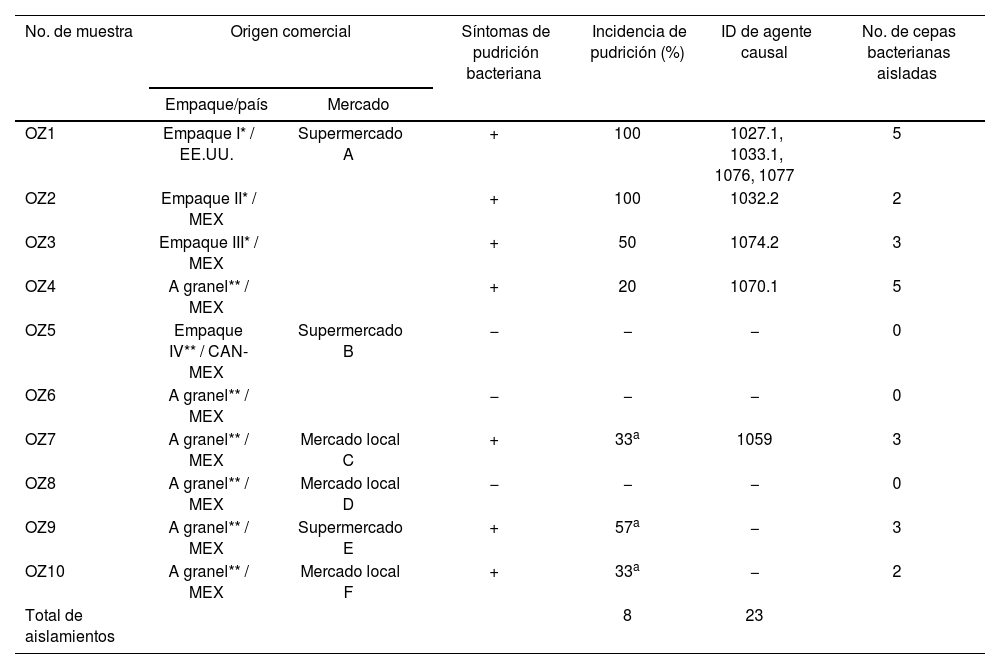
| No. de muestra | Origen comercial | Síntomas de pudrición bacteriana | Incidencia de pudrición (%) | ID de agente causal | No. de cepas bacterianas aisladas | |
|---|---|---|---|---|---|---|
| Empaque/país | Mercado | |||||
| OZ1 | Empaque I* / EE.UU. | Supermercado A | + | 100 | 1027.1, 1033.1, 1076, 1077 | 5 |
| OZ2 | Empaque II* / MEX | + | 100 | 1032.2 | 2 | |
| OZ3 | Empaque III* / MEX | + | 50 | 1074.2 | 3 | |
| OZ4 | A granel** / MEX | + | 20 | 1070.1 | 5 | |
| OZ5 | Empaque IV** / CAN-MEX | Supermercado B | − | − | − | 0 |
| OZ6 | A granel** / MEX | − | − | − | 0 | |
| OZ7 | A granel** / MEX | Mercado local C | + | 33a | 1059 | 3 |
| OZ8 | A granel** / MEX | Mercado local D | − | − | − | 0 |
| OZ9 | A granel** / MEX | Supermercado E | + | 57a | − | 3 |
| OZ10 | A granel** / MEX | Mercado local F | + | 33a | − | 2 |
| Total de aislamientos | 8 | 23 | ||||
OZ: origen de muestra; CAN: Canadá; EE.UU.: Estados Unidos de América; MEX: México.
Para determinar la sanidad de las zanahorias a través de la inducción de síntomas, se tomaron 14 piezas de cada muestra, se colocaron en cámaras húmedas estériles y se incubaron a temperatura ambiente (∼25°C) durante 5días.
Aislamiento de bacteriasDespués de la incubación de las zanahorias, del tejido con síntomas de pudrición se tomó exudado bacteriano con un asa bacteriológica estéril y se sembró directamente en placas con medio papa-dextrosa-agar (PDA) (BD Difco™, Francia) y en placas con agar nutritivo (BD Bioxon, México). Del crecimiento bacteriano, las colonias se reaislaron y purificaron (por estriado de una sola colonia) en placas con medio B-King. Las cepas aisladas se preservaron a −70°C en caldo nutritivo (BD Bioxon, México) y glicerol al 20% (vol/vol) y se depositaron en la colección del Colegio de Postgraduados.
Pruebas de patogenicidadPara determinar cuáles cepas bacterianas eran los agentes causales de la pudrición, cinco raíces de zanahorias estándar (variedad Nantes) visiblemente sanas se lavaron con agua corriente, se desinfectaron con etanol (70%) por 1min y después con hipoclorito de sodio (0,5%) por 5min, se enjuagaron con agua destilada estéril y se secaron al aire en una campana de flujo laminar14,22. De acuerdo con las pruebas de patogenicidad bacteriana establecidas y reportadas en zanahoria14,22, se siguieron los siguientes procedimientos. 1)En un primer ensayo, las zanahorias desinfectadas se cortaron en rodajas de 5mm de grosor con un cuchillo estéril y se colocaron en cámaras húmedas estériles (placas de Petri con papel absorbente y agua destilada estéril)14,22, tras lo cual las cepas individuales (n=23) se inocularon en el centro de las rodajas empleando 50μl de una suspensión celular a una concentración de 3×108 unidades formadoras de colonias (UFC)/ml, según la escala de turbidez de McFarland18,21. 2)En un segundo ensayo, cinco zanahorias sanas se desinfectaron por el método previamente descrito; a continuación, las mismas 23 cepas se inocularon sobre la superficie de las zanahorias enteras, sin heridas, con el mismo volumen y título bacteriano (50μl, 3×108 UFC/ml). Los controles negativos se prepararon aplicando 50μl de agua destilada estéril en lugar de inóculo. Las zanahorias de ambos ensayos se incubaron en cámaras húmedas estériles a 28±2°C y se evaluaron a las 24 y 48h después de la inoculación. Para cada ensayo se realizaron tres repeticiones.
Caracterización fisiológica y bioquímicaLas cepas bacterianas que causaron pudrición en zanahoria se caracterizaron por la tinción de Gram utilizando un kit de colorantes Gram (HYCEL®, México), siguiendo el protocolo del fabricante, y por el metabolismo oxidativo/fermentativo de la glucosa con el medio Hugh & Leifson, según lo descrito por Schaad et al.27. Para determinar la actividad enzimática de la catalasa, se tomó una asada de masa bacteriana de un cultivo de 24h y se mezcló con una gota de peróxido de hidrógeno (H2O2) al 3%; la formación de burbujas de gas indicó una reacción positiva27. La actividad de la citocromo-oxidasa se determinó empleando tiras de papel tratadas con una solución al 1% de sulfato de N,N-dimetil-p-fenilendiamina27. La actividad pectinolítica se evaluó en rodajas de papa (Solanum tuberosum L.) y la reacción de hipersensibilidad se probó en hojas de tabaco (Nicotiana tabacum L.), de acuerdo con los protocolos descritos por Schaad et al.27. Además, por API®20E (bioMérieux, EE.UU.) se evaluó la actividad de las enzimas β-galactosidasa, arginina dihidrolasa, lisina decarboxilasa, ornitina decarboxilasa, gelatinasa y ureasa, así como la utilización de citrato, la producción de H2S y la asimilación de D-glucosa, D-manitol, inositol, D-sorbitol, L-rhamnosa, D-sacarosa, D-melibiosa, amigdalina y L-arabinosa.
Extracción de ADNLa extracción de ADN de las cepas bacterianas se realizó por el método de CTAB al 2%24 a partir de cultivos puros con 48h de crecimiento a 28±2°C. La concentración y la pureza de las muestras de ADN se analizaron por espectrofotometría de luz UV a 260nm con un NanoDrop™ 2000 (Thermo Fisher Scientific); la integridad del ADN se evaluó por medio de una electroforesis en gel de agarosa (Invitrogen™) al 1% a 70V por 90min24.
Identificación molecularLa identificación molecular de las bacterias aisladas se realizó a partir de la amplificación por PCR y la secuenciación del gen 16S del ARN ribosomal (ARNr) utilizando los iniciadores 8F 5’-AGAGTTTGATCCTGGCTCAG-3’7 y 1492R 5’-GGTTACCTTGTTACGACTT-3’29. Las PCR se realizaron en un volumen total de 25μl, que contenían 1X del buffer de PCR, 2mM de MgCl2, 0,2mM de dNTP, 0,4μM de cada iniciador, 100ng de ADN y 1U de la enzima Platinum™ Taq DNA Polymerase (Invitrogen™). Las reacciones se llevaron a cabo en un termociclador Touch C1000 (Bio-Rad) con las siguientes condiciones: desnaturalización inicial a 95°C por 5min, 30 ciclos de desnaturalización a 95°C por 1min, hibridación a 58°C por 45s y extensión a 72°C por 1min, seguidos de una extensión final a 72°C por 8min.
Los productos de PCR se secuenciaron bidireccionalmente con los iniciadores 8F y 1492R por el método Sanger en la empresa Psomagen (Brooklyn, NY, EE.UU.). Las secuencias remitidas se analizaron y editaron con el programa BioEdit para generar las secuencias consenso de cada cepa (∼1300pb). Las secuencias consenso se compararon con las secuencias nucleotídicas de la base de datos del National Center for Biotechnology Information (NCBI), a través del programa Basic Local Alignment Search Tool (BLAST) (https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome). La especie se determinó de acuerdo con el porcentaje de similitud, mínimo >98,5%4.
Análisis filogenéticoEl análisis filogenético se realizó a partir de ocho secuencias nucleotídicas del gen 16S ARNr de las bacterias causantes de la pudrición de zanahoria y 39 secuencias de referencia: 27 del género Gluconobacter12,19 y 12 del género Paenibacillus13,23,35,37,38. La secuencia KP274903.1 de Pectobacterium carotovorum se incluyó como grupo externo. Los árboles filogenéticos se construyeron a partir de los alineamientos múltiples de las secuencias nucleotídicas (∼1300pb) generados por el programa CLUSTALW y se utilizó el método de máxima verosimilitud con 1000 réplicas y el mejor modelo de sustitución nucleotídica HKY+G11 en el programa MEGA1132.
Secuencias de ácidos nucleicosLas secuencias nucleotídicas obtenidas en el estudio se registraron en la base de datos GenBank de NCBI con los números de acceso PQ083548 a PQ083552 y PQ083678 a PQ083680.
ResultadosAislamiento de bacterias a partir de zanahorias con síntomas de pudrición inducidos en cámara húmedaDespués del periodo de incubación, se observó pudrición bacteriana en todos los empaques de las zanahorias baby (OZ1, OZ2 y OZ3) y en 4/6 muestras sin empaque (zanahorias a granel; OZ4, OZ7, OZ9 y OZ10). En algunas muestras se desarrollaron manchas húmedas (tabla 1 y fig. 1). Del tejido de las raíces sintomáticas en cámara húmeda (fig. 1) se aislaron 23 cepas bacterianas: 10 de zanahorias con empaque y 13 de zanahorias a granel (tabla 1).
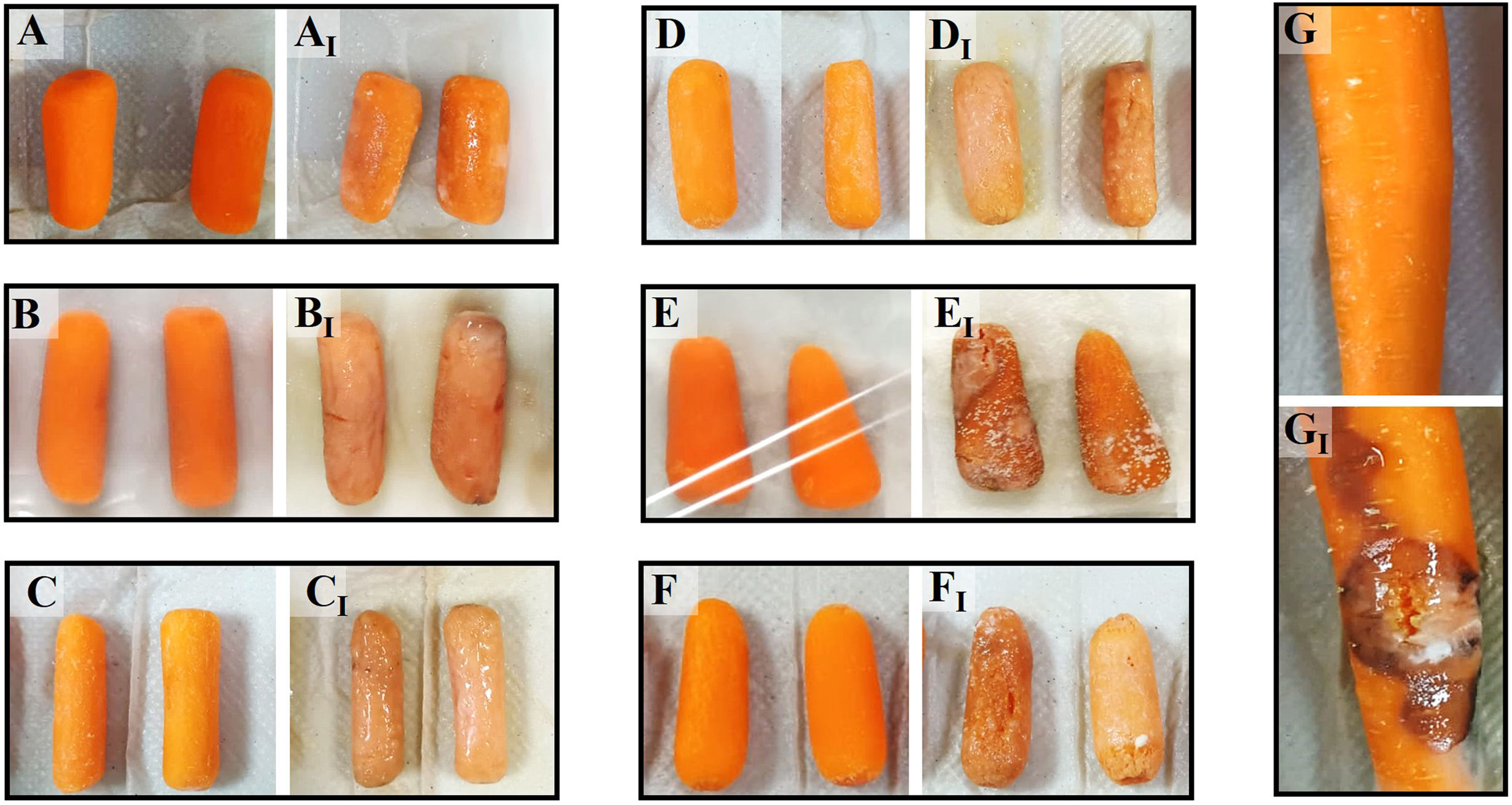
Síntomas de pudrición en zanahorias comerciales. Zanahorias sanas antes de la incubación (A, B, C, D, E, F y G) y zanahorias sintomáticas después de 5días de incubación a ∼25°C en cámara húmeda (AI, BI, CI, DI, EI, FI y GI).
AI, BI, CI y DI: ejemplares de la muestra OZ1, origen de las cepas 1027.1, 1033.1, 1076 y 1077, respectivamente; EI: ejemplares de la muestra OZ2, origen de la cepa 1032.2; FI: ejemplares de la muestra OZ3, origen de la cepa 1074.2; GI: ejemplar de la muestra OZ4, origen de la cepa 1070.1.
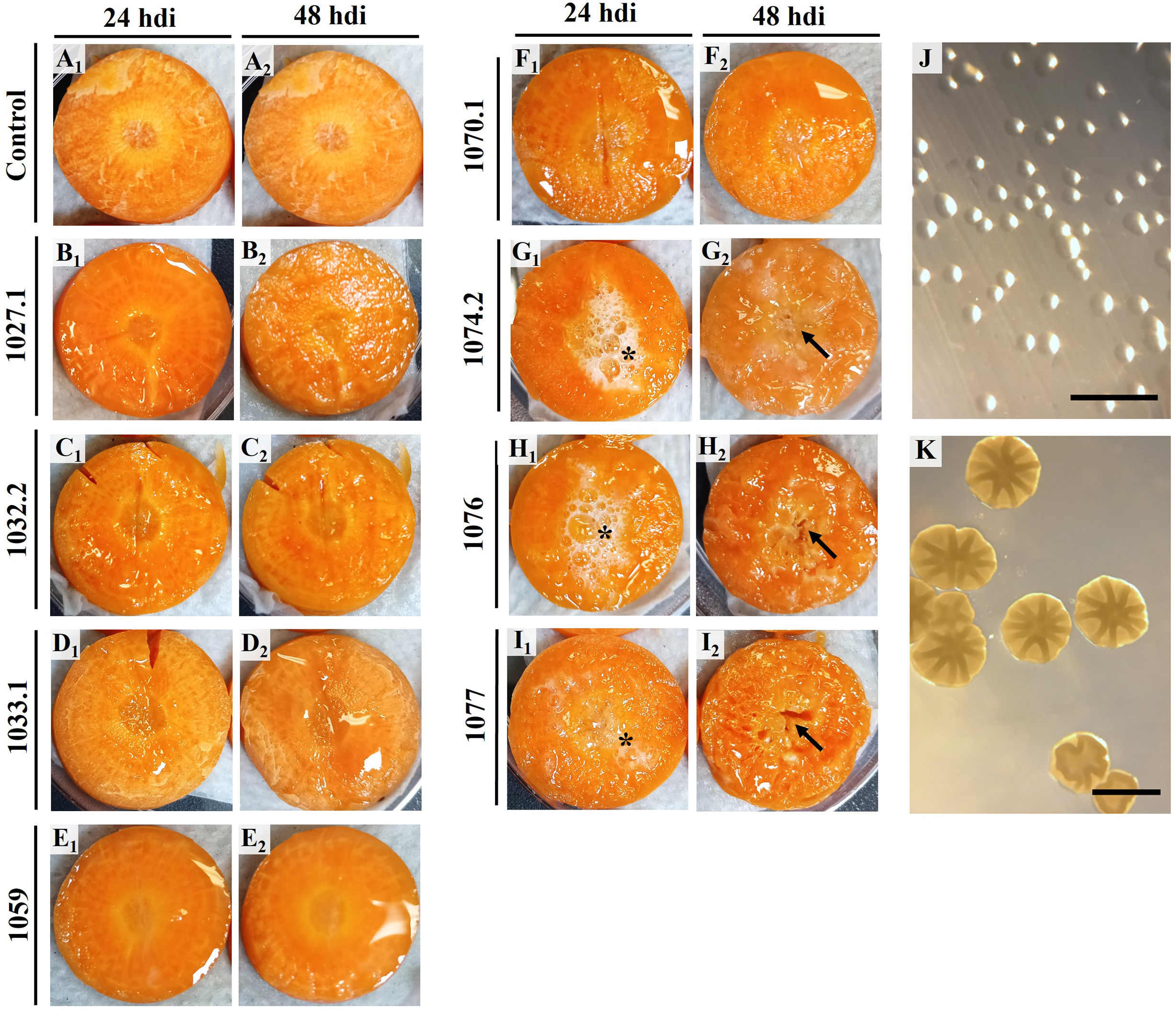
De los 23 aislados bacterianos inoculados para analizar la patogenicidad, 8 causaron pudrición tanto en rodajas de zanahoria como en zanahorias enteras sin heridas. En rodajas de zanahoria, la inoculación de las cepas 1027.1, 1032.2, 1033.1, 1059 y 1070.1 causó áreas húmedas a las 24h después de la inoculación (hdi) (fig. 2 BI, CI, DI, EI, FI), seguido del ablandamiento del tejido y olores no característicos u off odors (que recuerdan o se asocian al vinagre) a las 48hdi (fig. 2 B2, C2, D2, E2, F2). La inoculación con las cepas 1074.2, 1076 y 1077 causó pudrición blanda desde las 24 hdi, en la cual se observó fermentación (formación de burbujas) en el tejido degradado (fig. 2 G1, H1, I1), seguida de una maceración grave a las 48hdi (fig. 2 G2, H2, I2). En las rodajas de zanahoria del control negativo no se observó pudrición y el tejido permaneció íntegro hasta el final del ensayo (fig. 2 A1, A2). En el caso de las cepas bacterianas aisladas de las muestras OZ9 y OZ10, ninguna causó pudrición por sí sola.
Pruebas de patogenicidad en rodajas de zanahoria y morfología de las colonias bacterianas causantes de pudrición. Pudrición del tejido a las 24 y 48h después de la inoculación (hdi) con las cepas 1027.1 (B1 y B2), 1032.2 (C1 y C2), 1033.1 (D1 y D2), 1059 (E1 y E2), 1070.1 (F1 y F2), 1074.2 (G1 y G2), 1076 (H1 y H2) y 1077 (I1 y I2). Control negativo tratado con agua destilada estéril (A1 y A2). J)Morfología tipo de las colonias de las cepas 1027.1, 1032.2, 1033.1, 1059 y 1070.1 a las 48h de crecimiento en medio B-King. K)Morfología tipo de las colonias de las cepas 1074.2, 1076 y 1077 a las 48h de crecimiento en medio B-King.
*: Fermentación (formación de burbujas). Flechas negras: maceración grave del tejido. Las barras de escala corresponden a 0,5mm (J) y 2mm (K).
En medio de cultivo B-King, la morfología de las colonias de las cepas 1027.1, 1032.2, 1033.1, 1059 y 1070.1 se caracterizó por una apariencia traslúcida, superficie suave, elevación convexa, margen uniforme y brillo iridiscente (fig. 2J). En tanto que las colonias de las cepas 1074.2, 1076 y 1077 se observaron de color crema, textura granular, margen uniforme y elevaciones radiales en forma de asteriscos (fig. 2K).
Identificación molecularCon el análisis BLAST de las secuencias del gen ARNr 16S (∼1300pb) se identificó a la cepa 1027.1 como Gluconobacter kondonii Malimas et al., 2008 (99,12% de identidad con la secuencia NR_104680.1), a la cepa 1033.1 como Gluconobacter wancherniae Yukphan et al. 2010 (99,92% de identidad con NR_112952.1) y a las cepas 1032.2, 1059 y 1070.1 como Gluconobacter cerinus Yamada and Akita 1984 (99,93%, 99,78% y 99,48% de identidad con ON746595.1, MK883141.1 y PP386540.1, respectivamente). En tanto que las cepas 1074.2, 1076 y 1077 se identificaron como Paenibacillus polymyxa (Prazmowski 1880) Ash et al. 1994 (99,93%, 99,79% y 99,72% de identidad con EF532687.1, respectivamente).
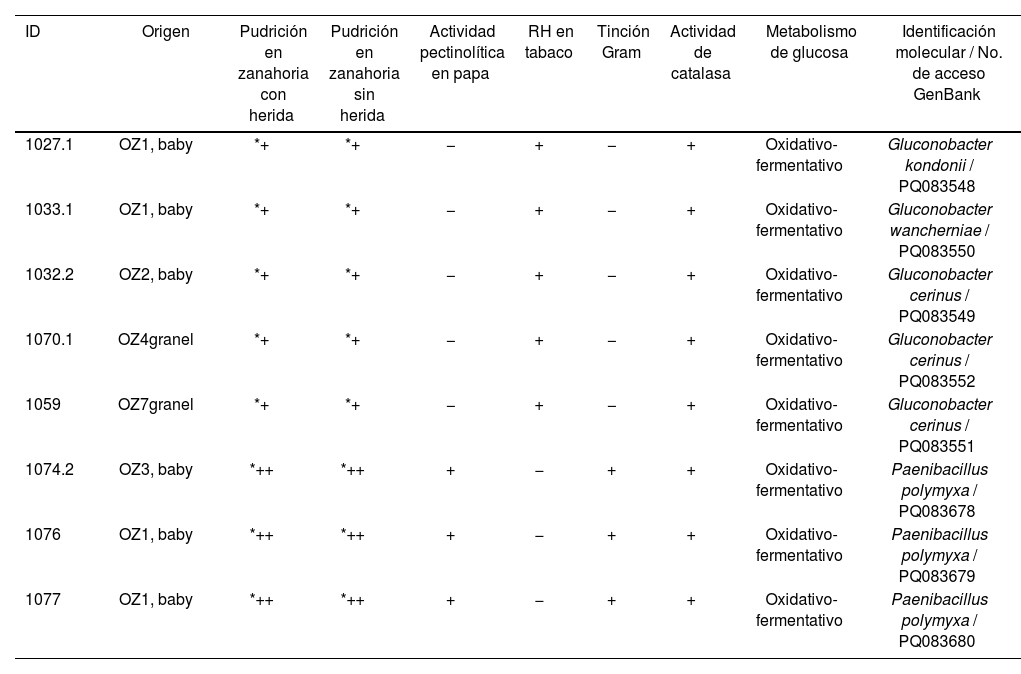
Caracterización fisiológica y bioquímicaLas cepas del género Gluconobacter (1027.1, 1032.2, 1033.1, 1059 y 1070.1) y P.polymyxa (1074.2, 1076 y 1077) mostraron un metabolismo oxidativo-fermentativo de la glucosa y actividad de la enzima catalasa positiva (tabla 2). En cuanto a las pruebas de patogenicidad vegetal, las cepas del género Gluconobacter indujeron reacción de hipersensibilidad en hojas de tabaco, mientras que las cepas de P.polymyxa mostraron actividad pectinolítica en tubérculo de papa (tabla 2). La caracterización fisiológica y bioquímica mediante API®20E mostró que las cepas del género Gluconobacter fueron negativas a la actividad de las enzimas citocromo-oxidasa, β-galactosidasa, arginina dihidrolasa, lisina decarboxilasa, ornitina decarboxilasa, gelatinasa y ureasa, pero fueron capaces de oxidar diferentes fuentes de carbono (tabla 3). G.cerinus presentó una oxidación más rápida de D-manitol y D-sorbitol respecto de G.kondonii. En contraste, las cepas de P.polymyxa no asimilaron ninguna de las fuentes de carbono evaluadas, pero resultaron positivas en las pruebas de β-galactosidasa y gelatinasa (tabla 3).
Identificación de las bacterias causantes de pudrición en zanahorias comerciales en Texcoco, México
| ID | Origen | Pudrición en zanahoria con herida | Pudrición en zanahoria sin herida | Actividad pectinolítica en papa | RH en tabaco | Tinción Gram | Actividad de catalasa | Metabolismo de glucosa | Identificación molecular / No. de acceso GenBank |
|---|---|---|---|---|---|---|---|---|---|
| 1027.1 | OZ1, baby | *+ | *+ | − | + | − | + | Oxidativo-fermentativo | Gluconobacter kondonii / PQ083548 |
| 1033.1 | OZ1, baby | *+ | *+ | − | + | − | + | Oxidativo-fermentativo | Gluconobacter wancherniae / PQ083550 |
| 1032.2 | OZ2, baby | *+ | *+ | − | + | − | + | Oxidativo-fermentativo | Gluconobacter cerinus / PQ083549 |
| 1070.1 | OZ4granel | *+ | *+ | − | + | − | + | Oxidativo-fermentativo | Gluconobacter cerinus / PQ083552 |
| 1059 | OZ7granel | *+ | *+ | − | + | − | + | Oxidativo-fermentativo | Gluconobacter cerinus / PQ083551 |
| 1074.2 | OZ3, baby | *++ | *++ | + | − | + | + | Oxidativo-fermentativo | Paenibacillus polymyxa / PQ083678 |
| 1076 | OZ1, baby | *++ | *++ | + | − | + | + | Oxidativo-fermentativo | Paenibacillus polymyxa / PQ083679 |
| 1077 | OZ1, baby | *++ | *++ | + | − | + | + | Oxidativo-fermentativo | Paenibacillus polymyxa / PQ083680 |
− negativo; + positivo; *+ pudrición ligera (ablandamiento de tejido en 48 horas); *++ pudrición grave (degradación del tejido en 24h); RH: reacción de hipersensibilidad.
Caracterización bioquímica de las bacterias causantes de pudrición en zanahorias comerciales en Texcoco, México
| Prueba | Gluconobacter kondonii(1027.1) | Gluconobacter cerinus(1032.2) | Paenibacillus polymyxa(1076) |
|---|---|---|---|
| Actividad enzimática de: | |||
| Citocromo-oxidasa | − | − | − |
| β-galactosidasa | − | − | + |
| Arginina dihidrolasa | − | − | − |
| Lisina decarboxilasa | − | − | − |
| Ornitina decarboxilasa | − | − | − |
| Gelatinasa | − | − | +* |
| Ureasa | − | − | − |
| Utilización de citrato | − | − | − |
| Producción de H2S | − | − | − |
| Oxidación de: | |||
| D-glucosa | + | + | − |
| D-manitol | +* | + | − |
| Inositol | +* | +* | − |
| D-sorbitol | +* | + | − |
| L-rhamnosa | +* | +* | − |
| D-sacarosa | +* | +* | − |
| D-melibiosa | + | + | |
| Amigdalina | +* | +* | - |
| L-arabinosa | + | + | - |
− reacción negativa; + reacción positiva a las 24h; +* reacción positiva a las 48h.
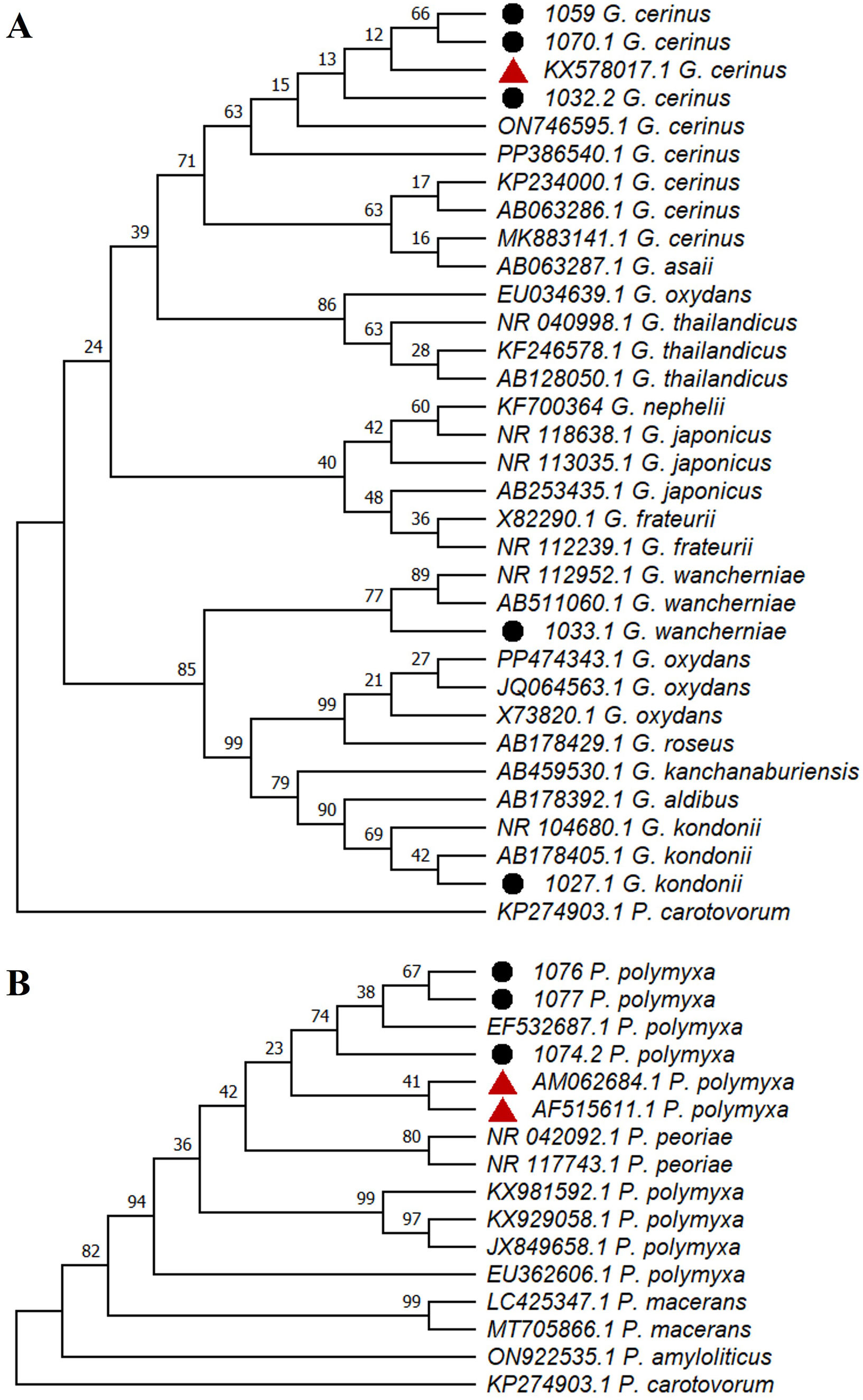
Con el análisis filogenético se corroboró la especie de las cepas causantes de pudrición en zanahoria y se determinó su relación filogenética con los miembros de los géneros Gluconobacter y Paenibacillus que han sido asociados con pudrición vegetal (fig. 3). Con base en las secuencias del gen ARNr 16S, las cepas 1032.2, 1059 y 1070.1 se agruparon en un mismo clado con la secuencia KX578017.1 de G.cerinus que causó pudrición en plátano (Musa paradisiaca L.)12, mientras que las cepas 1074.2, 1076 y 1077 resultaron filogenéticamente más cercanas a las cepas de P.polymyxa (AF515611.1) relacionadas con la pudrición de raíces de ginseng (Panax ginseng C.A. Mey.)13 y con la pudrición de tubérculos de cala (Zantedeschia spp., AM062684.1)23 (fig. 3).
Análisis filogenético de las cepas bacterianas 1027.1, 1032.2, 1033.1, 1059, 1070.1, 1074.2, 1076 y 1077, causantes de pudrición en zanahorias comerciales. Árboles filogenéticos consenso inferidos por el método de máxima verosimilitud (1000 réplicas) a partir de 32 secuencias nucleotídicas del gen ARNr 16S (∼1300pb) de especies del género Gluconobacter (A) y de 15 secuencias nucleotídicas del gen ARNr 16S (∼1300pb) de especies del género Paenibacillus (B). La secuencia KP274903.1 de Pectobacterium carotovorum se incluyó como grupo externo. Los árboles (A) y (B) se construyeron con el programa MEGA11. Los números en los nodos indican el porcentaje de réplicas en el que los taxones se asociaron en el mismo grupo. Los círculos negros indican las cepas aisladas en este estudio y los triángulos rojos indican las secuencias de Gluconobacter spp. (A) y Paenibacillus spp. (B) reportadas anteriormente como agentes causales de pudrición.
Desde el campo, el procesamiento y el almacenamiento inadecuados de las zanahorias puede afectar la calidad del producto y favorecer el desarrollo de enfermedades bacterianas que disminuyen su tiempo de vida en el anaquel34. Asimismo, las condiciones no asépticas del lavado y empacado pueden provocar contaminaciones bacterianas que podrían afectar la salud del consumidor. En esta investigación, las zanahorias de tipo baby presentaron pudrición bacteriana grave después de la incubación, a pesar de que los empaques indican que las zanahorias fueron desinfectadas y que no es necesario lavarlas para su consumo. Las zanahorias baby son un producto industrializado generado a partir del lavado, pelado y corte de zanahorias crudas2; por lo tanto, cabe considerar que las bacterias aisladas únicamente de estas muestras (P.polymyxa) podrían haber derivado de una contaminación originada durante alguna etapa del procesamiento de las zanahorias. Por otra parte, en zanahorias a granel las bacterias también afectaron la calidad para su consumo, ya que después de la incubación de las muestras, estas desarrollaron manchas húmedas y síntomas de pudrición, aunque la descomposición de las zanahorias fue menos frecuente y grave que la observada en las zanahorias baby industrializadas.
En este estudio, las bacterias identificadas con mayor frecuencia como causantes de pudrición de las zanahorias comerciales fueron del género Gluconobacter (familia Acetobacteriaceae) y se obtuvieron de una muestra con empaque en Estados Unidos y de tres muestras producidas en México. Las especies de Gluconobacter tienen la capacidad de oxidar etanol a ácido acético. Esta propiedad las hace importantes en la industria alimentaria y biotecnológica, principalmente en la producción de vinagre; sin embargo, también pueden promover la pudrición en diferentes frutas y causar pérdidas poscosecha9. Diversas especies del género Gluconobacter se han vinculado con la pudrición de uva, manzana, pera y piña9,10,26,33.
En uva, se ha evidenciado que la densidad y la diversidad de las poblaciones de Gluconobacter incrementan progresivamente durante la maduración del fruto y son mayores en las uvas podridas3,15, las cuales se caracterizan por tener un fuerte olor a vinagre asociado a altos niveles de ácido acético20. Las dos especies más frecuentemente vinculadas con la pudrición agria de las uvas son G.cerinus y Gluconobacter oxidans3. G.cerinus (syn. G.asaii) también causa pudrición húmeda en remolacha (Beta vulgaris L.)30. En manzana, la pudrición causada por Gluconobacter spp. se caracteriza por generar áreas acuosas en el punto de inoculación. En este estudio, la pudrición de las raíces de zanahoria causada por G.cerinus, G.kondonii y G.wancherniae también produjo áreas acuosas (fig. 2), además del ablandamiento del tejido y off odors que evocan el olor a ácido acético o vinagre. Este cambio de olor en frutos infectados con Gluconobacter spp. ha sido reportado frecuentemente. En piña, Ananas comosus (L.) Merr., G. oxydans puede causar «la enfermedad rosa», principalmente en frutos maduros25,26, y los tejidos podridos tienen un olor similar a melón (cantaloupe-like)26.
En cuanto a la fisiología, de las 23 cepas aisladas, solo las especies de Gluconobacter generaron reacción de hipersensibilidad en hojas de tabaco y así se evidenció su patogenicidad. Cabe recordar que esta reacción está relacionada con el sistema de secreción de tipoIII de bacterias gram negativas, a través del cual exportan factores de virulencia específica; además, se considera un indicador de virulencia y un rasgo común de muchas bacterias patógenas de plantas (Erwinia spp., Pseudomonas spp., Ralstonia spp. y Xanthomonas spp.)6 y de humanos (Escherichia coli, Salmonella spp., Shigella spp., Yersinia spp., etc.)6,31. Asimismo, las cepas de Gluconobacter y de P.polymyxa aisladas en este estudio presentaron una elevada actividad de la enzima catalasa. Esta enzima descompone el peróxido de hidrógeno en agua y oxígeno, lo cual resulta ser uno de los elementos más eficaces de protección de las bacterias fitopatógenas contra el estrés oxidativo generado por la defensa de las plantas5.
En relación con el perfil bioquímico, G.cerinus y G.kondonii (aisladas de zanahorias con pudrición en cámara húmeda) asimilaron todas las fuentes de carbono evaluadas en el sistema API®20E. En este sentido, se ha demostrado que las bacterias del género Gluconobacter prefieren ambientes ricos en azúcares, a diferencia de las acetobacterias de los géneros Acetobacter y Gluconacetobacter, que prefieren al etanol como fuente de carbono3. En uvas y manzanas, los daños mecánicos al fruto aumentan la accesibilidad del azúcar y permiten la infección por Gluconobacter; sin embargo, en pera, estas bacterias pueden causar descomposición sin la presencia de heridas3,33. En el caso de zanahoria, se observó que las diferentes cepas de Gluconobacter pueden descomponer las raíces sin la necesidad de que existan heridas.
Por otro lado, P.polymyxa también fue uno de los agentes causales de la pudrición de las zanahorias baby en muestras de dos empaques (uno de Estados Unidos y el otro de México); los resultados de las pruebas bioquímicas corroboran su patogenicidad. Debido a que P.polymyxa solo se aisló de zanahorias industrializadas, es posible que los procesos de lavado y corte hayan contaminado al producto con esta bacteria. Las especies de Bacillus y Paenibacillus raramente son consideradas como fitopatógenas, e incluso se consideran potenciales agentes de control biológico contra diversos patógenos de plantas. Sin embargo, se ha encontrado que las especies Bacillus cereus, Bacillus circulans, Bacillus pumilus, Bacillus subtilis, Paenibacillus amyloliticus, Paenibacillus macerans y P.polymyxa están implicadas en pudriciones blandas y pueden causar problemas de almacenamiento de algunos vegetales17,23,35. Así, P.polymyxa ocasiona pudrición blanda en tubérculos de papa y cala, tallos de Hylocereus undulates Britt. y raíces de ginseng13,23,37. En el caso de la pudrición de raíces de ginseng, se documentó que existen cepas no patogénicas y cepas patogénicas de P.polymyxa, y que la virulencia está relacionada con la capacidad de hidrolizar el almidón de dicha raíz13. En el caso de la pudrición blanda en tubérculos de cala causada por esta misma especie, la patogenicidad se debió a la actividad pectinolítica23. Además de la actividad pectinolítica, se ha descrito que P.polymyxa producen enzimas proteolíticas extracelulares, las cuales son influenciadas por los componentes del medio, como la presencia de azúcares (glucosa) fáciles de metabolizar o la relación de carbón/nitrógeno1. En cuanto a las cepas de P.polymyxa causantes de la pudrición de zanahoria en este estudio, se sugiere que su patogenicidad está dada por la actividad pectinolítica e incluso también por una actividad proteolítica, ya que resultaron positivas a la hidrólisis de gelatina.
ConclusionesLas zanahorias altamente procesadas y empacadas, a pesar de su desinfección industrial, pueden albergar bacterias causantes de pudrición que afectan su almacenamiento. Se reporta por primera vez la descomposición de zanahorias comerciales aparentemente sanas causada por las bacterias Gluconobater cerinus, Gluconobacter kondonii, Gluconobacter wancherniae y Paenibacillus polymyxa, principalmente en zanahorias baby. Preventivamente, para evitar el desarrollo bacteriano, se recomienda al consumidor conservar las zanahorias a una temperatura de 4-6°C. Sin embargo, los resultados de este estudio sugieren que es recomendable realizar una desinfección adicional de las zanahorias industrializadas antes de su consumo fresco y la remoción de la epidermis de las zanahorias sin procesar.
FinanciaciónEl presente trabajo ha sido financiado por el Consejo Mexiquense de Ciencia y Tecnología EDOMÉX (beca a Thelma J. Parra-Aguilar, folio ESYCA2023-1-7842) y por el Colegio de Postgraduados Campus Montecillo, a través del Laboratorio de Diagnóstico Integral Fitosanitario (LADIFIT) y de los Laboratorios de Bacterias y Hongos Fitopatógenos.
Conflicto de interesesLos autores declaran no tener conflictos de intereses.
Los autores agradecen el apoyo técnico brindado por Gema Jiménez, Paulina Romero y Martin Rivera del Laboratorio de Diagnóstico Integral Fitosanitario (LADIFIT).