Aeromonas spp. son patógenos oportunistas que producen infecciones intra y extraintestinales. El objetivo de este trabajo fue realizar la caracterización fenotípica y genotípica de una colección de aislamientos clínicos de Aeromonas, además de determinar su sensibilidad a distintos antimicrobianos. Se analizaron 37 aislamientos. El 54% fue de origen intraabdominal, el 22% se recuperó de piel y partes blandas y el 19% del torrente sanguíneo, entre otros sitios menos frecuentes. Por amplificación y secuenciación del gen gyrB, considerado método de referencia, el 37,8% de los aislamientos se identificaron como especies del complejo Aeromonas hydrophila, el 32,4% como especies del complejo Aeromonas veronii y el 29,7% como especies del complejo Aeromonas caviae. La identificación por pruebas bioquímicas tradicionales presentó una mejor correlación con la identificación molecular que la espectrometría de masas. Respecto de la sensibilidad antibiótica, cefotaxima (CTX), ceftazidima (CAZ), cefepima (FEP), piperacilina-tazobactam (PTZ), trimetoprima-sulfametoxazol (SXT), ciprofloxacina (CIP), amicacina (AK), gentamicina (CN) y nitrofurantoína (FF) mostraron actividad sobre más del 80% de los aislamientos ensayados. La sensibilidad y la especificidad de los métodos fenotípicos para determinar la presencia de carbapenemasas respecto de la detección del gen cphA (método de referencia) fueron del 60,9% y 100% con un ensayo colorimétrico (Blue Carba [BC]) y del 91,3% y 50% con el test de Hodge modificado (THM), respectivamente. La resistencia global a la colistina (CL) fue del 32,4%. El método automatizado para detectar dicha resistencia mostró un error muy mayor (very major error [VME]) del 16,2%, mientras que el método de screening rápido colorimétrico (TRCc) mostró una excelente correlación (VME 0%) con el método de referencia, la microdilución en caldo (MDC).
Aeromonas spp. are opportunistic pathogens that cause both intra- and extraintestinal infections. The objective of this work was the phenotypic and genotypic characterization of a collection of Aeromonas strains, in addition to determining their sensitivity to different antimicrobials. Thirty seven isolates were analyzed. 54% were of intra-abdominal origin, 22% from skin and soft tissues, 19% from the bloodstream, among other less frequent sites. By amplification and sequencing of the gyrB gene, which was considered the reference method, the following were identified: 37,8% as species of the Aeromonas hydrophila complex, 32,4% as species of the Aeromonas veronii complex, and 29,7% as species of the complex Aeromonas caviae. Identification by traditional biochemical tests presented a better correlation with molecular identification than mass spectrometry (MALDI TOF MS). Regarding antibiotic sensitivity, cefotaxime, ceftazidime, cefepime, piperacillin-tazobactam, trimethoprim-sulfamethoxazole, ciprofloxacin, amikacin, gentamicin and nitrofurantoin showed activity on more than 80.0% of the isolates tested. The sensitivity and specificity of the phenotypic methods to determine the presence of carbapenemases in relation to the detection of the cphAgene, the reference method, was 60,9% and 100%, respectively, for the colorimetric assay (Blue Carba), and of 91,3% and 50,0% respectively, for the modified Hodge test. The overall resistance to colistin was 32,4%. The automated method showed a very higher error (VME) of 16,2%, while the rapid colorimetric screening method (CRTc) showed an excellent correlation (VME 0%) with the reference method, broth microdilution.
Aeromonas spp. comprende bacilos gram negativos, anaerobios facultativos. Estas bacterias son oxidasa y catalasa positivas, reducen los nitratos a nitritos y muestran un metabolismo fermentativo de la glucosa26. Son microorganismos autóctonos del medio acuático; las infecciones en el ser humano pueden producirse por el consumo de agua o de alimentos contaminados, a través de traumatismos con heridas expuestas o por mordedura de algunas especies de reptiles y mamíferos. Estos agentes también pueden estar presentes en el medio hospitalario causando infecciones nosocomiales1. Se consideran microorganismos oportunistas emergentes que pueden causar desde infecciones leves, como gastroenteritis agudas, hasta procesos más complejos, como fascitis necrotizante y septicemia, en pacientes con enfermedad hepática u oncohematológica16,21.
Aeromonas es un género complejo que ha pasado por varias revisiones taxonómicas. Inicialmente fue ubicado en la familia Pseudomonadaceae y posteriormente en la familia Vibrionaceae, hasta que, en 1986, mediante métodos de secuenciación del gen 16S del ARN ribosómico (ARNr), se demostró su divergencia evolutiva que justificó la creación de la familia Aeromonadaceae16. Actualmente, se reconocen 53 especies dentro del género20. La identificación en el laboratorio puede llegar a ser un reto, dado que las pruebas bioquímicas convencionales, los sistemas automatizados, la espectrometría de masas Matrix Assisted Laser Desorption Ionization - Time of Flight (MALDI-TOF) e inclusive la amplificación y secuenciación del gen ARNr 16S presentan problemas para la identificación de las distintas especies28.
La identificación correcta a nivel de especie o de complejo tiene singular relevancia, ya que se ha observado que ciertas especies pueden producir una carbapenemasa cromosómica inducible tipo metalo-ß-lactamasa denominada carbapenem-hydrolysingAeromonas (cphA). Esta carbapenemasa puede no conferir resistencia a los carbapenems in vitro o expresarse en bajo nivel, por lo que su identificación es importante para evitar el fracaso terapéutico intratratamiento con dichos antibióticos29. En el mismo sentido, la importancia clínica de la identificación a nivel de especie de Aeromonas aisladas de bacteriemias monomicrobianas ya ha sido señalada en la literatura31.
Los objetivos del presente trabajo fueron los siguientes:
- 1.
Evaluar la efectividad de las pruebas bioquímicas convencionales y de la espectrometría de masas MALDI-TOF para identificar Aeromonas spp. como métodos alternativos a la identificación molecular.
- 2.
Determinar la sensibilidad a los distintos grupos de antimicrobianos, y, en particular, establecer la sensibilidad y la especificidad de distintas metodologías fenotípicas para detectar la resistencia a los carbapenems.
- 3.
Evaluar la capacidad de un sistema automatizado y de un método colorimétrico rápido para detectar sensibilidad (o resistencia) a colistina (CL).
Se estudiaron 37 aislamientos no consecutivos de Aeromonas spp. que habían sido obtenidos de diferentes materiales clínicos de pacientes atendidos en el Hospital de Clínicas «José de San Martín» (periodo 2007-2018). El 54% de los aislamientos era de origen intraabdominal (líquido abdominal/heces; n = 20), el 22% provenía de piel y partes blandas (n = 8), el 19% se recuperó de sangre (n = 7) y el 5% restante correspondió a otros sitios menos frecuentes (n = 2).
Todos los aislamientos se conservaron en caldo infusión cerebro-corazón (BHI) con glicerol al 20% a −70°C hasta el momento de su estudio. Antes de su uso en las pruebas descritas a continuación, fueron subcultivados en agar base Columbia con el agregado de sangre al 5%, con incubación de 24 horas a 37°C en atmósfera normal.
Identificación fenotípicaIdentificación por pruebas bioquímicas convencionalesEstas incluyeron coloración de Gram, catalasa, oxidasa (Ox), acidificación del agar Triple Sugar Iron (TSI), presencia de gas de glucosa, prueba de indol, hidrólisis de la esculina (ESC), prueba de Voges Proskauer (VP), prueba de ADNasa, descarboxilación de los aminoácidos lisina (LDC), ornitina (ODC) y arginina (ADH) en caldo Moeller y fermentación de los azúcares arabinosa (ARA), sacarosa (SAC) y celobiosa (CELB). Para la interpretación de los resultados, se usó el esquema propuesto por Janda et al16. Se evaluaron, además, sus características culturales en agar sangre, como la presencia de beta hemólisis
Identificación por espectrometría de masasLa identificación por espectrometría de masas (MALDI-TOF MS) se llevó a cabo con el espectrómetro Microflex MALDI-TOF MS (Bruker Daltonics, Bremen, Alemania) empleando el software FlexControl (Bruker Daltonics, Bremen, Alemania). Para ello, se depositó una colonia del aislado seguido de la extracción con fórmico al 70% (técnica on spot) sobre una tarjeta de análisis y se dejó secar a temperatura ambiente. Las muestras fueron cubiertas con 1 μl de matriz (α-ciano-4 hidroxicinámico) hasta su cristalización a temperatura ambiente. Los espectros de masas se analizaron en un rango de 2.000 a 20.000 m/z.
Los aislados fueron identificados mediante el software MALDI Biotyper versión 3.1. Bruker Daltonics, Biblioteca 11.0 (Bruker Daltonics, Bremen, Alemania). Para la interpretación de los resultados, se utilizaron los puntos de corte según las recomendaciones del fabricante: identificación confiable a nivel de género con un score entre 1,700 y 1,999 e identificación a nivel de género y especie con un score ≥ 2,000; los resultados obtenidos con un score <1,700 se catalogaron como no confiables.
Se utilizó el criterio de validez de interpretación entre la primera especie y la siguiente distinta de los 10 primeros resultados con una divergencia del 10%. Si no se cumplía esta condición, la identificación se consideró correcta solo a nivel de género. La identificación por MALDI-TOF MS se consideró correcta cuando el resultado obtenido estuvo de acuerdo con los resultados de identificación mediante la secuenciación del gen gyrB.
Identificación molecularSe llevó a cabo mediante la amplificación y posterior secuenciación del gen gyrB, luego de la extracción del ADN bacteriano por la técnica descrita en Green et al. (fenol-cloroformo-alcohol-isoamílico)15. En la amplificación, se utilizaron cebadores específicos y Taq ADN polimerasa (Promega). Esta metodología fue considerada el gold standard para la identificación de género y especie. Los productos de reacción en cadena de la polimerasa (PCR) fueron secuenciados utilizando el Big Dye Terminator v3.1 Cycle sequencing kit (Applied Biosystems, Foster City, CA, EE. UU.) en el secuenciador ABI PRISM 3100 Genetic Analyzer (PE Applied Biosystems, CA, EE.UU.). El alineamiento de las secuencias se realizó con el algoritmo BLASTN (The Basic Local Alignment Search Tool, version 2.0; National Center for Biotechnology Information, EE.UU.); estas se compararon con secuencias de cepas de referencia disponibles en la base de datos del GenBank.
Pruebas de sensibilidad a los antimicrobianosSe utilizó el sistema automatizado Vitek 2C (bioMerieux, Marcy l’Etoile, Francia) con el panel AST-082, que incluye los siguientes antibióticos: ampicilina (AMP), ampicilina-sulbactam (AMS), cefalotina (CF), cefotaxima (CTX), ceftazidima (CAZ), cefepima (FEP), piperacilina-tazobactam (PTZ), imipenem (IMI), meropenem (MEM), amicacina (AK), gentamicina (CN), ciprofloxacina (CIP), nitrofurantoína (FF), trimetoprima-sulfametoxazol (SXT) y CL. Se utilizaron los puntos de corte establecidos por el Clinical and Laboratory Standards Institute (CLSI) M45 3 ed., para Aeromonas spp.7, excepto en el caso de AMP, FF y CL, para los cuales se usaron los puntos de corte establecidos por el CLSI 32 ed. M100, para Enterobacterales9.
Detección de la resistencia enzimática a los carbapenemsDetección fenotípicaLa resistencia a los carbapenems se investigó por distintas metodologías, a saber: por un sistema automatizado (Vitek 2C; bioMérieux), por un método colorimétrico rápido para detección de carbapenemasas denominado Blue Carba (BC), descrito por Pires et al.25, y mediante el test de Hodge modificado (THM), según la técnica descrita en la guía del CLSI M100-S26, de 201610.
Evaluación genotípicaSe llevó a cabo mediante la amplificación del gen cphA luego de la extracción del ADN bacteriano, según se describió en un apartado anterior. Para la amplificación, se utilizaron los cebadores cphA R (5′-GCA TAG GTA CCT TAT GAC TGG GGT GCG GCC TTG–3′) y cphA F (5′-GCT TAG AGC TCC TAA GGA GCA AGA TGA AAG GTT GG-3′), para amplificar un fragmento de 720 pb, aproximadamente; los productos de PCR fueron revelados mediante electroforesis en un gel de agarosa al 1%, voltaje 110 V y un tiempo de corrida de 30 minutos. Esta técnica fue considerada el gold standard para la detección de resistencia a los carbapenémicos.
Evaluación de la resistencia a CLSe realizó mediante el método rápido de screening colorimétrico (TRCc) propuesto por Nordmann et al.23, por el sistema automatizado Vitek 2C y por el método de microdilución en caldo (MDC) como método de referencia. Los paneles de CL se prepararon según las recomendaciones de la guía CLSI M7-A10, de 20158.
Medidas de desempeño de los métodosSe calculó la sensibilidad y la especificidad de los métodos de identificación fenotípica por metodología convencional y por espectrometría de masas (MALDI-TOF) con relación a la identificación molecular (secuenciación del gen gyrB).
Se calculó la sensibilidad y la especificidad, el valor predictivo positivo (VPP) y el valor predictivo negativo (VPN) de los métodos fenotípicos de detección de resistencia a los carbapenems (BC y test de Hodge) en relación con la detección molecular del gen cphA.
Se calcularon el error muy mayor (very major error [VME]) y el error mayor (major error [ME]) de la determinación de la resistencia a la CL por el método automatizado y por el TRCc con referencia a la MDC.
ResultadosIdentificación por pruebas bioquímicasDe acuerdo con las características bioquímicas, 14 (37,8%) de los aislados correspondieron al complejo Aeromonas hydrophila, 12 (32,4%) al complejo Aeromonas veronii y 11 (29,7%) al complejo Aeromonas caviae. Un 73% de los aislados presentaron beta hemólisis.
Identificación por MALDI TOF MSMediante el análisis por espectrometría de masas MALDI-TOF, solo se obtuvo identificación correcta a nivel de especie en cinco aislados (13,5%), de género en nueve (24,3%), baja discriminación entre dos o más especies en 20 (54%) e identificación incorrecta en tres aislados (8,1%), los que fueron identificados como Aeromonas aquariorum (sinónimo de Aeromonas dhakensis) por secuenciación del gen gyrB. Con estos resultados, podemos inferir que la espectrometría de masas MALDI-TOF solo identifica correctamente Aeromonas a nivel de género, pero tiene deficiencias para identificar a nivel de especie.
Identificación molecularLa identificación molecular ubicó a 12 aislamientos (32,4%) como pertenecientes al complejo A. veronii (y dentro de este a uno como A. veronii bv. veronii), a 11 (29,7%) como pertenecientes al complejo A. caviae (y dentro de este, a uno como Aeromonas media) y a ocho (21,6%) como parte del complejo A. hydrophila, con uno como Aeromonas salmonicida y seis (16,2%) como A. dhakensis (A. aquariorum).
El análisis de correlación de las diferentes metodologías de identificación (pruebas bioquímicas y MALDI-TOF MS) con respecto al gold standard (secuenciación del gen gyrB) se muestra en la tabla 1.
Correlación entre métodos para la identificación de aislamientos clínicos de Aeromonas spp. a nivel de complejo
| Metodología | Secuenciación del gen gyrB | ||
|---|---|---|---|
| Comp. A. caviae (%) | Comp. A. hydrophila (%) | Comp. A. veronii (%) | |
| Pruebas bioquímicas | 100 | 85,7 | 75,0 |
| MALDI-TOF MS | 0,0 | 21,4 | 16,6 |
Comp.: complejo; MALDI-TOF: Matrix Assisted Laser Desorption Ionization - Time of Flight.
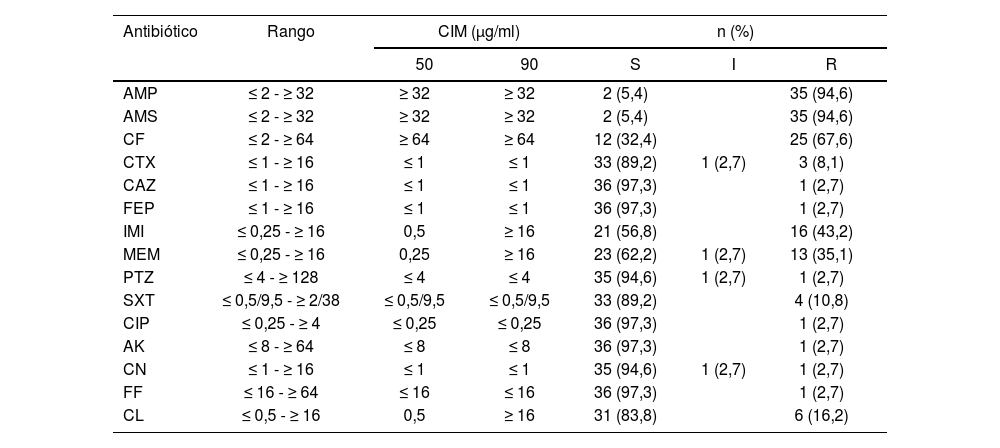
Los rangos de concentración inhibitoria mínima (CIM) y los valores de CIM50 y CIM90 para los antimicrobianos ensayados frente a los 37 aislados de Aeromonas spp. se presentan en la tabla 2. Por el método automatizado, la mayoría de los aislados mostraron resistencia (R+I) a AMP (94,6%), AMS (94,6%) y CF (67,6%), mientras que la resistencia a CTX, CAZ, FEP, PTZ, SXT, CIP, AK, CN y FF se detectó en menos del 20% de los aislados. Asimismo, fue variable la resistencia a carbapenems y a CL (IMI 43,2%; MEM 37,8% y CL 16,2%).
Sensibilidad antibiótica de aislamientos clínicos de Aeromonas spp
| Antibiótico | Rango | CIM (μg/ml) | n (%) | |||
|---|---|---|---|---|---|---|
| 50 | 90 | S | I | R | ||
| AMP | ≤ 2 - ≥ 32 | ≥ 32 | ≥ 32 | 2 (5,4) | 35 (94,6) | |
| AMS | ≤ 2 - ≥ 32 | ≥ 32 | ≥ 32 | 2 (5,4) | 35 (94,6) | |
| CF | ≤ 2 - ≥ 64 | ≥ 64 | ≥ 64 | 12 (32,4) | 25 (67,6) | |
| CTX | ≤ 1 - ≥ 16 | ≤ 1 | ≤ 1 | 33 (89,2) | 1 (2,7) | 3 (8,1) |
| CAZ | ≤ 1 - ≥ 16 | ≤ 1 | ≤ 1 | 36 (97,3) | 1 (2,7) | |
| FEP | ≤ 1 - ≥ 16 | ≤ 1 | ≤ 1 | 36 (97,3) | 1 (2,7) | |
| IMI | ≤ 0,25 - ≥ 16 | 0,5 | ≥ 16 | 21 (56,8) | 16 (43,2) | |
| MEM | ≤ 0,25 - ≥ 16 | 0,25 | ≥ 16 | 23 (62,2) | 1 (2,7) | 13 (35,1) |
| PTZ | ≤ 4 - ≥ 128 | ≤ 4 | ≤ 4 | 35 (94,6) | 1 (2,7) | 1 (2,7) |
| SXT | ≤ 0,5/9,5 - ≥ 2/38 | ≤ 0,5/9,5 | ≤ 0,5/9,5 | 33 (89,2) | 4 (10,8) | |
| CIP | ≤ 0,25 - ≥ 4 | ≤ 0,25 | ≤ 0,25 | 36 (97,3) | 1 (2,7) | |
| AK | ≤ 8 - ≥ 64 | ≤ 8 | ≤ 8 | 36 (97,3) | 1 (2,7) | |
| CN | ≤ 1 - ≥ 16 | ≤ 1 | ≤ 1 | 35 (94,6) | 1 (2,7) | 1 (2,7) |
| FF | ≤ 16 - ≥ 64 | ≤ 16 | ≤ 16 | 36 (97,3) | 1 (2,7) | |
| CL | ≤ 0,5 - ≥ 16 | 0,5 | ≥ 16 | 31 (83,8) | 6 (16,2) | |
S: sensible; I: intermedio; R: resistente.
AMP: ampicilina; AMS: ampicilina-sulbactam; CF: cefalotina; CIM: rangos de concentración inhibitoria mínima; CTX: cefotaxima; CAZ: ceftazidima; FEP: cefepima; PTZ: piperacilina-tazobactam; IMI: imipenem; MEM: meropenem; AK: amicacina; CN: gentamicina; CIP: ciprofloxacino; FF: nitrofurantoína; SXT: trimetoprima-sulfametoxazol; CL: colistina.
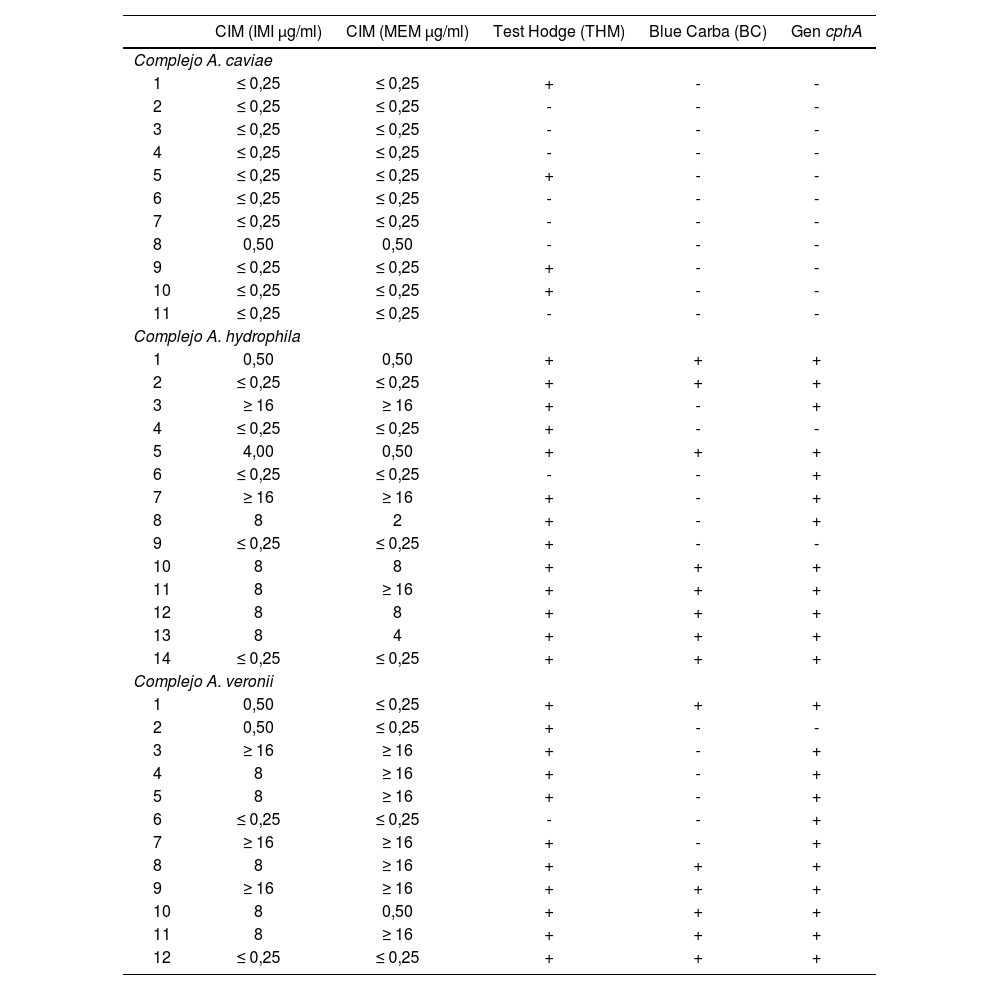
Se detectó el gen cphA en el 85,7% de los aislados asignados al complejo A. hydrophila (12/14); en el 91,7% de los asignados al complejo A. veronii (11/12) y no se lo detectó en ninguno de los pertenecientes al complejo A. caviae (ver la última columna de la tabla 3). En la tabla 3 también se informan los resultados obtenidos mediante los métodos automatizados, que pueden resumirse en los siguientes porcentajes de resistencia a carbapenems: IMI 66,7% y MEM 58,3% en el complejo A. veronii, IMI 57,1% y MEM 50% en el complejo A. hydrophila y 100% de sensibilidad a carbapenems en los miembros del complejo A. caviae. Se realizó el cálculo de la sensibilidad y la especificidad de los métodos fenotípicos para detectar carbapenemasas, tomando como gold standard la determinación de gen cphA (tabla 4).
Detección de carbapenemasas en aislamientos clínicos de Aeromonas spp. por métodos fenotípicos y moleculares
| CIM (IMI μg/ml) | CIM (MEM μg/ml) | Test Hodge (THM) | Blue Carba (BC) | Gen cphA | |
|---|---|---|---|---|---|
| Complejo A. caviae | |||||
| 1 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 2 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| 3 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| 4 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| 5 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 6 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| 7 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| 8 | 0,50 | 0,50 | - | - | - |
| 9 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 10 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 11 | ≤ 0,25 | ≤ 0,25 | - | - | - |
| Complejo A. hydrophila | |||||
| 1 | 0,50 | 0,50 | + | + | + |
| 2 | ≤ 0,25 | ≤ 0,25 | + | + | + |
| 3 | ≥ 16 | ≥ 16 | + | - | + |
| 4 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 5 | 4,00 | 0,50 | + | + | + |
| 6 | ≤ 0,25 | ≤ 0,25 | - | - | + |
| 7 | ≥ 16 | ≥ 16 | + | - | + |
| 8 | 8 | 2 | + | - | + |
| 9 | ≤ 0,25 | ≤ 0,25 | + | - | - |
| 10 | 8 | 8 | + | + | + |
| 11 | 8 | ≥ 16 | + | + | + |
| 12 | 8 | 8 | + | + | + |
| 13 | 8 | 4 | + | + | + |
| 14 | ≤ 0,25 | ≤ 0,25 | + | + | + |
| Complejo A. veronii | |||||
| 1 | 0,50 | ≤ 0,25 | + | + | + |
| 2 | 0,50 | ≤ 0,25 | + | - | - |
| 3 | ≥ 16 | ≥ 16 | + | - | + |
| 4 | 8 | ≥ 16 | + | - | + |
| 5 | 8 | ≥ 16 | + | - | + |
| 6 | ≤ 0,25 | ≤ 0,25 | - | - | + |
| 7 | ≥ 16 | ≥ 16 | + | - | + |
| 8 | 8 | ≥ 16 | + | + | + |
| 9 | ≥ 16 | ≥ 16 | + | + | + |
| 10 | 8 | 0,50 | + | + | + |
| 11 | 8 | ≥ 16 | + | + | + |
| 12 | ≤ 0,25 | ≤ 0,25 | + | + | + |
CIM: rangos de concentración inhibitoria mínima; IMI: imipenem; MEM: meropenem; THM: test Hodge modificado.
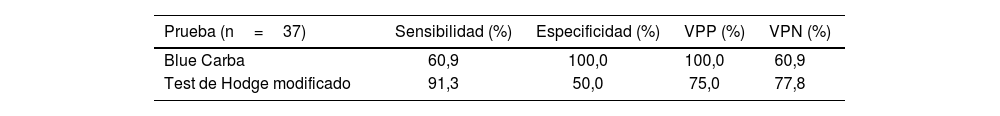
Parámetros de sensibilidad y especificidad de dos pruebas fenotípicas para detectar carbapenemasas en aislamientos clínicos de Aeromonas spp
| Prueba (n=37) | Sensibilidad (%) | Especificidad (%) | VPP (%) | VPN (%) |
|---|---|---|---|---|
| Blue Carba | 60,9 | 100,0 | 100,0 | 60,9 |
| Test de Hodge modificado | 91,3 | 50,0 | 75,0 | 77,8 |
Se estableció como método de referencia la detección del gen cphA, VPP: valor predictivo positivo; VPN: valor predictivo negativo.
Los resultados de sensibilidad a CL por las diferentes metodologías se muestran en la tabla 5, donde podemos observar que por el método automatizado Vitek 2C, el 16,2% (n = 6) de los aislados presentaron resistencia, con CIM superiores a 4μg/ml. Sin embargo, la determinación de la CIM por la técnica de referencia (MDC) reveló un 32,4% (n =12) de aislamientos resistentes, lo que demuestra que los métodos automatizados no son una alternativa confiable para evaluar la sensibilidad a CL (VME: 16,2%). Un método que tuvo una excelente concordancia con el método de referencia fue el método colorimétrico rápido (adaptado de Nordmann et al.22), dado que obtuvimos con aquel una correlación del 100% con el método de referencia (VME: 0%).
Cálculo de errores en la determinación de resistencia a colistina de aislamientos clínicos de Aeromonas spp. (método de referencia: MDC)
| Método evaluado | Resistencia a colistinan (%) | VMEn (%) | MEn (%) | mn (%) |
|---|---|---|---|---|
| Automatizado Vitek 2C | 6 (16,2) | 6 (16,2) | 0,0 | NA |
| TRCc | 12 (32,4) | 0,0 | 0,0 | NA |
TRCc: método rápido de screening colorimétrico; VME: very major errors; ME: major errors; m: minor errors; NA: no aplica, por la categoría del punto de corte; MDC: microdilución en caldo.
La taxonomía del género Aeromonas ha presentado cambios durante los últimos 100 años debido, principalmente, a la implementación de nuevas técnicas moleculares de identificación11,16.
La identificación mediante pruebas bioquímicas tradicionales es compleja a causa del comportamiento variable de cepas de una misma especie ante la misma prueba bioquímica, lo que puede causar confusión en la identificación1. En un trabajo publicado anteriormente3, solo el 35,5% de 90 aislamientos de Aeromonas se identificó correctamente por pruebas bioquímicas, por lo que los autores recomiendan confirmar la identidad mediante métodos moleculares. Sin embargo, en este estudio, una batería de pruebas básicas (TSI, indol, ESC, VP, LDC, ODC, ADH, ARA, SAC, CELB, DNAasa) nos permitió llegar a la identificación a nivel de complejo con una buena correlación respecto del método molecular: del 100%, el 85,7% y el 75% para los complejos A. caviae, A. hydrophila y A. veronii, respectivamente. Además, fue posible identificar todos los aislamientos de A. dhakensis (A. aquariorum) mediante pruebas bioquímicas tradicionales. Esto tiene impacto clínico, dado que la bacteriemia debida a esta especie es la que reviste mayor letalidad, especialmente en pacientes cirróticos o con inmunosupresión29. La espectroscopía de masas MALDI-TOF, en cambio, no logró identificar esta especie al no contar con espectros de A. dhakensis en su base de datos.
Para la identificación molecular de microorganismos se ha utilizado ampliamente el gen ARNr 16S, dado que su distribución es universal16; sin embargo, se ha observado que para la identificación de las distintas especies de Aeromonas, este gen presenta variaciones insuficientes de las secuencias, es decir, presenta una similitud entre especies del 96,7 al 100%. Esta característica, sumado a la presencia de mutaciones en posiciones específicas (heterogeneidad) de la secuencia en una de varias copias del gen, hacen que este no sea adecuado para la identificación de las distintas especies de Aeromonas24. Esto llevó a la utilización de genes internos (housekeeping genes), que presentaron menor grado de similitud entre especies. Dentro de estos genes se incluye la subunidad de ARN polimerasa B (rpoB), la subunidad de ARN polimerasa D (rpoD) y la subunidad B de la ADN girasa (gyrB)11,24. Como demuestran los datos obtenidos por Yáñez et al.33, las secuencias de nucleótidos del gen gyrB mostraron una gran calidad para la diferenciación de las especies de Aeromonas. En el presente estudio, la amplificación y secuenciación de dicho gen logró identificar los 37 aislamientos analizados.
El desarrollo de la tecnología de MALDI-TOF ha permitido la utilización de la espectrometría de masas en la identificación de microorganismos mediante el análisis del perfil proteico, principalmente de proteínas ribosomales, al detectar un espectro de masas específico para cada microorganismo19. Esta tecnología ha revolucionado las técnicas de identificación; sin embargo, presenta dificultades en la identificación de las especies de Aeromonas, como se demuestra en este trabajo, en el que solo se pudo identificar correctamente al 13,5% de los aislamientos a nivel de especie. Estos datos son concordantes con los publicados por Vávrová et al.30, Chen et al.5,6 y Lamy et al.17, quienes concluyen que es necesario actualizar la base de datos Biotyper para obtener mejores resultados en la identificación de las especies de Aeromonas mediante la tecnología de alta precisión de MALDI-TOF MS.
Sensibilidad a los antimicrobianosLa resistencia a los antibióticos β-lactámicos en el género Aeromonas está mediada, principalmente, por β-lactamasas cromosómicas inducibles2. Se han descrito tres clases de β-lactamasas: penicilinasa clase D, cefalosporinasa clase C y metalo- β-lactamasas (MBL) de clase B16. Estas β-lactamasas se han clasificado en cinco patrones principales: 1) cepas del complejo A. hydrophila que expresan β-lactamasas de clase B, C y D; 2) cepas de A. caviae que expresan β-lactamasas de clase C y D; 3) cepas de A. veronii que contienen β-lactamasas de clases B y D; 4) cepas de Aeromonas schubertii que albergan β-lactamasas de clase D; y 5) cepas de Aeromonas trota con β-lactamasas de clase C13,16. Con la excepción de A. trota que es sensible a la AMP13, la presencia de estas β-lactamasas generan la particular resistencia a aminopenicilinas (94,6% AMP), a β-lactámicos con inhibidores, como sulbactam (94,6% AMS) y clavulánico, y a las cefalosporinas de primera generación (67,6% CF).
Los aislamientos de Aeromonas productores de MBL de clase B son extremadamente problemáticos, como mencionan Janda et al.16, debido a la dificultad que conlleva su identificación en el laboratorio mediante los métodos de MDC, la difusión por discos, los métodos epsilométricos (E-test) o los métodos basados en inhibición por ácido etilendiaminotetraacético (EDTA)4. La MBL más ampliamente distribuida en las especies A. hydrophila y A. veronii es la de tipo cphA. En nuestro estudio, en los complejos de A. veronii y A. hydrophila, la presencia del gen cphA fue detectada con una frecuencia del 91,7% y 85,7%, respectivamente. Estos datos se aproximan a los reportados por Ruiz-Castillo et al.27: en esa investigación, el gen cphA resultó positivo en el 77,7% de los aislamientos de A. hydrophila, en el 85,7% de los de A. veronii y en ninguno de los de A. caviae.
La sensibilidad y la especificidad de los métodos fenotípicos para la detección de la carbapenemasa fue del 60,9% y 100%, respectivamente, con BC, y del 91,3% y 50% mediante el test de THM (tabla 4). En el estudio de Wu et al., 32, el THM mostró un buen desempeño en la identificación de esta carbapenemasa; sin embargo, cabe destacar que ese método puede presentar hasta un 47% de falsos positivos, como se puede observar en el trabajo de Ruiz-Castillo et al.27. El test de BC descrito por Pires et al.25 ha mostrado tener una muy buena correlación en la identificación de carbapenemasas tipo A, B y D en especies de Enterobacterales y en Pseudomonas spp. y Acinetobacter spp. En la literatura científica, no se encuentran trabajos en los que se haya utilizado este test para identificar carbapenemasas en especies de Aeromonas; sin embargo, Sinclair et al.29 encontraron una sensibilidad del 97,4% para la identificación de carbapenemasas en Aeromonas spp. empleando el test de Carba NP.
Como se observa en la tabla 3, la resistencia a los carbapenems es variable en los complejos A. veronii y A. hydrophila. La presencia del gen cphA no siempre se traduce en la expresión de resistencia a los carbapenems, pero puede presentar un efecto inducible ante la presencia del carbapenémico; esta carbapenemasa no afecta a penicilinas ni a cefalosporinas, ya que su sustrato específico son los carbapenems13. Este patrón de resistencia no se observa en las carbapenemasas adquiridas de tipo A (Klebsiella pneumoniae carbapenemasa, KPC), de tipo B (Verona integron-encoded metallo-β-lactamase [VIM], imipenemasa [IMP], New Delhi metallo-beta-lactamase [NDM]) o de tipo D (oxacilinasa, OXA-48), que confieren resistencia de alto nivel a carbapenems y a cefalosporinas de tercera o cuarta generación. Es por esta razón que, en el presente estudio, estos tipos de carbapenemasas no fueron investigadas4.
En concordancia con los resultados publicados por Aravena-Román et al.2, la resistencia a antibióticos como CTX, CAZ, FEP, PTZ, fluoroquinolonas, aminoglucósidos, SXT y FF se presentó en menos del 20% de los aislados, por lo que estos agentes podrían evaluarse como terapia de elección en el contexto clínico de cada paciente.
La aparición de cepas de Aeromonas multidrogorresistentes y la presencia de carbapenemasas cromosómicas inducibles ha propiciado el uso de CL como último recurso terapéutico; la resistencia a este antibiótico se presentó en un alto porcentaje en el complejo A. hydrophila, 71,4%; en el complejo A. veronii fue del 16,7% y no se encontraron aislamientos resistentes en el complejo A. caviae. Estos datos son coincidentes con los publicados por Fosse et al.12 y por Aravena-Román et al.2, quienes obtuvieron un alto porcentaje de resistencia en A. hydrophila y A. veronii, tanto en aislamientos clínicos como ambientales, asociado principalmente al uso excesivo del antibiótico en la acuicultura y en salud humana14. En un estudio reciente sobre los mecanismos de resistencia a CL en A. hydrophila, Liu et al.18 llegan a la conclusión de que el sistema de dos componentes EnvZ/OmpR es la forma principal de resistencia a la CL en este microorganismo, lo que genera una resistencia de nivel bajo a medio. A medida que las concentraciones de CL en el medio ambiente aumentan, la resistencia a los antibióticos mediada por este sistema se vuelve insuficiente para asegurar la supervivencia bacteriana. En consecuencia, A. hydrophila ha desarrollado una mutación mlaF que da como resultado una resistencia de alto nivel a la CL18.
La resistencia a CL por mecanismos plasmídicos mediada por el gen mcr ha sido descrita en los tres complejos de Aeromonas14; el presente estudio no realizó la búsqueda de este gen, pero su investigación debería ser tomada en cuenta en futuras investigaciones.
La determinación de resistencia (o sensibilidad) a la CL por métodos como la difusión por discos, el E-test o la CIM por sistemas automatizados ha presentado grandes problemas asociados a resultados de falsa sensibilidad debido a las características químicas del antibiótico. Por este motivo, se han buscado métodos alternativos; el TRCc propuesto por Nordmann et al.22 mostró en nuestro estudio una excelente correlación con el método de MDC, con una concordancia del 100% y un VME del 0%.
ConclusionesResaltamos la importancia de una identificación correcta de Aeromonas spp. para una interpretación adecuada de los mecanismos de resistencia. La secuenciación de gen gyrB permite una identificación correcta a nivel de especie, ya que es un gen con alto poder de resolución; sin embargo, se destaca la utilidad de las pruebas bioquímicas, al alcance de cualquier laboratorio de bacteriología de baja complejidad. Asimismo, podemos concluir que los métodos colorimétricos rápidos BC y TRCc son alternativas excelentes, de simple realización y bajo costo, para detectar carbapenemasas y resistencia a la CL, respectivamente, en el género Aeromonas. No obstante, el bajo número de cepas estudiadas, la evaluación de un solo sistema automatizado y con un único equipo de espectrometría de masas (de los que se encuentran disponibles en el mercado) marcan las limitaciones del presente estudio, un buen motivo para continuar con esta línea de investigación dada la importancia clínica que presenta este microorganismo.
FinanciaciónEl presente trabajo ha sido financiado con fondos del Proyecto UBACYT 2018 Modalidad I: Código 20020170100109BA.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.