Objetivo: El propósito de este estudio fue determinar la presencia de genes de virulencia en cepas de Streptococcus mutans (S. mutans) aisladas desde saliva de individuos de diferentes edades y asociarlas con el índice COPD y ceod según corresponda.
Material y método: A partir de un total de 120 individuos de ambos sexos, se conformaron 4 grupos de 30 personas, que se separaron de acuerdo con los siguientes rangos etarios: 3-5, 6-9, 12-15 y mayores de 18 años. A cada individuo se le determinó el índice COPD y ceod según correspondiera y se realizó recuento salival de S. mutans. La detección de los genes de virulencia: gtfB y spaP se realizó por reacción en cadena de la polimerasa convencional.
Resultados: Se estableció una asociación positiva entre el recuento bacteriano e índice COPD y ceod. El 100% de las cepas aisladas evidenciaron la presencia del gen gtfB y el 63,6% presentaron el gen spaP. No hubo evidencia estadísticamente significativa que relacionará un alto recuento bacteriano e índice COPD y ceod con la mayor presencia de genes que codifican factores de virulencia en cepas de S. mutans.
Aim: The aim of this study was to determine the presence of gtfB and spaP virulence genes in Streptococcus mutans strains isolated from saliva taken from individuals of different ages.
Material and method: A total of 120 individuals of both sexes were studied. They were assigned to one of 4 groups, with 30 individuals in each one, according to age; 3-5, 6-9, 12-15, and older than 18 years old. DMFT (decayed, missing, and filled teeth) and DMFT indexes were determined in each participant, depending on his/her age. S. mutans microbial counts were performed. The gtfB and spaP virulence genes were detected using conventional PCR.
Results: A positive association was found between microbial count and DMFT and DMFT indexes. All the isolated strains demonstrated the presence of gtfB, and 63.6% of the strains had spaP genes. No association was found between high bacterial counts or DMFT and DMFT indexes with the presence of genes that code for virulence factors in S. mutans strains.
Introducción
La caries es una enfermedad oral distribuida universalmente y de carácter multifactorial, siendo Streptococcus mutans (S. mutans) el agente bacteriano más comúnmente aislado. Este microorganismo posee diversos mecanismos virulentos que forman parte del inicio y desarrollo del proceso cariogénico1.
Los factores de virulencia ayudan y protegen al S. mutans de las defensas del hospedero, le permiten mantener su nicho ecológico en la cavidad oral y contribuyen con su capacidad de causar daño. Entre los principales factores de virulencia se encuentran: la habilidad de formar biofilms, la capacidad acidogénica, acidúrica, la síntesis de glucanos y fructanos por medio de las enzimas glucosil y fructosiltransferasas2-6. Streptococcus mutans, entre otras características, puede adherirse a las proteínas salivales, a receptores superficiales de otras bacterias, a la matriz extracelular y a receptores de las células epiteliales. Esta unión es mediada principalmente por proteínas de superficie y glucanos derivados de sacarosa. El gen spaP codifica el antígeno I/II. El gen de esta adhesina fue clonado por Lee et al. (1998), y su producto P1 es un polipéptido involucrado en la adherencia, independiente de sacarosa7. Mutantes deficientes en spaP no se adhieren in vitro y se relacionan con un menor número de lesiones cariosas4,8.
Se han desarrollado diferentes métodos para la detección de cepas cariogénicas de S. mutans. Algunos de ellos estiman el número de unidades formadoras de colonia (UFC/mL de saliva) y de acuerdo con él se establece el riesgo cariogénico9. En general, la identificación de pacientes de alto riesgo cariogénico se lleva a cabo mediante la cuantificación de UFC/mL de saliva de S. mutans en medios de cultivo10. La metodología de reacción en cadena de la polimerasa (PCR) permite la identificación de cepas de S. mutans con un alto grado de especificidad. Los genes gtfB, ftf y gbpB son los más utilizados para la identificación molecular de S. mutans10,11.
La patogenicidad bacteriana se fundamenta en la producción de diferentes tipos de elementos virulentos. En la mayoría de las especies patógenas para el ser humano, los genes de virulencia se agrupan en islas de patogenicidad. La presencia de factores de virulencia es un mecanismo regulado genéticamente12,13. De este modo, se podría argüir que cepas de S. mutans con una mayor dotación de genes que codifican factores de virulencia, se asocian con una mayor capacidad de inducir lesiones cariosas. El propósito del presente estudio es determinar la presencia de genes de virulencia en cepas de S. mutans aisladas desde saliva de individuos de diferentes edades y asociarlas con el índice COPD y ceod, según corresponda.
Material y método
Individuos y muestras
Fueron seleccionados un total de 120 pacientes de ambos sexos que acudieron de forma voluntaria a las clínicas Odontológicas de la Universidad de Talca. Se conformaron 4 grupos de 30 pacientes que se separaron de acuerdo con los siguientes rangos etarios (años): 3-5 (grupo A), 6-8 (grupo B), 12-14 (grupo C) y mayores de 18 años (grupo D).
Se excluyeron los pacientes que recibieron terapia antibiótica hasta 2 meses previos a la toma de la muestra, aquellos que utilizaran aparatos ortodóncicos o los que presentaran alguna patología sistémica.
Tanto el paciente como el adulto responsable en caso de los menores de edad fueron informados acerca del procedimiento que se iba a realizar, por lo que debieron firmar una carta de consentimiento informado, previa a la participación en la investigación.
A cada paciente se le determinó el índice COPD y ceod según correspondiera, y se registró en una ficha de recolección de datos. El procedimiento se realizó bajo los parámetros establecidos por la Organización Mundial de la Salud14, es decir expuesto a una fuente de iluminación adecuada y con las condiciones de asepsia requeridas para este tipo de procedimiento.
Durante la primera entrevista se indicó al paciente y/o a su apoderado que 2 h previas a la toma de muestra salival, no debería comer, lavarse ni enjuagarse los dientes.
La obtención de muestra consistió en la recolección de saliva estimulada mediante la masticación de un trozo de parafina sólida de 0,9 g durante 2 minutos15. La saliva fue vertida en tubos de ensayo estériles que fueron mantenidos a 4 ºC y trasladados al Laboratorio de Investigación Microbiológica de la Facultad de Ciencias de la Salud de la Universidad de Talca.
Cultivo y recuento de colonias
Las muestras de saliva fueron homogeneizadas utilizando un agitador de tubos Maxi Mix II (Thermolyne, Massachusetts, EE. UU.) durante 30 segundos. Se realizaron diluciones seriadas hasta una dilución final de la muestra de 1:1.000, con el propósito de obtener colonias bacterianas aisladas para facilitar el recuento. De esta última dilución se tomaron 50 μL que fueron sembrados en césped en placas de Petri con medio TYCSB.
Las placas fueron colocadas en jarras de anaerobiosis (bioMérieux, Marcy L'Etoile, Francia) con un sobre consumidor de oxígeno Gen box Anaer (bioMérieux) y un marcador de anaerobiosis Anaer Indicador (bioMérieux). Posteriormente, fueron incubadas a 37 ºC durante 48 h en una estufa Pasteur (Memmert, Schwabach, Alemania). Luego, a partir de cada placa fue seleccionada una colonia que fue mantenida a -20 ºC en un medio de guarda para su posterior procesamiento. Por otra parte, en cada placa se realizó un recuento de las colonias de S. mutans cultivadas durante 48 h en el medio TYCSB. Se consideraron para el recuento solo aquellas colonias adherentes, blanco-grisáceas, con superficie rugosa, apariencia de vidrio esmerilado, de consistencia dura, y que no pudieron ser disgregadas cuando se manipulaban con un asa de platino16. La cantidad total de colonias para S. mutans presentes en la placa de Petri se obtuvo utilizando el coeficiente de dilución y fueron expresadas en UFC/mL de saliva. Los recuentos bacterianos fueron expresados de acuerdo con el nivel de riesgo cariogénico considerándose un bajo riesgo recuentos < 105 UFC/mL de S. mutans; riesgo moderado, recuentos entre 105 a 106 UFC/mL, y alto riesgo, recuentos > 106 UFC/mL17.
Detección de los genes por reacción en cadena de la polimerasa convencional
Cultivos de S. mutans en 2 mL de caldo BHI fueron incubados a 37 ºC en ambiente anaeróbico durante 48 h. Para la obtención del ADN genómico, se utilizó el kit AquaPure Genomic DNA isolation (Bio-Rad, California, EE. UU.) y se siguió el protocolo entregado por el fabricante. Los tubos con el ADN se mantuvieron a 4 ºC hasta la realización de la PCR.
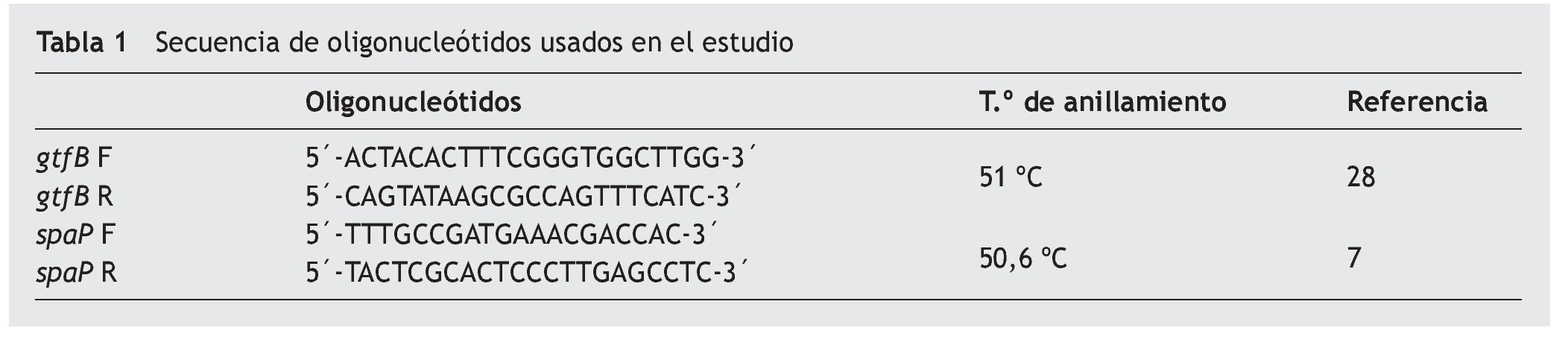
Cada mezcla de PCR consistió en un buffer Tris HCl 10 mM pH 8,3, 1,5 mM de MgCl2, 50 mM KCl, 200 μM de cada deoxinucleótido (Invitrogen, California, EE. UU.), 1 μM de oligonucleótido y 0,2 U/μL de Taq ADN polimerasa (Invitrogen). La PCR fue realizada en 30 ciclos en un termociclador DNAEngine (BioRad, California, EE. UU.), cada ciclo consistió en una denaturación a 94 ºC durante 4 minutos, anillamiento de acuerdo con la temperatura óptima para cada oligonucleótido que se detalla en la tabla 1, extensión a 72 ºC por 2 minutos y una extensión final a 72 ºC durante 5 minutos.
Los productos de la amplificación por PCR fueron analizados por electroforesis en gel de agarosa al 1,5%, teñidos en bromuro de etidio 0,5 μg/mL y fotografiados bajo luz ultravioleta.
Análisis estadístico
Los resultados obtenidos fueron introducidos en una base de datos en Excel y fueron analizados utilizando el software estadístico SYSTAT v15.0. Se realizó un análisis de Chicuadrado. La significancia estadística se considero al 95% de confianza (p < 0,05).
Resultados
Del total de participantes en el estudio, el 55,8% corresponde al género femenino y el 44,2% al masculino.
A partir de 120 muestras de saliva estimulada se obtuvo crecimiento bacteriano para 118 de ellas. Dos muestras no presentaron desarrollo bacteriano y correspondieron al grupo D (mayores de 18 años).
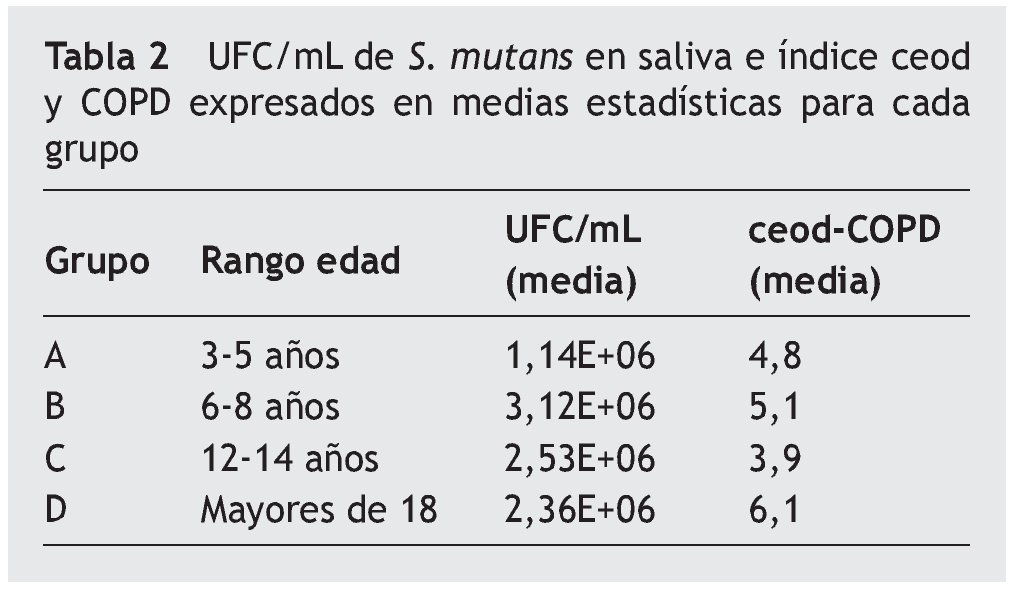
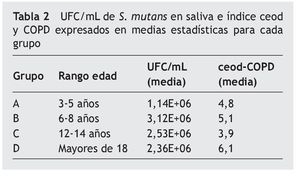
En la tabla 2 se observan las medias para cada grupo en relación con el recuento de UFC/mL de S. mutans en saliva e índice ceod y COPD. La media del las UFC/mL de S. mutans del grupo A presenta una diferencia significativa (p < 0,05) con la media de los otros grupos, siendo el recuento en este grupo menor.
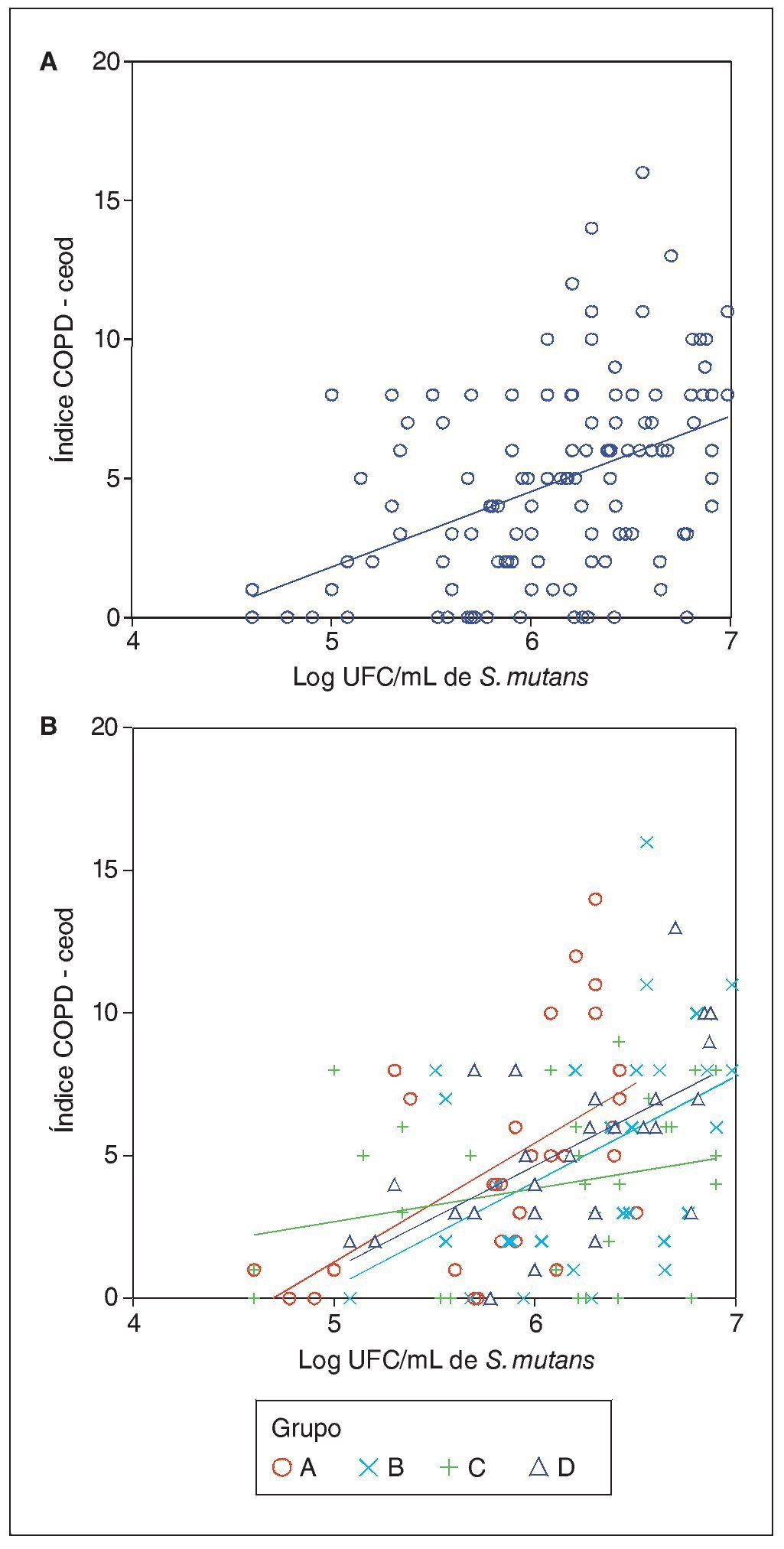
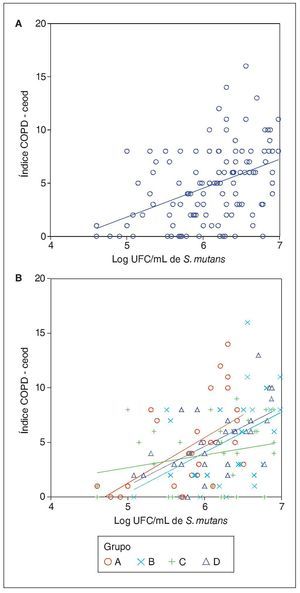
El Log UFC/mL de S. mutans presenta una tendencia positiva con respecto al índice ceod y COPD tanto en el análisis general de los datos (fig. 1A) como para cada grupo etario (fig. 1B).
Figura 1. (A) Tendencia de la relación entre Log UFC/mL de S. mutans e índice ceod y COPD. (B) Tendencia de la relación entre Log UFC/mL de S. mutans e índice ceod y COPD según grupo etario.
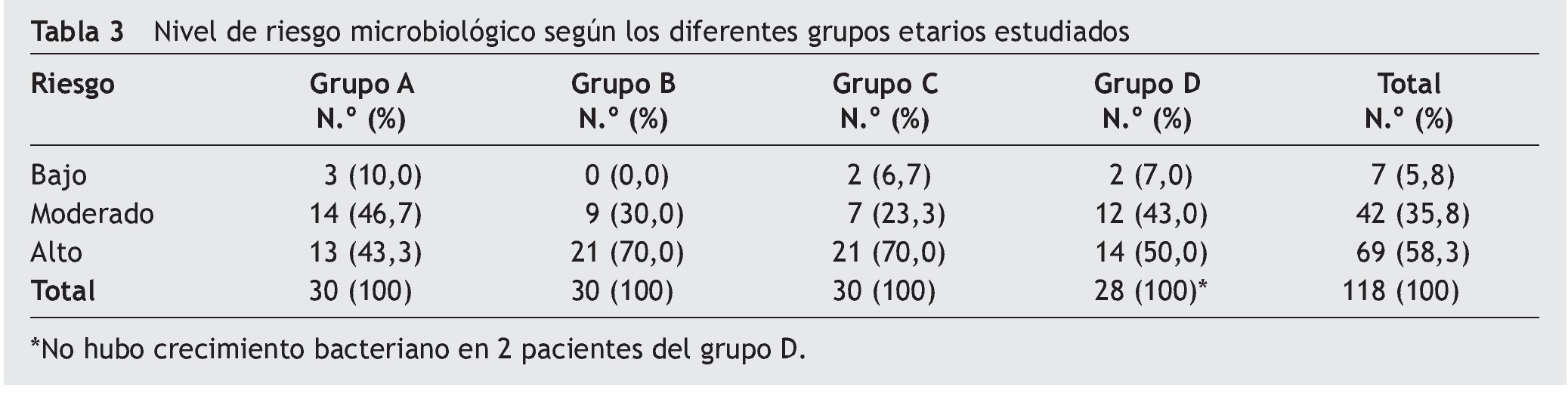
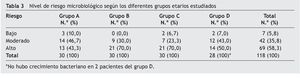
De acuerdo con el valor de las UFC/mL de S. mutans se establece un riesgo microbiológico para cada sujeto. El mayor porcentaje de la población estudiada presenta un riesgo considerado alto (> 106 UFC/mL de S. mutans en saliva), lo que coincide con el análisis realizado por grupo (tabla 3).
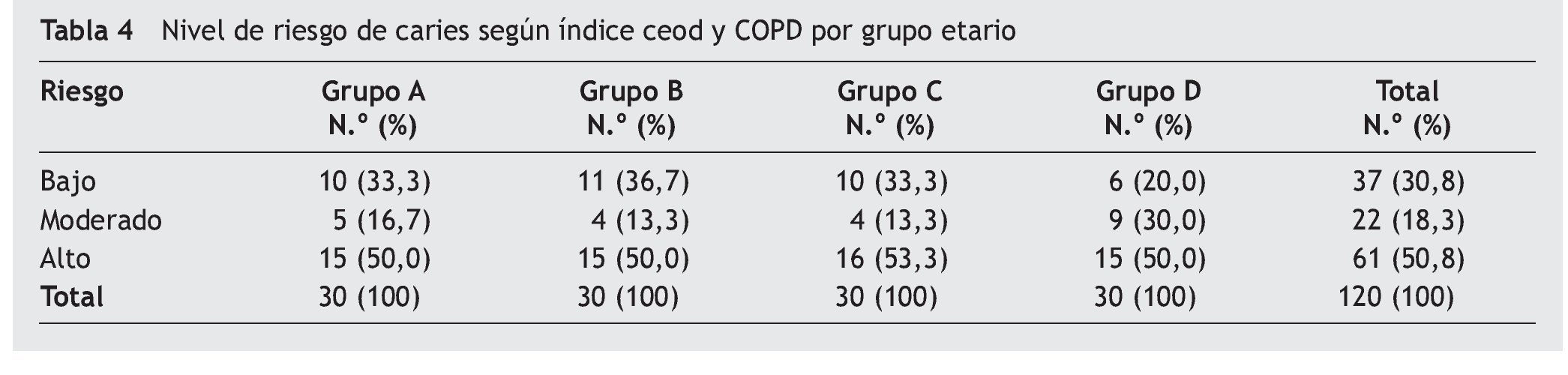
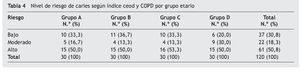
El nivel de riesgo de caries según índice ceod y COPD por grupo etario se presenta en la tabla 4. El 50% de los sujetos de cada grupo presenta un riesgo de caries alto.
En la figura 2A y 2B se observan los productos de PCR para los genes de virulencia gtfB y spaP, respectivamente. Los tamaños esperados para cada producto de PCR fueron de 750 pb para gtfB y 192 pb para spaP. En todos los casos en que se obtuvo amplificación se encontró que estas fueron específicas, mostrando solo una banda del tamaño esperado en cada caso.
Figura 2. (A) Productos de PCR para gen gtfB de S. mutans: carril 1 marcador de peso molecular (1Kb DNA ladder, Invitrogen); carril 17 control negativo; carriles del 2 al 16 bandas de ADN correspondientes al gen gtfB para cepas de S. mutans de los diferentes grupos etarios analizados. (B) Productos de PCR para gen spaP de S. mutans: carril 1 marcador de peso molecular (1Kb ADN ladder, Invitrogen); carril 17 control negativo; carriles 5, 9, 12 y 14 bandas de ADN correspondientes al gen spaP para cepas de S. mutans de los diferentes grupos etarios analizados.
Se determinó que el 100% de las cepas de S. mutans aisladas presentaban únicamente el gen de virulencia gtfB. El gen spaP fue evidenciado en el 63,6% de los individuos de los distintos grupos etarios. En su distribución por grupo etario, alrededor del 80% de las cepas presenta el gen, a excepción del grupo A en el que solo un 20% de las cepas lo presenta (p < 0,05).
Con respecto a la mayor presencia de genes que codifican factores de virulencia y el riesgo microbiológico de los sujetos, no existe asociación estadísticamente significativa entre los diferentes grupos etarios, al igual que con el riesgo de caries.
Discusión
La caries dental es una de las patologías orales más comunes. Presenta una alta incidencia y es la patología bucodentaria de mayor prevalencia en Chile18-20.
Respecto del índice ceod y COPD, los datos del presente estudio revelan que el 86,7% de la población estudiada muestra experiencia de caries con una media en el valor del índice de 4,8. Este último valor no concuerda con los datos entregados por otros autores como Urbina et al. (1997) que señalan en su investigación un índice de 10,0521. En lo que sí existe coincidencia con otros autores es en el alto porcentaje de la población que presenta caries22.
Se observó que en los distintos grupos etarios estudiados a medida que se incrementan las UFC/mL de S. mutans aumenta el valor del índice ceod y COPD. La excepción es el grupo C, que si bien muestra una media de UFC/mL de saliva mayor que los otros grupos, presenta una media del índice ceod y COPD menor que el resto. Lo anterior se podría deber a que los sujetos de 12-14 años se encuentran en una fase de dentición mixta, en la que probablemente piezas dentarias temporales afectadas por caries han sido reemplazadas por piezas sanas, lo que implica subestimar el índice. Por otro lado, las piezas permanentes, al ser mayor en número y tamaño que las temporales que las anteceden, representan una mayor superficie que servirá de hábitat para el establecimiento del S. mutans. La magnitud de la colonización aumenta a medida que se incrementa el número de piezas dentarias en boca, lo que se explica por el hecho de que S. mutans requiere de superficies no descamativas para colonizar23.
Es importante destacar que, de igual forma, en los resultados de la presente investigación hay coincidencia con otros estudios21 que sugieren la existencia de una asociación positiva entre caries y los niveles cuantitativos de S. mutans. Sin embargo, algunos investigadores han cuestionado que los niveles de S. mutans puedan ser usados para categorizar el riesgo de caries22.
De acuerdo con el valor de las UFC/mL de S. mutans se establece un nivel de riesgo microbiológico. El mayor porcentaje de la población estudiada presenta un riesgo considerado alto, lo que coincide con el riesgo de caries en relación con el índice ceod y COPD. Sin embargo, el porcentaje de pacientes con un bajo riesgo microbiológico (5,8%) difiere considerablemente en relación con el grupo de bajo riesgo de caries según el índice ceod y COPD (30,8%). De estos datos se podría inferir que el riesgo establecido por el índice ceod y COPD subestima el riesgo de caries de un individuo considerando que se basa en la historia o experiencia pasada de caries. Probablemente, un paciente de 20 años tenga una mayor experiencia de caries que un pequeño de 3 años. En este contexto, quizás resulte más recomendable utilizar el riesgo microbiológico que cuantifica las UFC/mL de S. mutans en saliva en el momento de ejecutar la toma de muestra, es decir en el minuto en que se está evaluando al paciente y no considerando lo sucedido con anterioridad, que podría no condecirse con lo que se observa clínicamente.
Para lograr establecer un indicador de riesgo potencial para el desarrollo de la caries dental, es necesario dilucidar entre otras cosas el rol que juega el microambiente de la cavidad oral, las estrategias del S. mutans para colonizar superficies y evadir las barreras de defensa del huésped y las relaciones que se establecen entre los diferentes microorganismos que conforman el biofilm dental.
Con respecto a los genes de virulencia estudiados, se evidenció que el gen gtfB se encontraba presente en todas las cepas aisladas3,24.
En la distribución del gen spaP, para cada grupo estudiado, alrededor del 80% de las cepas presentaba este gen a excepción del grupo A que mostró una relación inversa, es decir solo un 20% de las cepas tenía el gen. Lo anterior genera una interesante interrogante respecto de cómo se lleva a cabo la adhesión bacteriana en este grupo etario, pudiéndose argüir probablemente que la adhesión esté mayormente asociada a la síntesis de glucanos y fructanos a partir de dietas ricas en sacarosa. Es conocido que los componentes salivales juegan un rol importante en la adherencia bacteriana y también en la capacidad de los patógenos orales de formar biofilms. Por lo tanto, también se podría pensar que el gen spaP juega un rol importante pero secundario al gen gtfB, y que además el primero podría tener una regulación génica propia de una proteína inducible. De la misma forma, es importante destacar que la expresión o actividad biológica de P1 (Ag I/II) puede estar influenciada por otros múltiples productos genéticos25,26.
Son necesarias estrategias efectivas para prevenir y tratar las enfermedades orales donde intervienen biofilms27. Para lograr lo anterior, hay que considerar la participación de los múltiples factores involucrados en el inicio y evolución de la caries, para, de esa forma, desarrollar alternativas para su prevención y control.
Conflicto de intereses
Los autores declaran que no tienen ningún conflicto de intereses.
Recibido el 20 de agosto de 2013
y aceptado el 24 de marzo de 2014
* Autor para correspondencia.
Correo electrónico:svasquez@utalca.cl (S. Vásquez Ibarra).