La mucolipidosis tipo II alfa/beta, también conocida como I-cell disease (MIM 252500), es una enfermedad metabólica consistente en una alteración en el tráfico de las hidrolasas lisosomales, causada por la actividad deficiente de la N-acetilglucosaminil 1-fosfo (NAcGlc-1-P) transferasa, responsable del paso inicial en la generación del marcador molecular manosa-6-fosfato. NAcGlc-1-P-transferasa es una enzima multimérica compuesta de tres subunidades polipeptídicas codificadas por dos genes diferentes (α, β y γ), el gen que codifica las subunidades α y β (GNPTAB), localizado en el cromosoma 12q23.3, se encuentra alterado en MLII. Hasta la fecha, han sido descritas al menos 20 mutaciones missense/nonsense en GNPTAB relacionadas con esta enfermedad autosómica recesiva. En este estudio, caracterizamos las alteraciones moleculares de un nuevo caso de mucolipidosis II, que presentaba importantes defectos esqueléticos.
Materiales y métodosDeterminación de la actividad de las hidrolasas lisosomales en fibroblastos del paciente, plasma y medio de cultivo de los fibroblastos, mediante los correspondientes sustratos fluorogénicos. Secuenciación de todos los exones y de las regiones intrón-exón del gen GNPTAB, tras la amplificación del DNA genómico del paciente. Además, se analizó el exón 11 de todos los familiares por enzimas de restricción.
ResultadosLa actividad residual hallada de las hidrolasas lisosomales en los fibroblastos del paciente fue de aproximadamente 14% frente a los controles, mientras que la actividad de estas enzimas se multiplicó por 32 en plasma y por 9 en el líquido extra celular de los fibroblastos en cultivo del paciente, con respecto a los valores normales. Se identificaron dos nuevas mutaciones nonsense en GNPTAB asociadas con MLII, c.1383C>A (p.Cys461X) y c.3410T>A (p.Leu1136X), para las que el paciente fue heterocigoto compuesto.
ConclusionesCaracterización de los defectos moleculares de GNPTAB en un nuevo caso de MLII y la identificación de dos mutaciones noveles sin sentido, que facilitarán el diagnóstico prenatal de la enfermedad en la familia del paciente.
Mucolipidosis type II alpha/beta (MLII or I-cell disease) (MIM 252500) is a rare inborn lysosomal hydrolase trafficking disorder caused by the deficient activity of N-acetylglucosaminyl 1-phospho (NAcGlc-1-P) transferase, the enzyme responsible for the initial step in the generation of the mannose 6-phosphate recognition marker. NAcGlc-1-P-transferase is a multimeric enzyme composed of 3 polypeptide subunits (α, β and γ) encoded by 2 different genes. The gene encoding for the α/β subunits (GNPTAB), located on chromosome 12q23.3, is altered in MLII. To date, at least 20 missense/nonsense GNPTAB mutations have been described and incriminated in this autosomal recessive disorder. In this study, we characterized the molecular defect of a new case of MLII, presenting important skeletal abnormalities.
Material and methodsThe activity of lysosomal hydrolases in the patient's fibroblasts, plasma and cell culture medium was determined using appropriate fluorogenic substrates. All exons, as well as exon-intron boundaries, of the GNPTAB gene were sequenced after PCR amplification of the patient's genomic DNA. GNPTAB exon 11 was also studied by enzyme restriction analysis in the whole family.
ResultsIn the patient's fibroblasts, a residual activity of lysosomal hydrolases averaging 14% of control values was found, while a 32 and 9-fold increase in the activity of these enzymes was detected in plasma and the fibroblast culture medium, respectively. Two novel nonsense disease-associated GNPTAB mutations, c.1383C>A (p.Cys461X) and c.3410T>A (p.Leu1136X) were identified, the patient being a compound heterozygote.
ConclusionsCharacterization of the GNPTAB molecular defects in a new case of MLII and the identification of two novel nonsense mutations facilitated the prenatal diagnosis of this disease in the patient's family.
La mucolipidosis tipo II (I-cell disease) (OMIM 252500) es una enfermedad metabólica autosómica recesiva consistente en una alteración en el tráfico de las hidrolasas lisosomales, debido a la actividad deficiente de la enzima N-acetilglucosaminil 1-fosfotransferasa (NAcGlc-1-P-transferasa [IUBMB accession number EC 2.7.8.17]), una proteína multimérica compuesta por tres subunidades polipeptídicas (α, β y γ) responsable del paso inicial en la generación del marcador molecular manosa-6-fosfato1,2. Esta enfermedad se caracteriza por la presencia de numerosos cuerpos de inclusión en diferentes tipos celulares, ausencia de mucopolisacariduria, incremento de la actividad de las enzimas lisosomales en plasma, mientras que estas enzimas son deficientes en los fibroblastos3,4. Entre sus manifestaciones clínicas, destacan importantes alteraciones esqueléticas, retraso en el crecimiento y afectación del músculo cardiaco3,5. La esperanza media de vida de estos pacientes se sitúa entre los 5 y los 8 años de vida6. Hasta la fecha, han sido descritas al menos 20 mutaciones missense/nonsense en GNPTAB3−5,7–11 relacionadas con esta enfermedad autosómica recesiva. Actualmente, como herramienta bioquímica para el diagnóstico se utiliza la determinación de la actividad de múltiples hidrolasas lisosomales en fibroblastos y suero, y a nivel mundial se realiza en muy pocos laboratorios, el análisis mutacional de GNPTAB para el diagnóstico molecular.
En este estudio, presentamos un nuevo caso de MLII en un neonato prematuro con importantes deformidades óseas en extremidades, cuerpos de inclusión en linfocitos y elevaciones de la parathormona, diagnosticado de esta enfermedad por sus manifestaciones clínicas y determinación de la actividad de las hidrolasas lisosomales. Tras el exitus del paciente y el deseo de los padres de un nuevo embarazo, se caracterizaron las alteraciones moleculares en el gen GNPTAB en el paciente y en los padres, con el objetivo de posibilitar en un futuro el diagnóstico prenatal en esta familia.
Material y métodosPaciente a estudioEn el CHU de Poitiers (Francia) se realizó el seguimiento ecográfico del feto de una madre primípara, se detectó exceso de líquido amniótico y longitudes disminuidas en fémur y húmero. El cariotipo realizado al líquido amniótico no presentaba alteraciones (46,XY). En la semana 32 presentó amenaza de parto prematuro, que finalmente tuvo lugar a las 33 semanas y dos días por cesárea. El neonato requirió ventilación mecánica, presentó hipertrofia gingival, extremidades cortas, tórax más pequeño de lo habitual e hipoplasia pulmonar. Se observaron cuerpos de inclusión en los linfocitos del paciente y concentración elevada de parathormona en suero. El paciente falleció con 15 días de vida.
Análisis enzimáticoEn el laboratorio de «Maladies Métaboliques» Institut Fédératif de Biologie, CHU Purpan, Toulouse (Francia), se determinó la actividad de las enzimas de lipidosis (β-hexosaminidasa total, β-hexosaminidasa A, β-hexosaminidasa A [con sustrato específico], β-galactosidasa, β-glucocerebrosidasa, α-galactosidasa A y arilsulfatasa A), enzimas de mucolipidosis y oligosacaridosis (α-manosidasa, α-L-fucosidasa, β-manosidasa) y enzimas de mucopolisacaridosis (β-glucuronidasa y arilsulfatasa B) en leucocitos y fibroblastos en cultivo, y β-hexosaminidasa total, α-manosidasa, β-glucuronidasa en plasma y líquido extracelular de fibroblastos en cultivo del paciente y de un grupo control negativo para MLII. Las actividades enzimáticas se determinaron usando los correspondientes sustratos fluorogénicos o cromogénicos12.
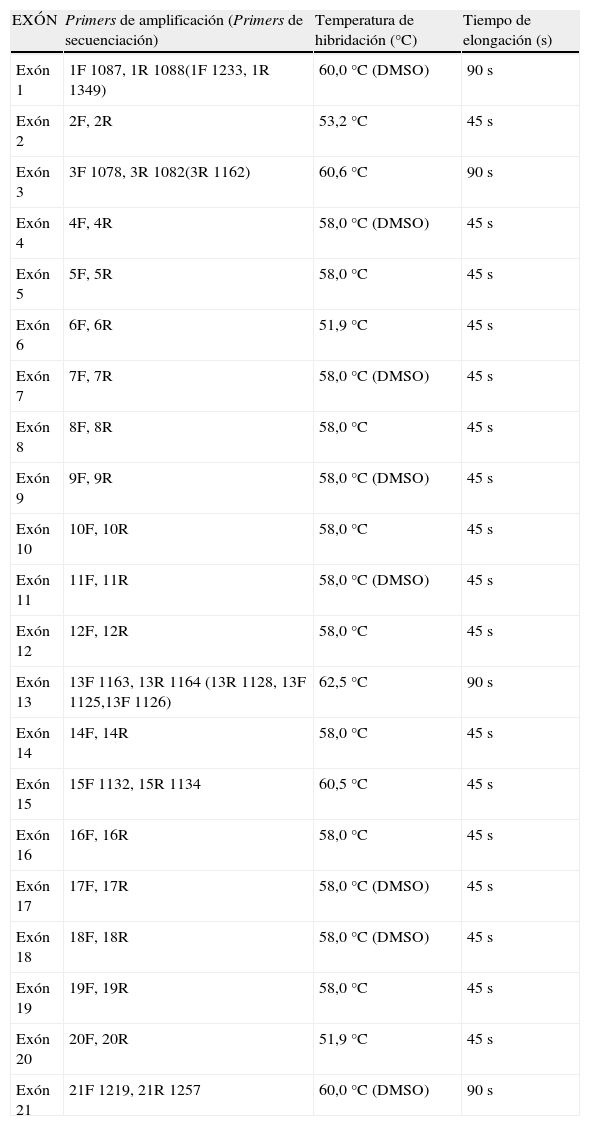
Análisis molecularEn el laboratorio de «Maladies Métaboliques» Institut Fédératif de Biologie, CHU Purpan, Toulouse (Francia), en colaboración con el Servicio de Bioquímica Clínica del HCU Lozano Blesa, Zaragoza (España), tras el exitus del paciente y con el objetivo de poder realizar en el futuro un diagnóstico prenatal en la familia, se llevó a cabo la implantación del análisis molecular de GNPTAB y el estudio de las alteraciones de este gen en el probando y sus progenitores. En el caso del paciente, se aisló DNA genómico de fibroblastos en cultivo (Wizard Genomic DNA Purification Kit, Promega). Se amplificaron individualmente los exones 1 al 21 del gen GNPTAB del paciente, incluyendo las uniones intrón-exón, en ambas direcciones usando los primers descritos en la tabla 11,13,14 y las condiciones especificadas en la tabla 2.
Secuencias de los primers usados.
| Primer | Secuencia del oligonucleótido 5’ 3’ |
| 1F 1087 | 5’ CGT CCG TCG CCG GAG CTG CAA TG 3 |
| 1R 1088 | 5’ GGC AAA ACC CCG TCT CTA ATA ATG 3’ |
| 1F 1233 | 5’ GGC GGT GAA GGG GTG ATG CTG TTC 3’ |
| 1R 1349 | 5’ ACA TAC TGT ATC GGG GCA TC 3’ |
| 2F | 5’ GAA AGT TAT ATA CTC TTA GTC 3’ |
| 2R | 5’ TGC TAA AGT GAA CAC ATC AGA 3’ |
| 3F 1078 | 5’ CATAATCTCTGGGTTTAAACCCTGTG 3’ |
| 3R 1082 | 5’ AACCCTCCCCAGTGCAGTGAAGC 3’ |
| 3R 1162 | 5’ GGGATTACAGGTGTGAGCCACC 3’ |
| 4F | 5’ TAC AGT GGG AGG TAT AGT AGC 3’ |
| 4R | 5’ CTA TGC ACT CAG CAC TGC AAA 3’ |
| 5F | 5’ AGC TTC TTG CTG ATT AAA 3’ |
| 5R | 5’ TCA AAC ATC CAA TGA TAA CAT 3’ |
| 6F | 5’ CAG ACA CCA TAG TTG AGT ATT 3’ |
| 6R | 5’ CTT GAA TCC ACA GTC ATT AAT 3’ |
| 7F | 5’ TCA GCT GTA GGT TTT CAC CAG 3’ |
| 7R | 5’ GTA AGG AGT GAG GCT CTT CTG 3’ |
| 8F | 5’ GGA ATT TAA AGG TGC TTC ATC 3’ |
| 8R | 5’ CTT ACA CAT TTT TAA CAT CTT 3’ |
| 9F | 5’ GAC AAA GAG GAG GAG GTA TGA 3’ |
| 9R | 5’ GAA GGC AAT GAA GAG CTA GAG 3’ |
| 10F | 5’ ACC CAA ACA GCT CTC ATT CA 3’ |
| 10R | 5’ GCT AAG TGA CTT CCA CGC TAG 3’ |
| 11F | 5’ TGT ATC AGA AGC CAG AAG TCA 3’ |
| 11R | 5’ TAG CAC ATG TTC AAT AAT GAT 3’ |
| 12F | 5’ ACT GTC TTT CAA AAT TGT AAT 3’ |
| 12R | 5’ TCT CTC TAC CTG TCA AGG ATG 3’ |
| 13F 1163 | 5’ TGC CTA CTT CAG CAG CAC ATA TAC 3’ |
| 13R 1164 | 5’ CCT GAG CAT GAG AAA GAA TGA GG 3’ |
| 13F 1125 | 5’ CAA GTG GAA AAC CAT CCA CC 3’ |
| 13F 1126 | 5’ TCC AAG TCA GCC TTG CTG AG 3’ |
| 13R 1128 | 5’ CTT CCT GGG CTC TCC TTG TTG 3’ |
| 14F | 5’ GTA CAC TTG TGC ACT AAA CAT 3’ |
| 14R | 5’ GGT AAA TAT GCA CAT TTC AAG 3’ |
| 15F 1132 | 5’ GTT TGC TTG TGT TCA GAA CTA AGG 3’ |
| 15R 1134 | 5’ TGT GAG CCA CTG CGC CTG ACC AG 3’ |
| 16F | 5’ CAC AGT CAT TAC TTA CAA TGC 3’ |
| 16R | 5’ AAA GAC ATA TAA AAC CAT AGA 3’ |
| 17F | 5’ GGA CTA AAT TGC CCT AGT TTC 3’ |
| 17R | 5’ CCA GAC CTT TGT GAT TAC TCT 3’ |
| 18F | 5’ GTG GAT GTT GAG TCC ACT ACG 3’ |
| 18R | 5’ CTA CTC CCT TCT TTC CAG TCC 3’ |
| 19F | 5’ AGG TCA GGA TTA TCC ATA TGT 3’ |
| 19R | 5’ GCT AAG TGA CTT CCA CGC TAG 3’ |
| 20F | 5’ CCA TAT ACA GAA GTA CAT AGT 3’ |
| 20R | 5’ CTA GTA TAC CAA GAA ATA CCA 3’ |
| 21F 1219 | 5’ GGC TCT TAG AAA GTT TGA TGC AC 3’ |
| 21R 1257 | 5’ GCA TCA CAT CAC AAA GAC ATC TC 3’ |
Condiciones de PCR.
| EXÓN | Primers de amplificación (Primers de secuenciación) | Temperatura de hibridación (°C) | Tiempo de elongación (s) |
| Exón 1 | 1F 1087, 1R 1088(1F 1233, 1R 1349) | 60,0°C (DMSO) | 90 s |
| Exón 2 | 2F, 2R | 53,2°C | 45 s |
| Exón 3 | 3F 1078, 3R 1082(3R 1162) | 60,6°C | 90 s |
| Exón 4 | 4F, 4R | 58,0°C (DMSO) | 45 s |
| Exón 5 | 5F, 5R | 58,0°C | 45 s |
| Exón 6 | 6F, 6R | 51,9°C | 45 s |
| Exón 7 | 7F, 7R | 58,0°C (DMSO) | 45 s |
| Exón 8 | 8F, 8R | 58,0°C | 45 s |
| Exón 9 | 9F, 9R | 58,0°C (DMSO) | 45 s |
| Exón 10 | 10F, 10R | 58,0°C | 45 s |
| Exón 11 | 11F, 11R | 58,0°C (DMSO) | 45 s |
| Exón 12 | 12F, 12R | 58,0°C | 45 s |
| Exón 13 | 13F 1163, 13R 1164 (13R 1128, 13F 1125,13F 1126) | 62,5°C | 90 s |
| Exón 14 | 14F, 14R | 58,0°C | 45 s |
| Exón 15 | 15F 1132, 15R 1134 | 60,5°C | 45 s |
| Exón 16 | 16F, 16R | 58,0°C | 45 s |
| Exón 17 | 17F, 17R | 58,0°C (DMSO) | 45 s |
| Exón 18 | 18F, 18R | 58,0°C (DMSO) | 45 s |
| Exón 19 | 19F, 19R | 58,0°C | 45 s |
| Exón 20 | 20F, 20R | 51,9°C | 45 s |
| Exón 21 | 21F 1219, 21R 1257 | 60,0°C (DMSO) | 90 s |
La purificación directa del producto de PCR se realizó con el kit Nucleospin Extract II (Macherey-Nagel, Alemania) para todos los exones, con la excepción del exón 3 que se purificó con QIAquick Gel Extraction kit (250) (Qiagen). El DNA se secuenció usando un secuenciador ABI3100 Applied Biosystems Automatic. En el caso de los padres, se aisló DNA genómico a partir de muestras de sangre EDTA, se amplificaron, purificaron y secuenciaron los exones 11 y 18 del modo anteriormente descrito. Para el análisis por enzimas de restricción del exón 11 del probando y sus padres, los productos de PCR se incubaron con Dde I (Roche Diagnostics, Alemania), y se analizaron en un gel de agarosa al 4%. Para la evaluación de las secuencias se utilizaron los programas Multalin, Bioinformatic Reverse Complement y Chromas.
ResultadosAnálisis enzimáticoEn leucocitos de sangre periférica del paciente se hallaron actividades casi normales frente al grupo control de las enzimas de lipidosis, mucopolisacaridosis, oligosacaridosis y actividades marcadamente elevadas de las hidrolasas lisosomales del probando en plasma (aprox. 3.200%). Se observó una actividad residual (14,7%) de todas las hidrolasas lisosomales (con la excepción de la beta-glucocerebrosidasa, cuyo transporte lisosomal es independiente del sistema de reconocimiento de la manosa-6-fosfato) en muestras de fibroblastos en cultivo, con la consiguiente elevación (951%) de su actividad en el líquido extracelular de fibroblastos. Estas determinaciones bioquímicas, junto con el estudio clínico del paciente, condujeron al diagnóstico de MLII.
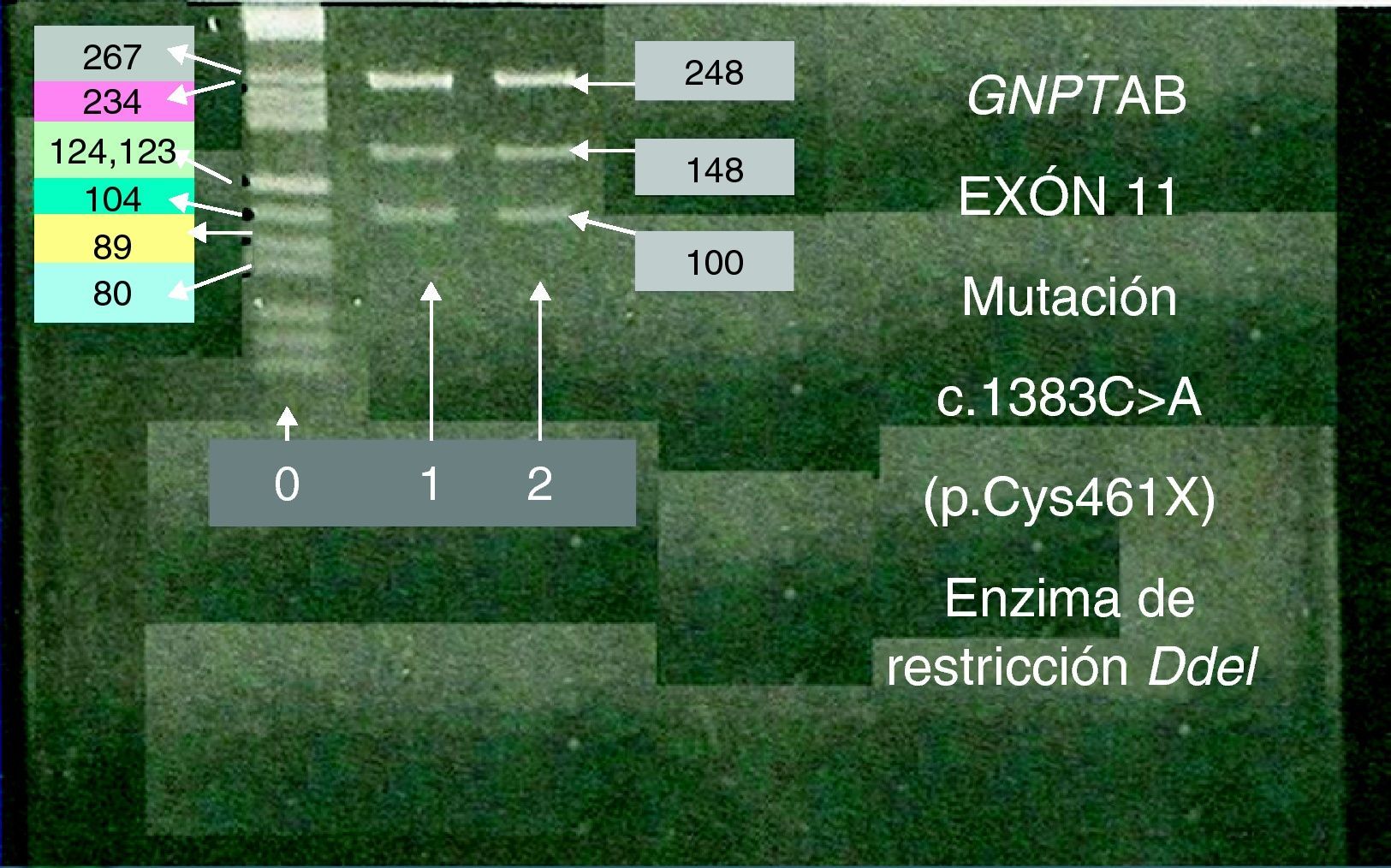
Análisis mutacionalLa secuenciación de los 21 exones y las correspondientes uniones intrón-exón del gen GNPTAB mostraron que el probando era heterocigoto compuesto para las mutaciones noveles: c.1383C>A (p.Cys461X) en el exón 11 y c.3410T>A (p.Leu1136X) en el exón 18. La madre del probando resultó ser heterocigoto para la mutación c.1383C>A (p.Cys461X) del exón 11, sin alteraciones en el exón 18, y el padre heterocigoto para la mutación c.3410T>A (p.Leu1136X) del exón 18, sin alteraciones en el exón 11. El status genético de la familia se confirmó por el enzima de restricción Dde I para la mutación del exón 11 (fig. 1). Además de las mutaciones citadas, el paciente presentaba la sustitución c.1932A>G (p.Thr644Thr) en el exón 13 en estado heterocigoto, y la deleción del-42_-40CGG común en la población control y no asociada con MLII. Ambas alteraciones también se encontraron en el padre en estado heterocigoto. Ni el probando ni sus progenitores presentaron ninguna otra mutación asociada a MLII que hubiera sido descrita con anterioridad.
La mutación c.1383C>A GNPTAB en estado heterocigoto en el probando y su madre.
El análisis por enzima de restricción del exón 11 en el paciente (1) y su madre (2).
Los productos de amplificación del exón 11 (fragmento de 248 pb) se incubaron con el enzima DdeI y fueron analizados por electroforesis en gel de agarosa.
Tras la digestión con DdeI del exón 11 en el probando (1) y su madre (2), se observaron tres fragmentos en ambos casos, fragmento de 248 pb (exón 11), y fragmentos de 148 pb y 100 pb (tras digestión del exón 11 con DdeI).
(0): Marcador molecular.
Hasta hace poco, el diagnóstico de MLII se basaba solamente en la determinación de las actividades de hidrolasas lisosomales en suero, mostrando una marcada elevación; o en muestras de fibroblastos (extraídos de la piel) en cultivo, donde se observaba una actividad deficiente de la mayor parte de estas enzimas. Sin embargo, este método no solo requiere una muestra no siempre disponible, como es el cultivo de fibroblastos extraídos por biopsia cutánea, sino que, además, con este método enzimático no es posible diferenciar entre la mucolipidosis tipo II y la mucolipidosis tipo III; por estos motivos el análisis mutacional de GNPTAB se ha convertido en el «gold standard» para el diagnóstico definitivo de MLII. De ahí la importancia de la implantación en nuestro laboratorio de enfermedades metabólicas, del diagnóstico molecular de MLII, mediante el análisis mutacional de GNPTAB, disponible en muy pocos laboratorios en el mundo.
Se realizó la caracterización molecular de un nuevo caso de MLII con características fenotípicas muy severas, se describieron en el probando dos mutaciones de codón de parada en heterocigosis compuesta, c.1383C>A (p.Cys461X) en el exón 11 y c.3410T>A (p.Leu1136X) en el exón 18, noveles y concordantes con las actividades de hidrolasas lisosomales halladas en el paciente. Puesto que las mutaciones halladas en el paciente son de tipo nonsense cabe esperar que las proteínas sintetizadas a partir de este DNA están truncadas, y que, por otro lado, sea posible que el mRNA correspondiente esté degradado por el proceso de «Nonsense-mediated mRNA Decay», así que por estos motivos cabría esperar un fenotipo de gran severidad, como es el caso del paciente presentado que fallece a los pocos días de vida.
Tras el posterior estudio de los padres, heterocigotos ambos, la madre para la mutación c.1383C>A (p.Cys461X) del exón 11 y el padre para la mutación c.3410T>A (p.Leu1136X) del exón 18, y puesto que ningún miembro de la familia presentó ninguna otra de las mutaciones asociadas a la enfermedad que hubieran sido descritas anteriormente, se infiere la implicación de estas dos nuevas mutaciones en MLII, lo que consideramos relevante en el conocimiento molecular de esta patología, debido al bajo número de mutaciones caracterizadas hasta el momento.
Además, este estudio facilitará el diagnóstico prenatal de MLII en un futuro embarazo de la madre del paciente; lo que consideramos de gran importancia por la gravedad de las manifestaciones clínicas y la corta esperanza de vida en pacientes afectos de esta enfermedad.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Este trabajo corresponde a una comunicación científica presentada y premiada en el III Congreso Nacional del Laboratorio Clínico celebrado en Valencia del 14 al 16 de octubre de 2009.