Las proteasas de serina son enzimas ampliamente distribuidas en la naturaleza, responsables de múltiples e importantes procesos biológicos. Durante las infecciones bacterianas los patógenos secretan y usan sus proteasas de serina como factores de virulencia para combatir contra el huésped, a través de diversos efectos como la desorganización de tejidos, la proteólisis de efectores inmunológicos o la inactivación de componentes relevantes para la fisiología del huésped; sin embargo, desde hace algunos años se ha observado que las proteasas de serina podían modular procesos fisiológicos por un mecanismo altamente específico, a través de la activación de los receptores activados por proteasas. En este artículo resumimos el conocimiento reciente sobre las proteasas de serina bacteriana y su relevancia en la fisiopatología de la infección, y destacamos la oportunidad de nuevas intervenciones antimicrobianas basadas en la inhibición de la interacción receptores activados por proteasas-proteasa.
Serine proteases are enzymes widely distributed in nature, and are responsible for multiple and important biological processes. During bacterial infection, pathogens secrete and use their serine proteases as virulent factors to combat against the host, through diverse mechanisms, such as tissue disruption, proteolysis of immunological effectors or inactivation of relevant components for the host physiology. However, some years ago it was observed that serine proteases could modulate physiological processes by a highly specific mechanism, through the activation of protease activated receptors (PARs). In this paper, we review recent knowledge about bacterial serine proteases and their relevance in the pathophysiology of infection. The opportunity for new antimicrobial interventions based on the inhibition of PAR-protease interaction, is also highlighted.
Las proteasas son enzimas que hidrolizan el enlace peptídico, con una vasta diversidad de implicaciones biológicas, desde la simple degradación de proteínas hasta la regulación de finos procesos fisiológicos1. Dentro de las enzimas proteolíticas las proteasas de serina comprenden más de un tercio de todas las enzimas proteolíticas conocidas, siendo el grupo más extenso2. Las proteasas de serina son enzimas que reciben su nombre basándose en el aminoácido nucleofílico serina (Ser) localizado en su sitio activo, responsable de llevar a cabo la escisión del enlace peptídico, y el cual está acompañado típicamente por los aminoácidos aspartato e histidina (His)2.
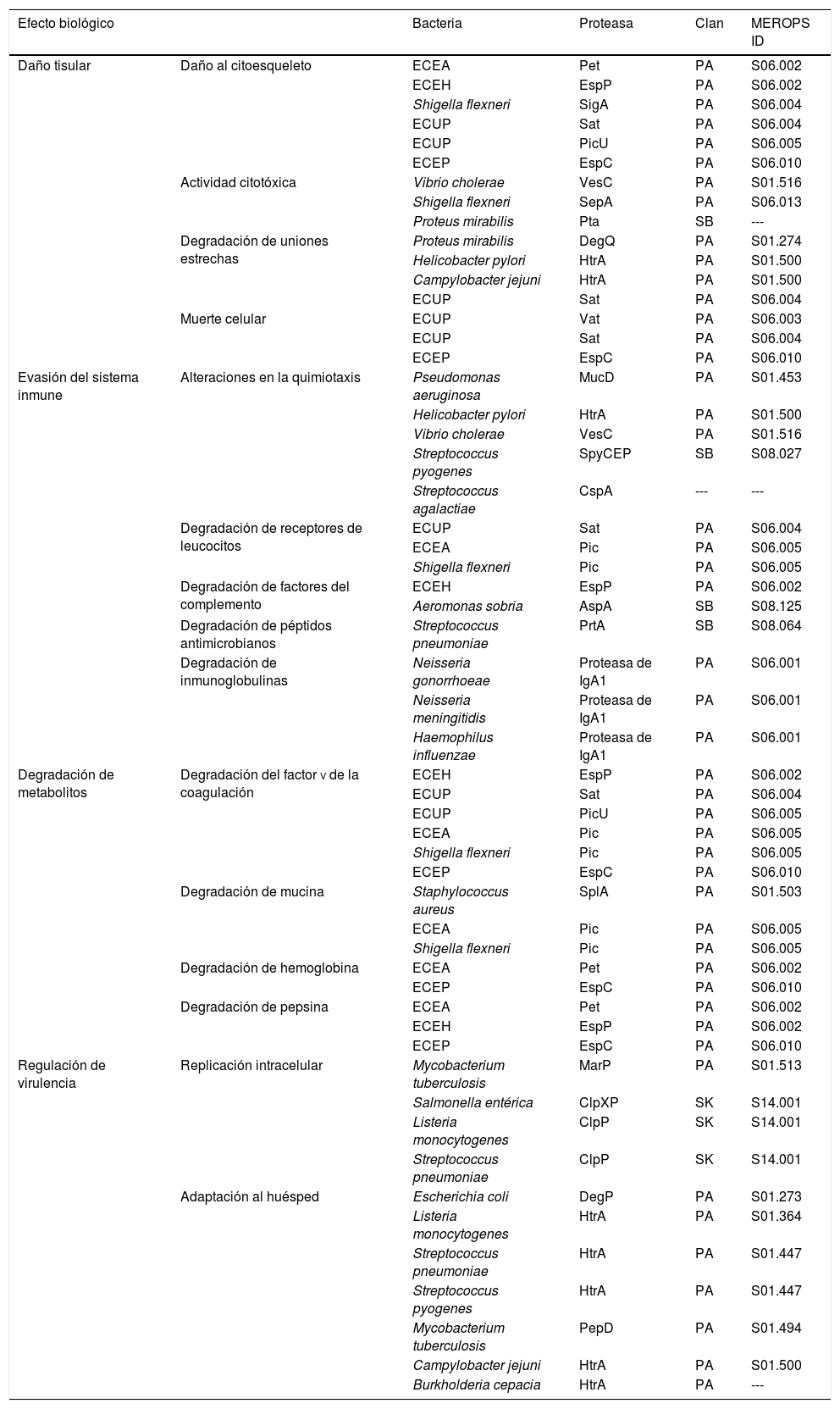
Las proteasas de serina tienen efectos directos e indirectos en la patogénesis del proceso infeccioso3. Lantz et al. en 1997 mencionan que las proteasas son consideradas factores de virulencia cuando estas actúan sobre el tejido del huésped al dañar las células y proteínas del mismo; al inducir la activación de proteasas endógenas del huésped que tienen como repercusión daño al propio organismo, al inactivar distintos mediadores del sistema inmune importantes para la defensa del huésped, y finalmente, por ser proteínas necesarias para la producción de otros factores de virulencia del microorganismo4. En la tabla 1 se resumen las proteasas de serina producidas por algunos microorganismos patógenos de importancia clínica, y en la cual se observa cómo algunas de ellas son conservadas entre diferentes especies, con la particularidad de presentar efectos específicos asociados a su patogénesis.
Proteasas de serina bacterianas con implicaciones en la virulencia
| Efecto biológico | Bacteria | Proteasa | Clan | MEROPS ID | |
|---|---|---|---|---|---|
| Daño tisular | Daño al citoesqueleto | ECEA | Pet | PA | S06.002 |
| ECEH | EspP | PA | S06.002 | ||
| Shigella flexneri | SigA | PA | S06.004 | ||
| ECUP | Sat | PA | S06.004 | ||
| ECUP | PicU | PA | S06.005 | ||
| ECEP | EspC | PA | S06.010 | ||
| Actividad citotóxica | Vibrio cholerae | VesC | PA | S01.516 | |
| Shigella flexneri | SepA | PA | S06.013 | ||
| Proteus mirabilis | Pta | SB | --- | ||
| Degradación de uniones estrechas | Proteus mirabilis | DegQ | PA | S01.274 | |
| Helicobacter pylori | HtrA | PA | S01.500 | ||
| Campylobacter jejuni | HtrA | PA | S01.500 | ||
| ECUP | Sat | PA | S06.004 | ||
| Muerte celular | ECUP | Vat | PA | S06.003 | |
| ECUP | Sat | PA | S06.004 | ||
| ECEP | EspC | PA | S06.010 | ||
| Evasión del sistema inmune | Alteraciones en la quimiotaxis | Pseudomonas aeruginosa | MucD | PA | S01.453 |
| Helicobacter pylori | HtrA | PA | S01.500 | ||
| Vibrio cholerae | VesC | PA | S01.516 | ||
| Streptococcus pyogenes | SpyCEP | SB | S08.027 | ||
| Streptococcus agalactiae | CspA | --- | --- | ||
| Degradación de receptores de leucocitos | ECUP | Sat | PA | S06.004 | |
| ECEA | Pic | PA | S06.005 | ||
| Shigella flexneri | Pic | PA | S06.005 | ||
| Degradación de factores del complemento | ECEH | EspP | PA | S06.002 | |
| Aeromonas sobria | AspA | SB | S08.125 | ||
| Degradación de péptidos antimicrobianos | Streptococcus pneumoniae | PrtA | SB | S08.064 | |
| Degradación de inmunoglobulinas | Neisseria gonorrhoeae | Proteasa de IgA1 | PA | S06.001 | |
| Neisseria meningitidis | Proteasa de IgA1 | PA | S06.001 | ||
| Haemophilus influenzae | Proteasa de IgA1 | PA | S06.001 | ||
| Degradación de metabolitos | Degradación del factor v de la coagulación | ECEH | EspP | PA | S06.002 |
| ECUP | Sat | PA | S06.004 | ||
| ECUP | PicU | PA | S06.005 | ||
| ECEA | Pic | PA | S06.005 | ||
| Shigella flexneri | Pic | PA | S06.005 | ||
| ECEP | EspC | PA | S06.010 | ||
| Degradación de mucina | Staphylococcus aureus | SplA | PA | S01.503 | |
| ECEA | Pic | PA | S06.005 | ||
| Shigella flexneri | Pic | PA | S06.005 | ||
| Degradación de hemoglobina | ECEA | Pet | PA | S06.002 | |
| ECEP | EspC | PA | S06.010 | ||
| Degradación de pepsina | ECEA | Pet | PA | S06.002 | |
| ECEH | EspP | PA | S06.002 | ||
| ECEP | EspC | PA | S06.010 | ||
| Regulación de virulencia | Replicación intracelular | Mycobacterium tuberculosis | MarP | PA | S01.513 |
| Salmonella entérica | ClpXP | SK | S14.001 | ||
| Listeria monocytogenes | ClpP | SK | S14.001 | ||
| Streptococcus pneumoniae | ClpP | SK | S14.001 | ||
| Adaptación al huésped | Escherichia coli | DegP | PA | S01.273 | |
| Listeria monocytogenes | HtrA | PA | S01.364 | ||
| Streptococcus pneumoniae | HtrA | PA | S01.447 | ||
| Streptococcus pyogenes | HtrA | PA | S01.447 | ||
| Mycobacterium tuberculosis | PepD | PA | S01.494 | ||
| Campylobacter jejuni | HtrA | PA | S01.500 | ||
| Burkholderia cepacia | HtrA | PA | --- | ||
Aunque en el pasado se ha planteado la importancia que representan las proteasas de serina en la virulencia de las infecciones bacterianas, desafortunadamente la investigación en este sentido sigue siendo limitada4, por lo que esta revisión tiene como objetivo revisar la actividad de las proteasas de serina bacterianas conocidas y las implicaciones fisiopatológicas que estas representan para el huésped, y a su vez destacar que el conocimiento básico ha servido para idear nuevas estrategias terapéuticas, de manera que se aliente la investigación en este campo.
Clasificación de las proteasasLas enzimas con actividad proteolítica se clasifican en 2 grandes grupos de acuerdo con la posición donde se lleva a cabo la hidrólisis del enlace peptídico en el sustrato, siendo las endoproteasas aquellas que rompen la estructura de las proteínas en los enlaces internos de la secuencia de aminoácidos, mientras que las exoproteasas realizan la hidrólisis en alguno de los extremos de la proteína blanco1.
Otra forma en la que se han catalogado las proteasas se fundamenta en el mecanismo de acción con el que llevan a cabo la escisión del enlace peptídico, distinguiéndose 7 tipos de proteasas, según el residuo de aminoácido catalítico en el sitio de acción, agrupados en 2 tipos de mecanismos: las que activan una molécula de agua responsable de hacer un ataque nucleofílico y poder hidrolizar el enlace peptídico (metalo-, aspartil-, glutamil- y asparagin- proteasa), y en las que el residuo aminoacídico realiza el ataque nucleofílico para unir de manera covalente a la enzima con la proteína sustrato, liberando a su vez la primera fracción de la proteína, y activando por acción del intermediario a una molécula de agua que hidrolice la unión proteasa-sustrato, liberando la segunda fracción (serin-, cisteín y treonin- proteasas).
En 1993 Barrett y Rawlings organizaron y clasificaron todas las proteasas de serina conocidas en una base de datos llamada MEROPS: la base de datos de enzimas proteolíticas, sus sustratos e inhibidores5, en la que estas enzimas se agrupan en clanes de acuerdo con su mecanismo de catálisis y la similitud de su estructura terciaria, y posteriormente en familias, basándose en la similitud de su secuencia primaria. Actualmente en la base de datos MEROPS se encuentran 334.149 secuencias relacionadas con proteasas de serina, de las que existen identificadas 1.703, las cuales son agrupadas en 15 clanes y 53 familias5.
Hablando exclusivamente de proteasas de serina, estas son reconocidas por poseer un residuo de Ser en su sitio activo, además de un aspartato y una His2. De acuerdo con la base de datos MEROPS los clanes PA, SB, SC y SK son las 4 agrupaciones que se categorizan como proteasas de serina al poseer esta tríada catalítica2, sin embargo, esta tríada de aminoácidos puede variar en el sitio activo dependiendo del clan de proteasas de serina del que se trate: los residuos Ser-Lis (lisina) en el clan SE y SM, Ser-Lis/His en SF, His-Ser-His en SH o Ser-His-Glu (glutamina) en SN6. Los clanes SC, PA y SE son los que ocupan los primeros 3 lugares respectivamente en cuanto a la cantidad de secuencias relacionadas, sin embargo son los clanes PA, SC y SB donde existe la mayor cantidad de proteasas de serina identificadas5.
De acuerdo con la base de datos MEROPS en las bacterias existen 194.093 secuencias de proteasas de serina pertenecientes a casi todos los clanes, con ausencia en algunas familias5. Los clanes de proteasas de serina que ocupan los primeros 6 lugares en cuanto a la cantidad de secuencias conocidas de estas enzimas son los clanes SC, SE, SK, SF, PA y SJ respectivamente5, encontrando en estos a algunas de las subfamilias de proteasas de serina, con una alta relevancia en cuanto a su implicación en la patogénesis bacteriana, como las proteasas HtrA (PA), Clp (SK) y Lon (SJ), por mencionar algunas3.
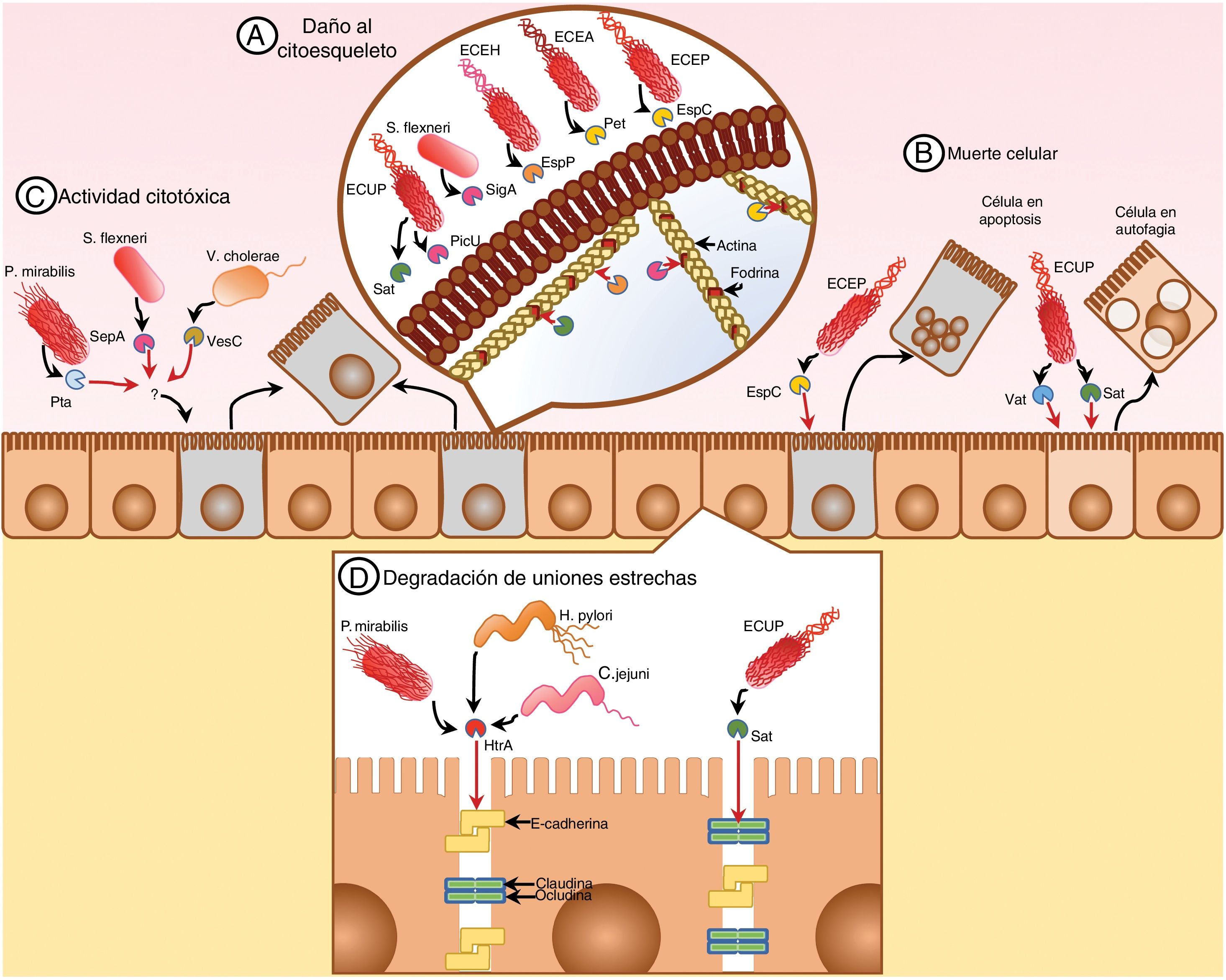
Las proteasas de serina como factores de virulenciaLos microorganismos patógenos tienen la capacidad de generar daño en el huésped de diferentes formas, ya sea alterando la arquitectura del tejido o la fisiología normal del organismo. Dentro de las proteasas de serina de origen bacteriano se ha demostrado cómo algunos patógenos tienen la capacidad de alterar el epitelio, producto de la citotoxicidad dependiente de la acción de sus proteasas (fig. 1A). Las proteasas SepA de Shigella flexneri, VesC de Vibrio cholerae y Pta de Proteus mirabilis son ejemplos de proteasas de serina que han mostrado actividad citotóxica en células epiteliales, en los que si bien se observa un desprendimiento celular, se desconoce el mecanismo de acción o el blanco específico sobre el cual actúan estas proteasas7,8. Por su parte, en Escherichia coli enterohemorrágica (ECEH) se describió cómo la proteasa EspP ocasiona daños al citoesqueleto de actina, mientras que las proteasas Pet, EspC y SigA de los enteropatógenos Escherichia coli enteroagregativa (ECEA), Escherichia coli enteropatógena (ECEP) y S. flexneri, respectivamente, también demostraron la capacidad de alterar el epitelio, evidenciando en estos casos particulares que su blanco se encuentra en la proteína fodrina, la cual mantiene la integridad del citoesqueleto, lo que sugiere la acción de estas enzimas como uno de los mecanismos involucrados en las alteraciones intestinales que se observan en los cuadros diarreicos causados por estos microorganismos, aunque su actividad en modelos in vivo no ha sido demostrada9,10. Este tipo de alteraciones celulares también se ha observado en el epitelio urinario a causa de Escherichia coli uropatógena (ECUP) con la producción de las proteasas Sat y PicU, las cuales comparten la fodrina como sustrato, generando un efecto citotóxico en el epitelio11 (fig. 1B).
Efecto de las proteasas de serina sobre la arquitectura del tejido. Los daños al tejido causado por las proteasas de serina de diferentes patógenos tienen diferentes mecanismos de acción, como los que degradan miembros esenciales del citoesqueleto desregulando la morfología celular como en el caso de ECEP, ECEA, ECEH, ECUP y S. flexneri (A), o inducen muerte celular, tal es el caso de ECEP y ECUP (B). Por otro lado, se encuentran aquellos microorganismos que también inducen un efecto citotóxico en células epiteliales, como en P. mirabilis, S. flexneri y V. cholerae (C). Finalmente, en un grupo de microorganismos se ha encontrado la capacidad de generar desarreglos en el epitelio al degradar las uniones estrechas intercelulares; estos microorganismos son H. pylori, C. jejuni, P. mirabilis y ECUP (D).
Las proteasas de serina también tienen la capacidad de inducir la muerte celular (fig. 1C) o la rotura de las uniones intercelulares en el epitelio (fig. 1D). Diferentes eventos de muerte celular han sido observados en modelos de infección, siendo la muerte celular por necrosis y apoptosis la relacionada con la proteasa EspC de ECEP en células HEp-2 y HT-2912, y la autofagia en el caso de las enzimas Sat y Vat de ECUP en células de epitelio humano de vejiga (CRL-1749) y riñón (CRL-1573)8,13. En ambos casos la evidencia fue producto de ensayos in vitro, por lo que su confirmación en modelos in vivo son necesarios para determinar la relevancia de estas enzimas en el contexto de la infección14. Por su parte, la rotura de las uniones intercelulares es un efecto que se ha evidenciado en distintas líneas celulares por acción de proteasas de serina del tipo HtrA en algunos microorganismos como Helicobacter pylori (MKN-28), Campylobacter jejuni (T84) y P. mirabilis (MKN-28/NCI-N87) al degradar la E-cadherina, mientras que UPEC rompe las uniones estrechas zonula occludens 1 (ZO-1), zonula occludens 3 (ZO-3) y ocludina en monocapas de células Caco-2/TC7. En ambos casos, logrando la invasión del tejido, estableciéndose en el espacio intercelular y permitiendo la replicación de los microorganismos13,15–17.
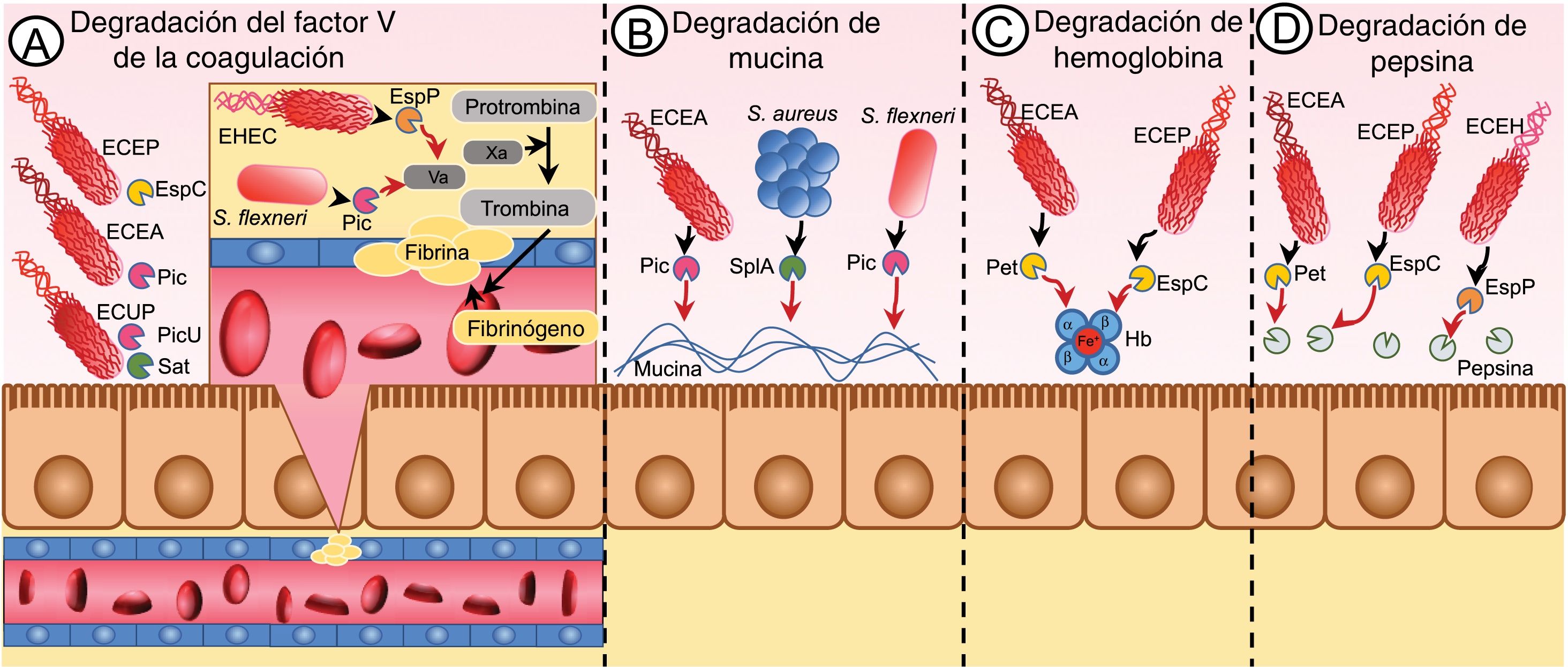
La degradación de múltiples sustratos por la actividad proteolítica de estas enzimas es una posible implicación en la virulencia de los microorganismos productores de proteasas. Uno de los ejemplos de mayor relevancia en este sentido, es la actividad que desempeñan las proteasas EspP de ECEH y Pic de S. flexneri en la alteración de la cascada de la coagulación al degradar al factor V de la misma, lo que se asocia a la presencia de hemorragia en los cuadros diarreicos ocasionados por estos enteropatógenos, sin embargo estas evaluaciones no han sido demostradas en ensayos in vivo8,18. Este efecto de interrumpir el proceso de coagulación dependiente de la actividad de proteasas de serina se ha visto replicado en otros microorganismos como ECEA, ECEP y ECUP gracias a las proteasas Pic, EspC y Sat/PicU, respectivamente (fig. 2A)19,20. Por otro lado, están aquellos microorganismos con la capacidad de establecerse en las mucosas en función de la degradación de la mucina, como hacen ECEA y S. flexneri en la mucosa intestinal por acción de la proteasa Pic o Staphylococcus aureus en el epitelio pulmonar gracias a SplA, como se mostró en un cultivo celular con células CalU-321,22 (fig. 2B).
Las proteasas de serina bacterianas alteran la fisiología normal del organismo degradando sustratos esenciales. Algunos patógenos cuentan con sustratos blanco de sus proteasas de serina, cuya relevancia en el organismo es tan alta que su reducción repercute en el correcto funcionamiento del organismo. Algunos patógenos como ECEH, S. flexneri, ECEP, ECEA y ECUP son conocidos por degradar el factor v de la coagulación, lo cual influye en el cuadro clínico de las infecciones causadas por estos patógenos (A). ECEA, S. flexneri y S. aureus degradan la mucina secretada por el epitelio para favorecer su colonización en el mismo (B). Otras bacterias hacen uso de sus proteasas de serina para obtener nutrientes como en el caso de ECEA y ECEP al degradar la hemoglobina para liberar el hierro y utilizarlo en su metabolismo (C). Finalmente, existen sustratos que son conocidos por ser degradados por las proteasas de serina de algunos microorganismos, pero cuya eliminación no se ha correlacionado con un mecanismo de patogenicidad; sin embargo, su eliminación no debe ser irrelevante; ejemplo de ello es la degradación de la pepsina por ECEA, ECEP y ECEH (D).
Entre otros procesos menos estudiados que alteran la fisiología del organismo y que dependen de proteasas de serina están la degradación de la hemoglobina (fig. 2C) y de la pepsina (fig. 2D) por diferentes proteasas de algunos patotipos de E. coli, entre las que resaltan Pet (ECEA), EspC (ECEP) y EspP (ECEH), sin embargo, estas implicaciones fisiopatológicas son meramente teóricas, dado que no existe evidencia de su relevancia en modelos in vivo8,19,23.
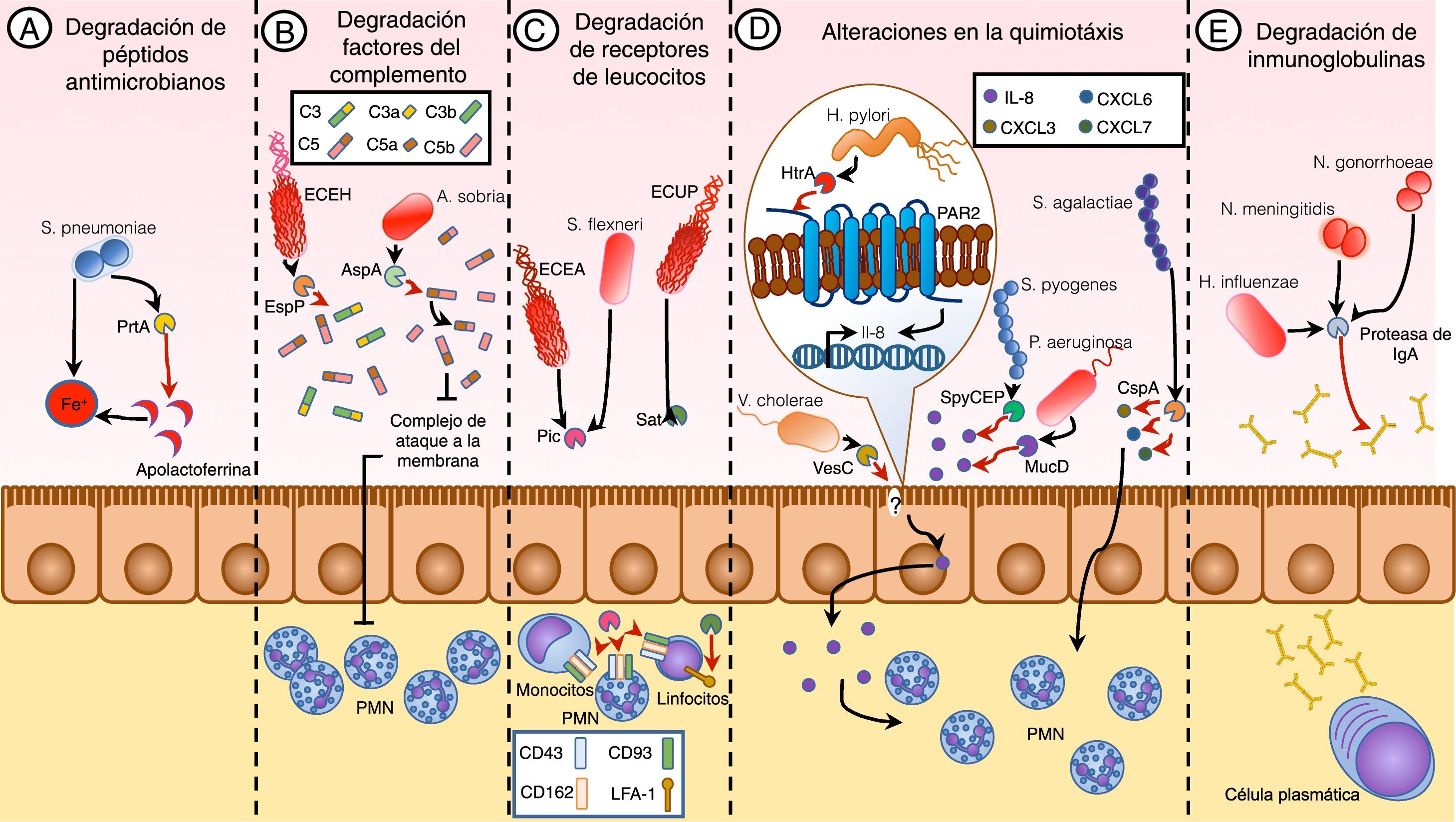
Proteasas de serina en la evasión de la respuesta inmuneLas proteasas de serina son enzimas que contribuyen a la virulencia de algunos patógenos al favorecer la evasión de la respuesta inmune, interfiriendo tanto a niveles de la inmunidad innata, como de la inmunidad adaptativa. Estas alteraciones van desde la degradación de compuestos antimicrobianos, como en el caso de Streptococcus pneumoniae, quien degrada la apolactoferrina por acción de la enzima PrtA24 (fig. 3A), hasta eventos de defensa más sofisticados como la inactivación de la cascada del complemento, en la que la destrucción de las proteínas C3, C3b y C5 por EspP de ECEH, o las alteraciones en C5 por la proteasa ASP de Aeromonas sobria favorecen la sobrevivencia25,26 (fig. 3B).
Las proteasas de serina como mecanismos de evasión inmunológica. Los mecanismos de evasión asociados a las proteasas de serina de patógenos comprenden diferentes vías, como la inhibición de la inmunidad innata al degradar péptidos antimicrobianos, como en el caso de S. pneumoniae (A). Además, se ha visto que la evasión del sistema de complemento puede ser desregulada por las proteasas de serina de algunos patógenos como ECEH y A. sobria (B). Otros patógenos como S. flexneri, ECEA y ECUP alteran la funcionalidad de las células del sistema inmune al degradar algunos receptores de membrana que median su interacción con otras células (C). La alteración de la quimiotaxis de células del sistema inmune, como en el caso de las proteasas de los patógenos H. pylori, V. cholerae, P. aeruginosa, S. pyogenes y S. agalactiae, puede ser estimulada para ser inducida o, por el contrario, la puede inhibir al degradar algunas quimiocinas (D). En el caso de la inmunidad humoral existen reportes que evidencian que H. influenzae, N. meningitidis y N. gonorrhoeae poseen una serín proteasa capaz de degradar los efectos de la IgA secretora (E).
Por otro lado, dentro la inmunidad celular se ha descrito cómo las proteasas de serina de algunos patógenos pueden degradar algunas moléculas de superficie (fig. 3C) o interferir en el reclutamiento de células efectoras (fig. 3D). En el primer caso ECEA y S. flexneri comparten la función de la proteasa Pic en la degradación de los receptores CD43, CD93 y CD162 expresados en linfocitos, neutrófilos, monocitos y otras células del linaje mieloide27, mientras que la proteasa Sat de ECUP degrada la proteína de adhesión celular LFA-1 en linfocitos11,19; en ambos casos esto provoca alteraciones en el proceso de adhesión celular, transmigración a los tejidos o comunicación celular. Por otra parte, la quimiotaxis es un fenómeno que se ha demostrado que puede ser regulado por la actividad de proteasas de serina de algunos microorganismos, induciendo el reclutamiento como lo hacen las proteasas HtrA de H. pylori y VesC de V. cholerae al inducir la producción de interleucina 8 (IL-8)28,29, sin embargo, existen proteasas de serina como SpyCEP de Streptococcus pyogenes, CspA de Streptococcus agalactiae y MucD de Pseudomonas aeruginosa cuya función es la degradación de quimiocinas como CXCL1, CXCL3, CXCL6, CXCL7 y CXCL8 (IL-8) evitando el reclutamiento celular y favoreciendo su sobrevivencia en el tejido30–32.
Los efectos sobre la inmunidad humoral, en los que se ha involucrado a las proteasas de serina de algunos patógenos, incluyen la degradación de los anticuerpos, en los que Neisseria gonorrhoeae, Neisseria meningitidis y Haemophilus influenzae son reconocidos por su actividad proteolítica contra la inmunoglobulina A (IgA) secretora33 (fig. 3E). Algunas de las observaciones que sugieren esta función son el hecho de que los patógenos productores de esta enzima tienen una gran especificidad por colonizar tejidos mucosos, la presencia de esta proteasa en el tejido de individuos infectados o el porqué en el suero existen inhibidores para esta proteasa34, además de saber que los fragmentos de unión al antígeno (Fab) obtenidos de la degradación de IgA puede enmascarar algunos epítopes de microorganismos patógenos, evitando su reconocimiento por el sistema inmune35.
Reconocimiento específico de las proteasas de serina por los receptores activados por proteasasLas proteasas de serina cumplen con funciones de regulación específica más allá de la degradación proteolítica generalizada de algunas proteínas, actuando como iniciadores de vías de señalización al activar proteolíticamente receptores celulares específicos, denominados receptores activados por proteasas (PAR)36.
Los PAR son una subfamilia de 4 miembros de receptores acoplados a proteínas G (RAPG), los cuales se constituyen de 7 dominios transmembranales, unidos por 3 crestas intra y extracelulares, con un dominio carboxilo terminal intracelular y una secuencia amino terminal extracelular37. Estos receptores son diferencialmente expresados en las células del organismo humano, presentando una diversidad de funciones dependientes del receptor, de la célula en la que se expresen o del tipo de estímulo que los active38. Clásicamente se reconoce la trombina como el activador del receptor PAR1, aunque ahora se sabe que junto con el receptor PAR3 y PAR4 también pueden ser activados por la tripsina o la catepsina G, mientras que la proteasa modelo en la activación del PAR2 es la tripsina, aunque también la triptasa y el factor Xa de la coagulación presentan el mismo efecto39.
El mecanismo por el cual ocurre la activación de estos receptores consiste en el reconocimiento de una secuencia específica, localizada en el dominio amino terminal extracelular, la cual una vez ubicada es escindida y expone un nuevo dominio aminoterminal, el cual es reconocido por la segunda cresta extracelular del receptor, iniciando su activación37. Los 4 miembros de la familia PAR cuentan con distintas secuencias de reconocimiento, lo que las hace selectivas para ser activados por ciertos tipos de proteasas de serina. El PAR1, el cual es activado por la trombina, cuenta con la secuencia de reconocimiento R41↓S42FLLRN, la cual al ser hidrolizada expone la secuencia de activación SFLLRN. Por su parte, el PAR2 que es activado por la tripsina cuenta con la secuencia R34↓S35LIGKV. En el caso del PAR3 la trombina realiza el corte proteolítico en la posición K38↓T39FRGAP, y en el PAR4, tanto la trombina como la tripsina entre la R47↓G48YPGQV40.
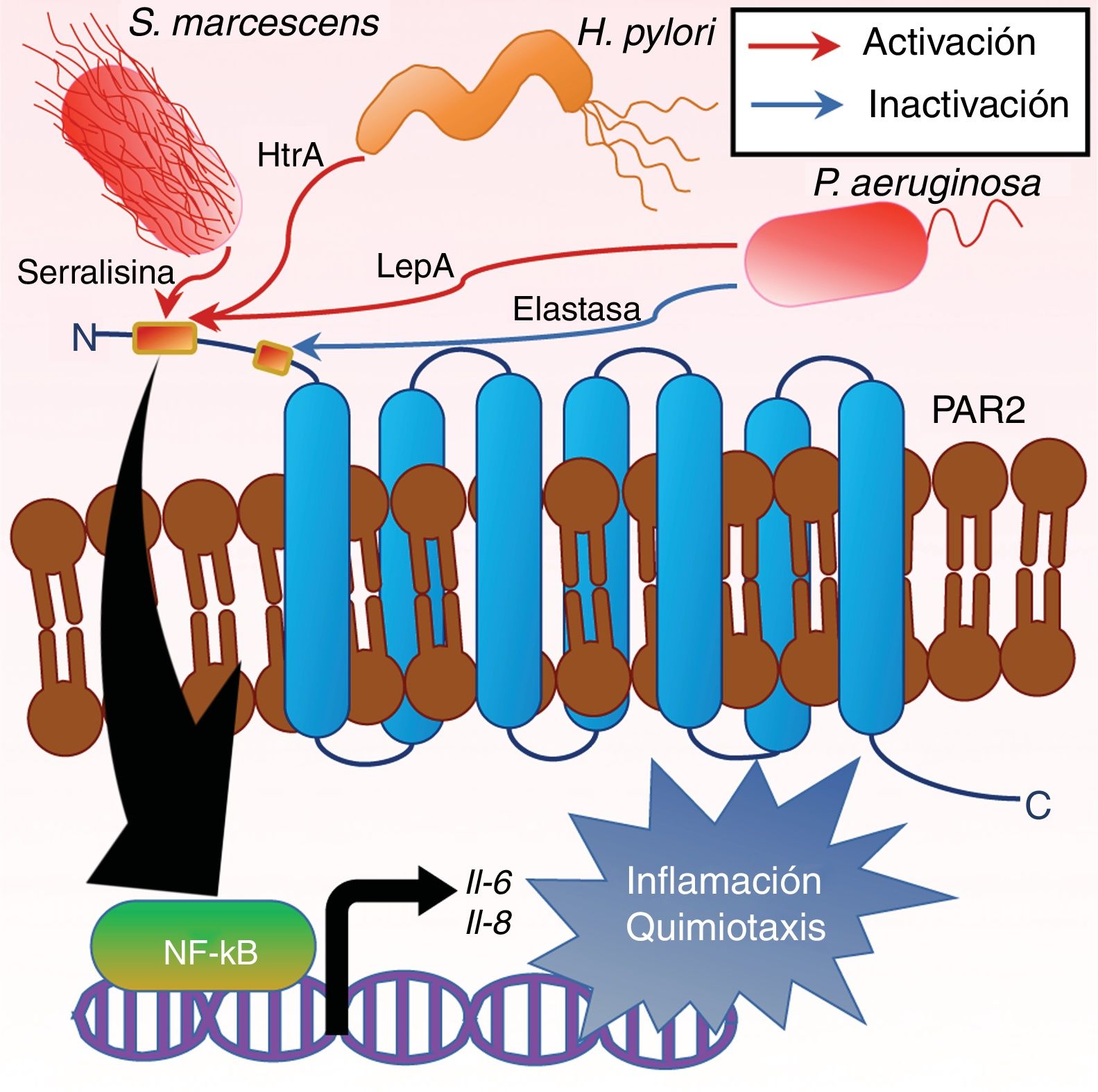
Actualmente se sabe que algunos microorganismos patógenos emplean sus proteasas de serina para la activación de los receptores PAR, generando con ello una serie de eventos con repercusión en el proceso de infección (fig. 4). Dentro de los estudios realizados en bacterias se observa una tendencia en el favorecimiento de la quimiotaxis, al observar por ejemplo a H. pylori cómo induce la expresión de IL-8 en células de la línea MKN4541, cómo Serratia marcescens emplea la serralisina para la expresión de IL-6 e IL-8 en células EBC-142 y finalmente cómo la proteasa de P. aeruginosa LepA también favorece la expresión de la IL-8 en células COS-743. Aunque la activación de estos efectos se observó en 3 distintas líneas celulares, hay que destacar que el PAR2 es el receptor que tienen en común estas 3 proteasas, y que en los 3 casos la inflamación es un común denominador. Sin embargo, no todas las proteasas de patógenos tienen por objetivo la activación de estos receptores, sino todo lo contrario; en P. aeruginosa también se observó cómo una elastasa tenía la capacidad de «desarmar» al receptor PAR2 en células de epitelio pulmonar (A549 y 16HBE), impidiendo su activación, lo que sugiere un mecanismo de evasión a la inmunidad innata en este órgano37.
Proteasas de serina bacterianas activadoras de receptores PAR. Los microorganismos S. marcescens, H. pylori y P. aeruginosa, por acción de las proteasas serralisina, HtrA y LepA, respectivamente, han mostrado la capacidad de activar al receptor PAR2, y con ello inducir la producción de citocinas inflamatorias y quimiotácticas como son la IL-6 y la IL-8. En el caso S. marcescens y P. aeruginosa se sabe que tales eventos tras la activación del receptor PAR2 involucran la activación del factor de transcripción NF-κB, y que en el caso de P. aeruginosa este efecto puede ser dependiente también de la activación de PAR1 y PAR4. Así mismo, en P. aeruginosa la proteasa de serina elastasa es reconocida por «desarmar» al receptor PAR2, realizando un corte proteolítico río abajo de su sitio de activación impidiendo la activación de este.
El empleo de inhibidores de proteasas como tratamiento es una estrategia que ha resultado bastante eficiente en múltiples enfermedades, al evitar un desbalance en la actividad proteolítica de estas enzimas, o simplemente evitando la alteración dependiente de la degradación de un sustrato específico44. Entre los padecimientos que han evidenciado una regulación dependiente de la inhibición de las proteasas involucradas en el proceso están la hipertensión, con la inhibición de la enzima convertidora de angiotensina45, o la infección por el virus de la inmunodeficiencia humana, al inhibir a la proteasa responsable en la activación del virión y, por ende, evitar la formación de nuevas partículas infectivas46.
De entre las proteasas producidas por microorganismos patógenos no todas desempeñan un papel de virulencia en el huésped, sino que algunas actúan al nivel del propio microorganismo para regular la expresión de otros factores de virulencia. Estos mecanismos de regulación, necesarios para permitir las condiciones de adaptación a su huésped, hace que estas proteasas sean consideradas también como factores de virulencia y probables blancos terapéuticos.
Las proteasas HtrA son enzimas del clan PA, que poseen un dominio tipo tripsina y uno o 2 dominios PDZ, asociadas con la sobrevivencia de algunos microorganismos cuando se desarrollan en ambientes hostiles, como temperaturas elevadas y en condiciones de estrés oxidativo47. E. coli es el modelo de estudio de las proteasas HtrA, en donde se ha descrito que los ortólogos periplásmicos DegP, DegQ y DegS48 tienen la función de degradar proteínas mal plegadas que sufrieron daño estructural, producto de las condiciones de estrés celular a las que se halla sometida la bacteria49. Tanto en bacterias gram negativas como en gram positivas se ha demostrado que las proteínas HtrA son importantes factores de virulencia, al encontrar que en ausencia de esta proteasa de serina la sobrevivencia de estos patógenos disminuye, y aunque el mecanismo que esta enzima desempeña en la fisiopatología de la infección a nivel de regulación no está del todo demostrado, se sugiere que emplea un mecanismo homólogo al observado en la enzima DegP de E. coli47. Entre los microorganismos en los que se ha observado una participación importante de esta proteasa en modelos de infección se encuentran C. jejuni, S. pneumoniae, S. pyogenes, Burkholderia cepacia, Listeria monocytogenes y Mycobacterium tuberculosis47. Por otro lado, en H. pylori y C. jejuni las proteasas HtrA han demostrado un efecto fisiopatológico más contundente en el que se observa la capacidad de interrumpir la arquitectura normal del epitelio, desorganizando las uniones estrechas, lo que permite su migración al espacio intercelular y favorece el establecimiento del nicho de infección17,50. Otros microorganismos patógenos en donde también se ha observado la capacidad de degradar las uniones estrechas dependientes de sus proteasas HtrA son ECEP, Salmonella enterica, S. flexneri, P. mirabilis y Yersinia enterocolitica15.
Estas proteasas tipo HtrA han sido consideradas por muchos autores como fuertes candidatos para realizar estrategias terapéuticas alternativas debido a la fuerte asociación que existe entre estas proteasas y la alta virulencia que presentan algunos de estos patógenos47. Hasta la fecha son pocos los reportes en los que se haya empleado el uso de inhibidores de las proteasas HtrA para mitigar la capacidad patogénica de algún microorganismo, siendo algunos de ellos el compuesto HHI (inhibidor de HtrA de Helicobacter) en el que se mostró cómo el bloqueo de la proteasa impedía la degradación de la molécula E-cadherina en un modelo de cultivo celular de epitelio gástrico MKN-2817. Otros compuestos son la clorometil cetona sintetizada para la inhibición de HtrA en E. coli51 o el tripéptido Val-Pro-Val (valina-prolina-valina) que bloquea el sitio activo en la HtrA de Chlamydia pneumoniae, impidiendo que esta última complete su ciclo de vida52.
Otra familia de proteasas vinculadas a la virulencia de bacterias patógenas durante el periodo de infección son las proteasas de serina Clp. Estas proteasas son igualmente reguladoras de estrés, degradando proteínas que han sido mal plegadas en consecuencia al choque térmico, estrés oxidativo o privación de nutrientes53. La actividad de proteasa de las enzimas Clp funciona en forma de complejo por la subunidad ClpP, la cual cuenta con la actividad de serín peptidasa, y con una ATPasa Clp la cual cuenta con la función de reconocer las proteínas destinadas a su degradación y transferirlas al dominio ClpP para su degradación54. En microorganismos como L. monocytogenes, S. aureus, S. pneumoniae, Bacillus anthracis y Salmonella enterica se ha descrito cómo las proteasas de serina Clp desempeñan un importante papel en su virulencia3. En L. monocytogenes y S. pneumoniae se mostró que al inhibir la acción de las proteasas ClpXP estos microorganismos presentaban una mayor susceptibilidad a la fagocitosis por macrófagos murinos derivados de médula ósea y de la línea celular RAW264.7, respectivamente, probablemente debido a su incapacidad de tolerar las condiciones intracelulares55,56. Por su parte, en S. aureus la implicación de las proteasas ClpXP está relacionada con la regulación en la expresión de otros factores de virulencia como hemolisinas, adhesinas de superficie y otras proteasas57, mientras que en B. anthracis la ATPasa ClpX está vinculada con la esporulación, la germinación y la resistencia al estrés58.
Al igual que las proteasas HtrA, la interrupción de la activad de las proteasas de la familia Clp también ha mostrado resultados favorables como una estrategia para la eliminación de microorganismos patógenos59. El inhibidor de proteasas de serina F2 mostró tener un efecto inhibitorio sobre las proteasas ClpXP de S. aureus y B. anthracis, mejorando además la susceptibilidad a antibióticos y al péptido antimicrobiano LL-37, con lo que se propone que este inhibidor usado complementariamente con el tratamiento adecuado puede funcionar sinérgicamente en una terapia antimicrobiana53. Por otra parte, están aquellos compuestos que tienen per se un efecto antimicrobiano, pero en los cuales su mecanismo de acción consiste en la inhibición de las proteasas de la familia Clp. Entre estos compuestos se encuentran los acil-depsipéptidos, los cuales fueron aislados de Streptomyces hawaiiensis y se sabe que actúan específicamente al inhibir la subunidad de peptidasa ClpP; estos compuestos mostraron un efecto bactericida en contra de S. aureus, Enterococcus faecalis y S. pneumoniae con una elevada tasa de sobrevivencia en modelos animales60.
En conjunto, esta información sugiere que la inhibición de la actividad de las proteasas de serina, la cual representa una participación vital en la fisiología y la fisiopatología de diferentes microorganismos patógenos, representa una serie de fuertes candidatos para la elaboración de nuevas terapias antimicrobianas.
ConclusiónLas proteasas de serina son reconocidas como el grupo de enzimas con actividad proteolítica más extendido en la naturaleza, al igual que en las bacterias1. Estas proteasas han representado una estrategia para las bacterias patógenas, permitiéndoles realizar procesos de colonización, invasión, adaptación a su nicho de colonización, favoreciendo la producción de otros factores de virulencia e inclusive lograr la evasión del sistema inmune, siendo todo esto lo que actualmente las cataloga como factores de virulencia en la patogénesis bacteriana3, sin embargo, el entendimiento sobre cómo estos determinantes en la virulencia llevan a cabo sus mecanismos a un nivel más específico, como en la activación de los PAR, es algo que permanece sin descripción actualmente para la mayoría de estas enzimas4.
El empleo de inhibidores específicos de proteasas como tratamiento es una estrategia que ha resultado bastante eficiente en múltiples enfermedades, al evitar un desbalance en la actividad proteolítica de estas enzimas o simplemente evitando la alteración dependiente de la degradación de un sustrato específico. Aunque la propuesta de emplear inhibidores de proteasas es una estrategia que se ha venido proponiendo desde hace algunos años, el utilizar a las proteasas de serina como blanco terapéutico en las enfermedades infecciosas es un campo que empieza a investigarse; sin embargo, para poder hacer de esta estrategia terapéutica una herramienta efectiva se necesita enriquecer el entendimiento sobre qué proteasas de origen bacteriano ejercen un papel determinante en la virulencia y cuáles son los mecanismos de acción que estas enzimas emplean en la patogénesis.
FinanciaciónEl presente trabajo forma parte de un Proyecto Apoyado por el Fondo Sectorial de Investigación para la Educación, financiado por el Consejo Nacional de Ciencia y Tecnología (CONACyT) y la Secretaría de Educación Pública (SEP), a través del Proyecto aprobado a JPPN (No. 238125). Gerardo García González es beneficiario del Programa de Becas Nacionales CONACyT (CVU 708895).
Conflicto de interesesLos autores manifiestan no tener conflicto de intereres que señalar.