El espermiograma tiene una capacidad diagnóstica limitada a la hora de predecir una gestación en las técnicas de reproducción asistida. Parámetros moleculares como los ARN mensajeros presentes en el esperma no se evalúan. Nuestro grupo ha realizado listados de genes diferencialmente expresados (GDE) de muestras de semen que lograban un embarazo (E) frente aquellas que no (NE), en pacientes que se sometían a ciclos de IAH o de ICSI. El objetivo era caracterizar los perfiles de expresión, usando la tecnología del microarray, de muestras de semen que logran o no embarazo en ciclos de IAH o ICSI.
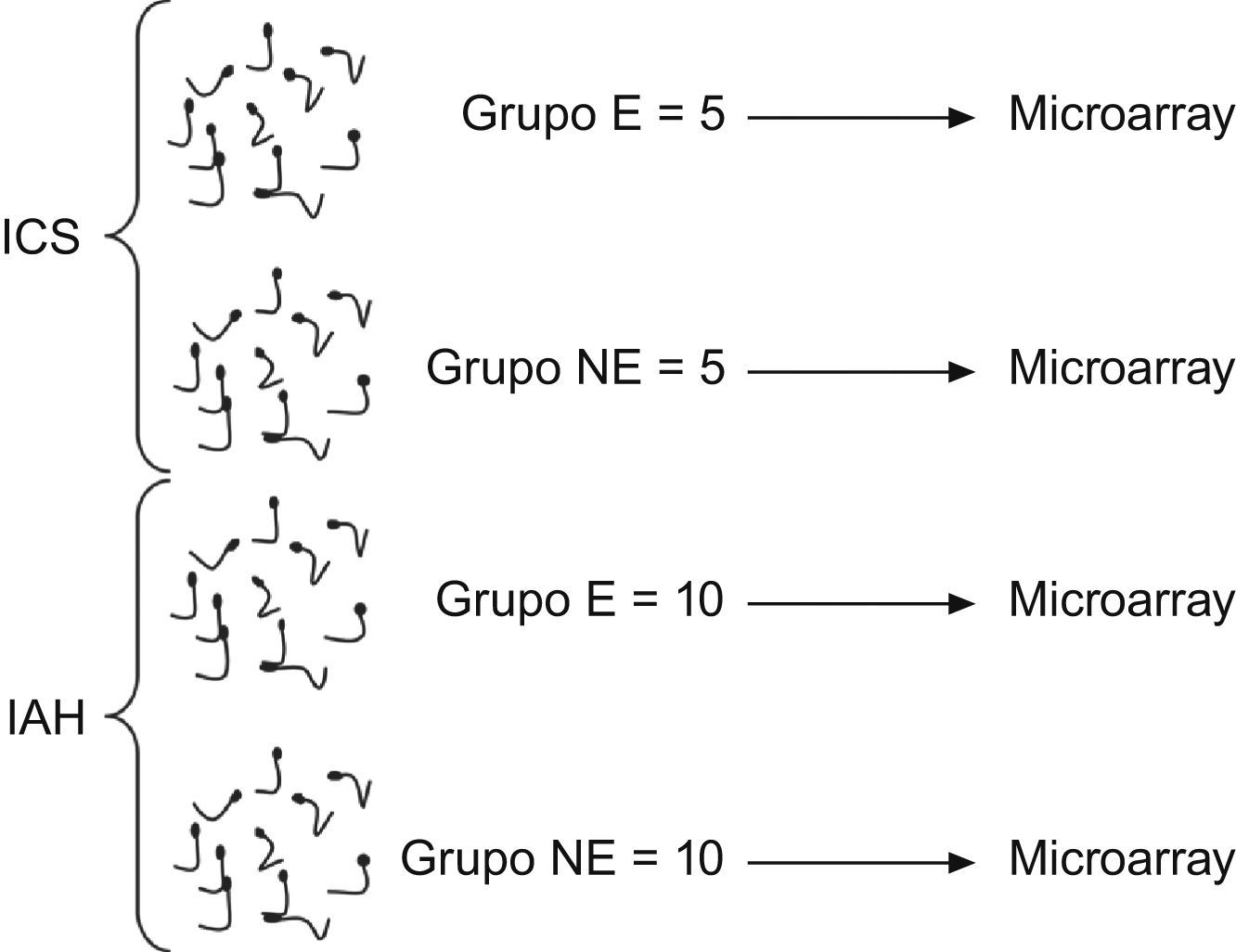
Material y métodos30 muestras de semen (10 lograron embarazo [E=10] y 10 no [NE=10]) se obtenían de pacientes que se sometían a un ciclo de IAH y 10 muestras de semen (E=5, NE=5) que se sometían a uno de ICSI con ovocitos de donantes. Finalmente se realizaron 4 microarrays con las muestras de semen que lograban embarazo frente aquellas que no para ambas técnicas evaluándose los GDE que había entre los grupos E y NE tanto en IAH como en ICSI.
ResultadosEl número total de genes (NTG) detectados en el microarray de IAH fue de 19.938 y 19.229 en ICSI. El número total de GDE fue de 950 en IAH y 49 en ICSI.
ConclusionesLos datos revelan que los factores moleculares requeridos para lograr embarazo son diferentes según la técnica utilizada.
Estas diferencias pueden ser potencialmente empleadas como marcadores de éxito en ambas técnicas y como futura herramienta terapéutica.
Basic sperm analysis has insufficient predictive power on pregnancy achievement in assisted reproductive techniques (ARTs). Molecular parameters such as as mRNA present within spermatozoa are not assessed. Our group listed differentially expressed genes (DEG) from sperm samples (SS) from that achieve pregnancy (group P) vs. those that do not (group NP) using microarray technology in couples undergoing homologous IUI and ICSI. The aim is to use microarray technology to characterise the different gene expression profiles (EP) between SS that achieve pregnancy or not in ICSI and IUI cycles.
Material and methodsTen SS (5 that achieved and 5 that did not achieve pregnancy) were obtained from IP undergoing ICSI and 20 (10 that achieved and 10 did not achieve pregnancy) IAH cycles with oocytes from young donors and their healthy female partners respectively. After freezing aliquots of the SS employed for both treatments and their respective mRNA expression profiles were compared. Finally 4 microarrays were performed in duplicate with sperm samples from group P vs. NP from both techniques and those DEG were evaluated at least twice, with statistically significant differences between P and NP SS for each group.
ResultsTotal number of genes (TNG) in IUI microarray was 19938 and 19229 in ICSI. The total number of DEG (950) in the IAH group was much greater than in the ICSI group (49).
ConclusionsThese data reveal that molecular features required for pregnancy are different in IUI and ICSI procedures.
These differences could be potentially employed to detect ICSI and IUI success markers or to improve pregnancy rates in these procedures.
La inseminación intrauterina homóloga (IAH) es la primera técnica de reproducción asistida (TRA) que se emplea con parejas que no logran embarazo tras un mínimo de un año manteniendo relaciones sexuales sin protección y con una anamnesis previa que no desvela patología alguna en cuanto a problemas de fertilidad1. La IAH consiste en el depósito de forma artificial de una preparación de semen tras un proceso de selección espermática, en el tracto reproductor femenino, siendo más frecuente su realización en el interior de la cavidad uterina. De esta manera se intenta acortar la distancia que separa óvulo y espermatozoide y facilitar su encuentro2.
Para que la inseminación artificial tenga éxito es imprescindible que al menos una de las trompas de Falopio sea permeable. Además, el semen del varón debe cumplir unos parámetros seminales mínimos2.
Normalmente la tasa acumulada de embarazo tras 4 ciclos de IAH es de un 20–40%. Un importante porcentaje de las parejas (30–40%), no logran un embarazo tras varios ciclos de IAH, incluso en esos casos en los que ningún miembro de la pareja presenta problemas para concebir2.
La inyección intracitoplasmática del espermatozoide (ICSI) es una técnica que se utiliza cuando los parámetros seminales son muy patológicos. Consiste en microinyectar un espermatozoide móvil y morfológicamente normal en un ovocito que previamente ha sido extraído del ovario de la mujer3. Tras la microinyección y después de haberse comprobado que ha habido fecundación y que las primeras divisiones embrionarias han sido correctas, se transfiere al útero de la paciente dentro los siguientes 3–6 días tras la microinyección4.
Esta TRA se prescribe en casos (entre otros) de oligoastenoteratozoospermia, fallo repetido de fecundación tras la in vitro convencional, cuando el semen presenta grandes cantidades de anticuerpos antiespermatozoide, en pacientes de cáncer que crioconservan sus muestras de semen previamente a un tratamiento oncológico, pacientes con daños en la médula ósea, aquellos hombres con problemas de eyaculación o eyaculación retrógrada o pacientes que conservaron muestras de semen antes de realizarse una vasectomía5–11. También puede ser usado en casos de diagnóstico genético preimplantacional para enfermedades monogénicas12 o en hombres con azospermia por ausencia bilateral congénita de vasos deferentes o por obstrucción inguinal de los conductos eyaculadores13. Aunque el ICSI se emplea en casos con factor masculino severo, también existen indicaciones femeninas como anormalidades en la zona pelúcida14.
Cuando una pareja acude a una clínica de reproducción asistida, a cada uno de los miembros se le somete a diferentes exámenes médicos.
La evaluación del potencial fértil de un varón suele basarse en una breve anamnesis y un espermiograma, y si no hay nada realmente patológico no se someten a más pruebas. El espermiograma consiste en un recuento de espermatozoides y valoración de su morfología y movimiento, siguiendo los criterios de la Organización Mundial de la Salud (OMS) establecidos en 199915.
Sin embargo, la experiencia diaria de la clínica nos muestra que hombres sanos con parámetros seminales dentro de la normalidad según la clasificación de la OMS15 son incapaces de dejar embarazadas a sus parejas, no presentando estas ningún tipo de patología (permeabilidad de las trompas, no endometriosis ni ovario poliquístico y niveles hormonales normales, por ejemplo) tras someterse a una IAH.
En el caso del ICSI, las tasas de fecundación, a pesar de tratarse de muestras de semen muy patológicas, alcanzan un 70–80%4, sin embargo y teniendo en cuenta que con esta técnica estamos forzando a entrar al espermatozoide en el ovocito y que a pesar de eso, entre un 3–5% de los ciclos de ICSI acaban en fallo de fecundación, podemos creer que, al igual que en las IAH hay factores moleculares que influyen en el potencial fértil.
Nuestro grupo planteó una hipótesis por la cual se suponen diferencias a nivel molecular (contenido de ARN mensajeros o perfil de expresión génico) entre las muestras de semen que consiguen un embarazo frente aquellas y que el espermiograma es incapaz de detectar. También planteábamos que los perfiles de expresión génico no solo diferirán entre aquellas muestras que logren embarazo o no, si no que esos perfiles, a su vez también serán diferentes dependiendo del tratamiento de reproducción asistida estemos estudiando, pues suponemos que según los requerimientos moleculares de las muestras, estas serán más aptas para una técnica u otra.
En la última década, conocida como la era posgenómica, las herramientas de análisis genético han sufrido una revolución técnica. La entrada en escena de sistemas que permiten el análisis masivo de genes en un solo experimento vs. aquellos en los que solo permitían el estudio gen por gen (o de un pequeño grupo de ellos) han proporcionado una nueva dimensión en a la hora de plantear y extraer conclusiones de los experimentos. Los microarrays, que es como se conocen estos sistemas, fueron descritos por primera vez por Schenna en 199516 y nos permiten estudiar, por ejemplo, qué genes se expresan en un situación biológica concreta y compararla con una patológica y ver en qué se diferencian, qué genes se expresan y en qué proporción, dándonos una visión global de una enfermedad y cuáles son los mecanismos que operan en dicho proceso patológico, además de ser un potencial papel en el diagnóstico de enfermedades.
El objetivo de nuestro trabajo era caracterizar el conjunto de ARNm (o perfil génico) con la ayuda de los microarrays que se generaban en muestras que lograban un embarazo frente aquellas que no tanto con la técnica de IAH como con la de ICSI.
Normalmente los experimentos de microarrays proporcionan una cantidad ingente de información y suelen derivar en largas listas de genes que están diferencialmente expresados en una condición biológica determinada respecto a otra. En un intento de darle un punto de vista funcional a los datos que obtuvimos quisimos saber cuántos de esos genes que nos daban diferencialmente expresados estaban ya descritos previamente en procesos relacionados con la reproducción. Esto se realizó gracias al Gene Ontology, una herramienta que permite organizar el conocimiento biológico. Hay que tener en cuenta que un gen por si mismo es difícil que explique una situación de enfermedad vs. salud ya que los genes interaccionan unos con otros formando redes complejas, como las rutas metabólicas, que a su vez interaccionan con otras, por lo tanto necesitamos un interpretación funcional (en este caso proporcionada por el Gene Ontology) de estos listados de genes.
Material y métodosMuestras biológicas humanasSe utilizaron un total de 30 muestras, 20 procedentes de pacientes que se sometían a su primer ciclo de IAH (10 que lograron embarazo [grupo E] y 10 que no [Grupo NE]) y 10 muestras procedentes de pacientes que se sometían a un ciclo de ICSI con ovocitos de donantes (5 que lograron embarazo [grupo E] y 5 que no [grupo NE]). Todas las muestras proceden de sémenes en fresco.
En el caso de las IAH los criterios de inclusión fueron los siguientes. Los varones debían presentar parámetros seminales normales y con una recuperación de espermatozoides móviles tras la selección espermática superior a 3 millones. Las mujeres eran menores de 36 años, presentaban permeabilidad tubárica y no presentaban, miomas, ovario poliquístico ni endometriosis, a parte de presentar parámetros hormonales normales.
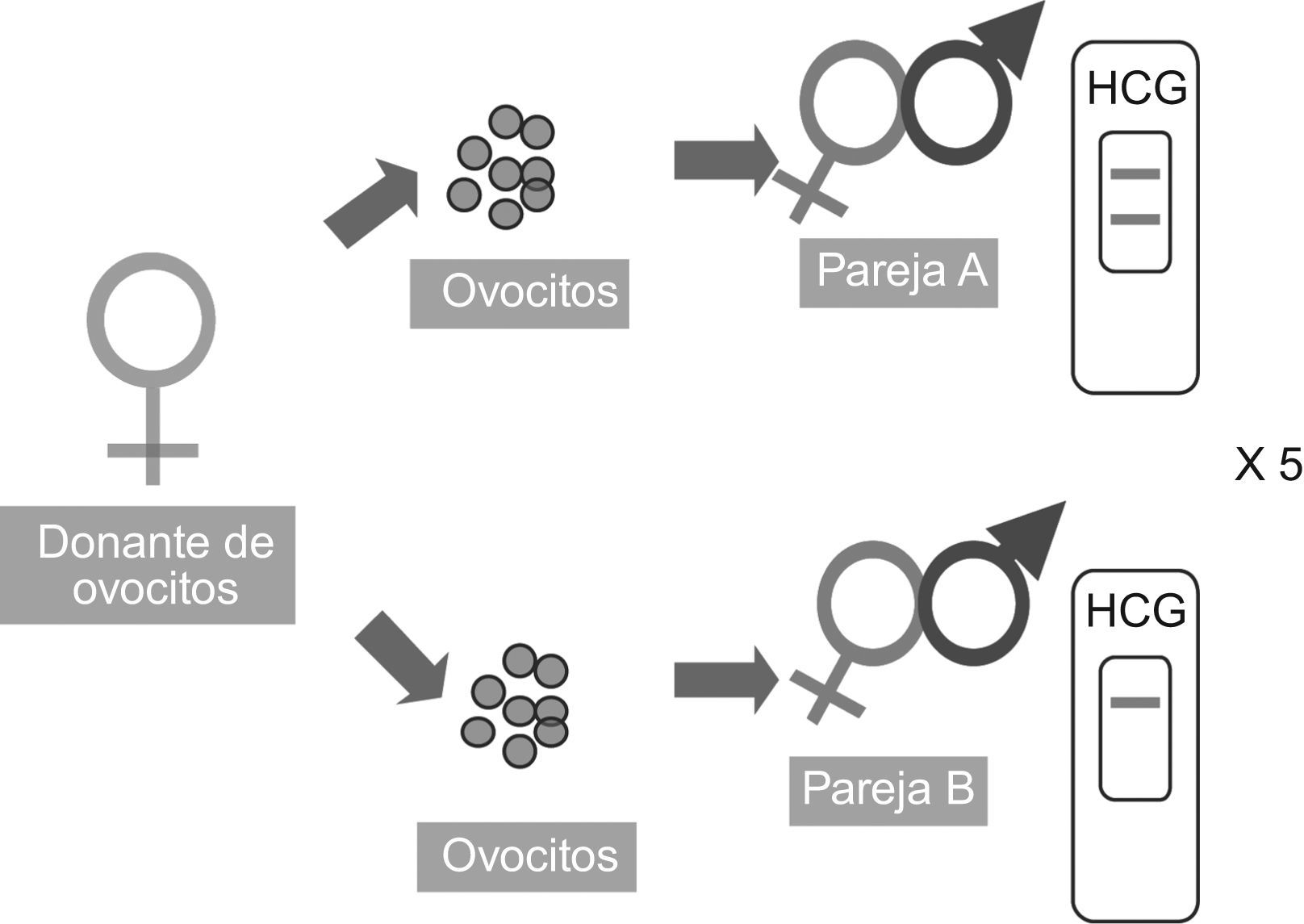
Con ICSI escogimos 5 casos. Cada caso estaba compuesto por 2 parejas que se sometían a un ciclo de ICSI con ovocitos de la misma cohorte donados por la misma donante consiguiendo una de las parejas un embarazo y la otra no (fig. 1). Finalmente se obtuvieron 5 muestras que embarazaban y 5 que no lo lograban.
Todas las muestras fueron obtenidas tras guardar entre 3–5 días de abstinencia sexual.
Este proyecto fue aprobado por el Comité Ético de Investigación Clínica del Instituto Valenciano de Infertilidad.
Análisis seminalLos parámetros seminales fueron evaluados tras 10min de liquefacción a 37°C y al 5% de concentración de CO2. La concentración y movilidad de las muestras fue evaluado en una cámara Makler® (Sefi Laboratories, Tel Aviv, Israel) siguiendo los criterios de la OMS15.
Preparación de las muestrasSe tomó aproximadamente una alícuota de 150 microlitros de las muestras de semen en fresco (antes de la selección espermática por swim-up) que posteriormente iban a ser empleadas en la TRA, para el pertinente análisis con microarrays. A continuación las muestras se sometían al proceso de selección espermática antes de usarlas para la TRA.
Estimulación ovárica para IAHEn los ciclos de IAH se estimuló tanto con FSH (Gonal-F®; Serono S.A., Spain or Puregon; Schering-Plough Española, Spain) como con hMG (Lepori®; Farma Laboratories, Spain; or Menopur; Ferring, Spain). La estimulación se inició en el día 3 del ciclo con dosis de FSH recombinante de entre 75–150UI. La foliculometría se monitorizó vía ecografía vaginal y siguiendo los niveles de estradiol. Cuando el tamaño del folículo dominante era de alrededor de 18 milímetros la paciente recibía 10.000 IU de hCG (Profasi® 10.000UI; Serono S.A., Spain) La paciente fue inseminada entre las 12–36h siguientes tras la inyección de hCG17.
Estimulación ovárica para ICSIPara los ciclos de ICSI, como son con ovocitos de donante, hemos de contemplar 2 tipos de tratamientos. El de la estimulación ovárica de la donante, que es diferente a la que se somete una paciente en un ciclo de IAH, y el de la preparación del endometrio de la paciente a la hora de transferirle el embrión.
Para la estimulación de las donantes se utilizaron tanto antagonistas como agonistas de la GnRH. Para estos últimos se utilizó el protocolo largo18. Este consiste en administrar 0,1 miligramos de leuprolide (Procrin®; Abbott S.A., Spain) o triptorelina (Decapeptyl®; Ipsen Pharma; Spain) a mitad de fase lútea del ciclo anterior. Una ecografía vía vaginal del ovario y unos niveles de estradiol determinados (niveles de estradiol en plasma <60 picogramos/mililitro (200 picomoles/litro) nos indicarán que el ovario está en reposo. En este momento es cuando se reduce la dosis de análogo a 0,05 miligramos hasta el día de administración de la hCG.
Los antagonistas de la GnRH se usaron siguiendo el protocolo de dosis baja19: a partir del día 6 de la estimulación se administró 0,25 miligramos de antagonista Cetrotide (Cetrorelix®; Serono S.A., Spain) hasta el día de la administración de la hCG.
Para la estimulación se usaron tanto la FSH recombinante (Gonal-F®; Serono S.A., Spain; or Puregon; Schering-Plough Española, Spain) como hMG (Lepori®; Farma Laboratories, Spain; or Menopur; Ferring, Spain).
Las dosis iniciales se determinaron según la edad de las pacientes y los niveles en plasma de FSH y estradiol. En el tercer día de la estimulación se midió el estradiol y la dosis de gonadotropina se ajustó según los resultados de dicha medición. La hCG (Profasi® 10.000UI; Serono S.A., Spain) se administró cuando 3 o más folículos alcanzaron los 18 milímetros de diámetro. La recuperación de los ovocitos se programó 36h tras la administración de la hCG11,20.
Preparación endometrial de las pacientesLa preparación del endometrio empieza con la desensibilización de la pituitaria con una dosis de 3,75 miligramos de triptorelina (Decapeptyl®; Ipsen Pharma, Spain) en la mitad de la fase lútea del ciclo anterior.
Las pacientes tomaron valeriato de E2 (Progynova®; Schering, Spain) desde el día 1 del ciclo, 2 miligramos del día 1 al 8, 4 miligramos del 9 al 11 y 6 miligramos del día 12 en adelante.
En el día 14 del ciclo, se evalúa la receptividad endometrial mediante ecografía vaginal y midiendo los niveles de E2. Las dosis de valeriato de E2 continuarán hasta que la donación sea posible.
El día de la donación, la paciente se aplicará por vía vaginal 800 miligramos/día de progesterona micronizada (Progeffik®; Laboratories Effik, Spain)11,20.
Extracción y aislamiento del ARNmLas alícuotas de las muestras de semen que se usarán posteriormente para hacer el microarray, se lavaron con 3 mililitros de PBS y se centrifugaron 10min a 400g. Se descartó el sobrenadante y el pellet fue resuspendido en 1 mililitro de Trizol, congelándolo posteriormente por inmersión directa en nitrógeno líquido y almacenándolo en el tanque de nitrógeno hasta su posterior utilización.
El ARN total se extrajo siguiendo el método del Trizol, según el protocolo propuesto por el fabricante (Life Technologies®, Inc., Gaithersburg, MD)21.
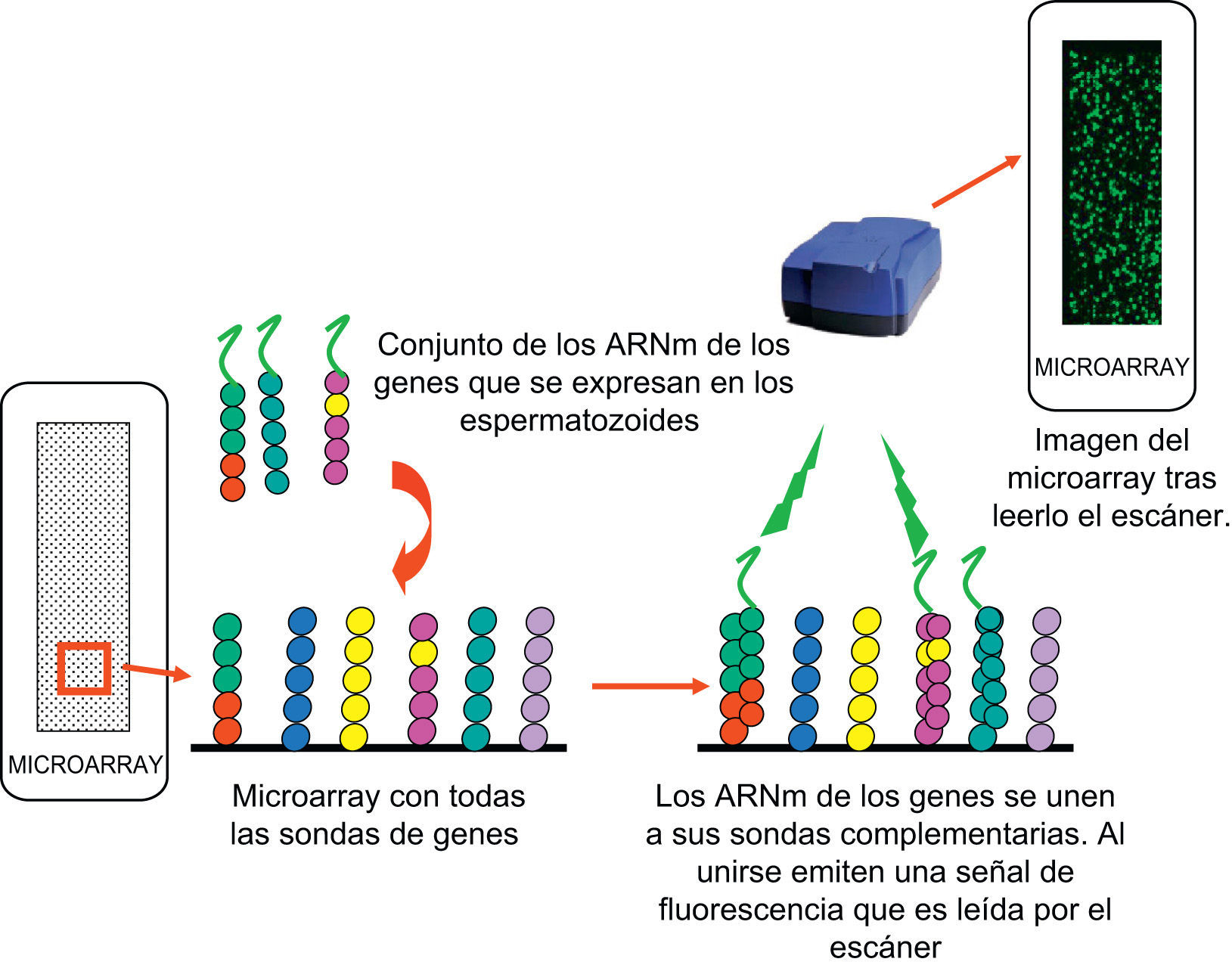
MicroarraysLos microarrays nos permiten evaluar la expresión de miles de genes en un único experimento. Básicamente se trata de una plataforma sólida en la que hay miles de sondas de genes que conocemos. Lo que hacemos es exponer estas sondas a nuestro ARNm problema (en este caso procedente de los espermatozoides que lograban embarazo o no, con cada una de las técnicas (IAH o ICSI) que previamente habíamos marcado con un fluorocromo. Los ARNm se unirán a sus sondas complementarias en el microarray emitiendo una señal de fluorescencia que será leída por un escáner y cuya imagen será posteriormente procesada (fig. 2).
Las muestras se hibridaron en microarrays de genoma humano completo de Agilent (WholeHuman Genome Oligo Microarray® de Agilent technologies, Madrid, Spain). Los protocolos de preparación e hibridación de las muestras se sacaron del Manual Técnico de Agilent.
Antes de realizar los microarrays, se procedió a agrupar las muestras en 4 grupos, las muestras que lograban embarazo (grupo E) y las que no (grupo NE) para cada una de las técnicas (IAH o ICSI). Todas las muestras que constituyen los grupos, contribuyen con la misma cantidad de ARNm22.
Finalmente se realizaron 4 microarrays (uno con cada grupo de muestras) comparando las muestras E frente NE para cada una de las técnicas (IAH o ICSI) y por último se compararon los perfiles de expresión génica entre ambas técnicas (fig. 3).
Análisis de los datosEl análisis de los datos se hizo con el soporte informático GenePix 6.0, que se encarga de medir la intensidad de fluorescencia que emiten las hibridaciones de nuestro ARNm problema al hibridar con las sondas del microarray.
Expresión diferencialEl perfil de expresión diferencial se determinó mediante la comparación del grupo E y NE usando test no paramétricos con dos criterios para definir los genes que presentaban una cantidad de ARNm para un tránscrito determinado alterada en un grupo respecto del otro: un cambio absoluto de 2 o más veces (esto es que la expresión de un gen sea el doble o más en un grupo que en el otro) y un p-valor menor de 0,05.
Estos genes que presenten un cambio igual o mayor de 2 veces con un p-valor<0,05 serán definidos como genes diferencialmente expresados (GDE).
Genes descritos en funciones reproductivas entre aquellos que están diferencialmente expresados entre los grupos E y NE para ambas técnicasNuestro grupo buscó de manera sistemática aquellos procesos biológicos que podrían tener cierta relación con la fertilidad masculina y confeccionó una lista de genes con la ayuda del Gene Ontology (GO). Posteriormente comprobamos si alguno de nuestro GDE de cualquiera de los 4 grupos (Grupo E, NE para IAH y Grupo E y NE para ICSI) estaba descrito en alguno de esos procesos.
Los procesos con los que confeccionamos esta lista fueron los siguientes:
Acrosoma (GO:0001669), reacción del acrosoma (GO:0007340), unión del espermatozoide a la zona pelúcida (GO:0007339), proceso reproductivo (GO:0022414), generación de gametos masculinos (GO:0048449), cópula (GO:0007620), implantación embrionaria (GO:0007566), desarrollo embrionario (GO:0009790),embarazo (GO:0007565), fecundación (GO:0009566), desarrollo de los genitales (GO:0048806), desarrollo para las células germinales (GO:0007281), desarrollo de las gónadas (GO:0008406), inseminación (GO:0007320), desarrollo de la placenta (GO:0001890), reproducción (GO:0000003), reproducción Sexual (GO:0019953), movilidad espermática (GO:0030317), desarrollo de las espermátidas (GO:0007286) y espermatogénesis (GO:0007283).
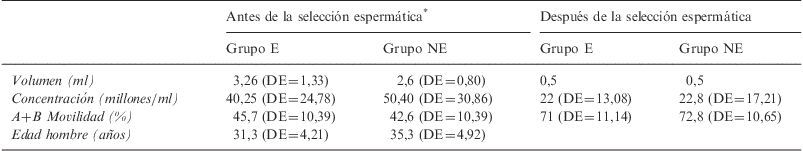
ResultadosAnálisis de semenTodas las muestras de semen incluidas en el estudio en el caso de las IAH presentaban parámetros similares normales y sin diferencias estadísticamente significativas entre ellos, tanto antes como después de la separación espermática por la técnica de swim-up (tabla 1).
Parámetros seminales de los grupos E y NE en el caso de las IAH
| Antes de la selección espermática* | Después de la selección espermática | |||
| Grupo E | Grupo NE | Grupo E | Grupo NE | |
| Volumen (ml) | 3,26 (DE=1,33) | 2,6 (DE=0,80) | 0,5 | 0,5 |
| Concentración (millones/ml) | 40,25 (DE=24,78) | 50,40 (DE=30,86) | 22 (DE=13,08) | 22,8 (DE=17,21) |
| A+B Movilidad (%) | 45,7 (DE=10,39) | 42,6 (DE=10,39) | 71 (DE=11,14) | 72,8 (DE=10,65) |
| Edad hombre (años) | 31,3 (DE=4,21) | 35,3 (DE=4,92) | ||
E: embarazo; IAH: inseminación intrauterina homóloga; NE: no embarazo.
En el caso de los ICSI, las muestras eran mucho más patológicas y ninguna de ellas cumplía con los criterios de normalidad establecidos por la OMS, en este apartado solo vamos a resaltar las indicaciones por las que estas parejas se sometía a ICSI.
La donación de ovocitos se indicó a sus respectivas parejas femeninas, en el grupo E, por edad (n=3), baja respuesta (n=1) y fallo ovárico precoz (n=1). En el grupo NE las indicaciones fueron similares: edad avanzada (n=2) y fallo ovárico precoz (n=3). La media de edad de las mujeres en el grupo E era de 42,4±5,8 y en el grupo NE 39±5 años.
La indicación masculina para ICSI fue en el grupo E, astenoteratozoospermia (n=3), oligoastenoteratozoospermia (n=1) y teratozoospermia severa (n=1).
En el grupo NE: oligoastenoteratozoospermia (n=2), oligoteratozoospermia (n=1) y astenozoospermia (n=2). Tanto las indicaciones para el grupo E como en el NE para ICSI suponen un factor masculino moderado o severo.
Análisis de los microarraysExpresión diferencialEl número total de genes que se expresaban en el caso de las IAH (esto es que daban una señal positiva en el microarray) era de 19.938, de estos 18.221 eran comunes tanto el grupo E como en el NE. De todos los genes que se expresaban en ambos grupo 756, estaban sobreexpresados en el grupo E y 194 en el grupo NE (un total de 950 GDE).
En los microarrays de ICSI el número total de genes que se expresaban era de 19.229, de los cuales 16.035 se expresaban tanto en el grupo E como el NE. De estos comunes, 44 se sobreexpresaban en el grupo E y 5 en el NE.
El 70% del número total de genes que se expresaban tanto en los microarrays de IAH como en los de ICSI, eran los mismos genes y casi el 50% de los GDE del grupo que embarazaba (Grupo E) con el ICSI, también estaban sobreexpresados en el grupo que embarazaba con las IAH.
Genes descritos en funciones reproductivas entre aquellos que están diferencialmente expresados entre los grupos E y NE para ambas técnicasExaminamos de manera sistemática nuestros GDE en la lista de genes descritos en procesos relacionados con la reproducción siguiendo las indicaciones descritas en la sección de material y métodos. Tras buscar los GDE en el caso de las IAH, encontramos que 34 genes, de un total de 750 GDE que teníamos en el grupo E, estaban descritos previamente en alguno de esos procesos. En el grupo NE de este mismo TRA solo había 9. En el caso de ICSI, 3 de los GDE del grupo E y 1 del NE estaban descritos en alguno de estos procesos (tabla 2).
Listados de los genes diferencialmente expresados que estaban descritos previamente en procesos relacionados con la reproducción tanto en el grupo que logra embarazo (E) como el que no (NE) para ambas técnicas (IAH e ICSI). Columna 1: grupo en el que GDE está expresado. Columna 2: número de acceso del gen y descripción del gen. Columna 3: procesos relacionados con la reproducción en los que el gen está descrito.
| IAH grupo E | Peptidase inhibitor 3, skin-derived (SKALP) (PI3), mRNA [NM_002638] | Copulación (GO:0007620) Apareamiento (GO:0007618) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) |
| Interleukin 8 (IL8), mRNA [NM_000584] | Proceso reproductivo (GO:0022414) | |
| Chemokine (C-C motif) ligand 4 (CCL4), transcript variant 1, mRNA [NM_002984] | Reproducción (GO:0000003) Reproducción (GO:0000003) | |
| Chemokine (C-C motif) ligand 3 (CCL3), mRNA [NM_002983] | Proceso reproductivo (GO:0022414) Reproducción (GO:0000003) | |
| v-fos FBJ murine osteosarcoma viral oncogene homolog (FOS), mRNA [NM_005252] | Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Vasodilator-stimulated phosphoprotein (VASP), transcript variant 1, mRNA [NM_003370] | Desarrollo embrionario (GO:0009790) | |
| mRNA for FLJ00028 protein, partial cds. [AK024438] | Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| Thrombomodulin (THBD), mRNA [NM_000361] | Desarrollo embrionario (GO:0009790) Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Regulator of G-protein signalling 2, 24kDa (RGS2), mRNA [NM_002923] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| B-cell CLL/lymphoma 6 (zinc finger protein 51) (BCL6), transcript variant 2, mRNA [NM_138931] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| LFNG O-fucosylpeptide 3-beta-N-acetylglucosaminyltransferase (LFNG), transcript variant 1, mRNA [NM_001040167] | Desarrollo de las gónadas (GO:0008406) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| v-maf musculoaponeurotic fibrosarcoma oncogene homolog B (avian) (MAFB), mRNA [NM_005461] | Desarrollo embrionario (GO:0009790) | |
| Insulin-like growth factor binding protein 7 (IGFBP7), mRNA [NM_001553] | Implantación embrionaria(GO:0007566) Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Notch homolog 2 (Drosophila) (NOTCH2), mRNA [NM_024408] | Desarrollo embrionario (GO:0009790) | |
| Adenosine deaminase (ADA), mRNA [NM_000022] | Copulación (GO:0007620) Desarrollo embrionario (GO:0009790) | |
| Prokineticin 2 (PROK2), mRNA [NM_021935] | Proceso reproductivo (GO:0022414) Reproducción Sexual(GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| mRNA; cDNA DKFZp686N17231 (from clone DKFZp686N17231). [BX648857] | Desarrollo embrionario (GO:0009790) Embarazo (GO:0007565) placenta development (GO:0001890) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Nuclear receptor subfamily 4, group A, member 3 (NR4A3), transcript variant 2, mRNA [NM_173198] | Desarrollo embrionario (GO:0009790) | |
| Granulin (GRN), mRNA [NM_002087] | Implantación embrionaria(GO:0007566) Desarrollo embrionario (GO:0009790) Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Quaking homolog, KH domain RNA binding (mouse) (QKI), transcript variant 4, mRNA [NM_206855] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) Reproducción Sexual(GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| Zinc finger, MIZ-type containing 1 (ZMIZ1), mRNA [NM_020338] | Desarrollo embrionario (GO:0009790) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) | |
| mRNA for CUGBP1 variant protein, partial cds, clone: fk00577 [AB210019] | Desarrollo embrionario (GO:0009790) Germ cell development (GO:0007281) Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) | |
| Transcription factor EB (TFEB), mRNA [NM_007162] | Desarrollo embrionario (GO:0009790) placenta development (GO:0001890) | |
| Nuclear autoantigen mRNA, partial cds; alternatively spliced. [L79989] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Zinc transporter 1 (ZnT-1) (Solute carrier family 30 member 1). [Source:Uniprot/SWISSPROT;Acc:Q9Y6M5] [ENST00000367001] | Desarrollo embrionario (GO:0009790) | |
| Muscleblind-like (Drosophila) (MBNL1), transcript variant 1, mRNA [NM_021038] | Desarrollo embrionario (GO:0009790) | |
| ADAM metallopeptidase domain 10 (ADAM10), mRNA [NM_001110] | Desarrollo embrionario (GO:0009790) | |
| Nuclear factor (erythroid-derived 2)-like 2 (NFE2L2), mRNA [NM_006164] | Desarrollo embrionario (GO:0009790) | |
| Carbohydrate (chondroitin 4) sulfotransferase 11 (CHST11), mRNA [NM_018413] | Desarrollo embrionario (GO:0009790) | |
| UDP-Gal:betaGlcNAc beta 1,4- galactosyltransferase, polypeptide 1 (B4GALT1), mRNA [NM_001497] | Reacción del acrosoma (GO:0007340) Unión del espermatozoide a la zona pelúcida (GO:0007339) Fecundación (GO:0009566) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) Reproducción sexual (GO:0019953) | |
| Ski oncogene (C-ski). [Source:Uniprot/SWISSPROT;Acc:P12755] [ENST00000378536] | Desarrollo embrionario (GO:0009790) | |
| Fc fragment of IgG, receptor, transporter, alpha (FCGRT), mRNA [NM_004107] | Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Solute carrier family 2 (facilitated glucose transporter), member 14, mRNA (cDNA clone MGC:71510 IMAGE:5297510), complete cds. [BC060766] | Proceso reproductivo (GO:0022414) Reproducción Sexual(GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| Lymphocyte antigen 6 complex, locus E (LY6E), mRNA [NM_002346] | Desarrollo embrionario (GO:0009790) | |
| IAH grupo NE | Cripto, FRL-1, cryptic family 1 (CFC1), mRNA [NM_032545] | Desarrollo embrionario (GO:0009790) |
| Cysteine dioxygenase, type I (CDO1), mRNA [NM_001801] | Proceso reproductivo (GO:0022414) | |
| Aldehyde dehydrogenase 1 family, member A3 (ALDH1A3), mRNA [NM_000693] | Desarrollo embrionario (GO:0009790) | |
| Homeobox B6 (HOXB6), mRNA [NM_018952] | Desarrollo embrionario (GO:0009790) | |
| Arachidonate 15-lipoxygenase, type B (ALOX15B), transcript variant d, mRNA [NM_001141] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Cylicin, basic protein of sperm head cytoskeleton 2 (CYLC2), mRNA [NM_001340] | Proceso reproductivo (GO:0022414) Reproducción Sexual(GO:0019953) Espermatogénesis (GO:0007283) Generación de gametos masculinos (GO:0048449) | |
| Isocitrate dehydrogenase 1 (NADP+), soluble (IDH1), mRNA [NM_005896] | Desarrollo de las gónadas (GO:0008406) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Shroom family member 3 (SHROOM3), mRNA [NM_020859] | Desarrollo embrionario (GO:0009790) | |
| CHK1 checkpoint homolog (S. pombe) (CHEK1), mRNA [NM_001274] | Proceso reproductivo (GO:0022414) Reproducción Sexual(GO:0019953) | |
| ICSI GDE E | Granulin (GRN), mRNA [NM_002087] | Implantación embrionaria(GO:0007566) Desarrollo embrionario (GO:0009790) Embarazo (GO:0007565) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) |
| Secreted phosphoprotein 1 (osteopontin, bone sialoprotein I, early T-lymphocyte activation 1) (SPP1), transcript variant 1, mRNA [NM_001040058] | Implantación embrionaria(GO:0007566) Embarazo (GO:0007565) placenta development (GO:0001890) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| Chemokine (C-X-C motif) receptor 4 (CXCR4), transcript variant 1, mRNA [NM_001008540] | Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) | |
| ICSI GDE NE | Ankyrin repeat domain 7 (ANKRD7), transcript variant 1, mRNA [NM_001077708] | Desarrollo de las gónadas (GO:0008406) Reproducción (GO:0000003) Proceso reproductivo (GO:0022414) |
GDE: gen diferencialmente expresado; E: embarazo; GO: Gene Ontology; IAH: inseminación intrauterina homóloga; ICSI: inyección intracitoplásmica; mRNA: ácido ribonucleico mensajero; NE: no embarazo.
Los datos revelan que el conjunto de ARNm presentes en los espermatozoides de las muestras de semen de pacientes difiere si estos han logrado un embarazo o no y también son diferentes según del tipo de TRA al que se han sometido.
La diferencias se encuentran pues a un nivel molecular, a nivel de los ARN mensajeros presentes en el escaso citoplasma del espermatozoide y que son depositados en el ovocito tras la fecundación y que parece que están implicados en el proceso de fecundación y en las primeras divisiones embrionarias23,23–27.
Asimismo se planteó que los requerimientos moleculares de las muestras de semen capaces de lograr un embarazo no son los mismos para todas las TRA, siendo más cuánto menos invasivas son, por ejemplo en una IAH, frente a las que más, como es el caso de ICSI. Suponemos que un espermatozoide que es capaz de embarazar a una IAH debe cumplir más «requisitos» que uno que logre un embarazo en ICSI. Este último, por ejemplo, no debe recorrer parte del tracto reproductivo, ni unirse ni atravesar la zona pelúcida del ovocito como en el caso de una IAH ya que aquí se introduce forzosamente el espermatozoide dentro del ovocito.
Además los resultados nos dejan entrever el desconocimiento que hay sobre el proceso reproductivo, ya que de un total de 999 GDE (756 grupo E IAH+194 grupo NE IAH+44 grupo E ICSI+5 grupo NE ICSI), solo 47 estaban descritos previamente en procesos biológicos relacionados con la reproducción. Cabría esperar que de esos genes diferencialmente expresados en espermatozoides tanto de muestras que han logrado o no un embarazo hubiera un mayor número descrito en funciones reproductivas.
Estas diferencias que hemos hallado a nivel molecular entre muestras que embarazan y no que no pueden ser detectadas por el espermiograma pueden ser potenciales marcadores de fertilidad en las diferentes técnicas de reproducción asistida28,29.
Por lo demás los datos revelan aspectos muy interesantes como es la coincidencia de un 70% de los genes entre pacientes que tienen una producción espermática aparentemente normal y otros que la tienen seriamente afectada, lo que nos deja ver la gran cantidad de genes expresados cuya función es desconocida en el espermatozoide y que probablemente no tienen ninguna relación con el proceso reproductivo incluidas las primeras etapas del desarrollo embrionario. Estas similitudes génicas también hablan a favor de la tecnología de los microarrays por su fiabilidad y reproducibilidad, a pesar de analizar más de 19.000 variables (genes). Esto las hace candidatas al salto desde la investigación básica al diagnóstico clínico aplicado30.
Puntualizar que nosotros hacemos el análisis con pequeñas alícuotas de muestras de semen en fresco. Consideramos que esa pequeña fracción de semen que separamos es representativa del resto de la muestra que se empleará para realizar la IAH o el ICSI. Posteriormente, el resto de la muestra es preparada mediante un proceso de lavado e incubación que elimina el plasma seminal y las células no espermáticas y selecciona espermatozoides móviles y de buena morfología y se emplea en el correspondiente TRA. Lo hacemos en fresco y no con los espermatozoides seleccionados, por que el objetivo es analizar la muestra de semen lo más «en bruto» que se pueda para poder buscar algún marcador que resulte de utilidad diagnóstica, al igual que se hace con un espermiograma. Además en esta fase inicial de nuestro estudio, lo importante era demostrar que existían diferencias entre los diferentes grupos31,32.
También nos gustaría destacar las ventajas que tiene el estudio en pacientes sometidos a donación de ovocitos. Con el modelo de la ovodonación usado en el ICSI conseguimos estandarizar al máximo el factor femenino, dejando más en evidencia el masculino, estudiando pacientes muy excepcionales en las que una misma donante es capaz de ceder sus óvulos a más de una receptora, con lo que se fecunda la misma cohorte ovocitaria mediante ICSI con semen de sus parejas, logrando una de ellas gestación y la otra no. Teniendo en cuenta que los ovocitos usados en ambas parejas son equiparables y que una logra embarazarse y otra no, deducimos que el peso del factor masculino debe ser más claro en estos casos.
El siguiente paso es realizar este tipo de análisis de manera individualizada, es decir, concretar los análisis reduciendo el número de genes a aquellos que hemos visto están directamente implicados en la fertilidad y el éxito reproductivo. Con ello aceleramos y abaratamos los análisis a fin de poder ofrecer a los pacientes una herramienta diagnóstica/clasificadora basada en la tecnología del microarray como se hace ya con otras patologías en otros campos33 Esta complementaría al clásico espermiograma (muy útil en los casos más patológicos) a la hora de evaluar el potencial fértil de una muestra de semen de pacientes que se vayan a someter a un tipo determinado de TRA.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.
Los autores quieren agradecer la colaboración de todos los técnicos de los laboratorios de Andrología y Semen FIV de Instituto Valenciano de Infertilidad de Valencia.
Este trabajo corresponde a una comunicación científica presentada y premiada en el III Congreso Nacional del Laboratorio Clínico celebrado en Valencia del 14 al 16 de octubre de 2009.